功能基因在皮肤微生物群落分析中的应用*
姚雪,李辉,张凯鑫,周怀谷,罗亚平△
(1.中国人民公安大学刑事科学技术学院,北京 100038;2.上海市刑事科学技术研究院,上海 200083)
1 引 言
人类皮肤微生物组指所有在人类皮肤上的微小生物,如细菌、真菌和病毒等[1]。针对细菌的分析主要是基于16S rRNA基因的测序结果,而这种分析方法存在一定的劣势。首先,该基因并非单拷贝基因,因此,其测序结果总体上来说仅仅是半定量的[2],会影响微生物群落的α多样性及β多样性的测度时,导致分析结果与实际情况间存在偏差;其次,16S rRNA基因的可变区变异程度不够大,在区分同一属内不同种时存在困难。已有的研究显示,与宏基因组shotgun测序相比,实验16S rRNA基因的V1-V3区测序获得的微生物群落结构分析结果与之相似,而V4区测序获得的微生物群落结构分析结果却不尽人意[3]。为了弥补16S rRNA基因存在的这些问题,开发新的管家基因引物是必需的。
在综合分析人类皮肤微生物群落组成的文献[4-6]的基础上,选择了4个最为普遍存在的属的细菌,从NCBI的Genome数据库中下载了这四个属60种细菌的全基因组并对其进行了分析,具体包括:Corynebacterium属24种细菌,Lactobacillus属12种细菌,Staphylococcus属8种细菌,Streptococcus属16种细菌。在对这些属的基因组分析的基础上,筛选出一些普遍存在的单拷贝功能基因[7],并针对其保守区域设计了一些具有通用扩增能力的引物。将这些引物成功地应用在分析不同年龄阶段、不同性别的36名志愿者的皮肤微生物群落结构特征分析上。结果证明这些新设计的引物对的扩增产物测序结果经分析,能够成功将细菌鉴定到种,在分析皮肤微生物群落方向具有良好的应用前景。
2 材料和方法
2.1 材料
2.1.1供试样品 试验样品采集自36名志愿者的双手手掌面,所有志愿者按年龄不同分为3个组别(15至24岁;25至34岁;35岁及以上),每个组别中男女数目各半。
2.1.2主要试剂及仪器 High Pure PCR Template Preparation Kit (Roche Diagnostics GMbH,德国),Q5 HighFidelity 2X Master Mix,(New England Biolabs,美国),JY88-II DN 超声波信号发生器(南京新辰生物科技有限公司,中国),Gene Amp PCR System 9700(Applied Biosystems,美国),Hiseq 2500测序仪(Illumina,美国)。
2.2 实验方法
2.2.1样品采集及DNA提取 使用尖头棉签蘸取0.1% Tween 20+0.15M NaCl,在志愿者手掌面上反复擦拭,将棉签头部剪入1.5 ml离心管中(含500 μL 0.1% Tween 20+0.15 M NaCl),涡旋震荡2 min。超声处理5 min(超声2 s,间隔3 s,功率18%)后使用Roche High Pure PCR Template Preparation Kit试剂盒提取DNA,最终使用100 μL ddH2O溶解DNA。(样品编号中L、R分别表示左手、右手,1、2、3、4、5、6、7、8等数字分别表示志愿者编号。)
2.2.2PCR扩增 PCR体系均为50μL体系:Q5 High-Fidelity 2X Master Mix 25 μL,上下游引物(每个处理所用引物均在5’端带有4bp barcode,引物浓度为10pMol)各1μL,溶解有DNA的ddH2O 23 μL。
rpsL基因引物对序列为rpslf (5’-ATGCCAACCATTAACCAATT-3’)(正向引物), rpslr (5’-GCAGTATCAAGGGCACCACG-3’)(反向引物)。PCR条件:预变性95℃ 5 min;变性95℃ 30 s,退火60℃ 30 s,延伸72℃ 30 s(35个循环);延伸7 min。
rpsH基因引物对序列为rpshf (5’-ATGGCTATGACTGATCCAAT-3’)(正向引物),rpshr (5’-TTACCAAACGTAGGCAATTACTTC-3’)(反向引物)。PCR条件:预变性95℃ 5 min;变性95℃ 30 s,退火50℃ 30 s,延伸72℃ 30 s(35个循环);延伸7 min。
dnaK基因引物对序列为dnakf (5’-TACTTCAACGATGCTCAACGTCAAGC-3’)(正向引物),dnakr (5’-TCCTTCTTAGCCTTTTCAGCAGCATC-3’)(反向引物)。PCR条件:预变性95℃ 5 min;变性95℃ 30 s,退火55℃ 30 s,延伸72℃ 30 s(35个循环);延伸7 min。
16S rRNA基因引物序列为F27(5’-AGAGTTTGATCCTGGCTCAG-3’)(正向引物),R338(5’-TGCTGCCTCCCGTAGGAGT-3’)(反向引物)。PCR条件:预变性95℃ 5 min;变性95℃ 30 s,退火55℃ 30 s,延伸72℃ 30 s(35个循环);延伸7 min。
2.2.3测序及数据分析 PCR产物使用Illumina Hiseq 2 500测序仪测序,测序结果使用QIIME 1.8.0软件分析[8]。选择质量>20且长度>200 bp的序列进行分析,先用FLASH软件根据overlap将双端序列合并成单端序列,得到完整的基因目标区域序列,然后根据barcode及引物进行分样,将测序数目少于1 000条的样品排除,不参与后续的分析运算;分样后使用QIIME 1.8.0软件包中的pick_otu.py中的uclust方法进行OTU聚类,之后在每个OTU中挑选一条代表序列,用blastx与对应功能基因的蛋白序列库进行比对,得到每个代表序列的物种信息,也就得到了对应的OTU的物种信息;根据每个OTU包含的序列数及对应的物种信息,可以计算出每个物种的丰度,并通过序列抽样的方法得到相关多样性指数的稀疏曲线(α多样性分析),通过计算样品间的距离(功能基因使用bray-curtis距离,16S rRNA基因使用weighted UniFrac 距离),对所有样品进行β多样性分析。
3 结果和分析
3.1 rpsH基因片段分析手掌面皮肤表面微生物群落
质量筛选后共得到871,953条序列(来自39只手掌面的皮肤),均一化后每个样品得到的OTU数目在61种至198种之间,含量最高的四个种分别是Streptococcus mitis,Lactobacillus crispatus,Abiotrophia defectiva 及 Streptococcus salivarius。由α多样性指数计算结果可见,参与实验的志愿者手掌面皮肤表面微生物群落的shannon指数在1.779到5.341之间,simpson指数在0.363到0.936之间。不同样品物种丰度聚类热图见图1,不同样品间的UPGMA建树见图2。

图1 不同样品物种丰度聚类热图(rpsH gene)
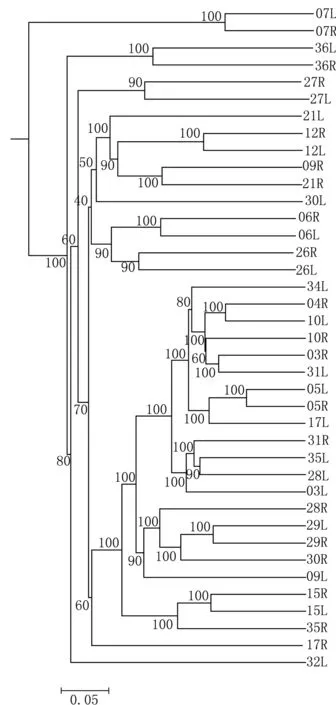
图2 不同样品间基于bray-curtis距离的UPGMA建树(rpsH gene)
3.2 rpsL基因片段分析手掌面皮肤表面微生物群落
质量筛选后共得到11,879,298条序列(来自62只手掌面的皮肤),均一化后每个样品得到的OTU数目在104种至632种之间,含量最高的四个种分别是Staphylococcus epidermidis,Streptococcus pneumoniae,Staphylococcus capitis及Staphylococcus hominis。由α多样性指数计算结果可见,参与实验的志愿者手掌面皮肤表面微生物群落的shannon指数在2.078到5.602之间,simpson指数在0.424到0.929之间。不同样品物种丰度聚类热图见图3,不同样品间的UPGMA建树见图4。
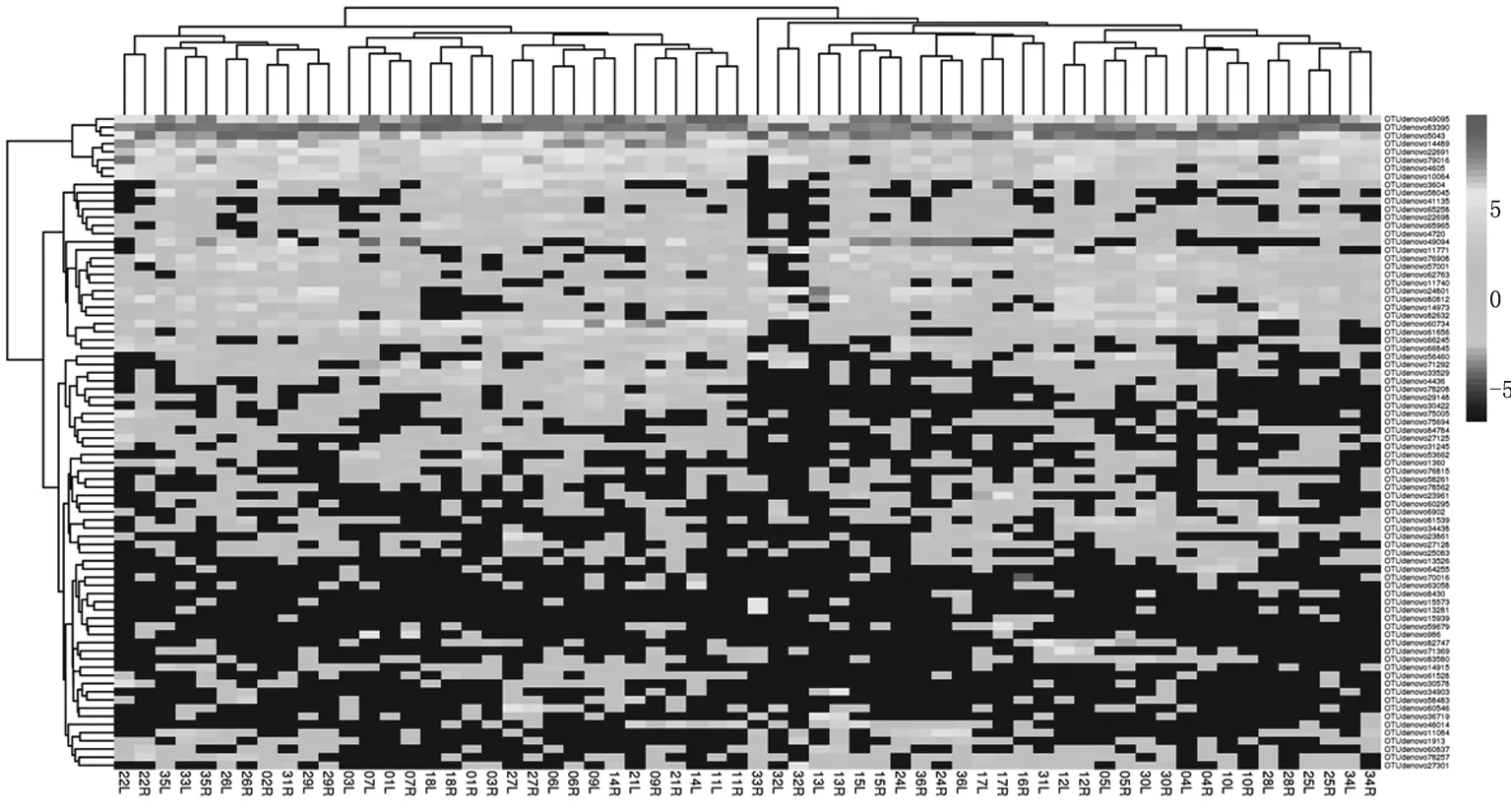
图3 不同样品物种丰度聚类热图(rpsL gene)

图4 不同样品间基于bray-curtis距离的UPGMA建树 (rpsL gene)
3.3 dnaK基因片段分析手掌面皮肤表面微生物群落
质量筛选后共得到6,633,964条序列(来自54只手掌面的皮肤),均一化后每个样品得到的OTU数目在39种至305种之间,含量最高的四个种分别是Prevotella sp. oral taxon 473, Bacillus subtilis,Streptococcus pneumoniae及Streptococcus sanguinis。由α多样性指数计算结果可见,参与实验的志愿者手掌面皮肤表面微生物群落的shannon指数在2.219到7.540之间,simpson指数在0.431 到0.983之间。不同样品物种丰度聚类热图见图5,不同样品间的UPGMA建树见图6。

图5 不同样品物种丰度聚类热图(dnaK gene)

图6 不同样品间基于bray-curtis距离的UPGMA建树(dnaK gene)

图7 不同样品物种丰度聚类热图(16S rRNAgene)

图8 不同样品间基于bray-curtis距离的UPGMA建树(16S rRNAgene)
3.4 16S rRNA基因片段分析手掌面皮肤表面微生物群落
质量筛选后共得到3,292,545条序列(来自48只手掌面的皮肤),均一化后每个样品得到的OTU数目在120 种到 251种之间,含量最高的四个属分别是Propionibacterium, Corynebacterium,Staphylococcus和Paracoccus。由α多样性指数计算结果可见,参与实验的志愿者手掌面皮肤表面微生物群落的shannon指数在 4.016 到 6.992之间,simpson指数在0.794 到 0.980之间。不同样品物种丰度聚类热图见图7,不同样品间的UPGMA建树见图8。
4 讨论
由16S rRNA基因测序结果统计得出的所有样品中含量最高的细菌分别是Propionibacterium,Corynebacterium和Staphylococcus等,该结论与Leung等人的研究结果相一致[4]。由其他三个功能基因的测序结果统计得出的含量较高的细菌的种类的结果存在差异,主要原因是不同的引物对针对不同的种属其扩增能力存在差异造成。虽然这些新设计的引物对的通用扩增能力不及16S rRNA基因的引物,但其鉴定能力更强,不仅能够将细菌准确地鉴定到种这一层级,而且也能实现对不同样品间的微生物群落结构特征进行比较。所以这些新设计的引物在皮肤微生物群落的分析中有较好的应用前景。

