鉴别伪狂犬病毒gE基因缺失疫苗毒与野毒套式PCR方法的建立
朱小甫,吴旭锦
(咸阳职业技术学院畜牧兽医研究所 动物疫病分子生物学诊断实验室,咸阳 712000)
·研究论文·
鉴别伪狂犬病毒gE基因缺失疫苗毒与野毒套式PCR方法的建立
朱小甫,吴旭锦
(咸阳职业技术学院畜牧兽医研究所 动物疫病分子生物学诊断实验室,咸阳 712000)
为建立一种能够鉴别伪狂犬病毒gE基因缺失疫苗毒与野毒套式PCR方法,根据GenBank上发表的伪狂犬病毒gD、gE基因序列,设计并合成了4对引物,对反应体系和条件进行优化,建立复合套式PCR方法。通过灵敏性试验、特异性试验、病料中PRV检测对比等方法验证建立方法的适用性。结果表明,该方法检测的极限为2.78×10-4pg/L,并且仅能从PRV野毒株扩增出354、217 bp目的条带,PRV疫苗扩增出了217 bp单条带,其他5种常见感染猪的病毒均为阴性。
伪狂犬病毒;gD基因;gE基因;鉴别诊断;套式PCR
伪狂犬病(pseudorabies,PR)又名奥叶兹基氏病,是由疱疹病毒科(Herpesvirdae),α-疱疹病毒亚科(Alpherpesvirinae)中的伪狂犬病毒(Pseudorabies virus,PRV)感染引起的一种或多种动物感染的急性传染病,迄今仍是我国主要的猪群传染病之一[1]。PRV可引起怀孕母猪流产、死胎、木乃伊胎;新生仔猪衰竭死亡,死亡率可达100%;成年猪多呈隐性感染,长期带毒排毒,危害巨大[2]。在我国,自1984年刘永纯首次报道伪狂犬病后,迄今为止已有20多个省份相继发生该病,给我国的养猪业特别是集约化养猪造成了巨大经济损失[3]。
随着分子生物学技术的飞速发展,PCR技术以其快速、敏感、易于操作等优点在动物疫病诊断中得到了广泛应用[4-6]。Harding等[7]针对gD基因设计引物,改进了PRV PCR检测方法。Jestin等[9]从鼻拭子抽提核酸,通过扩增PRV的gD基因序列检测出伪狂犬病毒。Talmage等[9]应用gB基因引物对活检的扁桃体细胞和实质组织进行扩增,能应用于潜伏感染的检查。国内娄高明等[10]、吕建强等[11]也分别建立了检测PRV的PCR方法,可用于伪狂犬病的诊断和流行病学调查。
疫苗免疫接种是防制乃至根除伪狂犬病的主要手段之一。西方许多国家制定的根除狂犬病计划均需依赖于基因缺失疫苗的免疫接种[12]。我国也一直采用疫苗预防为主的防控策略,广泛使用的Bartha-K61株弱毒疫苗基因中,具有整个gE和大部分gI基因序列缺失。由于猪群疫苗免疫覆盖率高,导致疫苗毒株在猪群中广泛存在。常规PCR技术不能区分疫苗毒和伪狂犬野毒,在诊断结果的判定上存在困难。本研究拟建立一种能够通过PCR技术直接区分检测样品(组织、血清及精液等)中的伪狂犬病毒为野毒还是疫苗毒,为临床控制伪狂犬病提供一种实用方法。
1 材料与方法
1.1材料
1.1.1病毒 参考毒株PRV SXHX株为离自户县某猪场伪狂犬野毒株;参考疫苗毒为勃林格生产的商品苗Bartha-K61株;猪圆环病毒2型(Porcine circovirus type 2,PCV2)SXXY13株分离自旬邑县某猪场;细小病毒(Porcine parvovirus,PPV)SXHZ株分离自汉中市某猪场;猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV)YLCH株分离自榆林市某猪场;分离毒株均由动物疫病分子生物学诊断实验室保存;猪瘟病毒(Classical swine fever virus,CSFV)和猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)均为商品疫苗毒株。
1.1.2病料 本实验室采集或猪场送检疑似PR组织病料(包括肺脏、脑组织和扁桃体)、病猪血清以及精液样品,分别来自咸阳市、宝鸡市、渭南市、和西安市猪场,共计22份,将组织材料研磨处理,12 000×g离心10 min,收集上清液-70℃保存备用。
1.1.3引物设计与合成 PRV gD基因序列(GenBank登录号:AY217094),PRV gE基因序列(GenBank登录号:AY170318),设计并合成了4对引物。PRVgD-1F:5'-TAAAATTGGGTCGGCGTCC-3' (2 6~4 6 b p),P R Vg D-1 R:5'-CGCCCTCAGGAATCGGAC -3'(712~731 bp);PRgE-1F:5'-GTTCAGAGCATGCGC CGG-3'(20~41 bp),PRgE-1R:5'- CGACA GCAGCCAGACGC -3'(1387~1368 bp);PRVgD-2F:5'- GACCGAGCACGAGGTGCG-3'(484~504 bp),PRVgD-2R:5'- GTCCACGCGCGCCTTGA -3'(6 8 0~7 7 0 b p);P Rg E-2 F:5'-ATCGCGATCGGCGAGTCA -3'(465~484 bp);PRgE-2R:5'- CGAGTCGAGCGTGCCACTA -3'(818~797 bp)。引物由生工生物工程(上海)有限公司合成,均用DEPC处理水稀释到20 μmol/mL。
1.2方法
1.2.1PRV DNA的提取 取PRV SXHX株细胞培养物200 μL置于无菌EP管,加入DNAzol Reagent 1000 μL,上下颠倒混匀,室温静置裂解10 min;4℃、10 800×g离心5 min,吸取600 μL上清液转移至另一无菌EP管,加入等体积冰冷无水乙醇沉淀10 min,4℃、10 800×g离心10 min;弃去上清液,加入1 mL冰冷的70%乙醇清洗2次,倒置干燥,用40 μL NaOH(8 mmol/L)充分吹打溶解,置沸水浴中10 min,取出后冰浴3 min,在核酸蛋白测定仪上测定DNA的含量。
1.2.2PRV nPCR方法的建立 取已知浓度的DNA溶液做为模板,摸索扩增条件。第1次扩增反应体系:DNA 2.0 μL,2×GC Buffer 12.5 μL,dNTP 1.0 μL,PRVgD-1F、PRVgD -1R、PRVgE-1F、PRVgE -1R各0.5 μL,改变rTaq DNA聚合酶用量(0.25~1.0 μL),用超纯水补足总体积25.0 μL。条件设定:95℃预变性5 min;94℃变性1 min,退火温度由60℃~65℃按1℃递增设定退火1 min,72℃延伸 1 min,共进行35个循环;最后72℃延伸10 min。第2次扩增时取2.0 μL第1次扩增产物作为模板,其他成份同第1次扩增进行设定摸索,只是引物更换为PRVgD-2F、PRVgD -2R、PRVgE-2F、PRVgE -2R。条件设定:95℃预变性5 min; 94℃变性1 min,退火温度由60℃~65℃按1℃递增设定退火1 min,72℃ 延伸1 min,共35个循环;最后72℃延伸10 min。摸索出反应体系和反应条件的最佳组合。
1.2.3PRV nPCR方法灵敏性试验 将DNA溶液做10倍梯度稀释至10-9倍,分别以建立的nPCR方法对各稀释后DNA溶液作为模板进行扩增,扩增完毕后取5.0 μL扩增产物,进行1.5%琼脂糖凝胶电泳,凝胶成像系统中照相观察。
1.2.4PRV nPCR方法特异性试验 提取CSFV、PRRSV和PEDV等病毒RNA基因组并反转录获得cDNA;按照DNAzol Reagent试剂说明提取PRV SXHX株、PCV2 SXXY13株以及PPV SXHZ株等DNA基因组病毒模板,用建立的nPCR方法检测,PCR产物用1.5%琼脂糖凝胶电泳,成像系统中观察照相。
1.2.5病料中PRV的检测 采用建立的检测方法对收集的22份病料进行检测。
2 结果
2.1PRV nPCR方法的建立 通过改变反应体系和反应条件,确定最优体系和条件。第1次扩增DNA 2.0 μL,2×GC Buffer 12.5 μL,超纯水7.0 μL,dNTP 1.0 μL,PRVgD-1F、PRVgD-1R、PRVgE-1F、PRVgE-1R各0.5 μL,rTaq DNA聚合酶0.5 μL,总体积25.0 μL。第一次扩增条件:95℃预变性5 min;94℃变性1 min,62℃退火1 min,72℃延伸1 min,共进行35个循环;最后72℃延伸10 min。第2次扩增反应体系:取2.0 μL第1次扩增产物作为模板,体系其余组分同第1次扩增,引物为PRVgD-2F、PRVgD -2R、PRVgE-2F、PRVgE -2R。第二次扩增条件:95℃预变性5 min;94℃变性1 min,65℃退火1 min,72℃延伸1 min,共进行35个循环;最后72℃延伸10 min。
2.2PRV nPCR方法的灵敏度试验 提取伪狂犬野毒SXHX株DNA,紫外分光光度计测定浓度为278 pg/ L,按照10倍梯度稀释至10-9,用建立的方法进行套式PCR,扩增完毕后电泳观察。结果显示,扩增出354、217 bp两个特异性条带,检测极限为2.78×10-4pg/L,表明建立的PCR方法灵敏度高(图1)。

图1 PRV nPCR方法的灵敏度检测Fig. 1 The sensitivity of nPCR for the detection of PRV
2.3PRV nPCR检测方法特异性试验结果 用所建立的方法对CSFV、PRRSV、PRV SXHX株、PRV疫苗、PEDV、PCV2 SXXY13株以及PPV SXHZ株cDNA/DNA进行扩增。结果发现,仅有PRV SXHX株扩增出了354、217 bp目的条带,PRV疫苗仅扩增出了217 bp条带,其他5种病毒均为阴性(图2),提示所建立的方法特异性好。
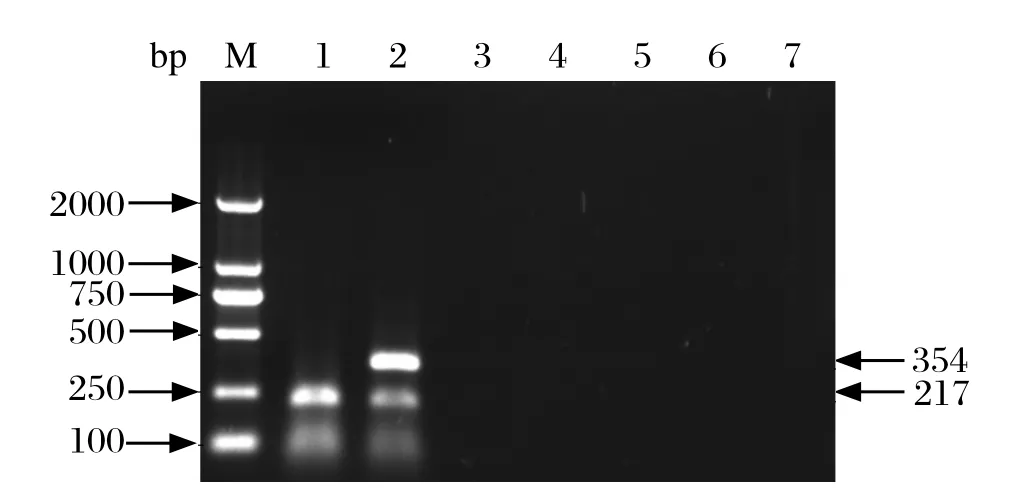
图2 PRV nPCR方法特异性检测Fig. 2 The specifi city of nPCR for the detection of PRV
2.4病料中PRV的检测对比结果 提取收集的22份疑似感染PRV的组织病料、血清以及精液总DNA,用建立的nPCR方法进行检测,nPCR方法在13份材料中扩增出了目的条带,阳性率为59%,其中2条带野毒株9份,野毒阳性率为40.9%,单条带疫苗毒株4份,疫苗毒阳性率为18.2%(图3)。
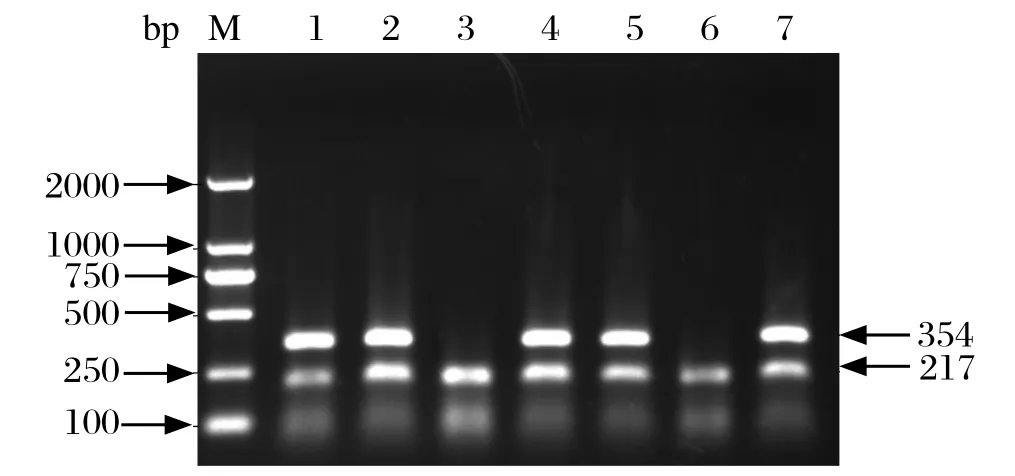
图3 部分待检样品中PRV检测结果Fig. 3 The detection result of PRV from some samples
3 讨论
我国使用的诊断猪伪狂犬的血清学方法有病毒分离鉴定、血清中和试验、琼脂扩散试验、乳胶凝集试验、酶联免疫吸附试验和免疫荧光技术,分子生物学方法有PCR技术和核酸杂交技术[13]。病毒分离鉴定是诊断伪狂犬最可靠的办法之一,但最大的缺点是耗时长,敏感性低。血清中和试验、琼脂扩散试验和乳胶凝集试验等检测方法是特异性好,但灵敏性较低。ELISA可区分疫苗免疫抗体和野毒感染抗体,且灵敏度高,目前应用广泛[14]。但是猪群野毒抗体阳性有可能是猪群曾经感染野毒所引起,因此,ELISA检测出野毒抗体阳性并不能代表猪群现在感染伪狂犬野毒,所以在伪狂犬野毒的定性诊断上尚需要依赖病原的检测。
国内有学者尝试建立相关的鉴别诊断方法,刘丽娜等[15]建立了多重PCR鉴别猪伪狂犬病野毒与疫苗毒诊断方法,可以检测到106 pg三基因缺失疫苗毒或756 pg PRV野毒的核酸模板量。但是多重PCR方法对病料模板的纯度和含量要求高于单一PCR,因此,将其应用于临床病理组织材料检测,确定其临床效果仍需进一步的研究。蔺芳等[16]也建立了多重PCR方法,设计了3对引物对4种伪狂犬疫苗样品DNA进行多重PCR扩增,其中1种疫苗得到与设计相符的1条特异性条带,其余3种样品为2条。建立的多重PCR仅检测了纯培养的细胞毒,没有进行临床样本的直接检测,这一方法能否应用于生产实际,是否不需细胞培养进行病毒分离纯化而直接检测组织或血清等材料,也需进一步的研究证实。
本研究针对gD、gE基因设计了4对引物,建立了能鉴别诊断PRV野毒株和疫苗毒株的nPCR方法。灵敏度实验结果表明,建立的方法检测极限为2.78 ×10-4pg/L,从野毒株可扩增出354、217 bp预期目的条带,疫苗毒仅出现217 bp条带,其他5种常见猪病毒均为阴性,结果提示所建立的方法特异性好,灵敏度高。从收集的22份材料(组织、血清和精液)中直接检出了野毒株9份,疫苗毒株4份,表明本方法可直接应用于检测临床不同材料,便捷直观,不需进行细胞培养,为临床快速准确诊断伪狂犬提供了有力的技术保障。
[1] 殷震, 刘景华. 动物病毒学[M]. 2版. 北京: 科技出版社,1997.
[2] 程晶, 荫硕焱, 盖新娜, 等. 2006年~2008年我国部分地区规模化猪场PRV血清流行病学调查[J]. 中国动物传染病学报, 2009, 17(1): 67-71.
[3] 张显浩, 陈瑞爱, 李冰, 等. 2012年-2013年我国集约化猪场猪伪狂犬病病毒感染情况的调查[J]. 动物医学进展,2015, 36(3): 133-136.
[4] 赵丽, 崔保安, 方忠意, 等. PCR检测猪伪狂犬病病毒方法的研究[J]. 中国预防兽医学报, 2007, 2 (2):142-146.
[5] 周斌, 苏鑫铭, 张素芳, 等. PCR快速检测伪狂犬病病毒野毒感染[J]. 中国病毒学, 2004, 19(6): 612-615.
[6] 赵丽, 崔保安, 陈红英. 等. 实时荧光定量PCR检测伪狂犬病病毒方法的建立与初步应用[J]. 中国兽医学报,2009, 29(4): 433-436.
[7] Harding M J, Prud homme I, Rola J. Specificity and nucleotyping studies of a gp50 based polymerise chain reaction assay for detection of pseudorabies virus[J]. Can J Vet Res, 1997, 61(2): 157-160.
[8] Jestin A, Foulon T, Pertuiset B, et al. Rapid detection ofpseudorabies virus genomic sequence in biological sampls from infected pigs using polymerase chain reaction reaction DNA amplification[J]. Vet Microbiol, 1990,23(1-4): 317-328.
[9] Talmage T B, Kwang O S, Frederick J F. Detection of PR viral DNA in tonsillar cells of latently infected pigs[J]. Am J Vet Kes, 1995, 56 (5): 587-594.
[10] 娄高明, 杜伟贤, 廖筱萍, 等. PCR检测伪狂犬病病毒DNA[J]. 中国生物化学与分子生物学报, 2001, 1(4): 519-523.
[11] 吕建强, 何云蔚, 卢体康, 等. 伪狂犬病病毒实时荧光PCR的建立和应用[J]. 动物医学进展, 2010, 31(3): 21-25.
[12] 金升藻, 陈焕春, 熊符. 伪狂犬病基因缺失疫苗研究进展[J].中国农业科学, 2002, 35(1): 89 -93.
[13] 张雪云. 猪伪狂犬病诊断方法研究进展[J]. 哈尔滨师范大学自然科学学报, 2104, 30(1): 71-75.
[14] 郝飞, 汤德元, 曾智勇, 等. 猪伪狂犬病诊断技术研究进展[J]. 猪业科学, 2013, (7): 92-94.
[15] 刘丽娜, 何启盖, 陈焕春, 等. 用多重PCR鉴别猪伪狂犬病野毒与疫苗毒的研究[J]. 中国兽医科技, 2005, 35 (2): 95-98.
[16] 蔺芳, 尹双辉, 尚佑军, 等. 多重PCR方法快速鉴别猪伪狂犬疫苗毒和野毒[J]. 安徽农业科学, 2009, 37 (13): 5889-5891.
DEVELOPMENT OF NESTED PCR FOR DIFFERENTIATION OF GE GENE DELETION VACCINE AND FIELD ISOLATES OF PSEUDORABIES VIRUS
ZHU Xiao-fu, WU Xu-jin
(Animal Epidemic Disease Diagnostic Laboratory of Molecular Biology , Institute of Animal Husbandry and Veterinary Medicine, Xianyang Vocational Technical College, Xianyang 712000, China)
In order to develop a nested PCR for differentiation of gE gene deletion vaccine and fi eld isolates of Pseudorabies virus(PRV),four pairs of primers were designed and synthesized according to gD, gE gene sequences of PRV published in GenBank. The reaction factors and conditions were optimized to establish a reliable nested PCR method. The results showed that the detection limit was 2.78×10-4pg/L. The PRV fi eld strains had 354 bp and 217 bp while PRV vaccine stain had 217 bp band only in agarose gel electrophoresis. There was no amplifi cation from other fi ve common swine viruses. The complex nested PCR developed here provided a reliable method for differentiation of gE gene deletion vaccine and fi eld isolates, which could be used for clinical diagnosis of pseudorabies.
Pseudorabies virus; gD gene; gE gene; differential diagnosis; nested PCR
S852.659.1
A
1674-6422(2016)04-0026-05
2015-12-23
咸阳市科学技术研究计划项目(2015k03-21)
朱小甫,男,硕士,主要从事动物疫病分子病原学与免疫学研究
吴旭锦,E-mail:zhuxiaofu2004@aliyun.com

