UDP-糖基供体的生物合成途径分析
尹森,孔建强
·综述·
UDP-糖基供体的生物合成途径分析
尹森,孔建强
糖基化是生物体中重要的生化反应,它可发生于大分子化合物,如蛋白质的糖基化[1];也可发生于小分子化合物,如各种天然产物苷元的糖基化[2]。执行化合物糖基化反应的催化酶为糖基转移酶。糖基转移酶的底物分为糖基受体和糖基供体。糖基受体可以为生物大分子化合物如蛋白质、核酸,还可以为小分子化合物如各种植物或微生物的次生代谢产物[3-5]。糖基受体通常含一些活性基团,如羟基、氨基、羧基、巯基等用于形成糖苷键,同时有少数糖基转移酶还可以催化 C-C 键的形成,生成碳苷类化合物[6-7]。糖基供体为糖的活化形式,通常糖基供体中存在一个或多个高能磷酸键,这些高能磷酸键在糖基转移酶执行催化反应时能通过能量转移而形成化合物与糖的糖苷键。常见糖基供体为尿苷二磷酸糖(UDP-糖)、胸腺苷二磷酸糖(TDP-糖)、鸟苷二磷酸糖(GDP-糖)、一磷酸糖等。这些糖基供体之间可以通过不同的代谢酶进行相互转化(图 1)。
生物体中最常见的糖基供体形式为 UDP-糖。常见的UDP-糖有两种类型,一种为六碳糖,包括 UDP-葡萄糖、UDP-葡萄糖醛酸、UDP-半乳糖、UDP-半乳糖醛酸和 UDP-鼠李糖等;一种为五碳糖,包括 UDP-木糖、UDP-阿拉伯糖和 UDP-芹菜糖等。其中 UDP-葡萄糖是其他 UDP-单糖形成的起始物,而 UDP-葡萄糖醛酸是 UDP-单糖由六碳糖转变为五碳糖的关键底物。
主要的催化 UDP-糖相互转化的相关酶可分为脱氢酶类[UDP-葡萄糖脱氢酶(UDP-glucose dehydrogenase,UGDH)]、脱羧酶类[UDP-木糖合酶(UDP-xylose synthase,UXS)、UDP-芹菜糖/UDP-木糖合酶(UDP-apiose/UDP-xyl synthase,UAXS)]、差向异构酶类[UDP-木糖差向异构酶(UDP-D-xylose epimerase,UXE)、UDP-葡萄糖差向异构酶(UDP-D-glucose epimerase,UGE)、UDP-葡萄糖醛酸差向异构酶(UDP-D-glucuronic acid epimerase,UGlcAE)]和还原酶类[UDP-鼠李糖合酶(UDP-rhamnose synthase,RHM 或 NRS/ER)][8]。
1 UDP-葡萄糖的来源及生物合成途径
由于 UDP-葡萄糖是其他 UDP-单糖形成的起始化合物,所以研究 UDP-葡萄糖的来源尤为重要。
目前主要有三个 UDP-葡萄糖的来源:①来源于生物体中的 6-磷酸葡萄糖;②来源于蔗糖;③来源于一些糖基转移酶的糖基交换。6-磷酸葡萄糖在葡萄糖磷酸变位酶(phosphoglucomutase,PGM)的作用下转变为 1-磷酸葡萄糖,然后又在 UDP-葡萄糖焦磷酸化酶(UDP-glucose pyrophosphorylase,UGPase)作用下形成 UDP-葡萄糖[9]。蔗糖合酶则可以在 UDP 存在的情况下催化蔗糖直接形成UDP-葡萄糖和果糖,同时蔗糖合酶还可以执行这一反应的逆反应[10]。在植物或微生物中存在一些糖基转移酶有糖基交换的功能,即糖基转移酶在 UDP 存在的情况下能催化葡萄糖苷分子中的糖基与另一种 UDP-单糖之间糖分子的相互交换而得到 UDP-葡萄糖[11]。
2 由 UDP-葡萄糖到 UDP-阿拉伯糖的生物合成途径
由 UDP-葡萄糖到 UDP-阿拉伯糖主要有三种代谢酶参与三步反应:脱氢、脱羧和差向异构。UDP-葡萄糖在UDP-葡萄糖脱氢酶(UGDH)的作用下形成 UDP-葡萄糖醛酸。UDP-葡萄糖醛酸在 UDP-木糖合酶的催化下形成 UDP-木糖。UDP-木糖在 UDP-木糖差向异构酶(UXE)的作用下形成 UDP-阿拉伯糖。
UGDH 是一种 NAD+依赖型的酶,目前已有多个UGDH 从多类生物和多种生物组织中被分离获得。植物中,UDP-葡萄糖脱氢酶的作用在豌豆中第一次被描述。在UDP-葡萄糖到 UDP-木糖或阿拉伯糖的代谢流中,后者对UGDH 存在反馈抑制的作用,即 UDP-木糖能够抑制UGDH 的活性[9]。
植物中的 UXS 存在两种类型,一种是膜结合型,一种以可溶形式存在于细胞质中[12]。根据系统进化分析,拟南芥和大麦中的 UXS 基因家族被分为三个亚型,其中一种亚型以可溶形式存在于细胞质中,另外两个为膜结合型[13-14]。两个膜结合型的 UXS 亚型的氨基酸序列主要区别在于跨膜区的 N 端前 100 个氨基酸[15]。植物中还存在一种双功能的 UDP-糖脱羧酶,可以催化 UDP-葡萄糖醛酸生成UDP-木糖,即 UDP-芹菜糖/UDP-木糖合酶(UAXS)。UAXS催化 UDP-葡萄糖醛酸所得产物除了 UDP-木糖,还有UDP-芹菜糖[16]。UAXS 与 UXS 虽然有相似的催化活性,但其酶学性质差别较大。UXS 偏好酸性条件进行反应[17],而 UAXS 在碱性条件下具有较高活性;UXS 在较低温度(20 ℃)活性较高,而 UAXS 则在较高温度(50 ℃)时具有更高活性。UAXS 对于 NAD+具有严格的依赖性,在没有外加 NAD+的情况下活性消失;UXS 对于 NAD+没有绝对的依赖性,非外加条件下仍具有活性,但在外加NAD+的情况下活性更高[18]。UXS 与 NAD+形成紧密的结合,NAD+能参与 UDP-GlcA 氧化形成 UDP-4-酮基-己糖中间体,进而脱羧形成 UDP-4-酮基-戊糖,然后构象变化最终形成 UDP-Xyl[19]。UXS 与 NADH 结合减少 UDP-4-酮基-己糖转化为 UDP-Xyl,导致 NAD+再生[20]。原始的UXS/UAXS 起源于早期的原核生物,然后再衍变成两个在功能上有明显区别的酶。早期的细菌 UXS 形成了现在的UXS 同系物:细菌、动物、真菌、植物。早期的细菌 UAXS变成了现在的植物 UAXS、细菌 ArnA 同系物和家族 B蛋白[21]。随着进化,植物的 UAXS 获得了与 RsU4kpxs 不同的功能,它可以通过 UDP-4-酮基-戊糖中间体形成 UDP-芹菜糖和 UDP-木糖两个产物[16]。
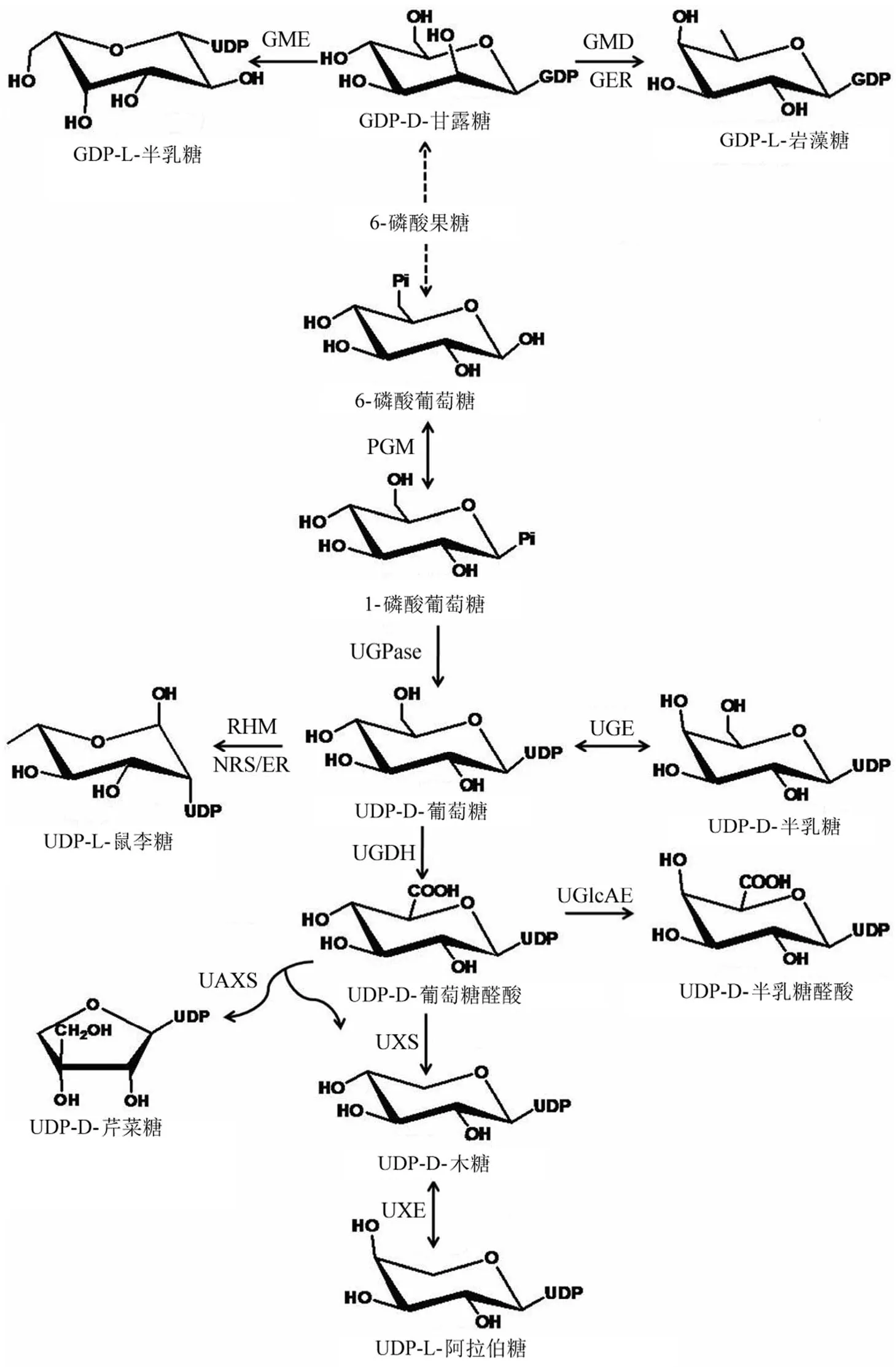
图1 核苷单糖的生物合成途径
在植物中,UDP-阿拉伯糖的生物合成主要由 UXE 执行。植物中的 UXE 为定位于高尔基体上的膜结合蛋白,其可以催化 UDP-葡萄糖 4 位羟基的差向异构化,从而形成UDP-阿拉伯糖[22]。
3 差向异构酶催化形成 UDP-糖的生物合成途径
生物体中主要有三对 UDP-糖的差向异构体,即 UDP-葡萄糖与 UDP-半乳糖、UDP-葡萄糖醛酸与 UDP-半乳糖醛酸以及 UDP-木糖与 UDP-阿拉伯糖。在微生物、动物中存在UDP-乙酰胺基葡萄糖和 UDP-乙酰胺基半乳糖[23-24]。
UDP-糖的 4 位差向异构酶是目前研究较多的一种类型[25]。UDP-葡萄糖和 UDP-半乳糖之间由 UDP-葡萄糖/半乳糖-4-差向异构酶催化,UDP-木糖和 UDP-阿拉伯糖之间的相互转化由 UDP-木糖/阿拉伯糖-4-差向异构酶催化[26],UDP-葡萄糖醛酸和 UDP-半乳糖醛酸由 UDP-葡萄糖醛酸/半乳糖醛酸-4-差向异构酶催化。此外在动物细胞中还发现催化 UDP-葡萄糖与 UDP-氮乙酰胺基葡萄糖的 4-差向异构酶[27]。这些 4 位差向异构酶具有相应的底物特异性。UGE 在维管植物中存在两种类型,一种类型除了能够催化UDP-葡萄糖和 UDP-半乳糖之间的转化之外,还能够催化UDP-木糖/阿拉伯糖的转化,而另一种 UGE 则对 UDP-木糖的催化效率很低,但这种植物中的 UGE 对于 UDP-氮乙酰胺基葡萄糖并不存在催化活性,与动物细胞中的双功能酶不同点在于其中的一个门控氨基酸残基的作用。而植物中的UXE 只能特异性识别 UDP-木糖和 UDP-阿拉伯糖,但不能催化 UDP-葡萄糖/半乳糖。UDP-葡萄糖醛酸差向异构酶只能特异性识别 UDP-GluAc 和 UDP-GalAc[28-29]。
对于 UDP-木糖与 UDP-阿拉伯糖之间的转化如前所述,由 UXE 执行。UDP-葡萄糖与 UDP-半乳糖之间的转化由 UDP-葡萄糖差向异构酶(UGE)执行。UGE 以可溶形式存在于细胞质中,根据系统进化关系可以分为两种亚型。其中一种亚型除了催化 UDP-葡萄糖与半乳糖之间的转化外,还具有较高的催化 UDP-木糖与阿拉伯糖之间相互转化的活性,而另一种 UGE 亚型则具有较低的此类活性(图 2)。同时,两种类型的 UGE 对于 UDP-半乳糖的催化活性较高,而对于 UDP-葡萄糖的催化活性较低,即 UGE能将大量的 UDP-半乳糖转化为 UDP-葡萄糖,却只能将少量 UDP-葡萄糖转化为 UDP-半乳糖[28, 30]。
UDP-葡萄糖醛酸差向异构酶(UGlcAE)是膜结合型的蛋白,定位于高尔基体上,UGlcAE 活性的发挥依赖NAD+[31]。目前对于 UGlcAE 的克隆与功能鉴定主要的方法有两种,一种是将其跨膜区截去并进行原核表达,另一种方法是使用真核表达[32-33]。
4 UDP-鼠李糖的生物合成途径
NDP-鼠李糖(在植物中主要为 UDP-鼠李糖,细菌中主要为 dTDP-鼠李糖)的生物合成在细菌、真菌和植物中由不同的酶参与催化。由 UDP-葡萄糖为底物最终生成UDP-鼠李糖,一共经历了 3 步反应:首先 UDP-葡萄糖在4,6-脱水酶(在细菌中为 rmlB)的催化下生成 NDP-4-酮基-6-去氧葡萄糖,然后再在 3,5-异构酶(在细菌中为 rmlC)的催化下生成 UDP-4-酮基-鼠李糖,最后在 4-还原酶(细菌中为 rmlD)的催化下生成 UDP-鼠李糖。其中第一步和第三步反应分别需要辅因子 NAD+和 NADPH 的参与[8]。
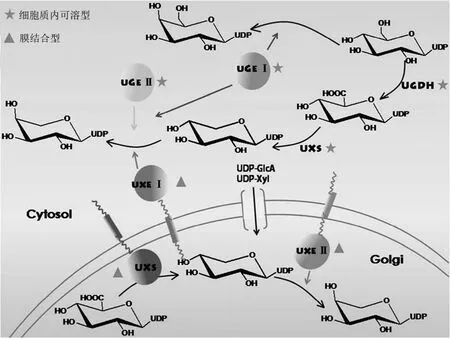
图2 植物中差向异构酶及其类型
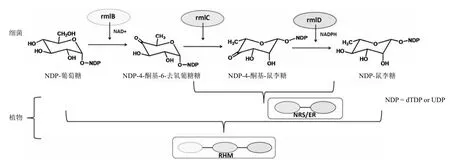
图3 NDP-鼠李糖合酶的进化关系
高等植物参与 UDP-鼠李糖生物合成的酶有两种,分别为 NRS/ER 和 RHM。NRS/ER 整合了细菌 rmlC、rmlD 两种酶的功能,即其能以 UDP-4-酮基-6-去氧葡萄糖为底物催化其形成 UDP-鼠李糖[34-35]。而 RHM 整合了细菌 UDP-鼠李糖生物合成途径中的三种酶,可以将 UDP-葡萄糖直接转化为 UDP-鼠李糖[36](图 3)。目前已有应用 RHM 与相应的鼠李糖糖基转移酶组合,通过生物催化的方法合成天然产物鼠李糖苷的研究[37]。
5 小结
UDP-糖基供体生物合成途径的研究是天然小分子化合物的糖基化研究的重要组成部分。不同糖基供体生物合成途径的组合,可以为糖基化过程提供不同种类的糖基供体。对于整合有多种糖基供体代谢途径的工程菌在与特定的糖基转移酶组合之后,可以合成特定天然小分子化合物的特定种类的糖苷。所以对于糖基供体生物合成途径的更深入的研究,可以为天然产物糖基化研究及其产业化提供更多的理论基础和指导原则。
[1] Lairson LL, Henrissat B, Davies GJ, et al. Glycosyltransferases:structures, functions, and mechanisms. Annu Rev Biochem, 2008,77:521-555.
[2] Thibodeaux CJ, Melancon CE 3rd, Liu HW. Natural-product sugar biosynthesis and enzymatic glycodiversification. Angew Chem Int Ed Engl, 2008, 47(51):9814-9859.
[3] Lim EK. Plant glycosyltransferases: their potential as novel biocatalysts. Chemistry, 2005, 11(19):5486-5494.
[4] Wang X. Structure, mechanism and engineering of plant natural product glycosyltransferases. FEBS Lett, 2009, 583(20):3303-3309.
[5] Vogt T, Jones P. Glycosyltransferases in plant natural product synthesis: characterization of a supergene family. Trends Plant Sci,2000, 5(9):380-386.
[6] Xie K, Chen R, Li J, et al. Exploring the catalytic promiscuity of a new glycosyltransferase from Carthamus tinctorius. Org Lett, 2014,16(18):4874-4877.
[7] Chen D, Chen R, Wang R, et al. Probing the catalytic promiscuity of a regio- and stereospecific C-glycosyltransferase from mangifera indica. Angew Chem Int Ed Engl, 2015, 54(43):12678-12682.
[8] Yin Y, Huang J, Gu X, et al. Evolution of plant nucleotide-sugar interconversion enzymes. PLoS One, 2011, 6(11):e27995.
[9] Bar-Peled M, O'Neill MA. Plant nucleotide sugar formation,interconversion, and salvage by sugar recycling. Annu Rev Plant Biol,2011, 62:127-155.
[10] Li LN, Kong JQ. Transcriptome-wide identification of sucrose synthase genes in Ornithogalum caudatum. RSC Adv, 2016, 6(23):18778-18792.
[11] Bode HB, Muller R. Reversible sugar transfer by glycosyltransferases as a tool for natural product (bio)synthesis. Angew Chem Int Ed Engl,2007, 46(13):2147-2150.
[12] Moriarity JL, Hurt KJ, Resnick AC, et al. UDP-glucuronate decarboxylase, a key enzyme in proteoglycan synthesis: cloning,characterization, and localization. J Biol Chem, 2002, 277(19):16968-16975.
[13] Harper AD, Bar-Peled M. Biosynthesis of UDP-xylose. Cloning and characterization of a novel Arabidopsis gene family, UXS, encoding soluble and putative membrane-bound UDP-glucuronic acid decarboxylase isoforms. Plant Physiol, 2002, 130(4):2188-2198.
[14] Hayashi T, Koyama T, Matsuda K. Formation of UDP-xylose and xyloglucan in soybean golgi membranes. Plant Physiol, 1988, 87(2):341-345.
[15] Pattathil S, Harper AD, Bar-Peled M. Biosynthesis of UDP-xylose:characterization of membrane-bound AtUxs2. Planta, 2005, 221(4):538-548.
[16] Ahn JW, Verma R, Kim M, et al. Depletion of UDP-D-apiose/ UDP-D-xylose synthases results in rhamnogalacturonan-II deficiency,cell wall thickening, and cell death in higher plants. J Biol Chem,2006, 281(19):13708-13716.
[17] Du Q, Pan W, Tian J, et al. The UDP-glucuronate decarboxylase gene family in Populus: structure, expression, and association genetics. PLoS One, 2013, 8(4):e60880.
[18] Mølhøj M, Verma R, Reiter WD. The biosynthesis of the branched-chain sugar d-apiose in plants: functional cloning and characterization of a UDP-d-apiose/UDP-d-xylose synthase from Arabidopsis. Plant J, 2003, 35(6):693-703.
[19] Gu X, Glushka J, Yin Y, et al. Identification of a bifunctional UDP-4-keto-pentose/UDP-xylose synthase in the plant pathogenic bacterium Ralstonia solanacearum strain GMI1000, a distinct member of the 4,6-dehydratase and decarboxylase family. J Biol Chem, 2010,285(12):9030-9040.
[20] Suzuki K, Watanabe K, Masumura T, et al. Characterization of solubleand putative membrane-bound UDP-glucuronic acid decarboxylase (OsUXS) isoforms in rice. Arch Biochem Biophys, 2004, 431(2):169-177.
[21] Williams GJ, Breazeale SD, Raetz CR, et al. Structure and function of both domains of ArnA, a dual function decarboxylase and a formyltransferase,involvedin4-amino-4-deoxy-L-arabinose biosynthesis. J Biol Chem, 2005, 280(24):23000-23008.
[22] Zhang Q, Shirley NJ, Burton RA, et al. The genetics, transcriptional profiles, and catalytic properties of UDP-alpha-D-xylose 4-epimerases from barley. Plant Physiol, 2010, 153(2):555-568.
[23] Dong S, Chesnokova ON, Turnbough CL Jr, et al. Identification of the UDP-N-acetylglucosamine 4-epimerase involved in exosporium protein glycosylation in Bacillus anthracis. J Bacteriol, 2009, 191(22):7094-7101.
[24] Guo H, Li L, Wang PG. Biochemical characterization of UDP-GlcNAc/Glc 4-epimerase from Escherichia c oli O86:B7. Biochemistry, 2006, 45(46):13760-13768.
[25] Fan DF, Feingold DS. Nucleoside diphosphate-sugar 4-epimerases. II. Uridine diphosphate arabinose 4-epimerase of wheat germ. Plant Physiol, 1970, 46(4):592-595.
[26] Burget EG, Verma R, Molhoj M, et al. The biosynthesis of L-arabinose in plants: molecular cloning and characterization of a Golgi-localized UDP-D-xylose 4-epimerase encoded by the MUR4 gene of Arabidopsis. Plant Cell, 2003, 15(2):523-531.
[27] Bengoechea JA, Pinta E, Salminen T, et al. Functional characterization of Gne (UDP-N-acetylglucosamine-4-epimerase), Wzz (chain length determinant), and Wzy (O-antigen polymerase) of Yersinia enterocolitica serotype O:8. J Bacteriol, 2002, 184(15):4277-4287.
[28] Kotake T, Takata R, Verma R, et al. Bifunctional cytosolic UDP-glucose 4-epimerases catalyse the interconversion between UDP-D-xylose and UDP-L-arabinose in plants. Biochem J, 2009,424(2):169-177.
[29] Guevara DR, El-Kereamy A, Yaish MW, et al. Functional characterization of the rice UDP-glucose 4-epimerase 1, OsUGE1: a potential role in cell wall carbohydrate partitioning during limiting nitrogen conditions. PLoS One, 2014, 9(5):e96158.
[30] Yin S, Kong JQ. Transcriptome-guided discovery and functional characterization of two UDP-sugar 4-epimerase families involved in the biosynthesis of anti-tumor polysaccharides in Ornithogalum caudatum. RSC Adv, 2016, 6(44):37370-37384.
[31] Gu X, Wages CJ, Davis KE, et al. Enzymatic characterization and comparison of various poaceae UDP-GlcA 4-epimerase isoforms. J Biochem, 2009, 146(4):527-534.
[32] Mølhøj M, Verma R, Reiter WD. The biosynthesis of D-Galacturonate inplants.functionalcloningandcharacterizationofa membrane-anchoredUDP-D-Glucuronate4-epimerasefrom Arabidopsis. Plant Physiol, 2004, 135(3):1221-1230.
[33] Gu X, Glushka J, Lee SG, et al. Biosynthesis of a new UDP-sugar,UDP-2-acetamido-2-deoxyxylose, in the human pathogen Bacillus cereus subspecies cytotoxis NVH 391-98. J Biol Chem, 2010,285(32):24825-24833.
[34] Martinez V, Ingwers M, Smith J, et al. Biosynthesis of UDP-4-keto-6-deoxyglucose and UDP-rhamnose in pathogenic fungi Magnaporthe grisea and Botryotinia fuckeliana. J Biol Chem, 2012,287(2):879-892.
[35] Watt G, Leoff C, Harper AD, et al. A bifunctional 3,5-epimerase/4-keto reductase for nucleotide-rhamnose synthesis in Arabidopsis. Plant Physiol, 2004, 134(4):1337-1346.
[36] Diet A, Link B, Seifert GJ, et al. The Arabidopsis root hair cell wall formation mutant lrx1 is suppressed by mutations in the RHM1 gene encoding a UDP-L-rhamnose synthase. Plant Cell, 2006, 18(7):1630-1641.
[37] Yonekura-Sakakibara K, Tohge T, Niida R, et al. Identification of a flavonol 7-O-rhamnosyltransferase gene determining flavonoid pattern in Arabidopsis by transcriptome coexpression analysis and reverse genetics. J Biol Chem, 2007, 282(20):14932-14941.
10.3969/j.issn.1673-713X.2016.04.013
中国医学科学院中央级公益性科研院所基本科研业务费(2016ZX350001)
100050 北京,中国医学科学院北京协和医学院药物研究所天然药物活性物质与功能国家重点实验室/卫生部天然药物生物合成重点实验室
孔建强,Email:jianqiangk@imm.ac.cn
2016-03-29

