人骨髓间充质干细胞诱导分化为汗腺样细胞过程中microRNA表达谱差异的分析
陈 艳 赵洪良 谭志军 赵焕军 张翠萍 付小兵2,(1.北京军区总医院药理科,北京 100700;2.解放军总医院基础医学研究所,北京 10085;.解放军总医院第一附属医院全军创伤修复与组织再生重点实验室暨皮肤损伤修复与组织再生北京市重点实验室,北京 100048)
论著
人骨髓间充质干细胞诱导分化为汗腺样细胞过程中microRNA表达谱差异的分析
陈 艳1,2赵洪良3谭志军3赵焕军3张翠萍3付小兵2,3
(1.北京军区总医院药理科,北京 100700;2.解放军总医院基础医学研究所,北京 100853;3.解放军总医院第一附属医院全军创伤修复与组织再生重点实验室暨皮肤损伤修复与组织再生北京市重点实验室,北京 100048)
【摘要】目的:利用微小RNA(microRNA)表达谱基因芯片检测新技术,寻找人骨髓间充质干细胞(MSCs)分化为汗腺样细胞(SGLCs)过程中的关键microRNAs及其介导的分子信号通路。方法:通过间接培养方法诱导MSCs分化为SGLCs后,利用microRNA基因芯片检测,分析MSCs诱导前后以及MSCs、SGLC与正常成人汗腺细胞(SGCs)之间microRNA差异表达谱,筛选出其中发生极其显著改变的microRNAs,通过microRNA靶点预测软件,进一步筛选出其中直接靶向与汗腺发育、干细胞分化相关基因的microRNAs及其靶基因。结果:microRNA表达谱的差异比对结果显示,SGLCs与MSCs相比,19 个microRNAs 上调,49个microRNAs 下调;SGCs与MSCs相比,120个microRNAs 上调,59个microRNAs下调。取SGLCs与MSCs差异表达谱和SGCs 与MSCs差异表达谱的交集,发现37个microRNAs在以上两个差异表达谱中均出现,其中12个microRNAs上调,25个microRNAs下调,它们可直接靶向与汗腺发育和干细胞分化相关的CEA等多个细胞因子,并参与EDA/ EDAR、NF-κB、Wnt、ERK等多个相关信号通路的调节。结论:通过生物信息学靶点分析,成功寻找到在MSCs分化为SGLCs的过程中,靶向汗腺发育和干细胞分化相关细胞因子和信号通路的关键microRNAs。
关键词骨髓间充质干细胞汗腺细胞microRNA表达谱差异性分析
本实验室前期研究建立了体外热休克汗腺细胞(SGCs)诱导人骨髓间充质干细胞(MSCs)分化为汗腺样细胞(SGLCs)的培养体系,并证明了无汗性外胚叶发育不良蛋白(EDA-A1)、重组人表皮生长因子(EGF)和胰岛素-转铁蛋白-亚硒酸钠(ITS)等相关汗腺诱导因子能促进汗腺的增殖发育和诱导分化[1-3]。最近研究表明,大于60%的编码基因翻译成蛋白质的过程都受微小RNA(microRNA)调控,检测这些关键调控节点的变化对于研究差异表达基因的生物学背景极为重要[4-5]。因此,寻找MSCs诱导成汗腺样细胞过程中的关键microRNAs及其靶基因表达的变化对阐明该诱导分化过程的分子机制意义重大。本实验采用Affymetrix® GeneChip® microRNA 4.0 Arrays芯片,全面检测miRBase Release 20数据库中所有成熟的microRNAs序列,并列出了宿主基因ID、microRNAs的靶基因以及microRNAs的聚类信息,通过表达谱的差异分析,找出关键调控的microRNAs、其可能的靶基因以及参与的信号调控网络。
1 材料与方法
1.1仪器与试剂人Affymetrix® GeneChip® microRNA 4.0 Arrays 芯 片 (Affymetrix芯片)、真核杂交试剂盒和杂交、清洗、染色试剂盒购自美国Affymetrix公司。mirVana™microRNA纯化试剂盒(AM1561)购自美国Ambion公司。FlashTag™ Biotin RNA 标记试剂盒购自美国 Genisphere 公司。Trizol 试剂购自美国Life Tech公司。Tris-Cl购自美国Capitalbio 公司。氯仿、异丙醇和乙醇购自北京化工厂。高速冷冻离心机(5900)生产厂家为日本Kubota公司。PCR 仪(PTC-225)为美国MJ公司生产。Hybridization Oven 640、Fluidics Station 450 和 GeneChip® Scanner 3000为美国Affymetrix公司生产。一次性RNase-Free吸头、离心管生产厂家为美国Axygen公司。
1.2实验分组当Transwell培养板下室的汗腺细胞生长达70%~80%融合后,上室中加入培养的第3代MSCs,利用Transwell透明聚酯膜培养板,按参考文献[3]的诱导方法,进行汗腺热休克、间接共培养,并添加EDA-A1、EGF和ITS等系列诱导因子。在MSCs加入到Transwell透明聚酯膜培养板上室的同时,取同批次MSCs,于6孔组织培养板中,以MSCs培养基常规培养,作为MSCs平行对照组;同样,在汗腺团加入到transwell透明聚酯膜培养板下室的同时,取同批次汗腺团,于6孔组织培养板中,以SGCs培养基常规培养,作为SGCs平行对照组。
1.3MicroRNA芯片检测样本制备热休克SGC 与MSCs间接共培养诱导10 d后,收集上室诱导后的SGLCs。同时,分别收集未经诱导的MSCs平行对照组和SGCs平行对照组细胞。用 1×PBS冲洗细胞3次,晾干。每 1×107个细胞加入 1 ml Trizol,利用氯仿-异丙醇-乙醇分别提取3组样本的总 RNA。
1.4MicroRNA表达谱芯片检测
1.4.1实验原理首先将提取的待测样品总RNA进行定量和完整性质检,然后利用microRNA纯化成套装置进一步纯化获取microRNA,再利用生物素进行microRNA标记、杂交、检测。MicroRNA表达谱检测的主要步骤包括:①poly A 加尾:通过 poly A 聚合酶的作用,在小RNA 的 3’端进行 poly A 加尾;②连接3DNA dendrimer:通过连接酶的引导作用,将带有生物素标记的3DNA dendrimer 连接到上述已加尾RNA 的 poly A 尾巴上;③对Affymetrix芯片进行杂交、清洗染色、扫描检测荧光及数据结果分析。
1.4.2 实验方法
1.4.2.1总RNA的质检和纯化按前述方法采用Trizol 试剂提取各样本的总 RNA,然后进行RNA定量(紫外分光光度计或Qubit等方法)和RNA完整性检测(琼脂糖凝胶电泳或Agilent2100检测);使用mirVana™ microRNA 纯化试剂盒(AM1561)对总 RNA进行纯化。
1.4.2.2Poly A加尾①稀释ATP mix:对于起始样本量为1 µg 的总RNA,取1支1.5 ml 的EP管,加入500 µl 1 mmol/L 的Tris-Cl和1 µl 的ATP mix,冰浴中放置。②配制RNA spike control混合物;另取200 µl EP 管1支,加入1 µg 经纯化处理的样本总RNA,用无RNA酶的水稀释至8 µl,再加入RNA Spike Control Oligos 2 µl,冰浴中放置。③配制Poly(A) Tailing Mix 5 µl:10倍体积的反应缓冲液1.5 µl,25 mmol/L MnCl21.5 µl, 上述稀释的 ATP mix 1 µl,PAP 酶1 µl,利用移液器将其加入至上述配置的10 µl RNA spike control的混合物中,冰浴中放置。④将盛有上述准备好样品的EP管放入PCR仪中,37 ℃ 15 min,4 ℃保存。
1.4.2.3生物素标记在上述反应后的15 µl 体系中,使用移液枪加入5×Flash Tag Ligation Mix Biotin 4 µl;然后往上述体系中加入T4 DNA Ligase 2 µl,这时终体积达到21 µl,使所有溶液管底集中;PCR反应:25 ℃ 30 min,4 ℃保存;向EP管中加入终止液2.5 µl终止反应。
1.4.2.4芯片杂交①预先取出芯片,室温下平衡若干小时。②配制杂交反应体系: 20倍浓缩液的真核生物杂交对照在使用前需进行65 ℃变性处理5 min。配制单张芯片微阵列所需的杂交混合液81.7 µl:2×杂交液50 µl,27.5%的甲酰胺15 µl,DMSO 10 µl,20倍浓缩液的真核生物杂交对照(bioB, bioC, bioD, cre)5 µl,寡核苷酸对照B2(3 nmol/L)1.7 µl。③将上述杂交体系于99 ℃恒温金属浴温育5 min, 45 ℃恒温金属浴温育 5 min;放入高速微量离心机中13 200 r/min离心5 min。④芯片加样:根据不同的芯片类型加入相应体积的杂交液,并用标签纸做好标记;芯片放在杂交炉中(保持水平放置)进行旋转杂交:48 ℃,60 r/min 16 h。
1.4.2.5芯片清洗、染色、检测及数据分析①杂交完成后,弃尽杂交液,注入Wash Buffer A 。②使用 Affymetrix® GeneChip® Commol/Land Console® Software(AGCC软件)的如下程序对芯片进行清洗、染色处理:清洗#1,25 ℃,清洗缓冲液A,10个循环,每个循环混合2次;清洗#2,50 ℃,清洗缓冲液B,8个循环,每个循环混合15次;染色#1,25 ℃,染料1,染色探针微阵列,10 min;染色后清洗,30 ℃,清洗缓冲液A,10个循环,每个循环混合4次;染色#2,25 ℃,染料2,染色探针微阵列,10 min;染色#3,25 ℃,染料3,染色探针微阵列,10 min;最后一次清洗,35 ℃,清洗缓冲液A,15个循环,每个循环4次;芯片维持缓冲液,维持缓冲液注入探针微阵列。 ③将上述染色清洗好的芯片放入扫描仪,点击Scan进行检测。扫描时间一般设定为6 min;芯片微阵列的荧光扫描结束后,将图像保存为“.DAT”类型的文件,以便后续进行数据分析。
1.5与汗腺发育、干细胞分化相关microRNA靶点的分析和验证筛选出MSCs诱导分化为汗腺细胞过程中发生显著变化的差异microRNAs后,利用miRBase并结合microRNA靶点预测软件DIANAmT、miRanda、miRDB、miRWalk、RNAhybrid、PICTAR5、PITA、RNA22、Targetscan,预测出其可能直接作用的靶基因,利用Microsoft Excel 2007建立microRNAs的靶基因数据库。打开该靶基因数据库,点击菜单按钮“筛选”,点击“靶基因”这一列列首右下角的三角形按钮,在下拉框中选择“文本筛选”,“包含”字段中分别输入已知的与干细胞分化、汗腺发育相关的基因,点击“确定”,即显示该数据库中可能靶向该基因的差异性表达的microRNAs。
1.6实验结果分析Affymetrix芯片扫描结果的文件类型为“.CEL”文件,与Affymetrix® Expression Console™ 软 件 相 互 兼 容, 便于数据结果分析。差异表达的数据分析可通过 Affymetrix® Transcriptome Analysis Console 实现。结果判定:SGLC组与MSC组检测数据的比值[SGLC/MSC(Ratio)] >2,表示显著高调的microRNA,记录Transcript ID(Array Design);SGLC/MSC(Ratio)<0.5, 表 示 显 著 低 调 的microRNA, 记 录Transcript ID(Array Design)。
2 结 果
2.1MSCs诱导前后MicroRNA表达散点图分析诱导组与MSC对照组的比对结果显示,显著上调的microRNAs位点有19个;显著下调的microRNAs位点有49个(图1A,见封三)。汗腺对照组与MSC对照组的比对结果显示,显著上调的microRNAs位点有120个,显著下调的microRNAs位点有59个(图1B,见封三)。我们发现图1B比图1A有更多的microRNAs位点发生了显著调高和调低的迁移,说明microRNA表达谱正由诱导后向汗腺转变。诱导后细胞与汗腺细胞比较,上调的miRNAs位点有38个,下调的microRNAs位点有131个(图1C,见封三)。
2.2MicroRNA数据比对分析microRNA Array 的检测结果纷繁复杂,为了缩小研究范围,筛选更有意义的microRNA位点进行下一步的验证分析,本研究中取图1A和图1B两个结果的交集,共得到37个microRNA位点(其中调高的12种,调低的25种,见表1和表2,这些microRNA在诱导分化后的SGLCs与MSCs对照组的microRNA表达谱比较中,以及成人汗腺与MSCs的microRNA表达谱比较中,均有显著变化;表示这些microRNAs既是MSCs诱导分化为汗腺样细胞过程中的关键microRNAs,也存在于成熟的汗腺细胞中并维持着显著的差异性表达,具有双重显著意义。这些筛选结果,为后期验证microRNAs作用靶点和分析信号通路,提供了研究范围和可靠的理论支持。
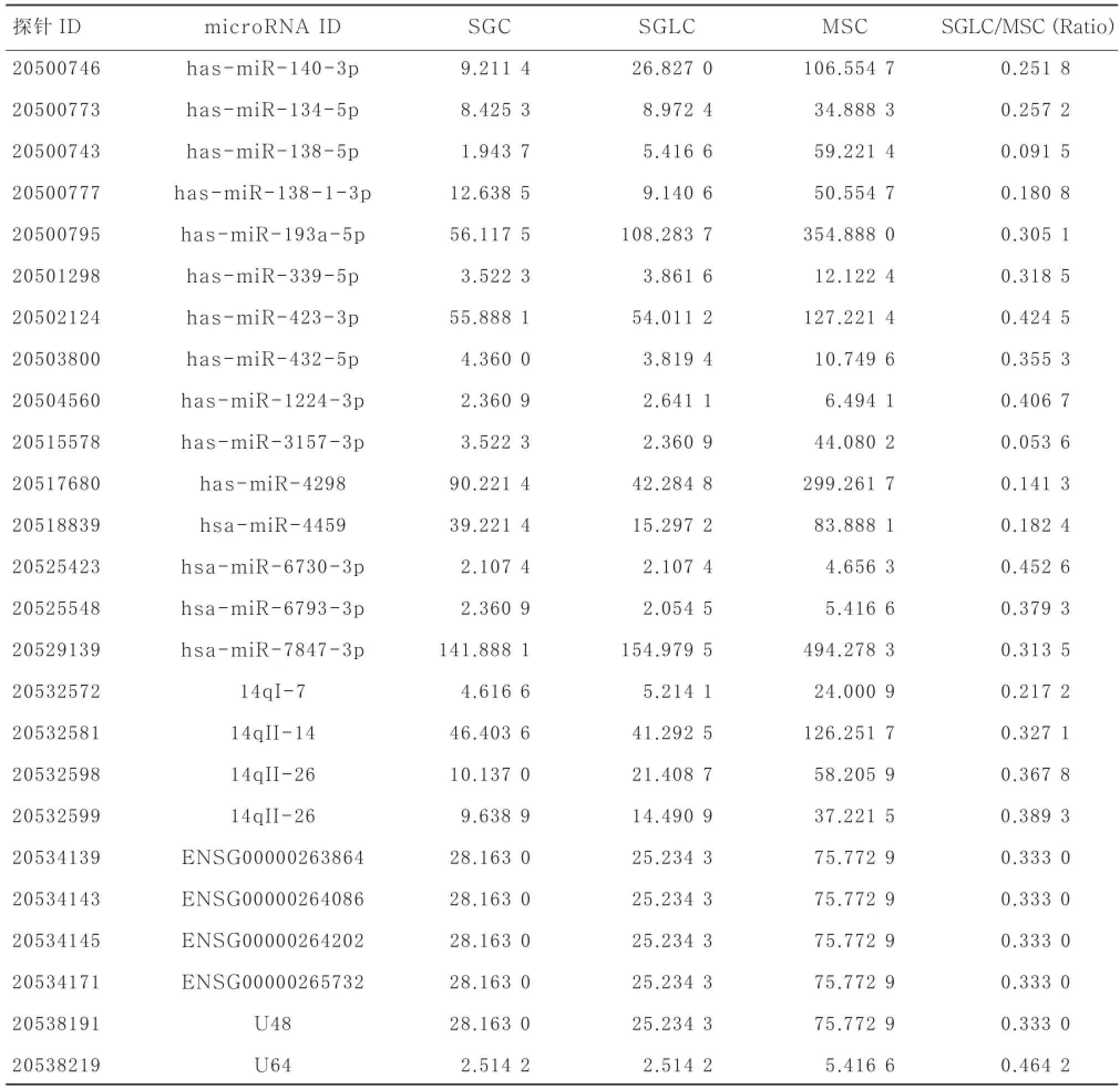
表1 37个microRNAs位点的microRNA Chip Array 的检测数据中显著低调的25个microRNAs检测数据
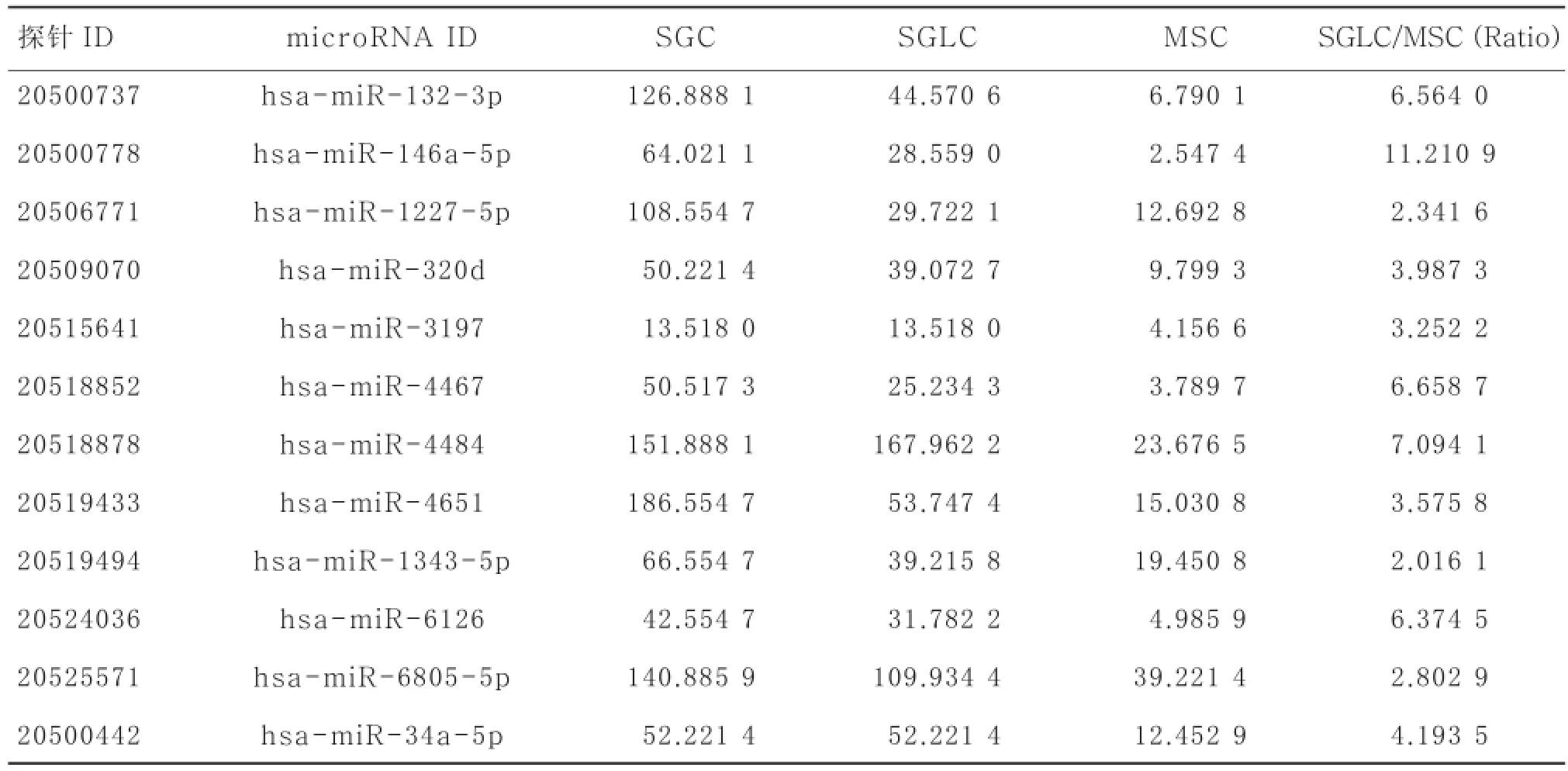
表2 37个microRNAs位点的microRNA Chip Array 的检测数据中显著高调的12个microRNAs检测数据
2.3与干细胞分化、汗腺发育相关的microRNA靶基因分析筛选出的37个microRNAs是MSCs诱导分化为汗腺细胞过程中发生显著变化的差异microRNAs,其直接靶向的靶基因可能成千上万。通过对与汗腺发育、干细胞分化相关microRNA靶点的分析和筛选,发现MSC诱导前后表达显著变化的差异性microRNAs主要靶向以下与干细胞分化、汗腺发育相关的细胞因子,通过参与EDA/EDAR、NF-κB、Wnt及ERK信号通路的调节促进MSCs向汗腺细胞转分化。
2.3.1与干细胞分化、汗腺发育相关的细胞因子EGF、肝细胞生长因子(HGF)、成纤维细胞生长因子(FGF)8、FGF10、癌胚抗原(CEA)、细胞角蛋白(KRT)7、KRT8、KRT14、KRT19、KRT79、Ptch、fox A1、shh、Bmp4、activin A。
2.3.2与干细胞分化、汗腺发育相关的信号通路① EDA/EDAR信号通路:EDA、EDAR、EDARadd等;② NF-κB信号通路:NFKB1、NFKB2、RELA、RELB、REL、IRAK1、TRAF2、TRAF5、TRAF6等;③Wnt/β-Catenin信号通路:β-Catenin、Lef1等;④ ERK信号通路:Ras-Raf-MEK-ERK。
另外,对37个关键microRNAs靶基因库筛选的结果表明,没有能直接靶向CD29、CD44、CD105、CD90、CD71、CD166这6种MSC分子标志基因,也没有能直接靶向NANOG、UTF1、 REX-1、SOX-2、OCT-4这5种MSC多能性分化潜能标志基因的microRNAs。但并不表示microRNAs一定不影响MSC分子标志的表达和MSC多能性分化潜能,microRNAs也可能通过间接作用,降低MSC分子标志的表达,促进MSC多能性分化潜能的增强,其作用还有待于进一步的研究考证。
3 讨 论
现阶段从分子水平对各种生理、病理改变的研究,主要包括编码基因、mRNA、蛋白表达和相关microRNAs调控等层面的协同研究[6]。最近研究显示,在MSCs自我更新和多向分化过程中,microRNA表达谱存在显著差异[7-8],这些显著差异性表达的microRNAs可能促使MSCs向特定方向分化,特异性microRNA表型也将成为区分特定细胞的分子标志[9]。例如,促进MSCs成骨分化的microRNAs有miR-196a、miR-210、miR-24等;促进MSCs成脂分化的microRNAs 有miR-143和miR-24;促进MSCs分化为神经元的microRNAs有miR-130a、miR-206等[10]。然而,参与干细胞诱导分化成汗腺细胞过程中的关键microRNAs尚未见研究和报道。因此,我们研究了该过程中的关键差异性microRNAs及其可能参与的信号通路调节。
MSCs诱导前后microRNA表达谱差异的比对分析结果显示,诱导后的SGLCs和成熟的SGCs中,has-miR-138-5p的表达量为诱导前MSCs的不到1/10(microRNA芯片检测结果显示,MSC: SGLC: SGC =59.221 4: 5.416 6: 1.943 6),为调低最为显著的microRNA;诱导后的SGLCs和成熟的SGCs中,has-miR-146a-5p的表达量为诱导前MSCs的 10倍多(microRNA芯片检测结果显示,MSC: SGLC: SGC =2.547 4: 28.558 9: 64.021 1),为调高最为显著的microRNA。这两种microRNAs的变化将显著影响其靶基因的表达,可能为MSCs向SGLCs诱导分化过程中的关键microRNAs。通过靶基因预测软件结合生物信息学分析,建立差异性microRNAs的靶基因库,再进一步分类筛选,我们找到了这些差异性microRNAs可能靶向、并参与调控MSCs诱导分化为SGLCs的关键靶基因及分子信号通路。这些通路多数已被证实是影响汗腺发生发育的关键信号通路,例如,ERK信号通路被认为是细胞因子促进干细胞分化的主要作用途径;EDA/EDAR通路可使外胚叶发育缺陷Tabby鼠实现毛发和汗腺的再生,且能刺激正常小鼠的外胚层组织和器官的发育;NF-κB作为下游通路,可被EDA/EDAR通路和MEKK1激活,促进皮肤附属器的发育。
本研究中,我们在众多预测到的靶基因中挑选出与汗腺发生、发育和干细胞分化相关的microRNAs靶基因,下一步我们将对这些靶基因进行表达量检测、靶基因验证和生物功能学验证,以确定所研究的microRNAs是否真正引起了靶基因表达的改变,是否影响了细胞表型和生物功能学的变化。
参考文献
[1]周岗,谢晓华,付小兵.汗腺细胞与骨髓间充质干细胞过程中基因表达变化及意义[J].中华实验外科杂志,2007,24(10):1186-1188.
[2]艾丽,李秀霞,张翠萍.ERK与NF-KB信号通路在汗腺发育过程中作用的研究进展[J].感染、炎症、修复,2009,10(3):190-192.
[3]陈艳,赵洪良,谭志军,赵焕军,张翠萍,付小兵. 人骨髓间充质干细胞向汗腺样细胞诱导分化的研究[J]. 感染、炎症、修复,2014,15(2):76-78.
[4]Friedman RC, Farh KK, Burge CB, Bartel DP. Most mammol/ Lalian mRNAs are conserved targets of microRNAs[J]. Genome Res, 2009, 19: 92-105.
[5]Love TM, Moffett HF, Novina CD. Not miR-ly small RNAs: big potential for microRNAs in therapy[J]. J Allergy Clin Immol/ Lunol, 2008, 121: 309-319.
[6]赵换军,赵洪良,张翠萍,付小兵. DNA甲基化对干细胞分化的影响及其作用方式[J]. 感染、炎症、修复,2013, 14(4): 246-249.
[7]Yu JM, Wu X, Gimble JM. Agerelated changes in mesenchymal stem cells derived from rhesusmacaque bone marrow[J]. Aging Cell,2011, 10: 66-79.
[8]Hackl M, Brunner S, Fortschegger K. miR-17, miR-19b, miR-20a, and miR-106a are down-regulated in human aging[J]. Aging Cell,2010, 9: 291-296.
[9]Lakshmipathy U, Hart RP. Concise review: microRNA expression in multipotent mesenchymal stromal cells[J]. Stem Cells,2008, 26: 356-363.
[10]Guo L, Zhao R. CH, Wu Y. The role of microRNAs in selfrenewal and differentiation of mesenchymal stem cells[J]. Experimental Hematol,2011, 39: 608-616.
Analysis on differential expression profiles of microRNA in the process of human bone marrow mesenchymal stem cells differentiated into sweat gland-like cells
Chen Yan*, Zhao Hongliang, Tan Zhijun, Zhao Huanjun, Zhang Cuiping, Fu Xiaobing. *Department of Pharmacy, General Hospital of Beijing Military Command, Beijing 100700, China; Institute of Basic Medicine, General Hospital of Chinese PLA, Beijing 100853, China
Corresponding author: Zhang Cuiping( E-mail: zcp666666@sohu.com)
Fu Xiaobing(E-mail: fuxiaobing@vip.sina.com)
AbstractObjective: To search the key microRNAs and investigate the microRNA-mediated regulating mechanisms on molecular signaling pathways by microRNA expressing profile gene chips detection in the process of human bone marrow mesenchymal stem cells (MSCs) differentiated into sweat gland-like cells (SGLCs). Methods: MSCs was induced to differentiate into SGLCs by the method of indirect culture. The profiles of microRNA differential expression in MSCs before and after differentiation, as well as in MSCs, SGLCs and adult matured sweat gland cells (SGCs) were arrayed by microRNA microarray detection, the markedly changed microRNAs were screened out. Subsequently, the significantly changed microRNAs which targeted the genes related to SGCs development and MSCs differentiation were screened out by microRNA target prediction software for further validation. Results: The comparison results of microRNA differential expression profiles showed that 19 microRNAs were up-regulated and 49 microRNAs down-regulated in SGLCs vs. MSCs. While 120 microRNAs up-regulated and 59 microRNAs down-regulated in SGCs vs. MSCs. Thirty-seven microRNAs were found simultaneously in the result of significantly changed microRNAs in SGLCs vs. MSCs and SGCs vs. MSCs, in which 12 up-regulated and 25 down-regulated. All the 37 microRNAs targeted directly to a number of cytokines related to the
Key words:Bone marrow mesenchymal stem cell;Sweat gland cell;MicroRNA expression profi les;Difference analysis
DOI:10. 3969/j. issn. 1672-8521. 2016. 01. 002
基金项目:国家“973”计划资助项目(2012CB518105);国家自然科学基金项目(81121004, 81230041, 81171798)
通讯作者:张翠萍,副研究员(E-mail: zcp666666@sohu.com) 付小兵,研究员,中国工程院院士(E-mail: fuxiaobing@vip.sina.com)sweat gland development and stem cells differentiation like CEA, and participated in the regulation of signaling pathways of EDA/EDAR, NF-κB, Wnt and ERK, etc. Conclusions: A series of differentially expressed microRNAs targeting to related cytokines and signaling pathways correlated with regulation of SGCs development and differentiation of MSCs have been found successfully by bioinformatics analysis..
(收稿日期:2015-11-16)

