结核分枝杆菌 Rv3607c T细胞表位分布情况预测及分析
王心倩,孙 妍,占卫红,雷 航,陈高瞻,余晓丽
(武汉轻工大学 生物与制药工程学院,湖北 武汉430023)
结核分枝杆菌 Rv3607c T细胞表位分布情况预测及分析
王心倩,孙妍,占卫红,雷航,陈高瞻,余晓丽
(武汉轻工大学 生物与制药工程学院,湖北 武汉430023)
摘要:为了预测和分析结核分枝杆菌( MTB) Rv3607c抗原的 CD4+T以及CD8+T 细胞表位的分布状况,首先在NCBI数据库中获取抗原的氨基酸序列,然后使用BLAST并与人类蛋白进行同源比对;之后使用NetMHC数据库预测并分析MTB的Rv3607c抗原的CD4+T细胞表位的分布状况;再使用SYFPEITHI、NETCTL、BIMAS和NetMHC 在线预测分析MTB的Rv3607c抗原CD8+T细胞表位的分布状况。结合两个表位预测结果,筛选出最终表位肽段,为后续验证试验做准备。其结果为:首先预测出MTB的Rv3607c抗原有4个CD8+T细胞候选表位;而CD4+T细胞表位的初步结果为强结合表位29个,弱结合表位183个。结合两个表位预测并分析,最终筛选出2个优势表位肽段。最后预测出的T 细胞候选表位将作为结核病特异性诊断和抗结核多表位疫苗的研究根基。
关键词:结核分枝杆菌;Rv3607c;T细胞表位分析
1引言
结核病是影响我国人民健康的最主要的重大传染性疾病,是由结核分枝杆菌(Mycobacteriumtuberculosis,MTB)引起的。据2015年WHO报[1],2014年估算全球有960万新发结核病病例,其中包括约540万男性、320万女性以及100万的儿童,其中150万人死于结核病,更不幸的是,HIV 共感染的不断扩散、MDR-TB患者的不断增多,愈加加剧了结核病的危害性。
目前,卡介苗( BCG)是唯一广泛使用的疫苗,BCG对儿童具有较强的保护作用[2],尤其是防止儿童重症结核病的发生,如结核性脑膜炎。但是卡介苗对成年人的保护作用在降低[3],并且有些人接种卡介苗后不能保证不感染结核病。随着耐药结核病的高发,以及各种抗结核药物的较大毒副作用,使得结核病的发病率以及死亡率不断攀升。理想的抗结核药物应当具有抗病高效性以及快速杀菌活性,并且价格低廉。
1998年,结核病的全基因序列被Cole等[4]破译,分子病理学检测技术也逐渐被应用于结核病的检查当中,基因检查技术也在结核分枝杆菌应用非常广泛,结核病研究也进入了基因组学新时代。抗原表位负责与抗体或者细胞表面的抗原受体结合而发挥免疫效应,即一个抗原分子的免疫活性区域。就目前所知,一个蛋白质抗原的表位,包含B细胞表位、辅助性T细胞表位和细胞毒性T细胞表位、自然杀伤细胞表位等一些与免疫识别密切相关的结构,并且也包含了一些不利于保护性免疫作用的结构。因此筛选和鉴定MTB蛋白抗原优势抗原表位对于通晓结合分支杆菌抗原引起的免疫应答反应、深入了解结核分枝杆菌的致病机制、改进免疫学诊断方法以及新型疫苗开发具有相当重要的意义[5]。
2材料与方法
2.1材料
在NCBI数据库中获得MTB的Rv3607c蛋白序列(Gene ID: 885345、133 aa)。
2.2同源性分析比对
在 NCBI中的 Blast (http://blast.ncbi.nlm.nih.gov/Blast.cgi )将Rv3607c的蛋白序列以及核酸序列与其他序列进行同源比对分析。登录NCBI选择protein Blast,然后选择非重复蛋白序列Non-redundant protein sequences ( nr) 数据库,并提交Rv3607c抗原序列,分析抗原序列与MTB复合群、非结核分支杆菌以及其他细菌的同源性;然后选择人类Human,进行分析比对Rv3607c抗原序列与人蛋白序列的同源性。
2.3Rv3607c抗原的CD4+T细胞表位进行预测
登录NetMHCⅡpan 3.1 Server ( http://www.cbs.dtu.dk/service/NetMHCⅡpan/),该软件可预测MHCⅡ中的三类分子HLA-DR、HLA-DR以及HLA-DR。预测分析时的限定条件选定为15个氨基酸残基长度的多肽,HLA-DR亚型,对MTB Rv3607c抗原进行在线预测。NetMHCⅡpan 3.1 Server预测结果输出依据亲和力的强弱判定,认定亲和值小于50 nm为强结合,亲和值大于50 nm小于500 nm为弱结合。
2.4Rv3607c抗原的CD8+T细胞表位预测
2.4.1NetMHC表位预测
登录NetMHC ( http://www.cbs.dtu.dk/service/NetMHC/),该软件可预测分析HLA-A、HLA-B、HLA-C以及HLA-E类别的MHC分子。由于绝大多数HLA分子偏向于与九个氨基酸长度的多肽有强结合,所以选择氨基酸长度为9个,并选择可能性最大的HLA-A 02 :01(A2 )以及HLA-A0301(A3 )两类HLA分子作为限定条件[6]。其预测结果评定方法同NetMHCⅡpan 3.1 Server 。
2.4.2SYFPEITHI表位预测
SYFPEITHI(http://www.syfpeithi.de/#userconsent#)是一个主要组织相容性复合体的数据库,可以进行针对MHC等位基因的产物的配体进行预测。进入网站“http://www.syfpeithi.de/#userconse nt#”,选择HLA- A* 02: 01、HLA- A* 03: 01两类,多肽长度选为9 个氨基酸,提交抗原序列,记录分析得分在前2%的多肽序列。
2.4.3BIMAS表位预测
进入BIMAS网站“http :// www - bimas.cit.nih.gov/molbio/hla_bin d/”,BIMAS是依据多肽分子与HLAⅠ类分子解离所用时间的一半进行排序,其分析结果是依据系数表推导的。选择 A_0201、A3两类HLA分子,多肽长度选为9 个氨基酸,而后输入Rv3607c的氨基酸序列并提交,进行预测分析[7]。
2.4.4NetCTL表位预测
登录NetCTL网站(http://www.cbs.dtu.dk/services/NetCTL/),从Supertype网页选择类别中,选定A2和A3两个超类型,Weight on C terminal cleavage值填写为“0.15”,以及Weight on TAP transport efficiency填写为“0.05”,Threshold for epitope identification填写为“0.75”,而后输入Rv3607c的氨基酸序列,进行在线预测。NetCTL 预测是通过人工网络神经操作的,结果是以得分高低来分别显示敏感度和特异度的高低。最后,对预测出的九个氨基酸的肽段,并且得分超过0.75的进行记录分析[8]。
3结果
3.1Rv3607c抗原同源性比对结果
Rv3607c抗原与结核分枝杆菌复合群(包括非洲分枝杆菌、减毒牛分枝杆菌、人型结核分枝杆菌、牛型结核分枝杆菌、田鼠分枝杆菌)的同源性都达到100%;与非结核分枝杆菌的同源性为65%—83%,其平均值为74%,与结核分枝杆菌复合群相比,二者之间的同源性不如前者的高;与其他细菌平均值为65%。Rv3607c抗原序列与人类蛋白同源性比对结果汇总可见表1。从表1中可以看得出,Rv3607c抗原与人类蛋白相似性较低,并且同源性仅为46%。
表1人类蛋白同源性与抗原Rv3607c比较结果

蛋白最高评分期望值同源性/%Rv3607c29.65.146
从以上结果可以得出Rv3607c抗原与结核分枝杆菌复合群的同源性相当高,另一方面Rv3607c抗原与人类蛋白的同源性却较低。因此认为,Rv3607c抗原有作为免疫性结核病疫苗的潜能,并且也有继续进行预测分析实验的必要性。
3.2Rv3607c抗原 T细胞表位结合预测结果
3.2.1Rv3607c抗原表位预测结果
使用NetMHCⅡ pan 3.1 Server对Rv3607c抗原的CD4+T细胞表位进行预测分析,过程中选择在中国HLAⅡ分子中分布频率较高的分子DRB1_0101、DRB1_0301、DRB1_0401、DRB1_0701、DRB1_0802、DRB1_0901、DRB1_1101、DRB1_1302、DRB1_1501作为限定条件,预测结果见表2。从预测结果可知,不同HLA分子结合数目大相径庭,具体来说Rv3607c抗原与DRB1_0101强结合的数目明显较多;与DRB1_1302结合的短肽数目最少。与不同HLA分子强弱结合的数目可见表2。
表2NetMHCⅡpan对Rv3607c抗原的CD4+T细胞表位分布的预测结果
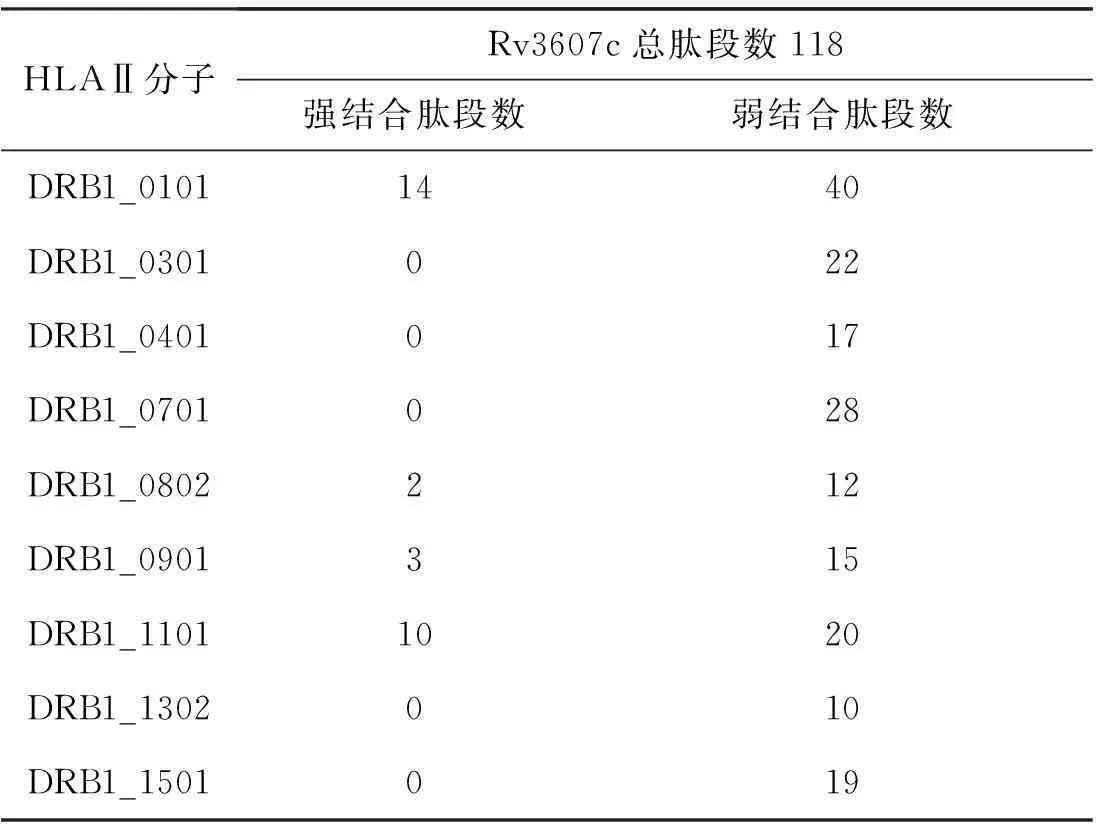
HLAⅡ分子Rv3607c总肽段数118强结合肽段数弱结合肽段数DRB1_01011440DRB1_0301022DRB1_0401017DRB1_0701028DRB1_0802212DRB1_0901315DRB1_11011020DRB1_1302010DRB1_1501019
3.2.2Rv3607c CD8+T细胞表位预测结果
选择在中国分布较广的HLAⅠ分子, HLA-0201 和 HLA-0301作为限定条件,对Rv3607c抗原使用 NETCTL、SYFPEITH、NetMHC和BIMAS这4种不同在线预测软件进行预测,并且综合HLA-0201和HLA-0301的预测结果,汇总结果见表3。最后,综合各种在线工具预测结果,Rv3607c抗原预测出4条抗原表位多肽。
表3不同在线预测工具对 Rv3607c抗原CD8+T 细胞表位的综合分析
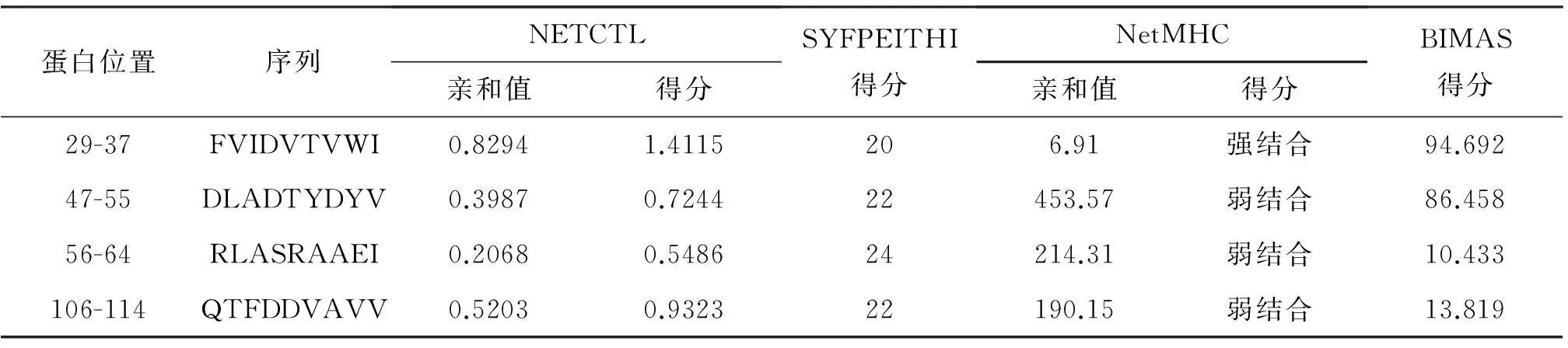
蛋白位置序列NETCTL亲和值得分SYFPEITHI得分NetMHC亲和值得分BIMAS得分29-37FVIDVTVWI0.82941.4115206.91强结合94.69247-55DLADTYDYV0.39870.724422453.57弱结合86.45856-64RLASRAAEI0.20680.548624214.31弱结合10.433106-114QTFDDVAVV0.52030.932322190.15弱结合13.819
3.2.3Rv3607c CD4+T和CD8+T细胞表位综合分析预测结果
通过五种在线预测工具分别预测,得出Rv3607c抗原的CD4+与CD8+T细肽段。最后依据CD4+T细胞表位的肽段包含CD8+T 细胞表位的肽段的原则,预测筛选出抗原 Rv3607c有2 条肽段,结果见表4 。
表4Rv3607c抗原的CD4+T和CD8+T细胞表位综合分析
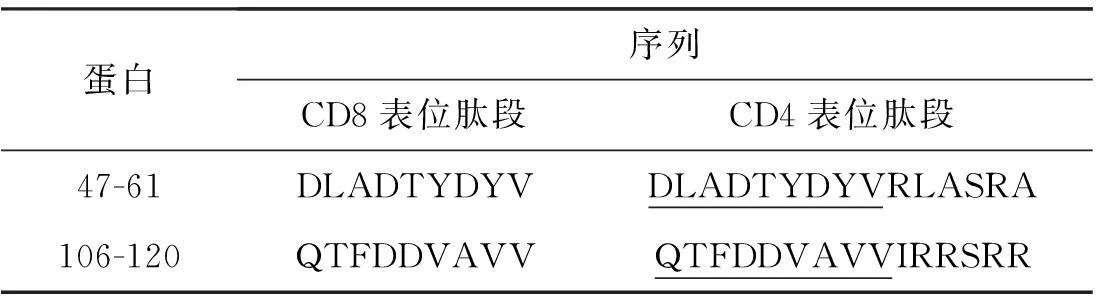
蛋白序列CD8表位肽段CD4表位肽段47-61DLADTYDYVDLADTYDYVRLASRA106-120QTFDDVAVVQTFDDVAVVIRRSRR
4讨论
在近几年的研究中,许多特异性结核抗原被研究人员发现[9],这一结果为结核病疫苗的研制提供了有价值的研究基础。许多新的疫苗也在动物实验中取得了较好的实验结果,但是仍然存在许多T细胞所识别的MTB蛋白抗原有待开发,这些抗原表位对于T细胞的激活和应答,包括参与抗MTB感染的免疫起到的重要作用是不容忽视的。在未来的研究中,我们还应当加大对其他MTB蛋白抗原的研究力度,并研制出更具有治疗效用、简单便捷的疫苗。
结核特异性免疫反应极其需要有效的T细胞免疫应答[10-11]。有研究表明,CD4+T细胞能够产生细胞因子IFN-γ,并且该细胞因子对于实验结核小鼠模型有极强的保护作用[12]。此外,通过对结核小鼠模型进行研究,实验人员也证实了CD8+T细胞对特异性免疫应答过程具有保护作用[13]。同时在艾滋病患者中,出现了代谢阻碍的CD4+T细胞会更容易感染结核病[14],即结核病合并感染艾滋病[15]。有部分研究报道,实验过程中发现受到结核分枝杆菌感染的患者体内存在MTB特异性CD8+T细胞[16-17],包括细胞毒性T细胞[18]以及自然杀伤性细胞[19]。到目前为止,还没有研究人员对结核分枝杆菌Rv3607c所编码的蛋白是否影响结核病的发病做出相关判断。本研究由于材料的局限性以及其他各方因素的限制,无法做出更加精准的判断,只有通过进一步实验,对Rv3607c的表位在结核病诊断所发挥的作用进行验证包括测定其免疫保护作用的强弱,才能给出更加让人信服的结论。
笔者预测分析是通过生物信息学的方法,并使用 SYFPEITH和INETCTL等在线预测软件对 MTB的Rv3607c的CD8+T以及CD4+T细胞表位进行预测,最终分析预测结果得出优势抗原表位肽段,并且为之后验证该抗原多肽段的特异性和敏感性做预备。预测过程中,使用NetMHC pan 2.1 Server 在线预测工具对CD4+T 细胞表位进行预测,再使用SYFPEITH对比其他3种在线预测工具预测结果得出Rv3607c抗原的CD8+T细胞表位有4个。最后,综合CD8+T细胞表位和CD4+T细胞表位预测分析的结果,选取出2条多肽段(DLADTYDYVRLASRA、QTFDDVAVVIRRSRR)进行人工合成,为进一步的验证实验做准备。
综合以上实验结果,笔者使用五种在线预测工具对MTB的Rv3607c抗原T细胞表位进行预测分析。这五种在线预测工具是根据Rv3607c的抗原表位多肽与不同等位基因结合能力强弱以及最终得分高低选取出最终优势抗原肽段。最终的实验结果将作为后阶段实验的理论依据以及实验基础,确定了该抗原的实验价值,为进一步的验证实验打下了良好的基础。
参考文献:
[1]Mario Raviglione. Global tuberculosis report 2015.[R]. Global Tuberculosis Report, 2015.
[2]熊昌辉,梁晓峰,王华庆. 卡介苗预防儿童结核性脑膜炎和粟粒性结核效果的系统评价[J]. 中国疫苗和免疫,2009(04):358-362.
[3]Li Wen-Chao ,Zhang Xu-ke ,Du Ling ,et al . Eimeria maxima : efficacy of recombinant Mycobacteri um bovis BCG expressing apical membrane antigen1 against homologous infection[J]. Parasitology Research, 2013,11:3825-3833.
[4]Cole ST,Brosch R,Parkhill J,et al.Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence[J].Nature,1998,393( 6685) :537-544.
[5]赵亚静,白雪娟,梁艳,等.结核分枝杆菌Rv3407蛋白抗原表位的预测[J].中国病原生物学杂志,2014(06):486-488.
[6]程良红,邹红岩,金士正,等. 中国人群人类白细胞抗原A、B和DRB1基因测序分型中模棱两可等位基因的鉴别[J]. 中国组织工程研究与临床康复,2009(31):6167-6171.
[7]Mommaas B,Kamp J,Drijfhout JW,et al.Identification of a novel HLA- B60- restricted T cell epitope of the minor histocompatibility antigen HA-1 locus[J].J Immunol,2002,169(6) : 3131-3136.
[8]Kesmir C,Nussbaum A K,Schild H,et al.Prediction of proteasome cleavage motifs by neural networks[J].Protein Eng,2002,15(4) : 287-296.
[9]叶娟,张舒林,刘文第. 结核分枝杆菌RD12区T细胞表位分布情况预测及分析[J]. 上海交通大学学报(医学版),2014(01):7-12.
[10]Lewinsohn DM,Swarbrick GM,Cansler ME,et al.Human CD8 T cell antigens /epitopes identified by a proteomic peptide library[J] PLoS One,2013,8( 6) : e67016.
[11]董毅,吴利先. T细胞免疫在抗结核杆菌感染中的研究进展[J]. 现代生物医学进展,2014(18):3593-3595.
[12]Daniela Santoro Rosa,Susan Pereira Ribeiro,Edecio Cunha-Neto. CD4+T Cell Epitope Discovery and Rational Vaccine Design[J]. Archivum Immunologiae et Therapiae Experimentalis,2010,2:121-130.
[13]Wang Xin-jing,Cao Zhi-hong,Jiang Jing,et al.Elevated expression of Tim-3 on CD8 T cells correlates with disease severity of pulmonary tuberculosis[J]. Journal of Infection,2011 ,4:292-300.
[14]Oldstone M B.The role of cytotoxic T lymphocytes in infectious disease: history,criteria,and state of the art[J].Curr Top Microbiol Immunol,1994,189: 1-8.
[15]崔中锋,史继静,王勇明. HIV感染合并肺结核患者CD4+T细胞计数与胸部影像特征的相关性研究[J].医药论坛杂志,2011(15):36-37.
[16]毛冬婷,罗燕芬,曹开源,等. 结核感染中PD-1调节CD8+T细胞免疫应答的研究[J]. 热带医学杂志,2015(05):587-591.
[17]Nobuhiro Nakamoto,David E Kaplan,Jennifer Coleclough,et al. Functional Restoration of HCV -Specific CD8 T Cells by PD-1 Blockade Is Defined by PD-1 Expression and Compartmentalization[J]. Gastroenterology,2008 (7):1927-1937.
[18]Li Fan,Yang Di,Wang Yi-qin,et al. Identification and modification of an HLA-A*0201-restricted cytotoxic T lymphocyte epitope from Ran antigen[J]. Cancer Immunology, Immunotherapy ,2009 (12):2039-2049.
[19]Grotzke J E,Lewinsohn D M.Role of CD8+T lymphocytes in control of Mycoba cterium tuberculosis infection[J].Microbes Infect,2005,7(4) : 776-788.
Prediction and analysis of T cell epitopes’s distribution of theMycobacteriumtuberculosisRv3607c
WANGXin-qian,SUNYan,ZHANWei-hong,LEIHang,CHENGao-zhan,YUXiao-li
(School of Biology and Pharmaceutical Engineering,Wuhan Polytecnic University,Wuhan 430023,China)
Abstract:To predict and analyze the distribution of CD4+T cell and CD8+T cell epitopes encoded by the Rv3607c ofMycobacteriumtuberculosis,We obtain the amino acid sequences by online NCBI database and analyze the homology between MTB complex and human proteins by BLAST,and predicting the distribution of CD8 T cell epitopes by SYFPEITHI、NETCTL、BIMASand NetMHC databases.After the predictiction,we can select the superior epitopes . For the results,there are 4 CD8+T cell epitopes candidates,29 strong and 138 weak candidate CD4+T cell epitopes candidates. In the end,two superior epitopes can be selected. Prediction of T cell epitopes will be the basis diagnosis of tuberculosis and the study of new tuberculosis vaccine.
Key words:Mycobacteriumtuberculosis;Rv3607c;T cell epitope analyze
收稿日期:2015-01-05.
作者简介:王心倩(1993-),女,硕士研究生,E-mail: wawawayyy@qq.com.
文章编号:2095-7386(2016)02-0036-04
DOI:10.3969/j.issn.2095-7386.2016.02.006
中图分类号:Q 93
文献标识码:A

