牦牛角性状候选基因的筛选
佘平昌,吴晓云,梁春年,禇 敏,丁学智,阎 萍*
(1.中国农业科学院兰州畜牧与兽药研究所,兰州 730050; 2.甘肃省牦牛繁育工程重点实验室,兰州 730050)
牦牛角性状候选基因的筛选
佘平昌1,2,吴晓云1,2,梁春年1,2,禇敏1,2,丁学智1,2,阎萍1,2*
(1.中国农业科学院兰州畜牧与兽药研究所,兰州 730050; 2.甘肃省牦牛繁育工程重点实验室,兰州 730050)
本研究旨在筛选牦牛角性状候选基因,为角发育机制的研究和无角牦牛的培育提供科学依据。采集牦牛角发育初期胎龄90 d的有角和无角牛的角基及额部皮肤组织,利用实时荧光定量PCR技术(qRT-PCR),比较牛角性状候选基因OLIG1、OLIG2、IFNAR1、IFNAR2、C1H21orf62、GCFC1、IFNGR2、SYNJ1、IL10RB、GART、RXFP2、FOXL2、TWIST1、TWIST2、ZEB2、E-cadherin和P-cadherinmRNA在角基间及皮肤间的相对表达量。结果显示,候选基因OLIG1、IFNAR1、C1H21orf62、SYNJ1、IL10RB、GART、RXFP2、TWIST2和E-cadherin在有角和无角牦牛角基间和皮肤间转录量差异不显著(P>0.05)。IFNGR2、FOXL2、TWIST1和P-cadherin在有角和无角牦牛角基间转录量差异显著(P<0.05)。IFNAR2和GCFC1在有角和无角牦牛角基间和皮肤间转录量差异都显著(P<0.05)。OLIG2和ZEB2在有角和无角牦牛角基间转录量差异极显著(P<0.01)。推测OLIG2和FOXL2可能是牛角性状的关键候选基因,同时ZEB2、TWIST1和IFNGR2基因可能对牛角的形成具有重要作用,而P-cadherin基因可能参与了牛角的发育。
牛角;无角性状;牦牛;候选基因;qRT-PCR
牛角是牛科动物特有的组织结构。在男耕女织的古代原始生产过程中,牛角是进行耕地过程中牵牛的基础,所以牛角在原始耕作时代中是一种生产力。但是进入集约规模化饲养后,牛群常发生争斗及伤害饲养人员的行为,造成了动物的伤害及养殖场的混乱,牛角变成了一种制约家畜集约化和设施养殖的性状。所以,很多养牛场将犊牛进行去角[1],然而去角增加了养殖成本,同时容易引起犊牛的应激反应,是违背动物福利的行为。动物福利问题已经在发达国家引起高度重视,欧盟启动了关于禁止动物阉割和去角的ALCASDE工程[2],而德国的动物福利组织也联合政府发表申明将积极推进无角公牛精液的使用。因此,无角牛品种的培育是未来牛育种的主要方向之一。
牛角根据生物学分类属于洞角,由角质鞘和骨心组成。牛角的发育较早,60胎龄的胎牛就细微可见角的形成,90胎龄时发育更加明显,基本可以通过肉眼判定角的有无,在角的生长部位可见真皮乳头状的突起形成角窦[3],同时出现表皮增厚的现象[4],角窦和皮肤间通过真皮乳头进行信号传导[5]。但是只有在出生时才可见螺纹状角的出现[6],出生后6月龄才开始出现角的空腔结构[3]。组织学观察发现胚胎期牛角与皮肤间的差异主要有4点:腺导管真皮细胞的集群不同、囊泡化的角质细胞、没有毛囊的发育[7]、真皮中出现大量的神经束[8],推测头皮层的神经纤维对牛角形成具有触动作用,所以神经发育相关基因可能会在角基部位高表达。在牛角性状候选基因的研究中,根据目前的研究进展,很多研究将无角突变定位在1号染色体(BTA1,Bostaurusautosome1)1.265~2.198 M上1 M左右的一段序列内,包括C1H21orf62、OLIG1、OLIG2、SYNJ1、GCFC4、GART、IL10RB、IFNAR1、IFNAR2、IFNGR2等基因[9-14]。而不在该序列内的一些基因也可能对牛角形成和发育具有重要作用,FOXL2和RXFP2基因在牛角形成和发育过程中高度表达[7,15],而ZEB2、TWIST1和TWIST2是畸角牛的候选基因[16-18],E-cadherin和P-cadherin对胚胎分化及表皮、毛囊、神经等的形成具有重要作用[19-21]。牛角是一种质量性状(Qualitative trait),其遗传方式可能为多基因遗传(Polygenic trait)。目前对牛角性状相关基因还没有进行精确定位,还需进一步研究。本研究通过荧光定量PCR (Real-time quantitative PCR,qRT-PCR)方法分析牛角性状候选基因mRNA的表达水平,初步揭示牛角发育和形成的分子机制,以期为无角牦牛品种的培育提供科学依据和理论支撑。
1 材料与方法
在青海省大通牦牛种牛场采集90 d胎龄的有角和无角牦牛的角基部及额部皮肤样品,液氮保存,并对其角基部及全身进行拍照,测量其顶臀长,根据胎儿顶臀长与胎龄的线性关系推算胎龄[22]。
1.1RNA提取及cDNA合成
采用Trizol (Invitrogen)法提取有角和无角牦牛的角基及额部皮肤组织的总RNA,按照TaKaRa公司试剂盒Prime Script RT reagent Kit with gDNA Eraser(Perfect Real Time)说明书合成cDNA。
1.2荧光定量PCR
使用Premier 5.0设计候选基因mRNA荧光定量PCR引物(表1),选择GAPDH作为内参基因。引物由上海英骏生物公司合成,产物经2%琼脂糖凝胶电泳检测。
定量PCR(CFX96)反应使用25 μL体系:SYBR®Premix Ex TaqTMⅡ (Tli RNaseH Plus) 12.5 μL,上下游引物各1 μL,cDNA 2 μL,ddH2O 8.5 μL。荧光定量 PCR 的反应程序:95 ℃预变性30 s;95 ℃变性5 s,53.5 ℃退火30 s,40个循环;之后进行熔解曲线分析,反应程序:65 ℃ 逐渐升温至95 ℃,每升温0.5 ℃读板1次。每个组织4个个体重复,每个样本3次试验重复。反应结束后以熔解曲线来判定qRT-PCR反应的特异性;获得每个样品的Ct值,运用2-ΔΔCt法计算相对转录水平。
1.3图像数据处理
本试验采用Excel 2013对数据进行统计,SPSS 17.0进行one-way ANOVA分析,Origin 7.5作柱状图。
2 结 果
2.1BTA1上候选区域内基因的相对表达量分析
为了分析候选基因的转录情况,本研究对比候选基因在有角牛和无角牛的角基间及额部皮肤间的相对转录水平,据此分析该基因是否参与了角的形成和发育(图1)。OLIG1、OLIG2、IFNAR1、IFNAR2、C1H21orf62、GCFC1、IFNGR2、SYNJ1、IL10RB和GART是1号染色体上1 M左右片段上的基因。研究发现,OLIG1、IFNAR1、C1H21orf62、SYNJ1、IL10RB、GART、PXFP2、TWIST2和E-cadherin在有角和无角牦牛的角基间及皮肤间的转录量差异都不显著(P>0.05);OLIG2在有角牛角基的转录量极显著高于无角牛角基(P<0.01),而在皮肤间差异不显著;IFNAR2在有角牛的角基和皮肤的转录量都要显著高于无角牛(P<0.05);相反,GCFC1在有角牛的角基和皮肤的转录量都要显著低于无角牛(P<0.05)。IFNGR2在无角牛角基中的转录量显著高于有角牛(P<0.05),而在皮肤间的转录差异不显著(P>0.05)。
表1荧光定量PCR引物序列
Table 1Primer sequences used for the real-time quantitative PCR
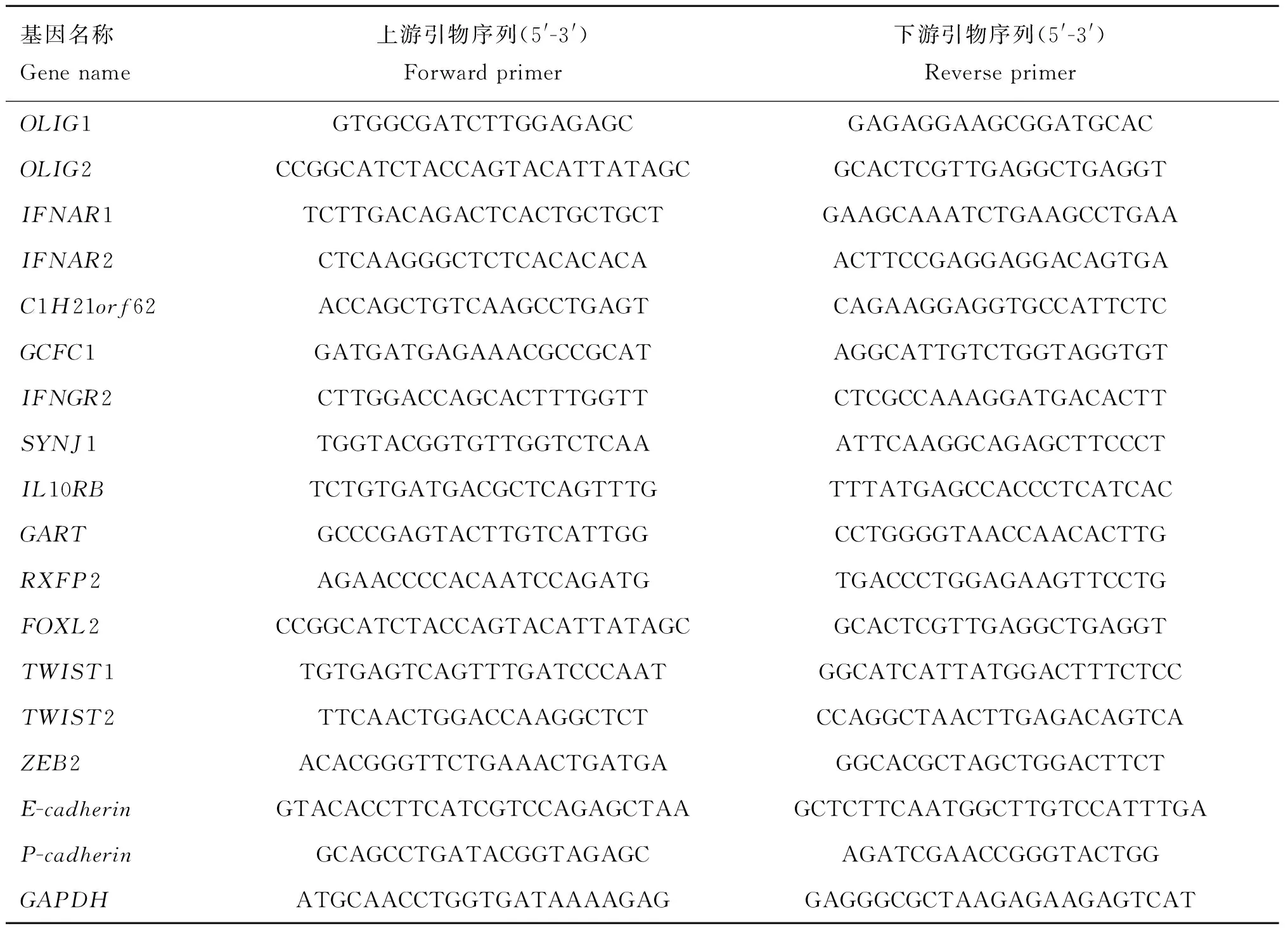
基因名称Genename上游引物序列(5'-3')Forwardprimer下游引物序列(5'-3')ReverseprimerOLIG1GTGGCGATCTTGGAGAGCGAGAGGAAGCGGATGCACOLIG2CCGGCATCTACCAGTACATTATAGCGCACTCGTTGAGGCTGAGGTIFNAR1TCTTGACAGACTCACTGCTGCTGAAGCAAATCTGAAGCCTGAAIFNAR2CTCAAGGGCTCTCACACACAACTTCCGAGGAGGACAGTGAC1H21orf62ACCAGCTGTCAAGCCTGAGTCAGAAGGAGGTGCCATTCTCGCFC1GATGATGAGAAACGCCGCATAGGCATTGTCTGGTAGGTGTIFNGR2CTTGGACCAGCACTTTGGTTCTCGCCAAAGGATGACACTTSYNJ1TGGTACGGTGTTGGTCTCAAATTCAAGGCAGAGCTTCCCTIL10RBTCTGTGATGACGCTCAGTTTGTTTATGAGCCACCCTCATCACGARTGCCCGAGTACTTGTCATTGGCCTGGGGTAACCAACACTTGRXFP2AGAACCCCACAATCCAGATGTGACCCTGGAGAAGTTCCTGFOXL2CCGGCATCTACCAGTACATTATAGCGCACTCGTTGAGGCTGAGGTTWIST1TGTGAGTCAGTTTGATCCCAATGGCATCATTATGGACTTTCTCCTWIST2TTCAACTGGACCAAGGCTCTCCAGGCTAACTTGAGACAGTCAZEB2ACACGGGTTCTGAAACTGATGAGGCACGCTAGCTGGACTTCTE-cadherinGTACACCTTCATCGTCCAGAGCTAAGCTCTTCAATGGCTTGTCCATTTGAP-cadherinGCAGCCTGATACGGTAGAGCAGATCGAACCGGGTACTGGGAPDHATGCAACCTGGTGATAAAAGAGGAGGGCGCTAAGAGAAGAGTCAT
2.2其他重要候选基因的相对表达量分析
RXFP2、FOXL2、TWIST1、TWIST2和ZEB2也是重要的角性状候选基因。研究发现,RXFP2和TWIST2在有角和无角牦牛的角基间和皮肤间转录量差异都不显著(P>0.05);FOXL2在有角牛角基的转录量显著高于无角牛(P<0.05),而在皮肤间差异不显著;TWIST1却在无角牛角基的转录量显著高于有角牛(P<0.05),皮肤间的转录量差异不显著。ZEB2在有角牛角基的转录量极显著低于无角牛(P<0.01),而在皮肤间同样差异不显著(P>0.05)。
2.3角发育相关基因的相对表达量分析
关于E-cadherin和P-cadherin与角性状的相关研究报道较少,但E-cadherin是表皮及毛囊的发育关键基因,P-cadherin的表达对表皮发育及神经分化具有重要作用,而据研究进展可知,角的发育是角基表皮分化的过程,中间也伴随着毛囊的发育,引入分析为试验提供参考。研究发现,E-cadherin在角基间和皮肤间的转录差异都不显著(P>0.05),而P-cadherin在有角牛角基中的转录量显著高于无角牛(P<0.05),皮肤间的转录量差异不显著(P>0.05)。


horn.角基组织;skin.皮肤组织;horned.有角牛群体;polled.无角牛群体;*.P<0.05;**.P<0.01horn.Horn bud;skin.Forehead skin;horned.Horned yaks;polled.Polled yaks;*.P<0.05;**.P<0.01图1 候选基因在有角和无角牦牛的角基间和皮肤间的相对转录水平Fig.1 Relative expression analyses of candidate genes in horn bud and skin tissues between horned and polled yaks by qRT-PCR
3 讨 论
本研究对BAT1上定位的无角性状基因座区域内的候选基因的表达量进行了分析。研究结果显示,OLIG1、IFNAR1、C1H21orf62、SYNJ1、IL10RB和GART在有角和无角牦牛的角基间及皮肤间的转录量差异不显著(P>0.05);OLIG2、IFNAR2、GCFC1和IFNGR2的mRNA转录量在角基间具有显著(P<0.05)或极显著(P<0.01)差异。候选基因中,OLIG2为少突胶质细胞转录因子,其主要参与少突胶质细胞的形成分化及髓鞘的形成,同时对哺乳动物的组织分化起重要调节作用[23]。本研究发现,OLIG2在角基间转录差异极显著,研究结果与在荷斯坦牛[7]和西门塔尔牛[15]中的研究结果一致。近期研究发现,OLIG2参与神经分化,推测该基因对牛角形成的作用机制可能与神经调控有关,说明OLIG2对角的发育有重要作用,因此将该基因作为荷斯坦奶牛、西门塔尔牛及牦牛无角性状的候选基因[7,13,15]。IFNAR2和GCFC1基因在角基间和皮肤间转录差异均显著,说明IFNAR2和GCFC1 的表达模式不具有角组织特异性,IFNAR2和GCFC1可能对表皮的发育过程具有关键作用。IFNGR2是参与T细胞活动的一个细胞因子,在无角荷斯坦牛中该基因存在突变位点AC000158:g.1390292G>A[24],但是S.Rothammer等[12]的研究又否定了该突变与无角性状的相关性,本研究发现,有角牛中IFNGR2基因的表达量显著低于无角牛,所以还不能排除IFNGR2可能为角性状候选基因。
本研究发现,其他候选基因RXFP2、TWIST2和E-cadherin在有角和无角牦牛的角基间和皮肤间转录量差异不显著(P>0.05);FOXL2、TWIST1、ZEB2和P-cadherin的mRNA转录量在角基间具有显著(P<0.05)或极显著(P<0.01)差异。候选基因中,FOXL2又名叉头框因子2,是叉头框转录因子家族成员之一,对哺乳动物早期胚胎的发育和生殖系统功能维持具有重要作用[25],本研究发现,FOXL2在角基间的表达量存在显著差异,在西门塔尔牛有角牛角基中该基因的表达量也显著高于无角牛[15],同时FOXL2基因是羊角性状的主效基因之一[26],所以FOXL2可能参与了牛角的形成和发育。TWIST1[16]和ZEB2[18]都是目前研究畸角牛的关键基因,TWIST1是在胚胎发育的一个转录因子,对诱导细胞移动及组织朔形具有重要作用,参与骨骼、肌肉和神经等的形成。ZEB2属于E盒结合锌指蛋白家族,在胚胎发育过程中对神经系统的发育具有重要作用。而牦牛中没有畸角牛,但是本研究发现其在角基中表达量差异显著,推测TWIST1和ZEB2可能与牛角的形成或发育具有调控作用,但是其功能仍需进一步明确。P-cadherin基因为钙粘蛋白家族成员,在胚胎发育过程中对表皮及神经的发育发挥着重要的作用,而角的发育为表皮增厚的过程,本研究中P-cadherin在角基间转录差异显著,推测该基因可能参与了牛角的发育。
本研究中,候选基因OLIG1、IFNAR1、C1H21orf62、SYNJ1、GART和RXFP2虽然在有角和无角牦牛的角基间和皮肤间的转录均不存在显著差异(P>0.05),但是根据本研究的内容不能完全排除这些基因可能参与角的形成或发育的可能。而IL10RB、TWIST2和E-cadherin基因的相关研究较少,且本试验结果显示这些基因在有角和无角牛角基及皮肤间的表达量都不存在差异,故推测IL10RB、TWIST2和E-cadherin基因可能不是牛角早期形成和发育的关键基因。
4 结 论
牦牛角形状候选基因的表达研究发现,候选基因OLIG1、IFNAR1、C1H21orf62、SYNJ1、IL10RB、GART、RXFP2、TWIST2和E-cadherin在有角和无角牦牛的角基和皮肤间相对转录水平差异不显著(P>0.05)。IFNGR2、FOXL2、TWIST1和P-cadherin基因在角基间转录量差异显著(P<0.05)。IFNAR2和GCFC1基因在角基和皮肤间转录量差异都显著(P<0.05)。OLIG2和ZEB2基因在角基间转录量差异极显著(P<0.01)。结合前期研究推测OLIG2和FOXL2基因可能是牛角性状的关键候选基因,同时ZEB2、TWIST1和IFNGR2基因可能对牛角的形成具有重要作用,而P-cadherin基因可能参与了牛角的发育。
[1]GRAF B,SENN M.Behavioural and physiological responses of calves to dehorning by heat cauterization with and without local anesthesia[J].ApplAnimBehavSci,1999,62(2):153-171.
[2]OLIVER M A,MIRABITO L,VEISSIER I,et al.Study on the improved methods for animal-friendly production in particular on alternatives to the castration of pigs and on alternatives to the dehorning of cattle[D].England:University of the West of England,2009:1-65.
[3]DOVE W F.The physiology of horn growth:A study of the morphogenesis,the interaction of tissues,and the evolutionary processes of a mendelian recessive character by means of transplantation of tissues[J].JExpZool,2005,69(3):347-450.
[4]任强,郁枫,王旭强,等.牦牛角的形态发生及Shh和β-catenin的时空表达[J].西北农业学报,2009,18(1):24-29.
REN Q,YU F,WANG X Q,et al.Temporal-spatial expression of Shh and β-catenin during horn morphogenesis in yak[J].ActaAgriculturaeBoreali-occidentalisSinica,2009,18(1):24-29.(in Chinese)
[5]HILZHEIMER M,LENGERKEN H.Natürliche rassengeschichte der haussäugetiere[M].Berlin:De Gruyter,1926:25-183.
[6]RUSSE I,SINOWATZ F.Lehrbuch der Embryologie der Haustiere[M].Berlin:Paul Parey,1991:304-344.
[7]ALLAIS-BONNET A,GROHS C,MEDUGORAC I,et al.Novel insights into the bovine polled phenotype and horn ontogenesis in Bovidae[J].PLoSOne,2013,8(5):e63512.
[8]WIENER D J,WIEDEMAR N,WELLE M M,et al.Novel features of the prenatal horn bud development in cattle(Bostaurus)[J].PLoSOne,2015,10(5):e0127691.
[9]GEORGES M,DRINKWATER R,KING T,et al.Microsatellite mapping of a gene affecting horn development inBostaurus[J].NatGenet,1993,4(2):206-210.
[10]SEICHTER D,RUSS I,ROTHAMMER S,et al.SNP-based association mapping of the polled gene in divergent cattle breeds[J].AnimGenet,2012,43(5):595-598.
[11]MEDUGORAC I,SEICHTER D,GRAF A,et al.Bovine polledness--an autosomal dominant trait with allelic heterogeneity[J].PLoSOne, 2012,7(6):e39477.[12]ROTHAMMER S,CAPITAN A,MULLAART E,et al.The 80-kb DNA duplication on BTA1 is the only remaining candidate mutation for the polled phenotype of Friesian origin[J].GenetSelEvol,2014,46:44.
[13]LIU W B,LIU J,LIANG C N,et al.Associations of single nucleotide polymorphisms in candidate genes with the polled trait in Datong domestic yaks[J].AnimGenet,2014,45(1):138-141.
[14]赵娟花,梁春年,裴杰,等.牦牛C1H21orf62 基因的克隆、生物信息学及表达分析[J].广东农业科学,2015(5):98-102.
ZHAO J H,LIANG C N,PEI J,et al.Cloning,bioinformatics and expression analysis ofC1H21orf62 gene in yak[J].GuangdongAgriculturalSciences,2015(5):98-102.(in Chinese)
[15]WIEDEMAR N,TETENS J,JAGANNATHAN V,et al.Independent polled mutations leading to complex gene expression differences in cattle[J].PLoSOne,2014,9(3):e93435.
[16]CAPITAN A,GROHS C,GAUTIER M,et al.The scurs inheritance:new insights from the French Charolais breed[J].BMCGenet,2009,10:33.
[17]ASAI M,BERRYERE T G,SCHMUTZ S M.The scurs locus in cattle maps to bovine chromosome 19[J].AnimGenet,2004,35(1):34-39.
[18]CAPITAN A,ALLAIS-BONNET A,PINTON A,et al.A 3.7 Mb deletion encompassing ZEB2 causes a novel polled and multisystemic syndrome in the progeny of a somatic mosaic bull[J].PLoSOne,2012,7(11):e49084.
[19]LARUE L,OHSUGI M,HIRCHENHAIN J,et al.E-cadherin null mutant embryos fail to form a trophectoderm epithelium[J].ProcNatlAcadSciUSA,1994,91(17):8263-8267.
[20]MCMILLEN P,HOLLEY S A.Integration of cell-cell and cell-ECM adhesion in vertebrate morphogenesis[J].CurrOpinCellBiol,2015,36:48-53.
[21]吴兴龙,王棉娟,胡春超,等.E-钙粘蛋白抗体对小鼠早期胚胎发育的影响[J].畜牧兽医学报,2012,43(8):1222-1229.
WU X L,WANG M J,HU C C,et al.Effects of E-cadherin antibody on mouse early embryo development [J].ActaVeterinariaetZootechnicaSinica,2012,43(8):1222-1229.(in Chinese)
[22]LIU B,CUI Y,YANG B,et al.Morphometric analysis of yak placentomes during gestation[J].AnatRec(Hoboken),2010,293(11):1873-1879.
[23]ZHOU Q,ANDERSON D J.The bHLH transcription factors OLIG2 and OLIG1 couple neuronal and glial subtype specification[J].Cell,2002,109(1):61-73.
[24]GLATZER S,MERTEN N J,DIERKS C,et al.A single nucleotide polymorphism within the interferon gamma receptor 2 gene perfectly coincides with polledness in Holstein cattle[J].PLoSOne,2013,8(6):e67992.
[25]BATISTA F,VAIMAN D,DAUSSET J,et al.Potential targets of FOXL2,a transcription factor involved in craniofacial and follicular development,identified by transcriptomics[J].ProcNatlAcadSciUSA,2007,104(9):3330-3335.
[26]PAILHOUX E,VIGIER B,CHAFFAUX S,et al.A 11.7-kb deletion triggers intersexuality and polledness in goats[J].NatGenet,2001,29(4):453-458.
(编辑郭云雁)
Screening of Candidate Genes for Horn Trait of Yak
SHE Ping-chang1,2,WU Xiao-yun1,2,LIANG Chun-nian1,2,CHU Min1,2,DING Xue-zhi1,2,YAN Ping1,2*
(1.LanzhouInstituteofHusbandryandPharmaceuticalSciencesofChineseAcademyofAgriculturalSciences,Lanzhou730050,China;2.KeyLaboratoryofYakBreedingEngineeringofGansuProvince,Lanzhou730050,China)
This study screened the candidate genes of horn trait which provides scientific basis for the horn developmental mechanism and the polled yak breeding.The horn bud and forehead skin tissue from horn and polled yaks with 90 d gestational age were collected,and the mRNA different expression ofOLIG1,OLIG2,IFNAR1,IFNAR2,C1H21orf62,GCFC1,IFNGR2,SYNJ1,IL10RB,GART,RXFP2,FOXL2,TWIST1,TWIST2,ZEB2,E-cadherin,P-cadheringenes were analyzed using real-time quantitative PCR(qRT-PCR).The results showed that expressions ofOLIG1,IFNAR1,C1H21orf62,SYNJ1,IL10RB,GART,RXFP2,TWIST2 andE-cadheringenes showed no significant differences in horn and skin between horned and polled yaks (P>0.05).There were significant difference at transcript levels ofIFNGR2,FOXL2,TWIST1 andP-cadheringenes in horn buds between horned and polled yaks(P<0.05).IFNAR2 andGCFC1 had significant transcription difference in the expression of horn bud and skin between horned and polled yaks (P<0.05).OLIG2 andZEB2 had highly significant transcription difference in horn bud expression between horned and polled yaks (P<0.01).We concluded thatOLIG2 andFOXL2 might be the key candidate genes.ZEB2,TWIST1 andIFNGR2 might also involve in the formation of horn.P-cadherinmight participate in the development of horn.
horn;polled trait;yak;candidate genes;qRT-PCR
10.11843/j.issn.0366-6964.2016.06.009
2015-11-02
牦牛遗传资源与育种(CAAS-ASTIP-2014-LIHPS-01);科技部科技支撑计划(2012BAD13B05)
佘平昌(1989-),男,云南普洱人,硕士生,主要从事家畜分子生物学研究,E-mail:sheemail@163.com
阎萍,研究员,博士生导师,主要从事动物遗传育种与繁殖研究,E-mail:pingyanlz@163.com
S823.85;S813.3
A
0366-6964(2016)06-1147-07

