微流控法DNA片段化研究进展
水玲玲, 李岚慧, 金名亮, 周国富
(华南师范大学华南先进光电子研究院,彩色动态电子纸显示技术研究所,广州 510006)
微流控法DNA片段化研究进展
水玲玲*, 李岚慧, 金名亮, 周国富
(华南师范大学华南先进光电子研究院,彩色动态电子纸显示技术研究所,广州 510006)
摘要:DNA分子的片段化技术对于下一代基因测序技术和疾病检测技术意义重大,文章回顾了现有的DNA片段化方法,着重介绍了基于微流控芯片技术的流体动力学剪切法DNA片段化技术,总结了影响微流控芯片中DNA片段化过程的因素,并且展望未来微流控技术在DNA片段化和生物芯片领域的应用.
关键词:微流控; DNA片段化; 流体剪切; 基因测序
基因测序也称DNA测序,是现代生物学研究中重要的手段之一,其关键是快速获得随机分布长短一致的DNA片段[1-3].通常,DNA片段长度的范围是选取接近相关生物体内1个单基因的平均长度,或者基于为整个数据库自动生成序列数据的方法来选取[4].因此,如何得到片段大小可控且长度分布集中的DNA片段成为大家普遍关心的热点研究课题之一.
DNA是由核苷酸组成的长链分子聚合物,易溶于水,在外力作用下,DNA长链会被拉伸,当外力大于分子间的化学键时,DNA组成单元间的共价键断裂从而形成随机分布的DNA片段[5].目前,常用的DNA片段化方法有以下几种:DNA限制酶切法[6]、超声波降解法[7-8]、喷射雾化法[9-10]和水动力剪切法[11-16].这些方法均被用于DNA片段的生成,并各有优点和缺点.基于水动力剪切DNA的方法是目前大家公认的比较有效的方法之一,可以较好地得到随机分布并且分散度较小的DNA片段,并且不会对DNA造成损伤;但是目前的实验设备,对密闭性和压力要求严格,器件相对昂贵.Digilab公司生产的全自动基因组DNA剪切仪(Hydrodynamic DNA Shearing Instrument),利用水动力剪切可以获得长度为40~4 000 bp的DNA片段[17].针对便携式医疗器械和下一代基因测序技术来说,操作更容易、样品用量更少、无规片段化、片段长度可控和对样品无损害的方法,仍然是科学家们追求的梦想.
微流控芯片实验室(Lan-on-a-Chip)或称微全分析系统(Micro Total Analysis System),是有由MANZ与WIDMER[18]在1990年提出的. 通过这种技术可以把生物和化学领域中所涉及的样品制备、生物与化学反应、分离检测等基本操作单位集成或基本集成与一块几平方厘米的芯片上,用以完成不同的生物或者化学过程,并对其产物进行分析的一种技术[19].随着20世纪微纳米加工技术的飞速进步,微纳米器件已经广泛的应用于各个领域当中,包括电子信息、医疗器械、生物和化学分析以及药物筛选等[20].从微流控技术的特征来看,它可以精密地操控微米级的流体,是一种非常适合用来实现少量DNA样品处理(包括片段化)的方法[8, 21];并且这种技术的器件小(mm~cm)、便携性好、样品使用量少.在微流控的微通道中,流体的流速以及流动的变化不但可以整体控制流体的速度,而且可以通过对微流控通道的几何结构的设计来控制流体流速在微通道中的变化(加速或者减速).因此,可以控制流体对DNA样品的流体剪切力,从而快速剪切溶液中的DNA分子,得到短链的DNA片段.微流控芯片可以通过现有的微加工方法来简单地实现,因此也可以比较容易地与其他功能的芯片集成,从而可以实现快速高效的诊疗流程,实现个人诊疗系统的器件化.
1DNA片段化微流控芯片材料和结构
在微流控芯片器件中,流体的控制主要通过外加力场来控制,因此流体的流速和外加力场的压力差成正比,可表示为[22]264:
Q=∫vidA,
(1)
其中,Q(m3/s)、vi(m/s)、A(m2)和P(Pa)分别代表流体的体积流速、特定位置点i处流体的线性速度、微通道的横截面积和通道两端的压力差;Rhy代表微通道中流体流动产生的阻力.由式(1)可知,微流控通道中流体对DNA分子的剪切力来源于流体流动过程,因此,流体的流速越快或者外加力场越大,DNA片段化的效果越好.
基于目前常用的微米加工技术和材质的选择,玻璃/硅基微流控芯片可以耐受比较高的压力,因此被常用于DNA片段化的芯片器件(图1A)[21]3;但是为了降低剪切压力,通过分段和连续的方法可以提高剪切效率,同时降低速度,因此高分子材料的微流控芯片也可以被用于DNA片段化的微流控芯片(图1B)[12]1045.
2微流控芯片DNA片段化技术中DNA样品的处理和要求
片段化的DNA分子对于基因测序和疾病检测技术非常关键,因此要求DNA片段化的技术对样品

图1用于DNA片段化微流控芯片的玻璃和高分子材料
Figure 1DNA fragmentation microfluidic chips made of glass and polymer materials
不挑剔,不会带来污染和损害.大量的流体力学DNA片段化实验结果表明,这种技术对DNA样品基本不挑剔.human genomic DNA[12]、salmon sperm DNA、restriction fragments、cosmids、BACs、YACs[13]等不同种类的DNA样品均可以在微流控芯片中实现无规的有效DNA片段化,不同剪切速度下得到的DNA片段分布和DNA的来源无关.
DNA溶液中离子强度会导致溶液的粘度增加,通常DNA分子在高离子强度下更趋于形成团状的卷曲结构,因此,随着离子浓度增加,要将同一种DNA分子剪切到相同大小的片段所需要的剪切力(流体速度)越大(图2A);随着DNA浓度的增加,其粘度也增加,但是研究结果表明,在相同剪切力(流体速度)条件下,所产生的DNA片段差别不明显(图2B).
3流体速度和微通道尺寸的影响
众所周知,流体力学DNA片段化主要由流体对DNA分子的快速剪切作用造成,因此流体的剪切速度决定了最终DNA片段化的尺寸.大量实验表明,随着流体流速的增加,DNA片段化的尺寸减小;而在一定剪切速度下,DNA片段的大小随着时间的增加而减小,经过一段时间以后将达到平台不再变化. 即在一定的流体速度下,DNA分子能够被剪切得到稳定的最小尺寸的片段(图3)[21]4.
由式(1)可知,流体的体积流速和施加在微通道两端的压力差成正比,与流体在微通道中的流体阻力成反比,因此,在相同的体积流速下,微通道尺寸越小,其中的流体线性速度越大,可以将相同DNA分子片段化得到的尺寸越小.表1显示,在同一种微流控芯片中,当微通道尺寸从50 μm减小到10 μm,体积流速稳定在3 mL/min时,DNA片段从9 000 bp降低到1 870 bp,而Human DNA也从较大尺寸降低到2 320 bp[12]1045.
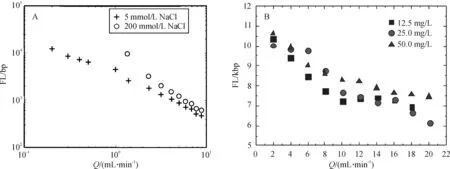
图2 DNA样品溶液离子强度(A)[12]3883和DNA浓度(B)[21]5对微流控片段化的影响
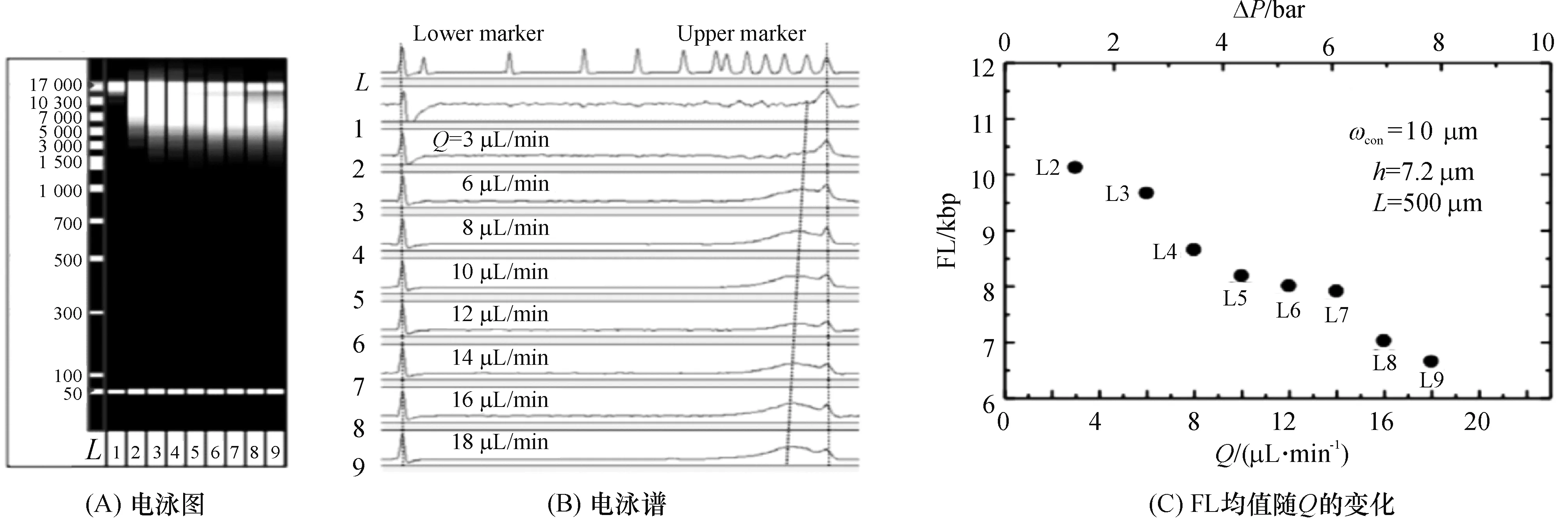
图3 微流控芯片中流体流速变化对剪切获得的DNA片段长度的影响[21]4
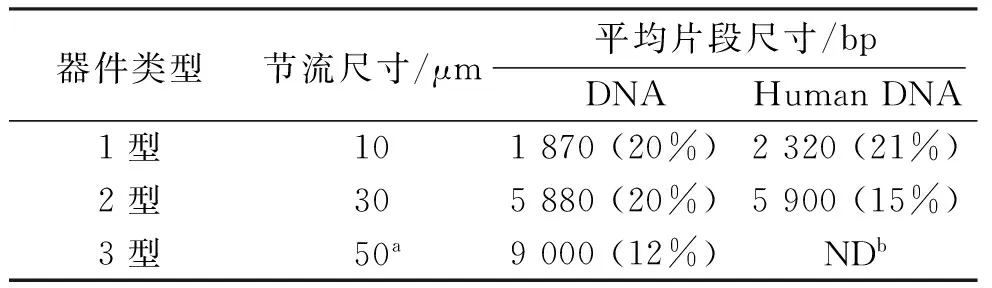
表1 不同尺寸微通道对DNA片段化结果的影响[12]1045
注:a表示流速为3 mL/min. NDb表示未检出.括号中的百分数为相对标准偏差.
4循环次数对微流控片段化的影响
虽然在微流控芯片中,流体的快速剪切可以将
DNA分子片段化,但是微流控通道的尺寸有限.在微流控芯片中,流体的阻力与通道的长度成正比,因此通道越长,阻力越大.一般来说,玻璃或者硅基的微流控芯片可以耐受106Pa数量级的压力,而塑料芯片一般只能耐受105Pa数量级的压力.通常情况下,微流控通道的长度可以控制在mm到cm级之间;但是为了减低达到片段化要求的高速流体流动,通道的尺寸不能太长,一般在mm到cm级之间.总之,当DNA溶液从微流控芯片的入口到出口流动的时间都在μs至ms级之间.溶液中存在大量的DNA分子,快速短暂的流动可以让部分DNA分子片段化,而大量的DNA分子来不及片段化就已经到达出口.研究发现,在流体力学剪切DNA片段化器件或设备中,可以通过多次循环样品来达到降低DNA片段尺寸和尺寸分布的效果.图4为样品在微流控通道中,相同流速下循环1~10次对DNA片段化结果的影响,循环次数的增加,不但可以降低DNA片段尺寸,还可以提高样品的回收率.
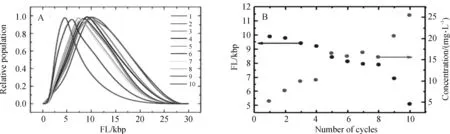
图4 固定流速下1~10次循环的DNA片段尺寸分布(A)、平均尺寸和质量浓度(B)[21]6
Figure 4Length distribution (A), average length and collected DNA concentration (B) of DNA fragments produced by shearing DNA solution through a microfluidic device at a fixed flow rated with 1 to 10 cycles[21]6
5温度对微流控片段化的影响
DNA溶液在低温下一般很稳定,可长时间保存;但是在高温下则会发生变性,核酸双螺旋碱基对的氢键断裂,双链变成单链,同时溶液的粘度降低.DNA解链的温度与分子中所含的碱基对成分有关,G和C越多则解链温度越高.THORSTENSON等[15]的研究表明,当工作温度在22~60 ℃时,流体力学剪切样品DNA获得的结果区别不大,而当温度>60°以后,相同剪切条件下DNA片段的尺寸明显减小.这可能是由于DNA分子由双螺旋结构解旋变成单链结构,因此在相同剪切速度下,更加容易被剪切变成更小的DNA片段(图5).
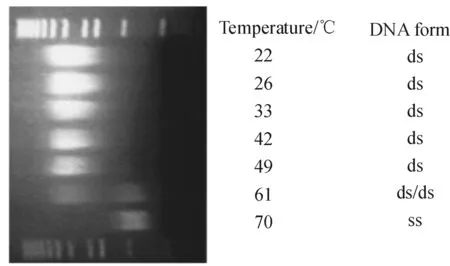
图5 温度对流体剪切DNA片段化的影响[15]851
Figure 5Effect of temperature on DNA fragmentation by hydrodynamic shearing[15]851
6结论
微流控技术可以精密准确地控制流体在微观尺度的流动和变化,流体快速流过微流控通道会对溶液中的分子产生剪切力作用,因此,这种技术可以用于DNA片段化研究.本文总结了微流控流体力学DNA片段化的影响因素和效果.DNA样品的来源、DNA的浓度对微流控DNA片段化影响不明显;溶液的离子强度可以增加溶液粘度和使DNA分子处于团簇状态,因此需要更大的剪切力才能获得相同尺寸的DNA片段;溶液在微流控芯片中多次循环可以降低DNA片段尺寸和尺寸分布,同时可以提高回收产率;工作温度在DNA解链温度以下,温度对片段化结果影响较小,当温度大于解链温度后,DNA解旋成单链因此更加容易被片段化.微流控流体剪切DNA片段化具有无规剪切、尺寸可控、分子破坏小和污染少等特点,结合微流芯片器件的尺寸小和用量少的优势,是一种非常适合于下一代基因测序和便携式医疗的新技术,正受到越来越多的关注.
参考文献:
[1]EL MUSTAPHA B, STAMBROOK P J. Next-generation sequencing technologies: breaking the sound barrier of human genetics[J]. Mutagenesis, 2014, 29(5):303-310.
[2]HEAD S R, KIYOMI K H, LAMERE S A, et al. Library construction for next-generation sequencing: overviews and challenges[J]. Biotechniques, 2014, 56(2):61-77.
[3]LIU L, LI Y H, LI S L, et al. Comparison of next-generation sequencing systems[J]. Journal of Biomedicine and Biotechnology, 2012,2012: Art 251364,7pp.
[4]HENGEN P N. Shearing DNA for genomic library construction[J]. Trends in Biochemical Sciences, 1997, 22(7):273-276.
[5]MANNEVILLE S, CLUZEL P, VIOVY J L, et al. Evidence for the universal scaling behaviour of a freely relaxing DNA molecule[J]. Europhysics Letters, 1996, 36(6):413-418.
[6]HOHEISEL J D, NIZETIC D, LEHRACH H. Control of partial digestion combining the enzymes dam methylase and MboI[J]. Nucleic Acids Research, 1989, 17(23):9571-9582.
[7]KASOJI S K, PATTENDEN S G, MALC E P, et al. Cavitation enhancing nanodroplets mediate efficient DNA fragmentation in a bench top ultrasonic water bath[J]. Plos One, 2015, 10(7): Art 0133014,15pp.
[8]TSENG Q, LOMONOSOV A M, FURLONG E E, et al. Fragmentation of DNA in a sub-microliter microfluidic sonication device[J]. Lab on A Chip, 2012, 12(22):4677-4682.
[9]LENTZ Y K, WORDEN L R, ANCHORDOQUY T J, et al. Effect of jet nebulization on DNA: identifying the dominant degradation mechanism and mitigation methods[J]. Journal of Aerosol Science, 2005, 36(8):973-990.
[10]SAMBROOK J, RUSSELL D W.Fragmentation of DNA by nebulization[J]. Cold Spring Harbor Protocols, 2006, 2006(4):1-3.
[11]JONEJA A, HUANG X H.A device for automated hydrodynamic shearing of genomic DNA[J]. Biotechniques, 2009, 46(7):553-556.
[12]NESTEROVA I V, HUPERT M L, WITEK M A, et al. Hydrodynamic shearing of DNA in a polymeric microfluidic device[J]. Lab on A Chip, 2012, 12(6):1044-1047.
[13]OEFNER P J, HUNICKE-SMITH S P, CHIANG L, et al. Efficient random subcloning of DNA sheared in a recirculating point-sink flow system[J]. Nucleic Acids Research, 1996, 24(20):3879-3886.
[14]SHUI L, SPARREBOOM W, SPANG P, et al. High yield DNA fragmentation using cyclical hydrodynamic shearing[J]. RSC Advances, 2013, 3(32):13115-13118.
[15]THORSTENSON Y, HUNICKE-SMITH S, OEFNER P, et al. An automated hydrodynamic process for controlled, unbiased DNA shearing[J]. Genome Research, 1998, 8(8):851.
[16]YEW F F H, DAVIDSON N. Breakage by hydrodynamic shear of the bonds between cohered ends of lambda-DNA molecules[J]. Biopolymers, 1968, 6(5):659-679.
[17]DIGILAB Inc. The best way to rragment DNA for genomic and paired end library construction, high throughput & long reads sequencing![EB/OL].[2016-01-12]. http://www.digilabglobal.com/--NEXTGEN-SHEAR.[18]MANZ A, GRABER N, WIDMER H M. Miniaturized total chemical analysis systems: a novel concept for chemical sensing[J]. Sensors & Actuators B: Chemical, 1990, 1(90):244-248.
[19]VO-DINH T.Development of a DNA biochip: principle and applications 1[J]. Sensors & Actuators B: Chemical, 1998, 51(1/2/3):52-59.
[20]CHEN G, WEI LI, DU WD, et al. Development of a DNA biochip for detection of known mtDNA mutations associated with MELAS and MERRF syndromes[J]. Hereditas, 2008, 30(10):1279-1286.
[21]SHUI L L, BOMER J G, JIN M L, et al. Microfluidic DNA fragmentation for on-chip genomic analysis[J]. Nanotechnology, 2011, 22(49):Art 494013,7pp.
[22]SHUI L, EIJKEL J C T, BERG A V D. Multiphase flow in micro- and nanochannels[J]. Sensors & Actuators B: Chemical, 2007, 121(1):264.
【中文责编:谭春林英文责编:李海航】
Recent Progress of DNA Fragmentation Based on Microfluidics
SHUI Lingling*, LI Lanhui, JIN Mingliang, ZHOU Guofu
(Institute of Electronic Paper Displays, South China Academy of Advanced Optoelectronics, South China Normal University, Guangzhou 510006, China)
Abstract:DNA fragmentation is one of the key technologies for gene library and disease detection. Normal DNA fragmentation technologies are reviewed focusing on microfluidic based DNA fragmentation technology. Effective parameters are discussed and summarized according to the resulted DNA fragments. The importance and perspective of microfluidic DNA fragmentation are summarized.
Key words:microfluidics; DNA fragmentation; fluidic shearing; gene sequencing
收稿日期:2015-12-31 《华南师范大学学报(自然科学版)》网址:http://journal.scnu.edu.cn/n
基金项目:国家自然科学基金项目(51405165);教育部“长江学者和创新团队发展计划”项目(IRT13064);广东省引进创新科研团队计划项目(2011D039)
*通讯作者:水玲玲,教授,国家“青年千人计划”入选者, Email:shuill@m.scnu.edu.cn.
中图分类号:TN27
文献标志码:A
文章编号:1000-5463(2016)01-0023-05

