台东苏铁中叶绿体功能基因RNA编辑的分析
沈成才
(北京师范大学克拉玛依附属学校,新疆 克拉玛依 834000)
台东苏铁中叶绿体功能基因RNA编辑的分析
沈成才
(北京师范大学克拉玛依附属学校,新疆 克拉玛依 834000)
摘要:台东苏铁是一种古老的裸子植物,有关苏铁的RNA编辑的研究很少。为此,我们分析了台东苏铁的RNA编辑位点和分布,为探究RNA编辑的功能和机制以及高等植物的起源和进化提供依据。本次以台东苏铁(Cycas taitungensis)为材料,采用异硫氰酸胍法提取台东苏铁总RNA。DNaseⅠ处理过的RNA 逆转得到的cDNA进行PCR扩增条带并测序。通过对测序结果分析比对,初步确定在台东苏铁中的ndhD2,PetB,ndhB2存在C-U 的编辑。研究表明,ndh基因有较高的编辑频率,而丝氨酸相比其他氨基酸有更高的编辑频率。结果显示,ndhB2中有C-Y的部分编辑现象,ndhA2存在沉默编辑现象。
关键词:台东苏铁;RNA编辑;RNA 提取;异硫氰酸胍法
RNA编辑是指转录后RNA成熟过程中的碱基的插入、缺失和替换等修饰和加工的过程,使得 RNA所携带的遗传信息发生改变,从而导致蛋白质序列的改变的过程[1]。RNA编辑广泛存在于动植物及微生物中,在叶绿体、线粒体和细胞核基因中均可发生,根据编辑效率的不同可分为沉默编辑、部分编辑和完全编辑三种[2]。叶 绿 体 的 RNA 编辑在高等植物中是广泛存在的,并在叶绿体基因的表达与调控方面起着重要的作用,是扩展原有遗传信息、增加基因表达调控途径的一种有效方式[3]。RNA编辑 还能够通过提高氨基酸的保守性来影响蛋白质的结构,进而影响其功能的发挥[4]。叶绿体RNA编辑具有组织特异性和发育阶段特异性,并受外界环境影响。目前, 已经测定叶绿体RNA编辑位点的高等植物有豌豆[5]、烟草[6]、拟南芥[7]、番茄[8]、角苔[9]、铁线蕨[10]、蝴蝶兰[11]、黄瓜[12]、棉花[13]、小麦[14]、菠菜[15]、玉米、水稻 、甘蔗、黑麦、大麦[16]、粗山羊草等,研究发现高等植物叶绿体中的RNA编辑主要以胞嘧啶转换成尿嘧啶的形式存在,且主要发生在密码子的第一、二位碱基,往往造成亲水性氨基 酸转变为疏水性氨基酸。目前,叶绿体RNA编辑位点的鉴定方法主要有生物信息学预测、高通量测序和PCR扩增等方法[17]。
目前,有关叶绿体RNA编辑的模型以及相关进化问题取得了较大进展,但裸子植物中关于叶绿体RNA编辑的研究的报道仅见于黑松[18]和银杏[19]等少数植物。现代裸子植物约有800种,隶属5纲即苏铁纲、银杏纲、松柏纲、红豆杉纲和买麻藤纲。苏铁(CycasrevolutaThunb),苏铁科苏铁属,又名凤尾蕉、避火蕉、金代、铁树等,苏铁科植物是世界上最古老的裸子植物,被地质学家誉为“植物活化石”[20]。有关苏铁RNA编辑的研究不多,李璐等人在常规的CTBA法中,加入了硼砂和β-巯基乙醇来消除多酚和多糖的干扰,从而得到了一个从苏铁叶片中有效提取 RNA 的方法[21]。台湾生物中央研究所赵淑妙教授预测台东苏铁中psbL基因只有一个RNA编辑位点,其它基因中没有RNA编辑现象[22]。江媛、陆萍等人认为,台东苏铁的叶绿体基因组中有40个蛋白编码基因有RNA编辑现象,在植物进化中,RNA编辑正在逐渐消失且密码子的第一、三位消失的更快,台东苏铁的叶绿体基因组的RNA编辑位点比线粒体少三倍[23]。
现阶段,对苏铁RNA编辑的研究较少,并且倾向与理论预测水平。计算机预测快速方便,但结果可能与真实情况存在偏差,也无法预测第三位的沉默编辑。从已有文献报道以及银杏编辑位点的分析,表明裸子植物中有较多RNA编辑现象。因此,本次通过实验手段并结合生物信息学的方法,对苏铁叶绿体8个功能基因进行RNA编辑位点的分析,确定苏铁中是否有无RNA编辑现象,为进一步全面了解苏铁叶绿体中功能基因全部编辑位点及其特点奠定基础。
1材料与方法
1.1材料
1.1.1实验材料
台东苏铁(Cycastaitungensis),购买于西安三森花卉市场。
1.1.2实验试剂
GT溶液(4 mol/L异硫氰酸胍,25 mmol/L柠檬酸钠,0.5%十二烷基肌氨酸钠);
10×TBE电泳缓冲液(108 g Tris; 55 g硼酸;9.3 g EDTA2Na;1 000mL;PH8.0);1%琼脂糖凝胶 (0.2 g琼脂糖;1×TBE缓冲液20 mL;2 μ EB);3 mol/L NaAc(pp.8),2 mol/L NaAc (ph1.8),4 mol/L LiCl,0.1﹪ DEPC水。无水乙醇、液氮、氯仿、水饱和酚酸、RT-PCR Kit、DNA酶等。
1.1.3实验仪器
DYY-12型电泳仪;PB-10型PH计;BS-124S型电子天平;YDS-30型液氮生物容器;DSX-280A型不锈钢手提式压力蒸汽灭菌锅;JB-3定时恒温磁力搅拌器;DYCP-31DN电泳槽;ALS1296型PCR仪;Centrifuge 5804R型台式冷冻离心机;Centrifuge 5417C型台式高速离心机;AGX-7601型通风厨;精密微量移液器;GDS-8000型凝胶成像系统;UNICAM UV300型紫外分光光度计;HH-2B型数显恒温水浴锅; DW-86L368型超低温冰箱;GZX-9140MBE型数显鼓风干燥箱;PE手套,离心管,PCR管,枪头。
1.2方法
1.2.1异硫氰酸胍法提取植物叶片总RNA
(1) 称取0.1 g新鲜植物叶片,加入液氮研磨成粉末,移入预冷的1.5 mL Eppendorf管中。
(2)加入300 μL GT溶液,混匀;加入30 μL 2mol/L NaAc(pH 4.8),反复振荡混匀;加入300 μL酚酸与100 μL氯仿,混匀。
(3)4 ℃,8 000 r/min离心15 min,弃沉淀,取上清移至另一DEPC水处理过的干净离心管中,加入2.5倍体积无水乙醇,-20 ℃放置5~6 h。 (-20 ℃可放置过夜)
(4) 4 ℃,8 000 r/min离心15 min,弃上清液。在沉淀中加入100 μL 4 mol/L LiCl,使沉淀溶解。
(5) 12 500 r/min离心15 min,弃上清液;在沉淀中加入100 μL灭菌后的DEPC水,混匀后再加入50 μL氯仿,50 μL水饱和酚混匀。
(6)12 500 r/min离心5 min,吸上清液移至另一DEPC水处理过的1.5 mL Eppendorf管,加入等体积氯仿。
(7)12 500 r/min离心5 min。吸上清液移至另一DEPC水处理过的1.5 mL Eppendorf管,加入1/10体积3 mol/L NaAc (pH 5.8)和两倍体积无水乙醇,-20 ℃放置过夜。
(8)12 500 r/min离心5 min,小心吸去上清,RNA沉淀在室温下稍干燥。
(9)加入20 μL DEPC处理过的灭菌水溶解,取5 μL样品经1.0%琼脂糖凝胶电泳检测(100v,100 mA,20 min) RNA质量,并取2 μL样品加入498 μL DEPC处理过的水,用紫外分光光度计测定RNA浓度;其余样品-20℃保存备用。
1.2.2待扩增基因引物设计
确定RNA编辑位点较多的8个基因,psbL、psbF、psbE、PetB、PetD、ndhB、ndhA、ndhD,从NCBI上查出台东苏铁Cycastaitungensis(序列号[NC-009618])全部叶绿体DNA序列和cDNA序列,选取编码框前后100 bp处的核苷酸序列,用相关软件设计并合成引物。
引物名称及序列见表1。
设计引物所用到的软件有:
http://www.sigmagenosys.com/calc/DNAcalc.asp
http://www.bioinformatics.vg/bioinformatics_tools/reversecomplement.shtml
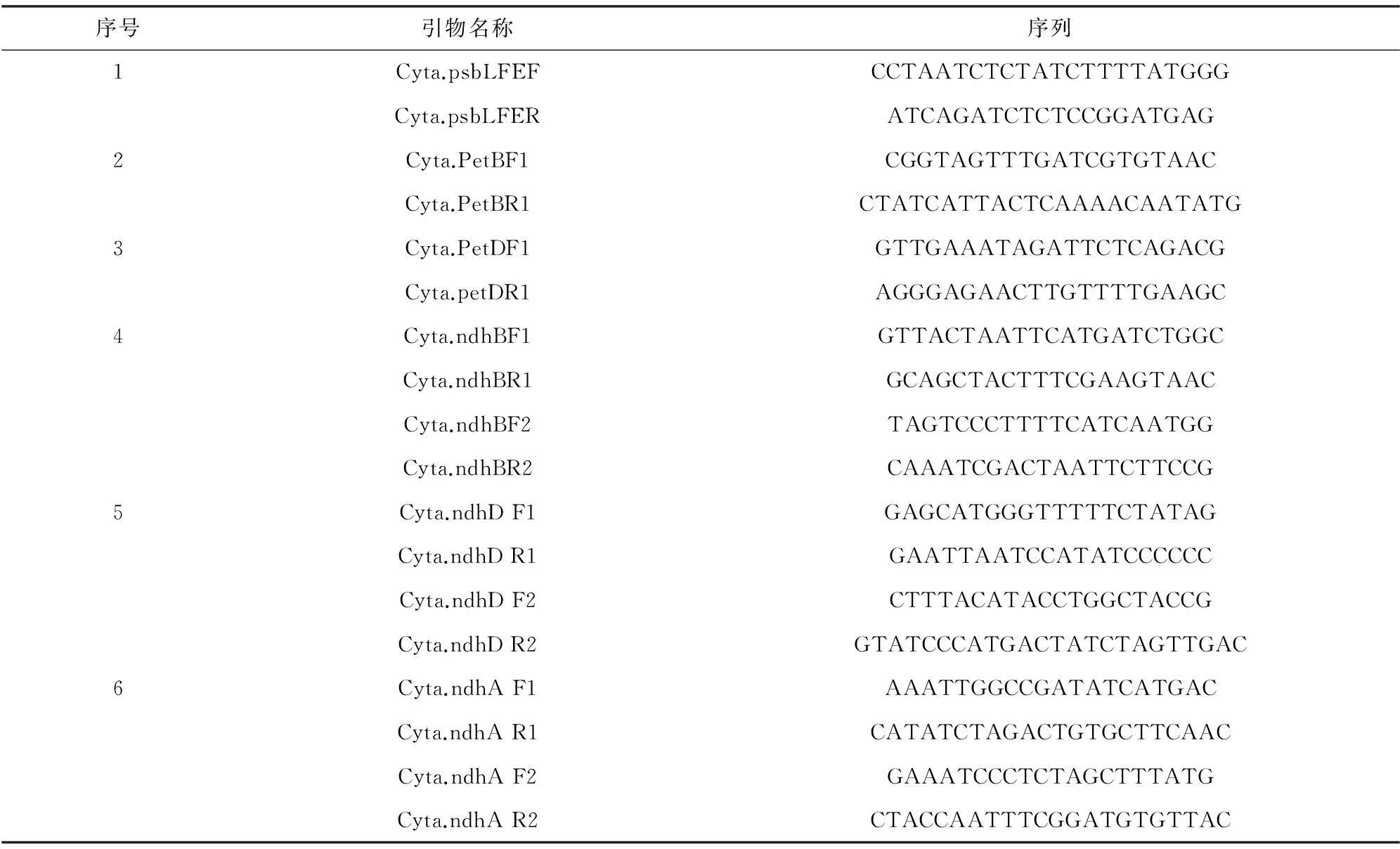
表1 引物名称及序列
1.2.3RT-PCR扩增
用RNase-free的DNaseI去除混杂在总RNA中的基因组DNA,采用逆转录试剂盒进行逆转录(具体操作按照试剂盒说明书进行),用3 μL的RNA为模板,对逆转得到的cDNA,进行PCR扩增。PCR反应体系2 μL 台东苏铁的cDNA, 12.5 μL 2 ×TaqPCR Mix,上、下游引物各2 μL,用水补足至25 μL。反应程序如下: 95 °C 3 m; 95 °C 1 m, 55 °C 1 m, 72 °C 1 m, 30个循环;72 °C延伸10 m。PCR扩增产物于4 °C保存。取2 μL样品经1.0%琼脂糖凝胶电泳检测(100 v,100 mA,20 min)PCR扩增条带。
1.2.4测序结果分析比对
采用Lesergene中seqman软件对测序结果拼接和分析,并用ClastW软件比对台东苏铁DNA和cDNA序列,确定RNA编辑位点。
ClastW 软件来自于http://www.ebi.ac.uk/Tools/clustalw/index.html
Translate Tool 软件http://ca.expasy.org/tools/dna.html
2结果和分析
2.1RT-PCR扩增
通过查阅国内外文献,确定了叶绿体中8个发生RNA编辑现象可能性比较大的基因,即:psbL、psbF、psbE、PetB、PetD、ndhB、ndhD和ndhA。并对这8个基因设计引物,进行RT-PCR扩增。结果psbL、psbF、psbE和ndhB1未扩增出条带,原因待查。图1显示其余基因均扩增结果良好,为单一条带。其中PetD虽然扩增出条带(图1泳道2),但分子量大小与预期结果不符,可能不是想要扩增的条带,故舍去。ndhD1和ndhA1在同等PCR扩增条件下得到的条带很淡(图1泳道4和7),在加大cDNA的用量之后,得到了符合测序的条带,但测序结果显示ndhD1和ndhA1的测序信号中断,无结果。可能是由于送去测序的样品中所含杂质较多,或测序本身的问题所造成。上述PCR扩增结果表明扩增条带好坏与引物设计,模板用量以及PCR体系的优化均有密切关系,而测序结果则与样品浓度,公司测序反应有关。
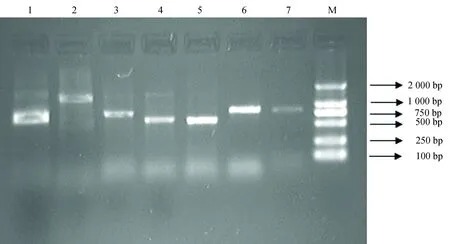
图1 PCR扩增条带的电泳结果Fig.1 Electrophoresis result of PCR amplification bands
2.2测序结果分析
经RT-PCR扩增和测序最终得到了PetB、ndhB2、ndhD2和ndhA2的cDNA测序结果。通过与NCBI所查到的台东苏铁DNA的序列分析比对,从表2可知在ndhD2的五个位( 34,400,469,570,706 bp)存在C-U的完全编辑现象,对应Pro-Leu,Ser-Phe,Ser-Phe,His-Tyr,Thr-Ile的氨基酸转变;在PetB的634bp位点存在 C-U的完全编辑,造成Pro到Ser氨基酸转变;在ndhA第二外显子350 bp位点存在U-Y沉默编辑;在ndhB2的203位点发生C-Y部分编辑, 同时404,509,533,695位点存在C-U的完全编辑,对应造成Ser-X,Ser-Leu,Ser-Leu,Pro-Leu,Thr-Ile氨基酸的转变。ndhB2位点533的完全编辑与203的部分编辑比较见图2和图3。经过对苏铁叶绿体中的ndhD2、PetB、ndhA和ndhB的编辑位点的分析,初步确定在台东苏铁中存在完全编辑、沉默编辑和部分编辑现象,且核苷酸的转换有C-U的转变方式。
3讨论与结论
本文采用异硫氰酸胍法获取台东苏铁的总RNA,成本低且效果好。经RT-PCR扩增和测序比对,得知台东苏铁叶绿体功能基因中存在完全编辑、沉默编辑和部分编辑现象。其中ndhD2的五个位点存在C-U的RNA完全编辑;在PetB的位点存在1个 C-U的完全编辑;在ndhA第二外显子350 bp位点存在U-Y沉默编辑;在ndhB2的203位点发生C-Y部分编辑, 同时有4个位点存在C-U的完全编辑。
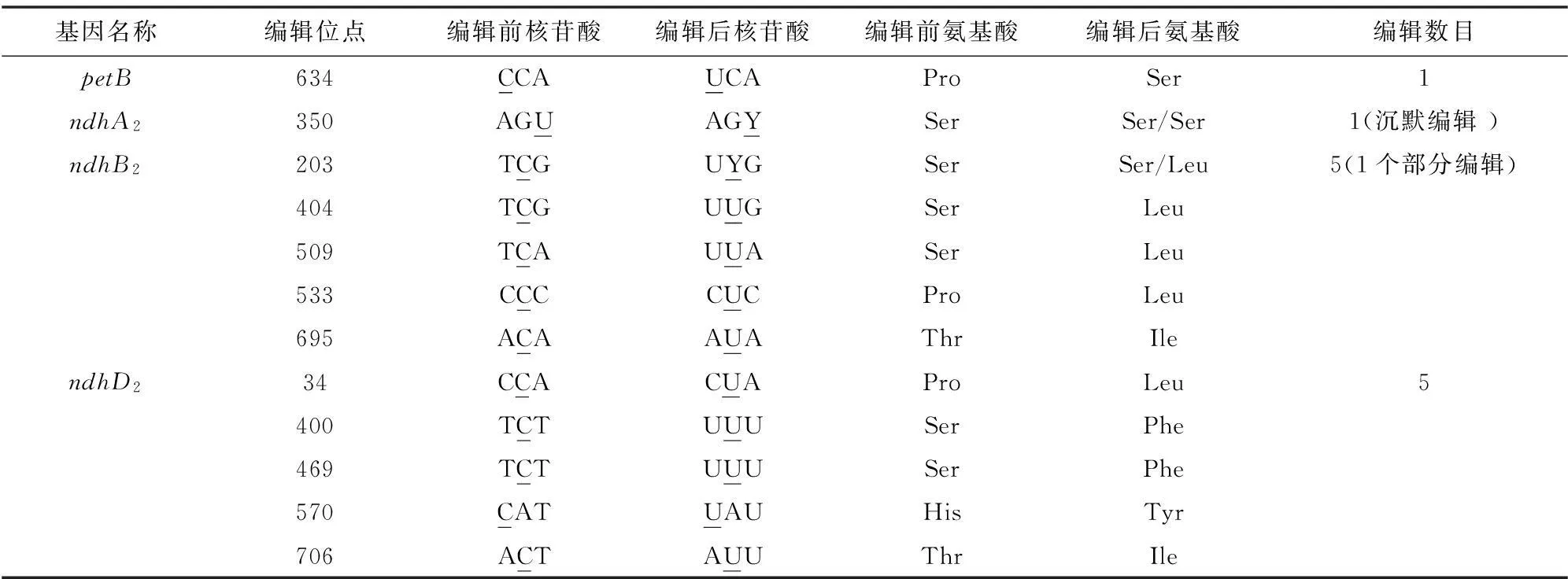
表2 台东苏铁中四个叶绿体基因的编辑前后核苷酸和氨基酸变化的统计结果
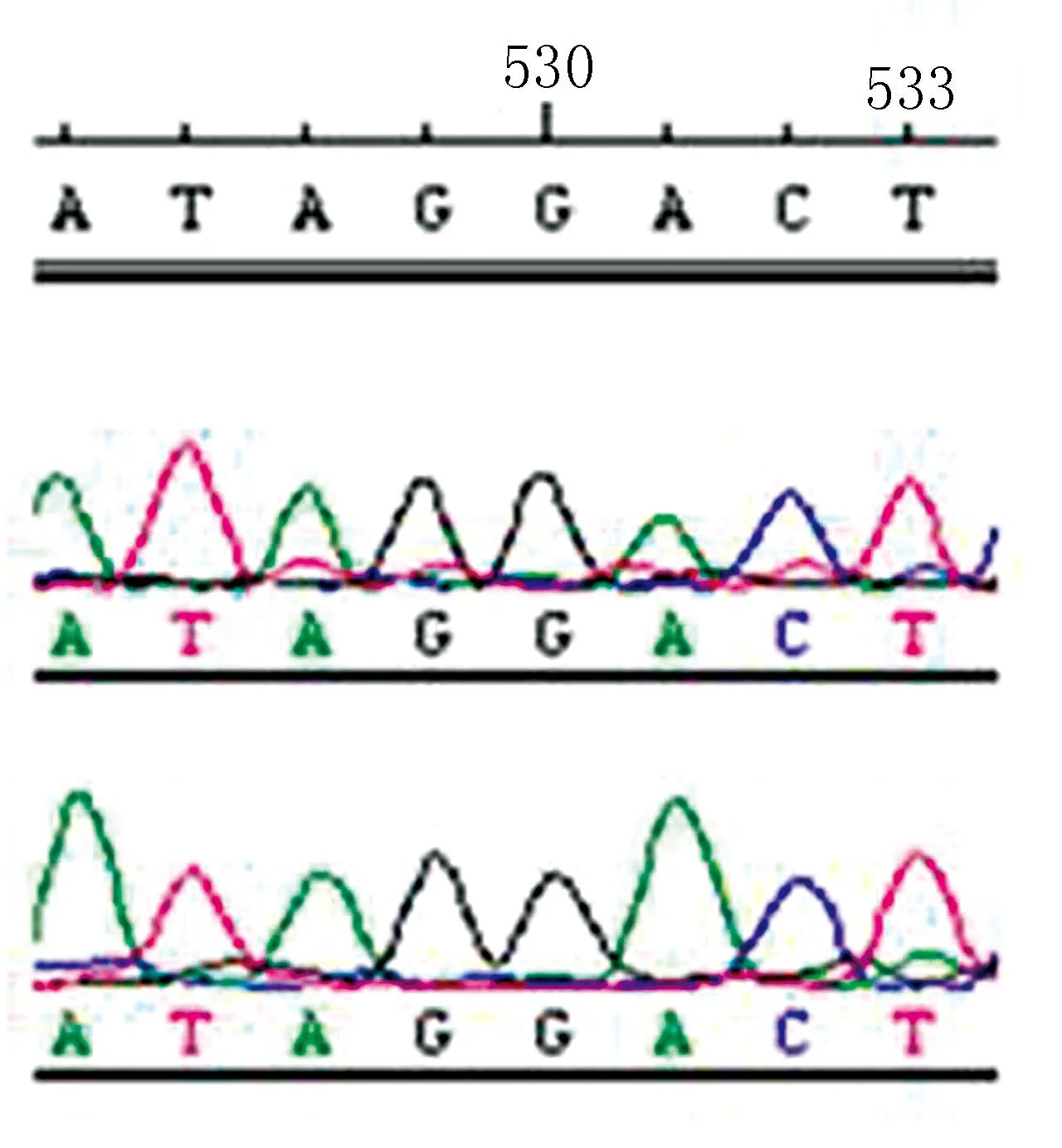
图2 ndhB2完全编辑Fig 2 Completely editing sites in ndhB2
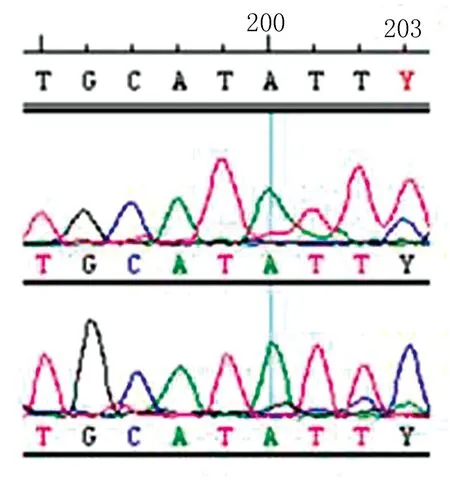
图3 ndhB2部分编辑 Fig 3 Partial editing sites in ndhB2
这说明台东苏铁叶绿体功能基因存在RNA编辑现象,大部分编辑呈现C-U的转变,ndh类基因中RNA编辑发生频率较高,并且丝氨酸相比其他氨基酸有更高的编辑频率。这一结论与江媛、陆萍等人的研究具有一致性,在petB的完全编辑和ndhD有5个完全编辑位点方面的研究结果也是具有一致性[23]。测序结果显示ndhB有4个位点完全编辑与1个位点部分编辑,ndhA有一个沉默编辑位点,这与陆萍等人的研究中提到ndhB仅有一个完全编辑位点且ndhA中未出现沉默编辑的结论不一致,这可能与材料的选择、不同发育阶段以及环境因素有关,有待进一步研究。
RNA编辑是高等植物叶绿体转录本成熟过程中的一种重要修饰和加工方式。RNA编辑使人们对生物的长期进化中所形成的遗传信息表达和调控机制有了一定的认识,也加深了对复杂的生命现象的认识。已有研究表明,RNA编辑位点在进化过程中呈减少趋势,但一些重要的位点会继续保留[24]。台冬苏铁中的ndhB的部分编辑位点是调控基因表达的一种方式,还是进化过程中会消失的位点, 有待进一步的研究。ndhA中存在沉默编辑位点,而近年来对于沉默编辑功能的认识也存在争议。Hirose等人认为沉默编辑与叶绿体的照光程度和发展阶段相关[25];Nakamura和Sugiura的研究表明, 叶绿体基因组存在密码子的偏好性, 沉默编辑会影响蛋白质翻译的效率[26];也有学者认为沉默编辑似乎是多余的,在今后的进化过程中会消除[27]。
总之,RNA 编辑不但使编码基因组的遗传信息得到了扩增,同时对基因的表达产物有所影响。目前对 RNA 编辑的机制还不完全清楚,尤其是台东苏铁属于较古老的裸子植物,对其叶绿体RNA 编辑的研究具有一定的生物学意义。为进一步全面了解苏铁叶绿体中功能基因全部编辑位点的特点和功能奠定了基础,也为研究生物进化方面提供一定的参考价值。
参考文献
[1]程水源,陈昆松,杜何为,等.银杏RNA的提取[J].果树学报,2005,22(4):428-429.
CHENG Shuiyuan ,CHEN Kunsong ,DU Hewei ,et al .Total RNA extraction of Ginkgo biloba leaves[J].Journal of Fruit Science,2005,22(4):428-429.
[2]MA Yanli,YU Jianing.The progress of RNA editing in higher plant chloroplast[J].Chinese Bulletin of Life Science,2009,21(3):438-443.
[3]马红战,张保军,樊虎玲.大麦叶绿体基因RNA编辑位点的预测与鉴定[J].麦类作物学报,2013,33(6):1071-1077.
MA Hongzhan,ZHANG Baojun,FAN Huling.Predication and identification of RNA editing sites in the Chloroplast genome of barley[J].Journal of Triticeae Crops, 2013 ,33(6) :1071-1077.
[4]石文清,邓平川,李变丽,等.二穗短柄草叶绿体RNA编辑位点的预测及其鉴定[J].麦类作物学报,2012,32 (1):28-35.
SHI Wenqing ,DENG Pingchuan,LI Bianli ,et al.Prediction and identification of RNA editing sits in Chloroplast transcripts of brachypodium distachyon[J].Journal of Triticeae Crops,2012,32 (1):28-35.
[5]INADA M,SASAKI T,YUKAWA M,et al. A systematic search for RNA editing sites in peachloroplasts: an editing event causes diversification from the evolutionarily conserved amino acid sequence[J].Plant & Cell Physiology,2004,45(11):1615-1622.
[6]HIROSE T,KUSUMEGI T,TSUDZUKI T,et al.RNA editing sites in tobacco chloroplast transcripts:editing as a possible regulator of chloroplast RNA polymerase activity[J]. Molecular General Genetics,1999,262(3):462-467.
[7]TSUDZUKI T,WAKASUGI T,SUGIURA M. Comparative analysis of RNA editing sites in higher plant hloroplasts[J].Journal of Molecular Evolution,2001,53(4):327-332.
[8]KAHLAU S,ASPINALL S,GRAY J C,et al. Sequence of the tomato chloroplast DNA and evolutionary comparison of solanaceous plastid genomes[J]. Journal of Molecular Evolution,2006,63: 194-207.
[9]KUGITA M,YAMAMOTO Y,FUJIKAWA T, et al. RNA editing in hornwort chloroplasts makes more than half the genes functional[J].Nucleic Acids Research,2003,31(9):2417-2423.
[10]WOLF P G,ROWE C A,HASEBE M. High levels of RNA editing in a vascular plant chloroplast genome: analysis of transcripts from the fernAdiantumcapillus-veneris[J]. Gene,2004,339:89-97.
[11]ZENG W H,LIAO S C,CHANG C C. Identification of RNA editing sites in chloroplast transcripts ofPhalaenopsisaphroditeand comparative analysis with those of other seed plants[J]. Plant & Cell Physiology,2007,48(2):362-368.
[12]GUZOWSKA-NOWOWIEJSKA M, FIEDOROWICZ E,PLADER W . Cucumber,melon,pumpkin,and squash: are rules of editing in flowering plants chloroplast genes so well known indeed[J].Gene,2009,434(1-2):1-8.
[13]江媛,范术丽,宋美珍,等.棉花叶绿体基因 RNA编辑位点的测定及分析[J]. 植物学报,2011,46(4):386-395.
JIANG Yuan ,FAN Shuli , SONG Meizhen,et al.Identification and analysis of RNA editing sites in Chloroplast transcripts of Gossypium hirsutum[J].Chinese Bulletin of Botany ,2011, 46 (4): 386-395.
[14]邓李坤,李妍,俞嘉宁.小麦叶绿体蛋白质编码基因RNA编辑位点的测定及与返白现象的关系[J].植物学报,2012,47(6):581-593.
DENG LIkun ,LI Yan ,YU Jianing .RNA editing sites in Chloroplast protein-coding genes in leaf white mutant of triticum aestivum[J].Chinese Bulletin of Botany,2012,47(6):581-593.
[15] BOCK R,HAGEMANN R,KöSSEL H,et al. Tissue-and stage specific modulation of RNA editing of the psbF and psbL transcripts from spinach plastids-a new regulatory mechanism[J].Molecular & Genetic General,1993,240(2): 238-244.
[16]DIEKMANN K,HODKINSON T R,WOLFE K H,et al.Complete Chloroplast genome sequence of a major allogamous forage species,perenial ryegrass[J]. DNA Research,2009,16(3):166-176.
[17]王梦醒,詹豪爽,吕萌荔,等.粗山羊草叶绿体基因RNA编辑位点的鉴定与分析[J].麦类作物学报,2014,34(10):1341-1349.
WANG Mengxing ,ZHAN Haoshuang ,LU Mengli ,et al .Identification and analysis of RNA editing sites in Chloroplast transcripts of aegilops tauschii[J].Iournal of Triticeae Crops,2014,34(10):1341-1349.
[18]EVI K,WERNER H , DIETER E.A simple and efficient protocol for isolation of functional RNA from plant tissues rich in secondary metbolites[J].Plant Molecular Biology Reporter,2000,18(1):33-39.
[19]马艳莉,陈海燕,王继刚,等.银杏叶绿体ndhF RNA编辑现象分析及C290位编辑对胁迫处理的响应[J].植物学报,2011,46(1):1-10.
MA Yanli ,CHEN Haiyan,WANG Jigang ,et al .Analysis of editing sites for ChloroplastndhFin Ginkgo biloba and the editing efficiency at C290 in response to different stresses[J].Chinese Bulletin of Botany,2011,46(1):1-10.
[20]TAKAHIKO T,TATSUYA W,MASAHIRO S. Comparative analysis of RNA editing sites in higher plant Chloroplasts[J].Journal of Molecular Evolution,2001,53(4-5):327-332.
[21]李璐,付乾堂,余迪求.一种从苏铁叶片中有效提取RNA的方法[J].云南植物研究,2008,30(5) :593-596.
LI Lu ,FU Qiantang ,YU Diqiu .An effective protocol for the isolation of RNA from Cycad leaves[J].Acta Botanica Yunnanica,2008,30(5):593-596.
[22]WU C S,WANG Yanan,LIU Shumei,et al. Chloroplast genome (cpDNA) of Cycas taitungensis and 56 cp protein-coding genes of Gnetum parvifolium: Insights into cpDNA evolution and phylogeny of extant seed plants[J]. Molecular Biology and Evolution,2007,24(6):1336-1379.
[23]CHEN Haiyan,DENG Likun,JIANG Yuan,et al.RNA editing sites exist in protein-coding genes in the Chloroplast genome of Cycas taitungensiss [J]. Journal of Integrative Plant Biology ,2011,53(12): 961-970.
[24]FIEBIG A,STEGEMANN S,BOCK R.Rapid evolution of RNA editing sites in a small non-essential plastid gene[J].Nucleic Acids Research,2004,32(12):3615-3622.
[25]HIROSE T,FAN H,SUZUKI J Y,et al.Occurrence of silent RNA editing in chloroplasts: its species specificity and the influence of environmental and developmental conditions[J].Plant Molecular Biology,1996,30(3):667-672.
[26]NAKAMURA M,SUGIURA M . Translation efficiencies of synonymous codons are not always correlated with codon usage in tobacco chloroplasts[J].The Plant Journal,2007,49(1):128-134.
Analysis of RNA editing sites in the chloroplast genome ofCycastaitungensis
SHEN Chengcai
(KaramaySchoolAttachedToBeijingNormalUniversity,KaramayXinjiangUygurAutonomousRegion834000,China)
Abstract:Cycas taitungensis is ancient gymnosperms,and the research of RNA editing in Cycas is limited.We analyzed the composition and distribution of RNA editing in Cycas taitungensis,which provided the useful information for exporing the function and mechanism of RNA editing,as well as for revealing the origin and evolution of higher plants.The thiocyanate method is appropriate for the RNA extraction of Cycas taitungensis. The cDNA was obtained by reverse transcribed the DNA-free RNA which treated with DNase.The RNA editing sites were found by RT-PCR,sequence and blast DNA and cDNA. Our results showed that the conversion of C-to-U and C-to-U editing events could be found in the ndhD2 and PetB transcripts of Cycas taitungensis as well as in ndhB(2 )transcript. We found that ndh genes have a higher rate of editing,while the serine codon was more frequently edited than the others.The results showed that there is a partially editing sites in ndhB(2 ) transcrip,and silence editing sites also could be found in the ndhA2 transcript.
Keywords:Cycas taitungensis;RNA editing;RNA extraction;Guanidine thiocyanate.
中图分类号:Q51
文献标志码:A
文章编号:1672-5565(2016)01-013-06
doi:10.3969/j.issn.1672-5565.2016.01.03
作者简介:沈成才,女,中教一级,研究方向:生物技术; E-mail: shencc_1@sina.cn.
收稿日期:2015-04-29;修回日期:2015-11-23.

