贵州省布依族和苗族HLA-C基因rs3130542位点多态性研究*
赵孝梅, 何 燕, 张旺德, 张 婷, 王婵娟, 吴昌学, 官志忠
(贵州医科大学 分子生物学重点实验室, 贵州 贵阳 550004)
贵州省布依族和苗族HLA-C基因rs3130542位点多态性研究*
赵孝梅, 何燕**, 张旺德, 张婷, 王婵娟, 吴昌学, 官志忠
(贵州医科大学 分子生物学重点实验室, 贵州 贵阳550004)
[摘要]目的: 研究HLA -C 基因rs3130542位点在贵州省布依族和苗族人群中的分布特征,探讨该位点在不同群体间的分布差异。方法: 应用TaqMan-MGB探针基因分型方法,检测贵州布依族(289例)和苗族(174例)群体HLA -C 基因rs3130542 SNP多态位点的基因频率及基因型频率,比较同一性别不同民族及同一民族不同性别人群间HLA -C 基因rs3130542 SNP多态位点的基因频率及基因型频率,并与人类基因组计划网站公布的中国北京人群、日本人群、欧洲人群及非洲人群的分布特点进行比对。结果: 布依族和苗族群体比较HLA-C 基因rs3130542位点等位基因频率和基因型频率差异有统计学意义(P<0.05),同一民族群体不同性别之间比较HLA-C 基因rs3130542位点多态性分布差异均无统计学意义(P>0.05);与人类基因组计划网站公布的其他人群相比,贵州苗族布依族人群HLA-C 基因rs3130542位点的基因型频率与中国北京人群间差异有统计学意义(P<0.05),等位基因和基因型频率与日本人群间差异均有统计学意义(P<0.05),但与欧洲人群间差异无统计学意义(P>0.05),等位基因频率与非洲人群间差异有统计学意义(P<0.01)。结论: HLA-C 基因rs3130542位点在不同地域、不同民族或种族之间分布存在一定的差异;但在贵州省苗族和布依族同一民族男女性别中的分布差异无统计学意义。
[关键词]HLA-C抗原; 基因多态性; 等位基因; 基因型; 少数民族
HLA-C是人类白细胞抗原(human leukocyte antigen, HLA)的一个基因座,属于经典的HLA-Ι类分子。HLA-C所含的等位基因相对较少但存在着高度的多态性[1]。近年来有关HLA-C基因多态性与疾病进展的研究不断地增加和深入,发现HLA-C基因多态性与许多疾病如HBV感染[2]、HIV感染[3]、神经纤维瘤病Ι型[4]、早期复发性流产[5]、梅毒[6]、人类T淋巴细胞白血病病毒I型(HTLV-1)[7]等均存在一定的相关性。由于特定的生活习俗和特殊的地理分布使得某些疾病在少数民族人群中发病率较高,因此本研究选取贵州省人口较多的2个少数民族(布依族和苗族)为研究对象,分析其群体中HLA-C 基因rs3130542位点的分布特征,为进一步研究HLA-C 基因与某些疾病的相关性及关联机制提供一定的参考,同时也为群体遗传学的研究提供一定的基础参考数据,现报道如下。
1对象与方法
1.1对象
根据知情同意原则,随机采集贵州省世居布依族和苗族无关个体外周血2 mL,所选的研究对象三代内均无族外通婚,彼此间无直接的血缘关系,无遗传病史。两个民族共463例。其中,布依族289例, 男134例,(50.17±9.19)岁,女155例,(48.12±3.54)岁;苗族174例,男70例,(49.99±19.80)岁,女104例,(49.14±19.80)岁。两个民族研究对象的年龄和性别比较差异无统计学意义(P>0.05)。
1.2方法
1.2.1DNA提取采用德国QIAGEN公司的FlexiGenen DNA Kit 全血基因组DNA提取试剂盒提取外周血DNA,并进行DNA浓度和纯度检测(DU 640蛋白质核酸定量分析仪,美国Beckman公司),DNA终浓度标化至30 g/L,于-20 ℃冰箱保存。
1.2.2基因分型采用美国ABI公司的StepOne plus 实时荧光定量PCR仪和TaqMan SNP Genotyping Assays对HLA-C基因rs3130542位点进行基因分型。PCR反应为10 μL体系:DNA溶液(30 g/L)1.5 μL,×Taqman Universal Master Mix 5.0 μL,20×Assay Working Stock 0.25 μL加ddH2O补足10 μL。PCR反应条件为:60 ℃ 30 s,95 ℃ 10 min,95 ℃ 15 s、60 ℃ 1 min, 共55个循环,60 ℃ 30 s。用Step One Software v2.1软件根据荧光发光的特点分析基因分型的结果。
1.2.3测序验证根据基因分型结果,每种基因型随机抽取5个样本送Life公司测序验证。测序引物如下:上游5′-GACTTCCCGCAACAACAG-3′,下游5′-CAT CTTTCCACCCATTCC-3′。
1.3观察指标
对采集的血液标本进行基因型和等位基因频率Hardy-Weinberg 平衡检验,观察布依族和苗族人群rs3130542位点基因型和等位基因频率,比较两个群体间不同性别个体、群体内部不同性别个体的rs3130542位点基因型和等位基因频率,并将贵州地区苗族布依族人群中HLA-C 基因rs3130542位点的多态性分布频率与人类基因组计划网站(http://www.hapmap.org)公布的不同地区人群的扫描结果进行比较。
1.4统计学分析
卡方检验分析rs3130542位点基因型频率分布是否符合Hardy-Weinberg平衡。采用SPSS19.0软件对结果进行分析,基因型和等位基因型频率用直接计数法计算,组间分布差异用卡方检验进行分析,P<0.05为差异有统计学意义。
2结果
2.1Hardy-Weinberg 平衡检验
本研究群体基因型和等位基因频率均符合Hardy-Weinberg 平衡(P>0.05),提示本研究所选取的样本具有较好的群体代表性。
2.2HLA-C基因rs3130542 SNP位点基因分型

注:A为HLA-C A/G 位点PCR扩增图;B 为SNP分型图,图中红点代表野生型HLA-C AA纯合子,蓝点代表纯合突变型HLA-C GG,绿点代表杂合型HLA-C AG,黑色方块代表空白对照(NTC)图1 人基因组rs3130542(HLA-C)SNP位点的TaqMan探针实时荧光定量PCR结果Fig.1 The genotyping results of rs3130542 (HLA-C) SNP loci by using TaqMan probe real-time quantitative PCR
2.2.1HLA-C基因分型人基因组rs3130542(HLA-C)SNP位点的TaqMan-MGB探针实时荧光定量PCR扩增曲线如图1A。根据人基因组rs3130542(HLA-C)TaqMan探针分型试剂盒(TaqMan® SNP Genotyping Assays)信息,并采用StepOne Software v2.1软件分析PCR 扩增后收集的荧光,等位基因分布图中X轴代表VIC探针(HLA-C AA),Y轴代表FAM 探针(HLA-C GG),样品集中分布在4个区域:3个空白对照组在接近原点的对角线上,靠近X轴的红点代表该样本为HLA-C AA野生型;靠近Y轴的蓝色点则代表该样本为HLA-C GG纯合突变型;处于对角线上的绿色点则为HLA-C AG杂合型(图1B)。2.2.2测序验证结果从HLA-C A/G(rs3130542)基因分型结果中随机选取5%的样本进行测序验证,测序结果显示与TaqMan-MGB探针实时荧光定量PCR对基因分型的结果完全符合。测序结果见图2。
2.3同一性别不同民族群体HLA-C A/G(rs3130542)基因分型和等位基因频率分布
布依族与苗族群体比较HLA-C A/G(rs3130542)基因型(χ2=7.987,P=0.018)及等位基因频率(χ2=8.742,P=0.003)差异有统计学意义,见表1。男性群体中,布依族、苗族人群比较HLA-C A/G(rs3130542)基因型及等位基因频率分布差异无统计学意义(P>0.05);女性群体中,布依族和苗族人群比较HLA-C A/G(rs3130542)等位基因频率分布差异有统计学意义(χ2=4.876,P=0.027),基因型分布差异无统计学意义(P>0.05),见表2。
2.4同一民族不同性别人群中HLA-C A/G(rs3130542)基因型及等位基因频率分布
贵州省布依族和苗族同一群体不同性别之间比较,HLA-C A/G(rs3130542)基因型分布和等位基因频率均差异无统计学意义(P>0.05),见表3。
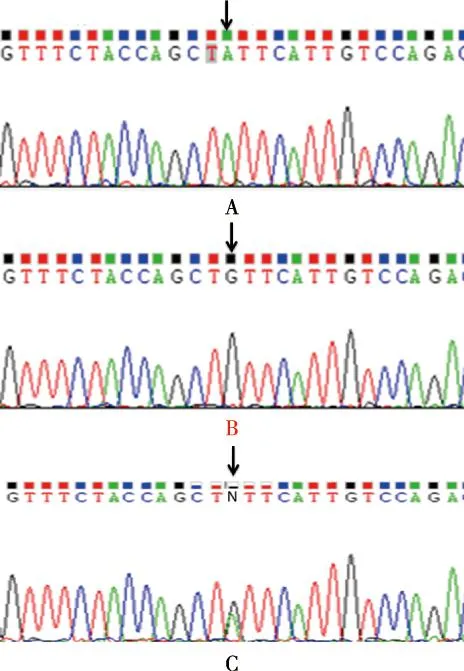
注: A、B、C分别代表测序结果为AA纯合子、GG纯合子和AG杂合子(箭头所指处)图2 HLA-C基因rs3130542(A/G)位点测序结果Fig.2 The sequencing results of HLA-C gene rs3130542 (A / G) locus
2.5贵州地区苗族布依族人群与其它地区人群HLA-C 基因rs3130542位点多态性的比较
将贵州地区苗族布依族人群中HLA-C 基因rs3130542位点的多态性分布频率与人类基因组计划网站(http://www.hapmap.org)公布的其它地区人群的扫描结果进行比较,结果显示,贵州地区苗族布依族人群HLA-C 基因rs3130542位点与与中国北京人群比较基因型分布差异有统计学意义(P<0.05),与日本人群(JPT)比较基因型和等位基因频率差异有统计学意义(P<0.05),与欧洲人群(CEU)比较差异无统计学意义(P>0.05),与非洲人群(YRI)比较等位基因频率差异有统计学意义(P<0.01),见表4。

表1 贵州省布依族和苗族群体HLA-C基因rs3130542位点基因型及等位基因频率分布(n,%)

表2 同一性别布依族和苗族群体HLA-C基因rs3130542位点

表3 贵州省布依族和苗族同一群体不同性别个体HLA-C基因rs3130542位点

表4 不同地区人群HLA-C A/G(rs3130542)基因型及等位基因频率比较(n,%)
3讨论
HLA-C可作为杀伤细胞免疫球蛋白样受体(killer-cell immunoglo-bulin-like receptor, KIR)的配体,并与之相结合影响NK细胞的活性[8-9],进而在免疫调节中发挥重要的作用。而国际人类基因组单体型图计划(Hap map project)指出人类基因组中约有300多万个单核苷酸多态性(single nucleotide polymorphism, SNP)位点[10]。SNP具有多态信息含量大、密度高、遗传稳定和易于检测等优点,因而非常适宜用于常见疾病的易感基因研究。
本研究的受检人群中,布依族和苗族比较HLA-C A/G(rs3130542)等位基因频率和基因型差异有统计学意义(P<0.05),因此可推测HLA-C基因rs3130542 SNP位点多态性分布存在民族差异性。虽然布依族和苗族同为汉藏语系,但二者分属不同的语族群体(分别为壮侗语族、苗瑶语族),随着历史的不断演变与人类的进化已使它们各自的遗传背景发生了一定的改变,进一步导致遗传多样性和特异性的存在。同一性别不同民族群体之间比较结果显示,女性群体中布依族和苗族人群比较HLA-C基因rs3130542 SNP位点等位基因频率分布差异有统计学意义(P<0.05);而同一民族群体不同性别之间比较HLA-C基因rs3130542 SNP位点多态性分布差异均无统计学意义;提示HLA-C基因rs3130542 SNP位点多态性分布的性别差异性不明显。
贵州地区苗族布依族人群与人类基因组计划网站公布的不同地区及种族的HLA-C基因rs3130542 SNP位点多态性进行比较可知:贵州苗族布依族人群和北京人群虽然种族相同、地理位置相对较近,但二者HLA-C 基因rs3130542位点等位基因频率的分布还是存在一定的差异性,提示可能为民族差异所致;从地域角度讲,贵州苗族布依族人群和日本人群在遗传学分类上同属东亚人群,但由于中国和日本之间存在地理隔离,影响了基因的交汇,从而造成了地区间基因的差异[11];贵州苗族布依族人群与非洲人群比较等位基因频率差异明显,说明HLA-C 基因rs3130542位点存在种族和地域的差异性;而贵州苗族布依族人群与欧洲人群虽存在种族和地域的差异,但二者HLA-C 基因rs3130542位点的分布却很相似,其原因还需综合所有可能相关的因素进一步分析探究。
中国是一个多民族的国家,而贵州又是一个拥有17种世居少数民族的多民族聚居的省份,其中苗族、布依族为全省人口数最多的两个民族,了解贵州苗族、布依族的HLA-C基因多态性的分布情况可为接下来进一步研究HLA-C基因多态性与一些疾病的发生与进展的关联提供一定的理论依据,从而有助于深入了解某些疾病在不同种族及地区之间的遗传背景的差异,以达到了解、预防和治疗疾病的目的。
4参考文献
[1] Mak TW, Sauders ME. Primer to the immune response[M].2nded.London:Academic Press, 2011:11-15.
[2] Hu ZB, Liu Y, Zhai XJ, et al. New loci associated with chronic hepatitis B virus infection in Han Chinese[J]. Nature Genetics, 2013(45): 1499-1503.
[3] 何宁, 徐丽丹, 于景翠. 人类白细胞抗原C与HIV-1感染[J]. 实用医学杂志, 2014(9): 1498- 1499.
[4] 林泉,成先桂,钟永明,等.广西壮族人群HLA-C 基因多态性与神经纤维瘤病Ⅰ型相关性[J]. 现代中西医结合杂志, 2012(26): 2858-2859.
[5] 尹璐, 郭媛. 绒毛滋养细胞HLA-C基因多态性与早期复发性流产关系研究[J].白求恩军医学院学报, 2012(3): 192-193.
[6] 刘丽, 庄云龙, 乔文本. 山东地区汉族人群HLA-C基因分组与梅毒的相关性研究[J]. 中国输血杂志, 2013(5): 450-452.
[7] Blais ME, Dong T, Rowland-Jones S.HLA-C as a mediator of natural killer and T-cell Activeation:spectator or key player[J]. Lmmunology, 2011(1): 1-7.
[8] Guerini FR, Clerici M. NK cells in human disease:An evolving story[J]. Clin Immunol, 2012(3): 203-206.
[9] O'Connor GM, Guinan KJ, Cunningham RT, et al. Functional polymorptaism of the KIR3DL1/S1 receptor on human NK cells[J].J Immunol, 2007(1):235-241.
[10]Frazer Ka, Ballinger DG, Cox DR, et al. A second generation human haplotype map of over 3.1 million SNPs[J]. Nature, 2007(7164): 851-861.
[11]裴利国, 屈蕾, 贾飞, 等. 宁夏回、汉族群体LIN28B基因多态性[J]. 解剖学报, 2014(1): 128-133.
(2015-10-15收稿,2015-12-05修回)
中文编辑: 周凌; 英文编辑: 刘华
A Study ofHLA-C gene rs3130542 SNP Polymorphism in Buyi and Miao Ethnics Groups in Guizhou
ZHAO Xiaomei, HE Yan, ZHANG Wangde, ZHANG Ting, WANG Chanjuan, WU Changxue, GUAN Zhizhong
(TheKeyLaboratoryofMolecularBiology,GuizhouMedicalUniversity,Guiyang550004,Guizhou,China)
[Abstract]Objective: To investigate distribution characteristics of rs3130542 site of HLA-C gene Buyi and Miao ethnics and explore its distribution differences in different minorities. Method: TaqMan-MGB based on real-time PCR was used to determine the gene frequencies and genotype frequencies of HLA-C gene rs3130542 SNP polymorphism in Buyi ethnics(289 cases) and Miao ethnics(174 cases) of Guizhou. The gene frequencies and genotype frequencies of HLA-C gene rs3130542 SNP polymorphism were compared between the same sex people in different nationalities, between the same nationalities in different sexes and between Guizhou population and other populations of different regions and ethnic groups whose gene information was published by Human Genome Project. Results: There were significant differences of allele gene frequencies and genotype frequencies of HLA-C gene rs3130542 SNP polymorphism between Buyi nationality and Miao nationality (P<0.05). No statistically significant difference was found between male and female in the same ethnic group about HLA-C gene rs3130542 polymorphism distribution (P>0.05). There were statistically significant differences of genotype frequencies of HLA-C gene rs3130542 SNP polymorphism between the Buyi, Miao ethnic groups in Guizhou and the Beijing Chinese people (P<0.05). There were significant differences of allele frequencies and genotype frequencies of HLA-C gene rs3130542 SNP polymorphism between Buyi, Miao nationality and Japanese population (P<0.05) and there were significant differences of allele gene frequencies of HLA-C gene rs3130542 SNP polymorphism between Buyi, Miao nationality and African population (P<0.05). However, no significant difference was found in allele frequencies and genotype frequencies of HLA-C gene rs3130542 SNP between the Buyi , Miao ethnic groups and European population. Conclusion: The allele and genotypes frequencies of HLA-C gene rs3130542 SNP locus show significant differences among different ethnic groups, regional groups and racial groups. No significant difference is found between the male and female of the Buyi and Miao ethnic groups in Guizhou about HLA-C gene rs3130542 polymorphism distribution.
[Key words]HLA-C antigens; gene polymorphism; alleles; genotype; minority groups
[中图分类号]R342
[文献标识码]A
[文章编号]1000-2707(2016)01-0012-05
*[基金项目]国家科技支撑计划项目(2013BAI05B03); 贵州省“2011协同创新中心”[黔教合协同创新中心(2014)06号]
**通信作者 E-mail:annieheyan@gmc.edu.cn网络出版时间:2016-01-07
网络出版地址:http://www.cnki.net/kcms/detail/52.5012.R.20160107.2118.062.html

