大麦抗纹枯病种质资源的鉴定及遗传分析
楚单单,崔 君,金 越,戴欢欢,李 菲,胡梦娜,薛大伟,张晓勤
(杭州师范大学生命与环境科学学院,浙江 杭州 310036)
大麦抗纹枯病种质资源的鉴定及遗传分析
楚单单,崔君,金越,戴欢欢,李菲,胡梦娜,薛大伟,张晓勤
(杭州师范大学生命与环境科学学院,浙江 杭州 310036)
摘要:对31个来自不同地区的大麦品种进行了纹枯病抗性鉴定,共筛选出9份对纹枯病抗性较强和4份对纹枯病几乎无抗性的大麦.用21对SSR标记对这31种大麦进行遗传多样性分析,共扩增81个等位变异,每个位点上有2~5个变异,平均每个位点上的变异为3.857 1;位点多态性信息量(PIC)变幅为0.170 5~0.686 1,平均0.473 9.聚类分析可将这些供试材料分成2个大类和5个亚类.研究结果表明,大麦种质中抗纹枯病材料丰富且蕴含了丰富的遗传变异,可为大麦抗纹枯病的遗传改良提供重要的基因资源.
关键词:大麦;纹枯病;SSR;遗传多样性;聚类分析
大麦(HordeumvulgareL.)是禾本科作物,其种质资源由于长期的驯化栽培、自然进化和遗传定向改良,造成遗传基础比较单一.纹枯病是影响麦类作物产量的主要病害之一,能侵染27科以上的植物,包括许多重要的粮食作物如水稻、大麦、玉米等,因而被植物病理学家所广泛关注.纹枯病作为大麦上比较严重的真菌病害,对其进行研究有助于大麦的抗病性育种.大麦纹枯病一般从苗期至穗期均可发生,主要危害麦类的叶鞘,影响麦类作物的结实率和千粒重,严重时将会导致叶片早衰、植株倒伏造成大麦减产20%~30%[1-2].对大麦纹枯病的防治,主要采用栽培技术和药剂防治相结合的措施[3],但选育和推广抗病种质是防治大麦病害最安全、最行之有效的途径[4].同时,大麦纹枯菌致病力及大麦抗病性的研究对其它禾本科植物的相关研究具有重要的借鉴意义,也可为寻找新的抗性相关基因提供有效途径.遗传多样性研究是大麦抗病种质资源发掘、保护和利用的重要环节.SSR(simple sequence repeat)分子标记由于具有操作简单、重复性好、染色体位置已知等优点,在种质和遗传多样性分析中广泛运用,已成为遗传多样性研究中最常用的一种方法.
本研究利用纹枯菌株对收集自不同地区的31份大麦种质进行接种,通过比较不同种质间的病情指数,筛选具有一定抗性的大麦种质.同时,运用SSR分子标记技术对不同抗性的大麦种质进行遗传背景分析,明确不同抗性的大麦种质的遗传背景差异,为大麦纹枯病抗性育种提供理论参考.
1材料与方法
1.1 试验材料
供试的31份材料是来自不同地区的大麦种质(表1),引自浙江省农业科学院.鉴定所用的大麦纹枯病菌为立枯丝核菌,由浙江省农业科学院提供.研究中采用PDA培养基对纹枯菌进行扩大培养.
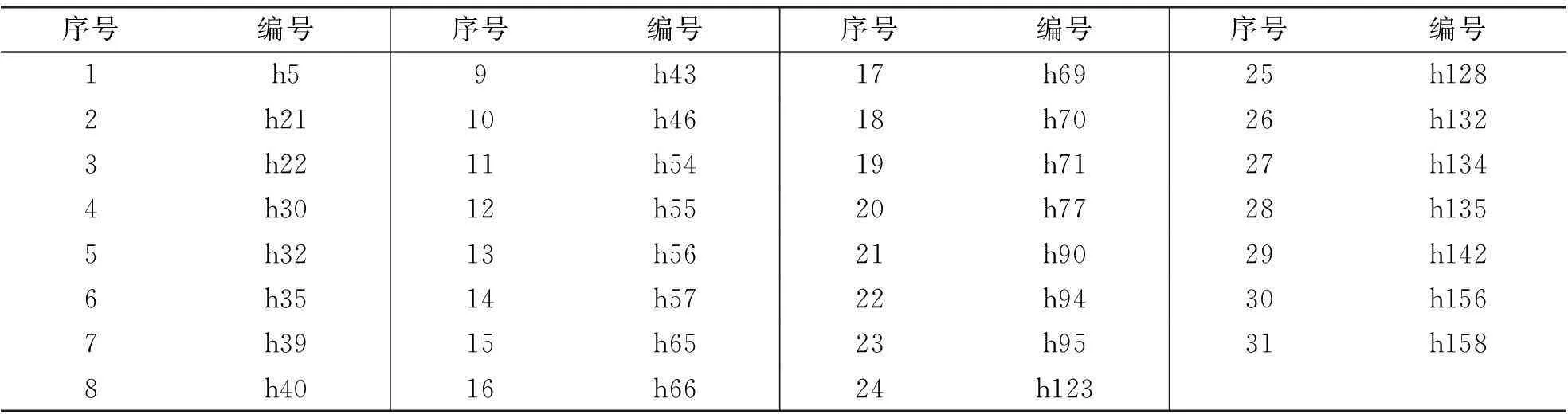
表1 供试材料
1.2 抗性鉴定方法
将PDA培养基上的病原菌菌丝体连同培养基切成小块一起置入元麦粒培养基中,相同条件下继续培养至菌丝长满元麦粒培养基,经过9次扩大培养获得足够的立枯丝核菌菌落.将待测材料播种于塑料育苗盘中,每种材料播种20粒,并设置3组重复.待其生长至四片真叶期,在大麦麦苗基部周围摆入长满菌丝的元麦粒,每组6粒左右,接种后用灭菌土覆盖,加水湿润后,放入人工智能培养箱内培养,定期喷施营养液.病菌在穿透寄主之前产生侵染垫、侵染圈以及单附着胞等侵染结构菌丝,直接侵染寄主表皮后,快速在受侵细胞内呈网状扩展,损害细胞壁,产生病斑[5].接种30 d后,大麦幼苗基部产生不同程度的侵染,统计病斑高度,进行抗病性鉴定.
1.3 SSR分析方法
1.3.1DNA提取各大麦种质幼苗叶片DNA提取参考Murray等[6]的方法.
1.3.2SSR引物的筛选从大麦的7条染色体中各选取5对相应的引物进行SSR分子标记实验,结果显示,每组染色体上有3对引物的标记结果可以用于分析.因此,每种材料采用21对SSR标记进行扩增.
1.3.3PCR扩增反应体系为10 μL,含2 μL引物,2 μL模板DNA和5 μL 2XMix、1 μL H2O.PCR的扩增反应程序为:94 ℃预变性5 min,94 ℃变性35 s,55 ℃退火30 s,72 ℃延伸40 s,共40个循环;72 ℃延伸10 min.
1.3.4琼脂糖凝胶电泳PCR扩增产物用3%的琼脂糖凝胶电泳的方法进行检测,凝胶成像仪进行拍照分析记录.
1.3.5数据处理使用SPSS20和Excel统计31个大麦种质的病斑高度数据,计算其平均数和标准差,并进行感病程度分析.采用Anderson等[7]的方法计算等位位点的多态性信息量(PIC,ploymorphism information content);采用Nei等[8]的方法计算相似系数(GS):GS=2Nij/(Ni+Nj),其中Ni和Nj分别为i,j两材料总基因数,Nij为i和j两种材料同有的等位基因数,遗传差异(GD)=1-GS.使用DPS5.02软件计算欧氏距离,按照UPGMA(Unweighted Pair Group Method using arithmetic Averages)方法对31份供试材料进行聚类分析.
2结果
2.1 大麦苗期纹枯病的抗性鉴定
SSR标记技术是一种以特异引物PCR为基础的分子标记技术,是由1~5个核苷酸为重复单位簇集而成的串联重复序列,可分布在整个基因组的不同位置上,而且在基因组中的分布是随机的[5].
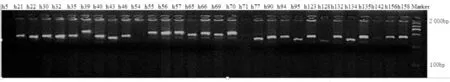
M:Marker 1~31,表1中的31份材料.图1 HVP93在供试材料的扩增结果Fig. 1 The results of PCR amplification of HVP93 in test materials
本研究所选取的35对SSR标记中,有21对在31个大麦品种中扩增出重复性好、多态性高的条带(图1),占总引物的60%,其他引物在供试大麦中无扩增产物.利用这21对标记,对31份大麦种质进行多态性研究,共检测到81个等位变异,单个位点上有2~5个变异,平均每个位点上的变异为3.875 1.多态性最高的为HVP80、HVP172等5个标记,有5个变异位点,HVP332、HVP3262标记多态性最低,只有2个变异位点.位点多态性信息量(PIC)变幅为0.170 5~0.686 1,平均为0.473 9(表2).
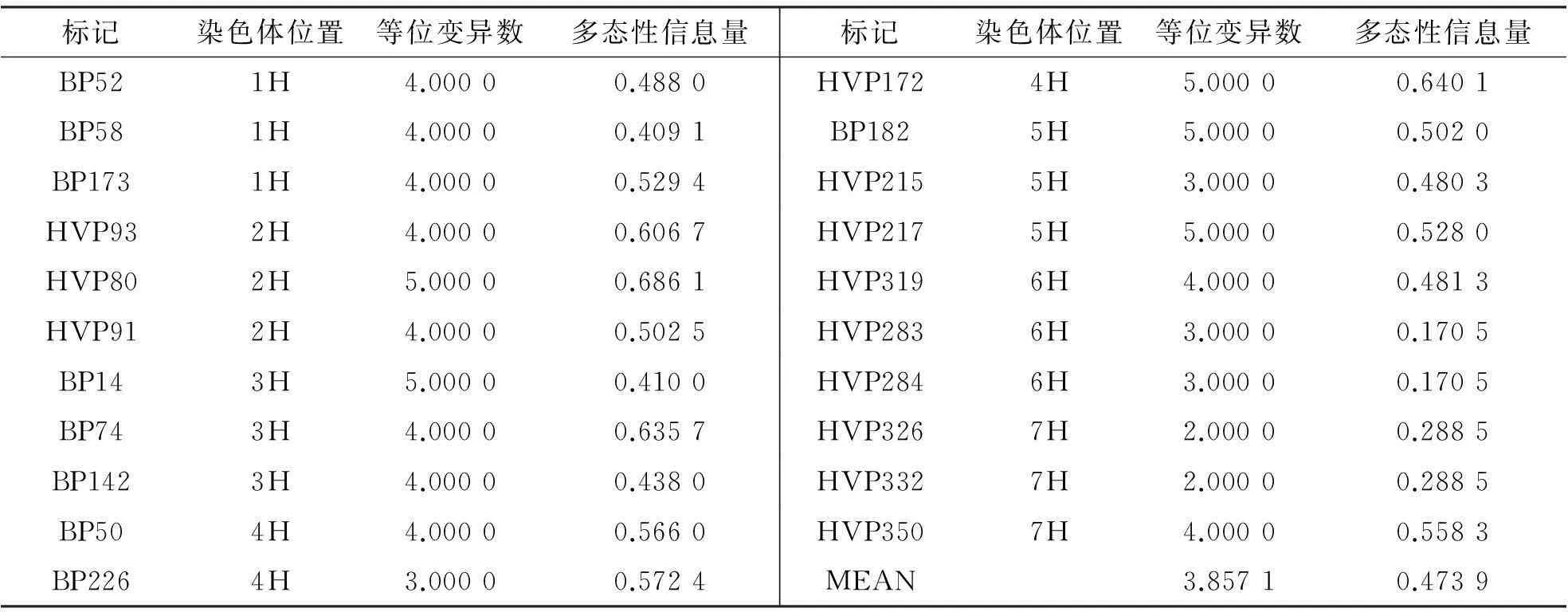
表2 SSR标记名称、所在染色体、检测到的等位基因数目及多态信息量
植株发病后,根据病斑高度、发病部位、病斑大小以及是否侵入叶鞘和茎秆,对感病情况进行分级.目前国内未有统一的分级标准[9].本研究对31个大麦品种的幼苗进行纹枯病抗性鉴定,并且在Ruhs的病情评价标准[10]基础上,结合大麦纹枯病病情特征,对病情等级间的差异进行划分,提出了一种改进的大麦苗期病情划分标准(表3).结果表明不同大麦种质对纹枯病的抗性存在显著差异,其中有9个品种具有较高抗性,占供试材料的29%;有4个品种无抗性,占供试材料的13%,其余18个大麦品种(58%)表现出不同程度的抗性(表4).

表3 改进的大麦苗期纹枯病病情调查标准
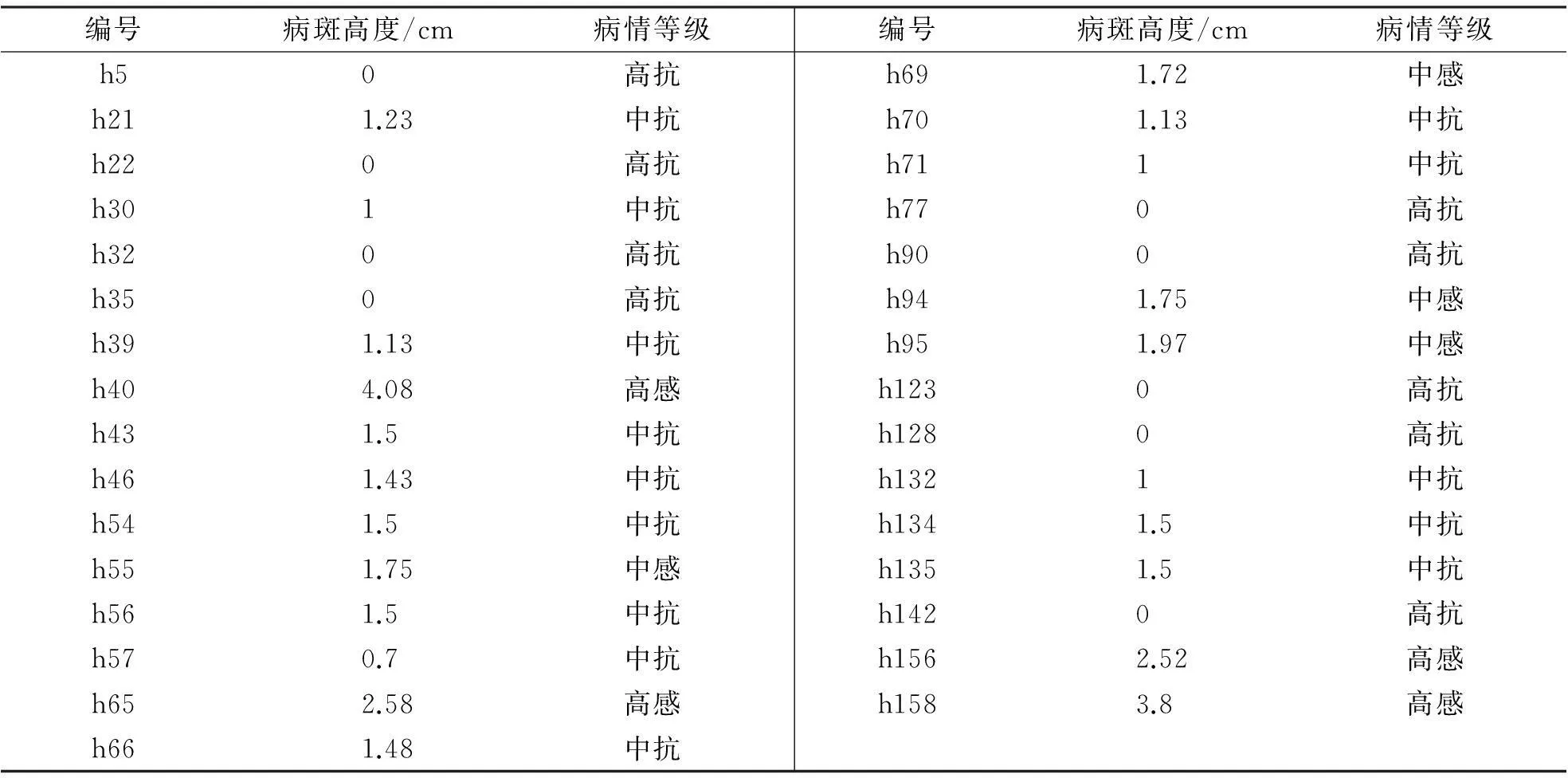
表4 31份材料的病情等级
2.2 聚类分析及遗传距离分析
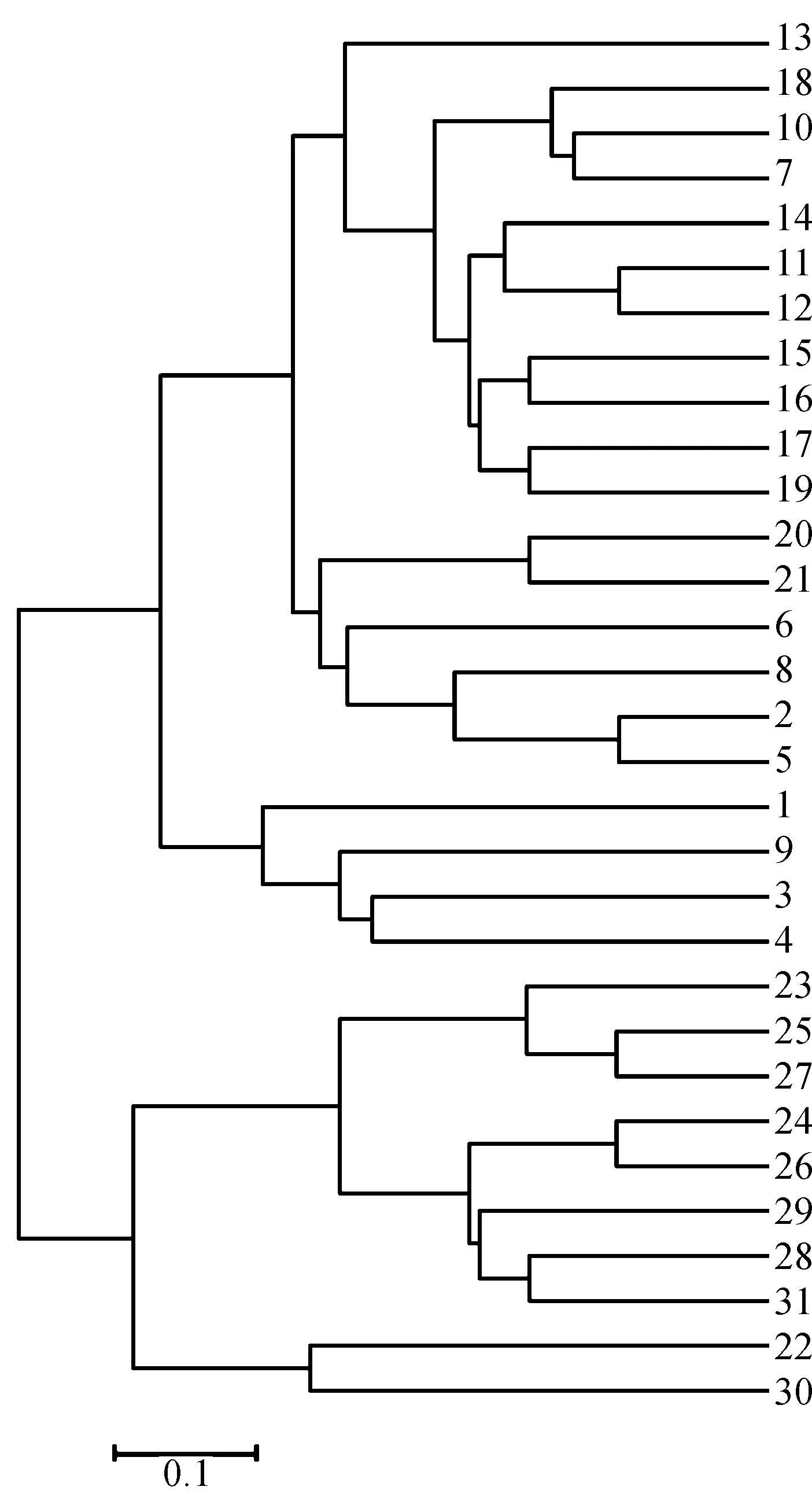
图2 31种大麦材料UPGMA聚类图Fig. 2 UPGMA dendrogram of 31 barley varieties
为分析31份大麦材料间的遗传背景,利用21对SSR标记等位变异计算不同材料间的遗传距离.31份大麦材料的平均遗传距离为0.810 9.利用SSR遗传距离矩阵按照UPGMA进行聚类分析,构建各个供试材料的亲缘关系图,结果表明,在遗传图距为0.348的水平上31份材料被聚分为2大类、5个亚类群.如图2所示,13、18号到4号21种大麦为第一大类群,余下10种大麦为第二大类群,其中第一大类群被聚分为3个亚类,第二大类群被聚分为2个亚类.
3讨论
近年来,关于纹枯病抗性遗传分析的研究逐渐增多,并鉴定出一些纹枯病抗性材料[11].不同种质对纹枯病的感病程度存在明显差异,在鉴定中也发现许多感病较轻材料.但是在中国各麦区生产上大力推广的品种、高代新品系等种质对纹枯病的抗性普遍较差,这也正是近年来麦类作物纹枯病大面积发生的原因[9].前人的研究表明,大麦有多种感染纹枯病的途径,通过合理的种植和管理,也能降低大麦纹枯病的发生频率.如,前茬为水稻的大麦田,纹枯病病株率约为43.1%,而前茬为棉花的大麦田,病株率约为18.4%.另外,早播与氮肥过量也将增加大麦纹枯病的发病率[12].而对大麦纹枯病的防治,目前主要采用栽培技术和药剂防治相结合的综合防治方法[3],常见的防治方法还有推广抗病种质、农业防治及化学防治等[13].本研究对31份大麦材料进行了SSR分子标记分析,在选取定位在大麦7条染色体的标记中得到21个重复性较好、多态性较高的标记.其中,HVP93等存在重复性较好的扩增结果,并且存在多态性.聚类分析表明31份材料可以被聚分为2大类,5小类.13、18、4号等21种大麦为第一大类群,余下10种大麦为第二大类群,其中第一大类群被聚分为3个亚类,第二大类群被聚分为2个亚类.通过对31份大麦材料的研究,发现与其他种质相比,h5、h22、h32等9个品种对纹枯病的抗性较好,占供试材料的29%,有4个品种无抗纹枯病性,占供试材料的13%.当前中国大麦品种抗纹枯病的抗原单一、抗病性差,本研究鉴定出的大麦纹枯病抗性品种可作为纹枯病抗性基因研究的遗传材料.
现代育种模式导致作物种质间遗传多样性降低、相似系数增大,因此丰富作物育种材料是当前农作物育种的主要任务之一.本研究的遗传多样性分析和纹枯病性的鉴定进一步证明了h5等大麦种质携带丰富抗性基因,未来这些筛选出的大麦种质资源将在大麦抗纹枯病育种中发挥重要作用.
参考文献:
[1] 陈光喜.水稻纹枯病的发生及综合防治技术[J].四川农业与农机,2013(6):40.
[2] 居为民,高苹,武金岗.气候条件对麦类纹枯病发生趋势影响的研究[J].植物保护,2000,26(2):20-22.
[3] 赵桂东,刘荆,李清铣.大麦种质资源抗纹枯病性鉴定及纹枯病菌致病性的研究[J].大麦与谷类科学,1993(4):20.
[4] 张增艳,姚乌兰,李宏涛,等.大麦和小麦抗病性的分子基础研究进展[J].麦类作物学报,2005,25(6):139-143.
[5] 张小村.小麦抗纹枯病基因的遗传和分子标记分析[D].泰安:山东农业大学,2004.
[6] MURRAY M G, THOMPSON W F. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Research,1980,8(19):4321-4325.
[7] ANDERSON J A, CHURCHILL G A, AUTRIQUE J E, et al. Optimizing parental selection for genetic linkage maps[J]. Genome,1992,36(1):181-186.
[8] NEI M, LI W H. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proceedings of the National Academy of Science,1979,76(10):5269-5273.
[9] 陈江.小麦抗纹枯病QTL分析[D].北京:中国农业大学,2014.
[10] WASANO K, ORO S, KIDO Y. The syringe inoculation method for selection rice plants resistant to sheath blightRhizoctoniasolani[J]. Jpn J Trop Agric,1953,27:131-134.
[11] HOLLINS T W, SCOTT P R. Differences between wheat cultivars in resistence to sharp eyespot cansed byRhizoctoniacerealis[J]. Tests Agrochem Cult,1985,6:166-167.
[12] 赵桂东,刘荆.江苏省大麦纹枯病研究进展[J].大麦科学,1994(2):32-33.
[13] 崔苗青,赵金枝,王树杰.大麦纹枯病的发生与防治[J].中国农村小康科技,2005(1):33.

第15卷第1期2016年1月杭州师范大学学报(自然科学版)JournalofHangzhouNormalUniversity(NaturalScienceEdition)Vol.15No.1Jan.2016
Identification and Genetic Analysis of Barley Germplasm against Sheath Blight
CHU Dandan, CUI Jun, JIN Yue, DAI Huanhuan, LI Fei, HU Mengna, XUE Dawei, ZHANG Xiaoqin
(College of Life and Environmental Sciences, Hangzhou Normal University, Hangzhou 310036, China)
Abstract:In order to identify sheath blight resistance of barley germplasm, 31 barley varieties from different areas were used in this study, and 9 varieties with strong sheath blight resistance and 4 varieties with no sheath blight resistance were chosen. 21 pairs of SSR (single sequence repeat) markers were used to characterize the genetic diversity of 31 varieties, and 81 alleles were amplified, with a range of 2~5 alleles per locus, and there was an average of 3.857 4 alleles per locus. Loci polymorphism information content (PIC) ranged from 0.170 5 to 0.686 1 with an average of 0.473 9. By using cluster analysis method, the 31 barley accessions could be classified into 2 groups and 5 sub-groups. The research showed that the materials in barley germplasm against sheath blight were rich, and had abundant genetic diversity, which could provide important gene resource for the genetic improvement of sheath blight resistance of barley germplasm.
Key words:barley; sheath blight; SSR; genetic diversity; cluster analysis
文章编号:1674-232X(2016)01-0025-05
中图分类号:S5
文献标志码:A
doi:10.3969/j.issn.1674-232X.2016.01.005
通信作者:张晓勤(1976—),女,副教授,博士,主要从事作物学研究.E-mail:xiaoqinzhang@163.com
基金项目:浙江省自然科学基金项目(LY14C130007);杭州市科委基金项目(20140432B03);浙江省大学生科技创新活动暨新苗人才计划(2014R421029);杭州师范大学教改项目.
收稿日期:2015-06-03

