大鼠肝脏半乳糖凝聚素-3 cDNA分子多样性分析
大鼠肝脏半乳糖凝聚素-3 cDNA分子多样性分析
方恩浩1,2,张珊珊3,刘倩芸3,周建钟4,杨仙玉1*
(1.浙江农林大学动物科技学院,浙江 临安 311300;
2. 浙江农林大学集贤学院,浙江 临安 311300;
3. 浙江农林大学林业与生物技术学院,浙江 临安 311300;
4. 浙江省林业生物质化学利用重点实验室,浙江 临安 311300)
摘要:半乳糖凝聚素-3(Galectin-3,Gal-3)是一种多功能蛋白,参与机体内的多种生命活动过程。为分析大鼠Rattus norvegicus gal-3 cDNA分子多样性,在分析GenBank注册的大鼠gal-3 cDNA 序列的同时,从三只不同大鼠个体的肝脏第一链cDNA,共计9个独立实验中克隆gal-3 开放阅读框OFR (Open reading frame)。分析结果表明,在已注册的序列中存在1个单核苷酸多态性 (Single nucleotide polymorphism,SNP)。本实验从1-3号大鼠肝脏分别获得118、95和120个克隆,新发现1处缺失和5处SNP,共克隆17个不同的 gal-3 ORF,其中仅1个克隆与已报告大鼠gal-3 cDNA 的ORF完全相同。从1号和3号个体肝脏中分别克隆11和14个不同的gal-3 ORF,揭示大鼠gal-3 cDNA存在个体水平的分子多样性。
关键词:大鼠;半乳糖凝聚素-3;cDNA多样性;单核苷酸多态性
中图分类号:S443.5;Q533+.3文献标志码:A
收稿日期:2015-01-22;修回日期:2015-04-15.
作者简介:宋东光,男,副教授,研究方向:生物信息学与植物分子育种;E-mail:3dsong@163.com.
doi:10.3969/j.issn.1672-5565.2015.02.04
Livergalectin-3 cDNA diversity inRattusnorvegicus
FANG Enhao1,2, ZHANG Shanshan3,LIU Qianyun3,ZHOU Jianzhong4,YANG Xianyu1*
(1.CollegeofAnimalScienceandTechnology,ZhejiangAgriculturalandForestryUniversity,Lin′an311300,China;
2.JixianHonorsCollege,ZhejiangAgriculturalandForestryUniversity,Lin′an311300,China;
3.SchoolofForestryandBiotechnology,ZhejiangAgriculturalandForestryUniversity,Lin′an311300,China;
4.ZhejiangProvincialKeyLaboratoryofChemicalUtilizationofForestryBiomass, 88HuanchengBeilu,Lin′an311300,China)
Abstract:Galectin-3 (Gal-3) is a multi-functional protein involved in many biological processes. To clarify the gal-3 cDNA diversity of Rattus norvegicus, cloning of gal-3 open reading frame (ORF) from liver first strand cDNA of three individuals was performed totally from 9 independent experiments. Consequently, 118, 95 and 120 clones were obtained from three individuals, respectively. Alignment of all these sequences indicated 1 deletion and 5 single nucleotide polymorphism (SNP) sites, and 17 different clones were obtained. Nine out of the 17 clones appeared in two or more independent experiments, and 11 and 14 different clones were obtained from the first and the third individuals, respectively. These results indicated the objectivity of liver gal-3 cDNA diversity at individual level in R. norvegicus.
Keywords:Rattus norvegicus; Galectin-3; cDNA diversity; SNP
近年来,高通量DNA测序技术的发展,基因组和转录组的数据在不断的积累,学者们逐渐发现转录子的多样性以及转录产物 (RNA)与基因组DNA不相匹配的现象,而且这种多样性出现于个体水平[1-2]。半乳糖凝集素(Galectins, Gal)作为动物类凝聚素,通过其高度保守的糖识别结构域 (Carbohydrate recognition domain, CRD)特异性识别并结合β-半乳糖苷,从而发挥其生物学功能[3]。在哺乳类,已发现15个家族成员,分为原型、串联重复型和嵌合型三类[3]。半乳糖凝聚素-3(Gal-3) 是该家族唯一嵌合型代表,具有多种生物学功能,参与细胞增殖与分化、血管发生及肿瘤发生与转移等多种生命过程,在正常细胞和肿瘤细胞中均有表达[4-7]。Gal-3由单拷贝基因LGALS3 编码,包含5 个内含子和6 个外显子,约由250 个氨基酸残基组成,相对分子质量约31 kD,由3个结构域组成[8]。(1) N端结构域(N-terminal domain):由12个氨基酸残基组成,Ser6磷酸化位点具有细胞靶向调节作用;(2) 串联重复序列:由富含 Gly、Tyr和 Pro的串联重复序列的类胶原结构组成(Repetitive collagen-like sequence);(3) CRD:由一个糖识别结构域组成,包含凋亡基因Bcl-2家族同源序列BH1中的保守基序NWGR(Asp-Trp-Gly-Arg)[8]。在前期研究工作中,作者发现中华大蟾蜍Bufogargarizans皮肤第一链cDNA中gal-3开放阅读框(Open reading frame,ORF)存在个体水平的分子多样性[9-11]。鉴于Gal-3是一种多功能蛋白,分析哺乳动物gal-3 cDNA分子个体水平的多样性具有重要的生物学意义。本文选择哺乳类模式动物大鼠Rattusnorvegicus为研究材料,对其肝脏gal-3 ORF进行克隆,并对其多样性进行分析和讨论。
1材料与方法
1.1试验动物和主要试剂
SD大鼠 (Rattusnorvegicus) 购自浙江省实验动物中心,取其肝脏剪成小块投入液氮中快速冷冻,再转入-70 ℃超低温冰箱保存备用。
RNA提取试剂盒购自上海博彩生物技术公司,Quantscript RT kit、pGM-T载体、大肠杆菌E.coli感受态细胞DH5α购自北京天根生化科技有限公司,DNA聚合酶链式反应PCR (Polymerase chain reaction) 试剂盒购自TaKaRa,引物合成与DNA测序委托上海桑尼生物科技有限公司。
1.2引物设计
为克隆大鼠gal-3 ORF,参考GenBank已注册的大鼠gal-3 cDNA序列 (Accession number: FQ220174, BC089054, FQ210101, FQ214799, FQ219929, FQ219987, FQ220157, FQ220215, FQ220224, FQ220313, FQ220348, FQ220361, FQ220369),在ORF上、下游分别设计引物P1 (5′-AGGAGGAGCACTAACCAGGAAA-3′)和P2 (5′-TTTCCCGCTCATAACACACAG-3′)。
1.3大鼠肝脏第一链cDNA的合成及gal-3 ORF克隆
首先按试剂盒使用说明,纯化大鼠肝脏总RNA,通过琼脂糖电泳和分光光度计检测其浓度和完整性后,合成其第一链cDNA。然后以大鼠肝脏第一链cDNA为模板,以1.2中设计的 P1与P2为引物,按照试剂盒说明实施PCR。经1%琼脂糖凝胶电泳检测后,将PCR产物与T载体进行连接、转化。然后通过蓝白斑筛选和菌落PCR筛选阳性菌落及回收其重组质粒,并委托公司进行测序。为排除在反转录、PCR或DNA测序等实验过程中可能人为导入的碱基变化,本实验对每一个体的肝脏第一链cDNA均实施3组独立的PCR及克隆实验,将每个实验编号为x-y(x表示大鼠个体编号1-3,y表示实验组号)。
1.4缺失及SNP分析
将重复出现于两个或更多独立实验中的碱基变化(包括单核苷酸多态性SNP (Single nucleotide polymorphism,SNP)和缺失)确定为客观存在的;反之,为实验过程中导入的。
1.5同源性分析
利用EditSeq寻找已测序样品的ORF,利用NCBI Blast功能在GenBank中查找下载其他物种同源蛋白的氨基酸序列(http: //blast.ncbi.nlm.nih.gov/Blast. cgi),利用ClustalX 2.0 进行同源性分析。
2结果
2.1大鼠gal-3 ORF克隆及其多样性分析
从1号个体三个独立实验中分别克隆31、36和51个gal-3 ORF克隆 (表1),即从1号个体共计克隆118个gal-3 ORF。从2号和3号个体分别获得95和120个gal-3 ORF克隆 (表1)。将所有序列进行比对分析,发现1处缺失 (△301-321 bp) 和5处SNP 18(A-G)、43(T-C)、356 (T-A)、441 (A-G) 和488(A-G) (表1)。与大鼠结构基因对比分析发现,SNP 18(A-G)发生于第2外显子3′末端,SNP 48(T-C) 和356(T-A) 以及△301-321 bp 位于第3外显子内部,SNP 441 (A-G) 和488(A-G)分别位于第4、第5外显子内部 (见图1)。进一步分析发现,本文共计克隆17个不同的大鼠gal-3 ORF (见表2,CL1-17)。其中9个克隆 (CL1, 2, 3, 7, 8, 9, 12, 16, 17) 重复出现于2个或更多独立的实验中,CL1出现于所有独立实验(见表3),并与GenBank已注册大鼠gal-3 cDNA (Accession number: FQ220174)的ORF完全相同 (见图2),CL16在1号和3号个体的所有实验中均有出现(见表3)。从1号和3号个体分别获得11和14个不同的克隆,2号个体中由于没有发现缺失和SNP,因此仅有1个克隆(见表3)。尽管个体间存在一定的差异,但是这些实验结果仍然揭示大鼠肝脏gal-3 cDNA个体水平的分子多样性。
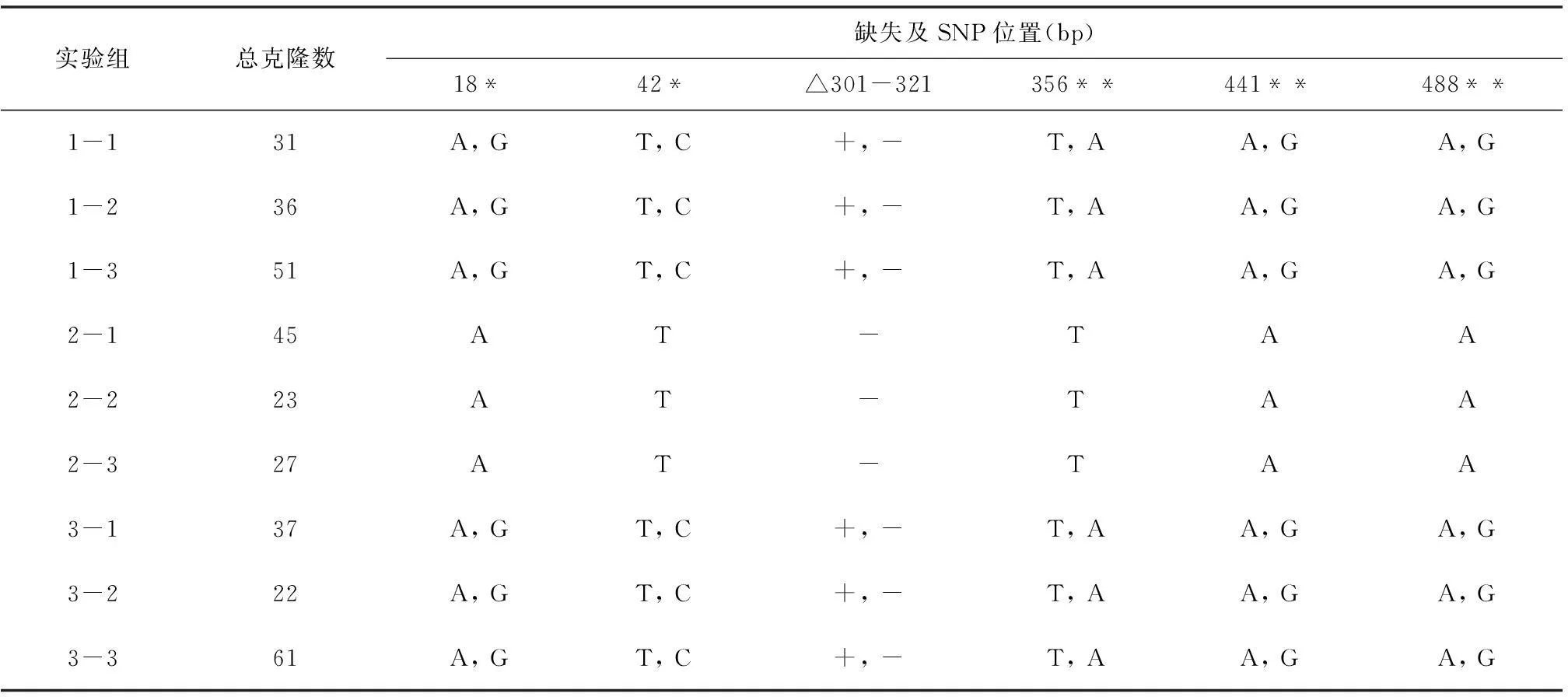
表1 大鼠 gal-3ORF的缺失及单核苷酸多态性
注:*: 示该SNP 导致同义突变; **: 示该SNP 导致错义突变; bp: 碱基对; A, T, C, G: 代表4种脱氧核糖核苷酸; △: 缺失; -, +: 分别代表缺失的有无。
Notes:*:Show the SNP resulting in synonymous mutation; **: Missence mutation,respectively;bp:Base pair; A,T,C,G:Represent 4 kinds of deoxynucleotides; △: Deletion;-,+:Indicate no deletion or the existence of deletion,respectively.
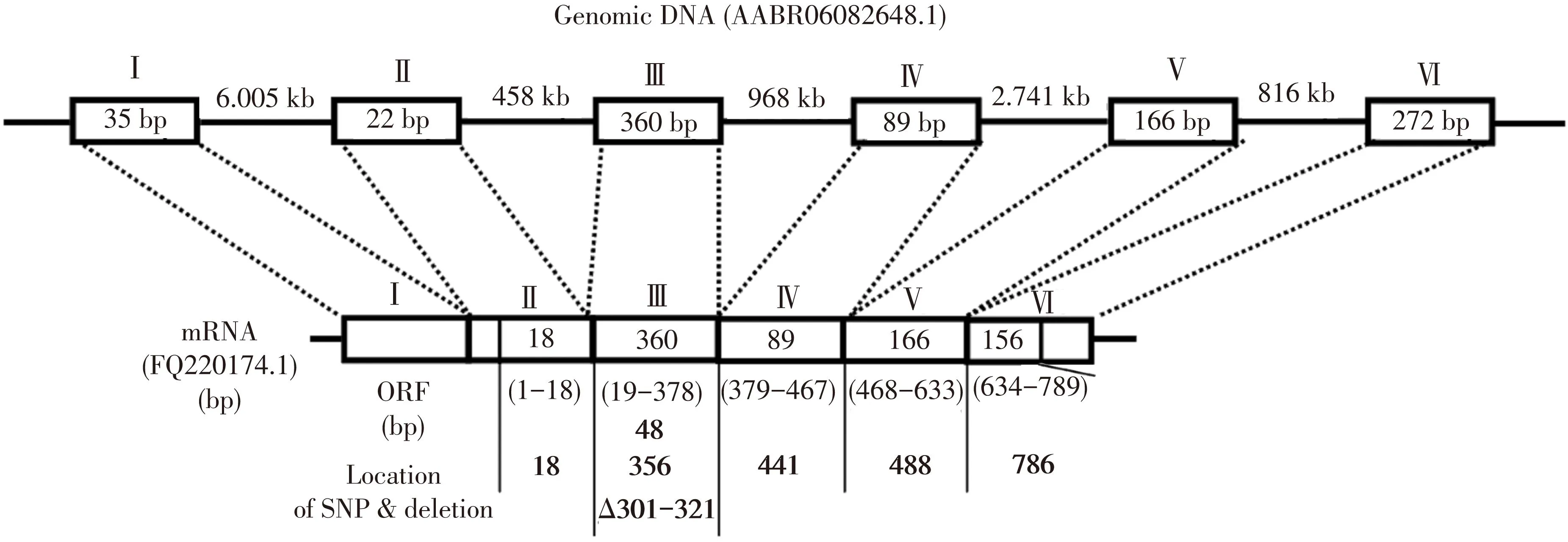
图1 大鼠LGALS3基因及肝脏SNP在ORF中的位置及两者的对应关系
注:长方形: 外显子; 线段: 内含子; 罗马数字: 外显子序号; bp, kb: 碱基对, 千碱基对;△: 缺失;正常数字:外显子和内含子的长度;加粗数字:示缺失或SNP的位置。
Notes: Rectangle: Exon; Line: Intron; Roman numerals: Serial number of exon;bp,kb:Base pair and kilobase pair. △: Deletion; Number in regular: Length of exon and intron; Bold number: Deletion or the location of SNP.
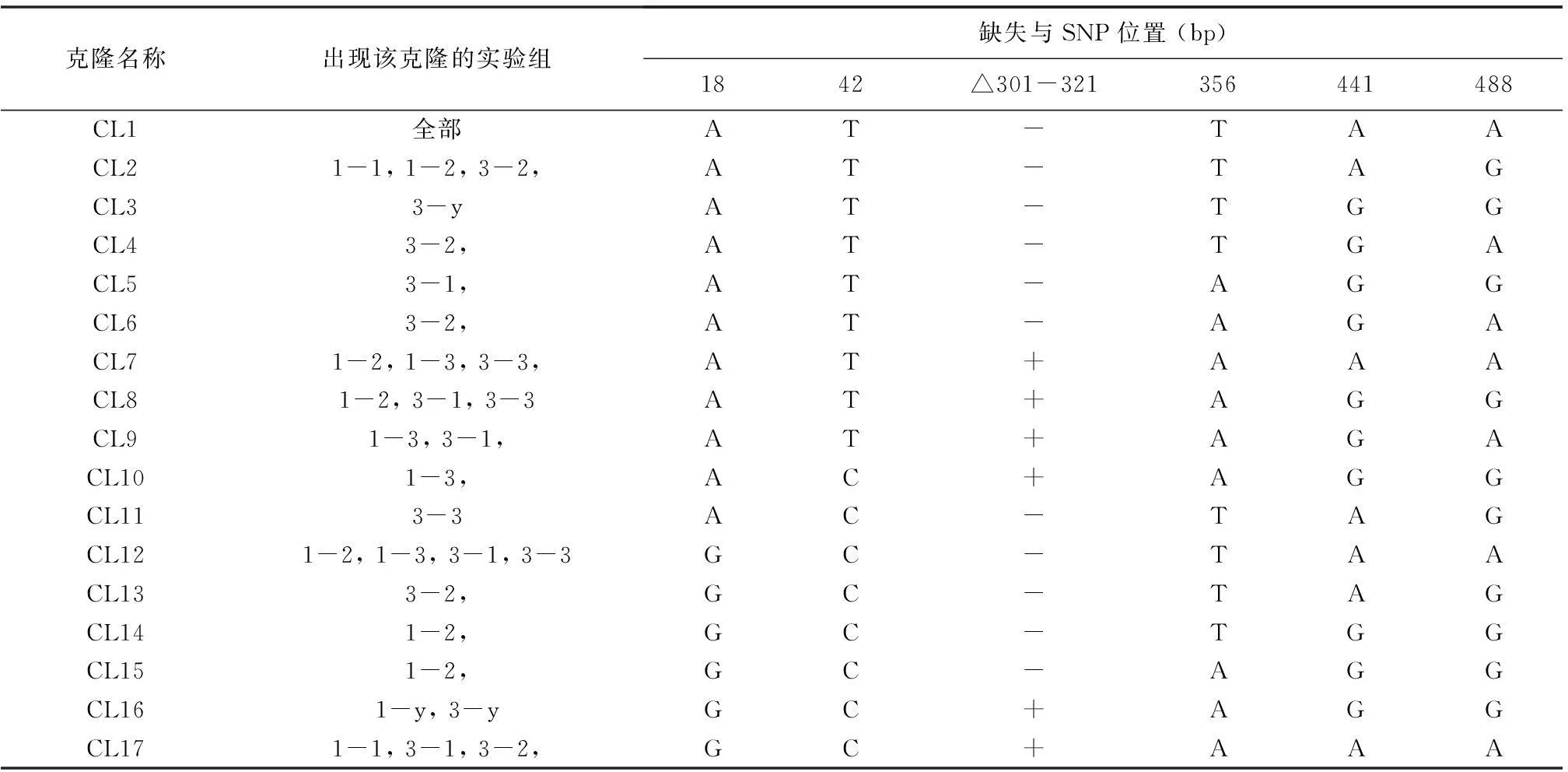
表2 大鼠不同gal-3ORF克隆在不同实验中的出现情况
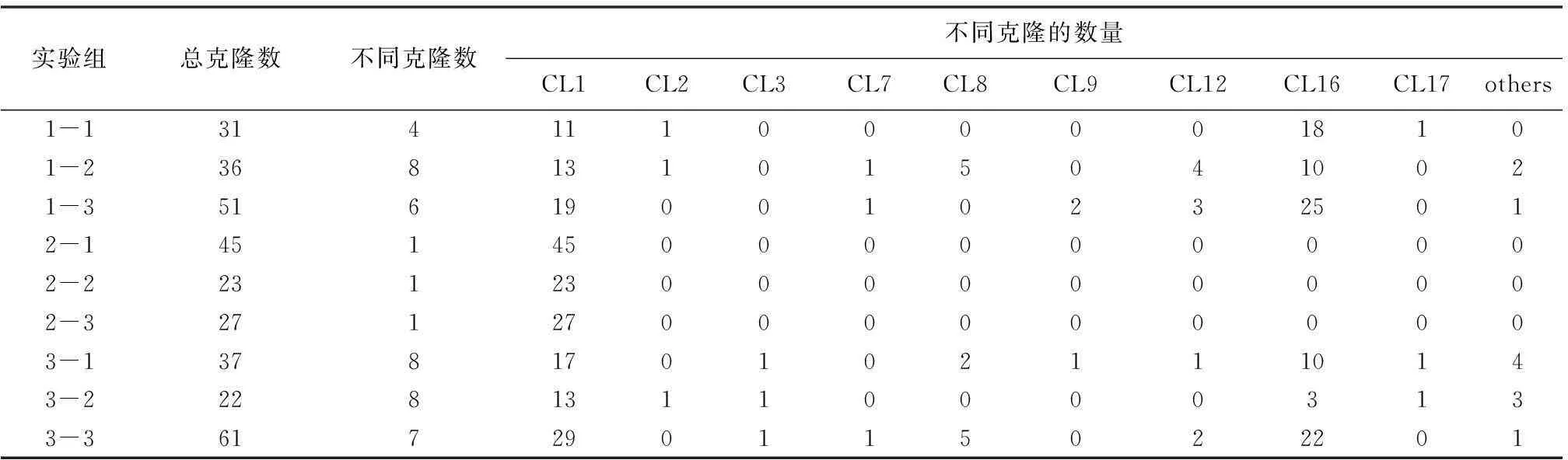
表3 不同克隆在不同实验组中的出现频次
2.2大鼠GenBank注册gal-3 ORF分析
为简化数据分析的复杂性,作者从GenBank注册的大鼠gal-3 cDNA序列中,首先选择了无插入和缺失的具有完整ORF(789 bp)的所有序列,共计13个(见材料与方法1.2)。其次,在序列分析过程中,针对其ORF 进行了比较。分析结果发现1个SNP:786 (C-T),与本实验中发现的5个SNP均不同,位于第6外显子内部(见图1),将13个cDNA分为两组。第一组包括5个克隆,其ORF与本文报告的CL1完全相同,第二组包括8个克隆,在本实验中尚未发现。
2.3大鼠Gal-3 氨基酸变化位点分析
上述18个不同的gal-3 ORF (2个源自GenBank,16个为本文新发现) 实际编码9个不同多肽链,表明cDNA分子多样性导致其编码蛋白的多样性。多肽链之间产生差异的原因有两个:一是21 bp的缺失使17个不同的多肽链分为两组,即789 bp的长型ORF (long type,L type) 和768 bp的短型ORF (short type,S type),分别编码由262和255个氨基酸残基组成的大鼠Gal-3;二是6处SNP (1个源自GenBank,5个源自本实验)中,18(A-G)、43(T-C) 和786 (C-T)三处SNP为同义突变,356(T-A)、 441 (A-G) 和488(A-G) 三处为错义突变(见表1),导致其对应编码氨基酸的改变(见图3)。结果CL1和12编码相同的肽链,CL2、11和13,CL3和14,CL5和15,CL8、10和16,CL7和17分别编码相同肽链,CL4、6和9各自编码不同的肽链。但是,这些氨基酸的变化并没有发生在磷酸化位点、糖基结合位点以及与Gal-3生物学功能关系密切的两个保守基序上(见图3),暗示氨基酸的替换或减少并没有改变Gal-3与β-半乳糖苷结合的生物学特征。

图2 大鼠gal-3 ORF多序列比对
注:·: 与第一行的相应碱基相同; 实线矩形:碱基缺失; 虚线矩形: SNP。
Notes: ·: The same as that in the reference sequence;Line enclosed rectandle:Deletion;Dot line enclosed rectangle:SNP.
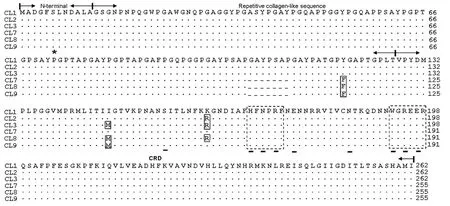
图3 大鼠几种Gal-3变异体的氨基酸序列比对分析
注: ·: 与第一行的氨基酸相同; - : 缺少相对应的氨基酸;_:β-半乳糖苷结合位点;* : 磷酸化位点; 实线矩形: SNP导致对应氨基酸的改变; 虚线矩形: 识别β-半乳糖苷的重要基序。
Notes:·: The same as that in the reference sequence; -:Absence of the corresponding amino acid; _:β-galactoside binding site; * : Phosphorelation site; Line enclosed rectangle:Amino acid subsititution due to SNP;Dot line enclosed rectangle:Important motif for β-galactoside binding.
2.4大鼠Gal-3 氨基酸序列同源性分析
为分析大鼠Gal-3 氨基酸序列同源性,选择CL1和CL8的编码蛋白分别作为L type和S type代表,与其他动物Gal-3进行了同源性分析(见图4)。L type 与五种哺乳动物小鼠Musmusculus(EDL20744)、猪Susscrofa(NP_001090970.1)、人Homosapiens(AAA88086)、苏门答腊星星Pongoabelii(XP_002824813)和兔Oryctolaguscuniculus(NP_001075807.1)的同源性分别为87%、81%、80%、80%和74%。与家鸡Gallusgallus(ABO25859) 、家鸽Columbalivia(EMC80882)、鸭Anasplatyrhynchos(XP_005029449), 热带爪蟾Xenopus(Silurana)tropicalis(NP_988986)以及斑马鱼Daniorerio(AAR84191)的同源性介于46%~64%。S type比L type的同源性略高,如与小鼠的同源性为88%,与猪的同源性为84%等。
注: - : 缺少相对应的氨基酸; _:β-半乳糖苷结合位点;* : 磷酸化位点; 实线矩形: 因△301-321 导致S type Gal-3缺少的7个氨基酸在其它动物的Gal-3中亦不存在; 虚线矩形: 识别半乳糖苷的重要基序。
Notes:-: Absence of the corresponding amino acid;_:β-galactoside binding site; * : Phosphorelation site; Line enclosed rectangle:Seven amino acid missing due to △301-321; Dot line enclosed rectangle:Important motif for β-galactoside binding.
3讨论
本文从3只大鼠肝脏第一链cDNA,9个独立的克隆实验中共计得到333个gal-3 ORF克隆(见表1),除去重复出现的克隆后,发现17个不同的大鼠gal-3 ORF(见表2)。在1号和3号个体中分别出现11和14个不同的克隆(见表2),表明大鼠中确实存在个体水平的gal-3 ORF分子多样性。
那么这种多样性是客观存在还是人为因素导致的呢?如果是客观存在,又如何解释该现象呢?
在进行cDNA克隆的实验中,如在反转录、PCR以及DNA测序过程中均有可能人为导入一些碱基的变化。为了排除这些因素的干扰,本实验使用了3只不同的大鼠个体的肝脏第一链cDNA,针对每一个体实施3组独立的PCR及其克隆。在分析数据的过程中,严格确认重复出现于2个或更多独立实验中的碱基变化,最后确定本次实验新发现1处缺失和5处SNP。因此,尽管有些克隆仅出现一次,基于这些缺失和SNP而克隆的17个不同的大鼠gal-3 ORF应该是客观存在的。另外,S type ORF缺失21 bp,其编码蛋白比L type ORF编码蛋白少7个氨基酸残基,同源性比对分析揭示其它动物的Gal-3中不存在与此对应的7个氨基酸(见图4中矩形框),支持大鼠S type ORF的客观存在。
对于cDNA多样性的原因至少需要分析以下几点。(1)物种的染色体倍数;(2)多拷贝基因;(3)RNA选择性剪切(RNA alternative splicing);(4)RNA编辑(RNA editing);(5)人为因素。目前,关于大鼠为二倍体以及Gal-3编码基因LAGAL3为单拷贝基因不存在任何争议。本文对新发现的1处缺失和5处SNP进行的结构基因上的定位分析(见图1)难以得出大鼠gal-3 ORF多样性与RNA选择性剪切有关。那么会不会与RNA编辑有关呢?
RNA编辑是mRNA前体的一种加工方式,通过脱氨基作用使腺嘌呤转变为次黄嘌呤(A-I)或者使胞嘧啶转变为尿嘧啶(C-U),导致mRNA与染色体上的编码基因不匹配[2,12]。上述的6处SNP中,除了356(A-T)为颠换以外,其他5处SNP均为转换,表明这5处的SNP很可能由RNA编辑造成。实际上,在α-银环蛇毒素(α-bungarotoxin,α-BGT)的研究中,个体水平上的cDNA多样性早有报道,可是对其多样性产生的原因仍存在争议,有学者认为与RNA编辑有关,有学者则完全否定RNA编辑[13-15]。但是大鼠肝脏gal-3 ORF中的356(A-T)以及21 bp (301-321)的缺失目前尚无理论可以对其进行合理的解释。目前采用基因组和转录组测序结果进行比对分析的报道越来越多,发现大量的RNA序列与基因组DNA不匹配,被称为RDD (RNA-DNA differences),难以用现有的理论包括RNA编辑的机制进行解释,因此有学者认为生物体内很可能存在未知的尚未发现的调控RNA多样性的机制[1]。大鼠的gal-3 cDNA的多样性产生的机制可能包括RNA编辑及尚未发现的调控机制,有待于今后更加深入细致的研究。至于大鼠gal-3 cDNA的多样性的生物学意义,很可能与Gal-3作为多功能蛋白在生物体内广泛行使功能的性质有关。虽然目前得此结论为时尚早,但是,基因的多样性总体上是生物进化的特征,有利于基因的表达调控及其生物学功能的实现。
参考文献(References)
[1]LI M, WANG I X, LI Y, et al. Widespread RNA and DNA sequence differences in the human transcriptome [J]. Science, 2011, 333(60338): 53-58.
[2]LI J B, LEVANON E Y, YOON J K, et al. Genome-wide identification of human RNA editing sites by parallel DNA capturing and sequencing [J]. Science, 2009, 324(5931): 1210-1213.
[3]YANG R Y, RABINOVICH G A, LIU F T. Galectins: structure, function and therapeutic potential [J]. Expert Reviews in Molecular Medicine, 2008, 10: e17.
[4]DUMIC J, DABELIC S, FLGEL M. Galectins: an open-ended story [J]. Biochim Biophys Acta, 2006, 1760(4): 616- 635.
[5]KRZESLAK A, LIPINSKA A. Galectin-3 as a multifunctional protein [J]. Cellular & Molecular Biology Letters, 2004, 9(2): 305-328.
[6]TAKENAKA Y, FUKUMORI T, RAZ A. Galectin-3 and metastasis [J]. Glycoconjugate Journal, 2004, 19(7-9): 543-549.
[7]NAKAHARA S, OKA N, RAZ A. On the role of galectin-3 in cancer apoptosis [J]. Apoptosis, 2005, 10(2): 267-275.
[8]ARGÜESO P, PANJWANI N. Focus on molecules: galectin-3 [J]. Experimental Eye Research, 2011, 92(1): 2-3.
[9]徐跃, 宋敏国, 杨仙玉, 等. 中华大蟾蜍galectin-3基因的克隆、序列分析及其原核表达载体的构建[J]. 西南大学学报(自然科学版), 2012, 34(4): 46-52.
XU Yue, SONG Minguo, YANG Xianyu, et al. Cloning and sequencing ofBufogargarizansgalectin-3 cDNA and the construction of the vector for its prokaryotic expression [J]. Journal of Southwest University (Natural Science Edition), 2012, 34(4): 46-52.
[10]周品品, 万文彬, 袁进强, 等. 比较中华大蟾蜍不同个体间galectin-3 cDNA分子多样性[J].安徽农业科学, 2013, 41(9): 3938-3943.
ZHOU Pinpin, WAN Wenbin, YUAN Jinqiang, et al. Comparison of the skin Galectin-3 cDNA diversity among different individuals ofBufogargarizans[J]. Journal of Anhui Agricultural Sciences, 2013, 41(9): 3938-3943.
[11]潘李念, 万文彬, 张姝芳, 等. 中华大蟾蜍gal-3基因多样性的原因分析[J]. 华东师范大学学报(自然科学版), 2013, 11(6): 171-180.
PAN Linian, WAN Wenbin, ZHANG Shufang, et al. Analysis of gal-3 gene diversity ofBufogargarizans[J]. Journal of East China Normal University (Natural Science), 2013, 11(6): 171-180.
[12]BRENNICKE A, MARCHFELDER A, BINDER S. RNA editing [J]. FEMS Microbiology Reviews, 1999, 23(3): 297-316.
[13]LIU L F, CHANG C C, LIAN M Y, et al. Genetic characterization of the mRNA encoding α-bungarotoxin: isoforms and RNA editing inBungarusmulticinctusgland cells[J]. Neucleic Acids Research, 1998, 26(24): 5624-5629.
[14]汪芳,王义权,童宗中. 银环蛇心脏毒素类似物cDNA的克隆[J]. 遗传, 2003, 25(6): 672-676.
WANG Fang ,WANG Yiquan, TONG Zongzhong. Cloning and characterization of cDNAs of cardiotoxin-like protein inBungarusmulticinctusvenom gland [J]. Hereditas, 2003, 25(6): 672-676.
[15]林鲁萍, 林群, 王义权. 两个蛇毒基因克隆及cDNA序列多态性再分析[J]. 动物学研究, 2007, 28(5): 457-464.
LIN Luping, LIN Qun, WANG Yiquan. Cloning of two toxin related genes and analysis of their cDNA polymorphism [J]. Zoological Research, 2007, 28(5): 457-464.

