致敏性植物几丁质酶研究进展
相启森,时国庆,马云芳,董吉林,申瑞玲
(郑州轻工业学院食品与生物工程学院,河南省食品生产与安全协同创新中心,河南郑州450002)
致敏性植物几丁质酶研究进展
相启森,时国庆,马云芳,董吉林,申瑞玲*
(郑州轻工业学院食品与生物工程学院,河南省食品生产与安全协同创新中心,河南郑州450002)
几丁质酶(EC 3.2.1.14)是一类能够催化水解几丁质结构中β-1,4糖苷键的水解酶,广泛存在于植物、动物和微生物等多种生物中。植物几丁质酶的高水平表达能够增强植物对害虫、病原菌和环境胁迫的抵抗能力。最近的研究表明,来源于植物性食品的几丁质酶是一种重要的食品过敏原,与乳胶-水果综合症密切相关。致敏性植物几丁质酶的潜在健康危害引起了广泛的关注。本文总结了致敏性植物几丁质酶的分布、结构、分类等方面的研究进展,探讨了致敏性植物几丁质酶对食品安全的影响,以期为食品中致敏性植物几丁质酶的分离、结构鉴定、过敏原性评价和控制技术等方面的研究提供参考。
食物过敏,植物几丁质酶,过敏原
近年来,世界范围内食物过敏性疾病的发病率持续上升,严重威胁了人类健康。研究表明,约有5%的成年人发生食物过敏,而婴幼儿及儿童的发病率相对更高,约为8%[1-2]。食物过敏(Food allergy)主要是免疫球蛋白E(IgE)介导的I型超敏反应(即过敏反应),还有一小部分属于非IgE介导的IV型超敏反应(即迟发型超敏反应)。食物过敏反应发生迅速,能够损伤多种组织器官,可导致过敏性休克、低血压、心律失常等症状,严重时甚至危及生命[3-4]。据估计,美国每年约有100人死于食物过敏反应[5]。目前,食物过敏已成为全球关注的食品安全问题和公共健康问题。
食品中能使机体产生过敏反应的抗原分子即为食品过敏原,食品过敏原大部分是蛋白质[6]。分离和鉴定食品过敏原是研究食物过敏性疾病发生机制和预防措施的重要基础。植物性食品是食品过敏原的重要来源,国内外学者对来源于蔬菜、水果、小麦、大豆等植物性食品的过敏原进行了大量研究[7-8]。几丁质(Chitin)又名甲壳素,是由N-乙酰基葡萄糖胺以β-1,4糖苷键聚合而成的线型高分子聚合物,广泛存在于自然界中。几丁质酶(Chitinase)能够催化水解几丁质生成N-乙酰葡糖胺,广泛存在于植物、动物、细菌、真菌和病毒等生物中[9-10]。大量研究表明,植物几丁质酶是一类重要的食品过敏原,其潜在的健康危害引起了广泛的关注[9-12]。本文总结了植物几丁质酶在植物中的分布及其分类、结构和过敏原性等方面的研究进展,并探讨了植物几丁质酶对食品安全的影响,以期为食品中致敏性植物几丁质酶的分离、结构鉴定、过敏原性评价和控制技术等方面的研究提供参考。
1 几丁质酶分类
几丁质酶的分类方法主要包括以下3种。
1.1根据国际生物化学和分子生物学联合会(ⅠUBMB)酶学委员会(EC)分类
由于几丁质酶和纤维素酶有许多相同之处,且同属于糖基水解酶类,根据国际生物化学和分子生物学联合会(IUBMB)酶学委员会(EC)的命名原则,几丁质酶被命名为EC.3.2.1.14[13]。
1.2根据催化部位氨基酸序列相似性分类
根据催化部位氨基酸序列的相似性,Henrssat将糖基水解酶分为57个家族,其中几丁质酶(EC.3.2.1.14)被分为18和19家族,而同样参与几丁质降解的β-N-乙酰己糖胺酶(EC 3.2.1.52)被分为20家族[14-16]。18家族由病毒、真菌、昆虫、植物、动物等不同来源的几丁质酶构成[17];而19家族几丁质酶几乎全部由来源于植物,只有很少一部分来源于灰色链霉菌等微生物[17-18]。
1.3根据基因序列和结构差异分类
根据基因序列和结构差异,植物几丁质酶可分为5类,即I~V类。其中I、II和IV类植物几丁质酶属于19家族,而III和V类植物几丁质酶属于18家族[19-20]。
2 植物几丁质酶结构特点
如图1所示,植物几丁质酶前体一般含有一个N-端信号区(前导肽区,Presequence module,PM)和一个催化区(Catalytic module,CM),I和IV类几丁质酶在PM区之后有富含半胱氨酸的几丁质结合域(Chitinbinding modules,CtBM),CtBM与CM之间由可变交联区(Linker module,LM)连接[10,20]。

图1 植物几丁质酶结构示意图Fig.1 Schematic representation of the structure of plant chitinase
I类植物几丁质酶由CtBM、CM和LM组成,且与烟草I类几质酶同源性大于50%;II类植物几丁质酶与烟草I类和II类几丁质酶的同源性大于50%,但缺少CtBM结构[21-22]。III植物类几丁质酶与烟草III类几丁质酶和溶菌酶的同源性大于50%[10,23],属于植物病程相关蛋白8(Pathogenesis-related proteins 8,PR-8)[22-23]。IV类几丁质酶与菜豆(Phaseolus vulgaris)PR-4蛋白在CM区的同源性大于50%,且其CM区比I类和II类植物几丁质酶约少50个氨基酸残基[10]。I和II类植物几丁质酶在CM区含有5个环结构(Loop),IV类植物几丁质酶仅含有两个环结构,而III和V类植物几丁质酶结构中均不含有环结构[10,20]。
3 植物几丁质酶致敏性
几丁质酶广泛存在于植物的根、茎、叶、果实以及种子等组织中,在抵抗防御病原物侵染和不良环境胁迫等方面具有重要作用[24-25]。植物病程相关蛋白(PR蛋白)是植物在病理或病理相关环境下诱导产生的一类蛋白,具有抵抗真菌、细菌、病毒、昆虫等病原物侵染和干旱、盐渍等逆境胁迫等多重作用[25]。根据结构特点,PR蛋白可分为17类,其中PR-3、-4、-8和-11具有几丁质酶活性[26]。大量研究证实,植物几丁质酶是一种重要的食品过敏原。此外,由于几丁质酶具有增强植物抗真菌病害的能力[22,25],几丁质酶基因被广泛应用于抗真菌病害植物的基因工程育种研究中[27-28],但经基因工程导入的外源几丁质酶基因表达产物是否具有致敏性尚不清楚。
3.1Ⅰ类植物几丁质酶
绝大部分具有致敏性的植物几丁质酶属于I类。I类植物几丁质酶存在于鳄梨(Persea americana)[29-30]、欧洲栗(Castanea sativa)[30-31]、香蕉(Musa paradisiaca)[32-33]、猕猴桃(Actidinia chinensis)[34]等植物果实中。Sowka等从鳄梨中果皮分离得到一种分子量为32ku的蛋白质(Prs a 1)。该蛋白能与20份乳胶过敏患者(其中7人对鳄梨过敏)血清中的15份发生免疫反应,表明该蛋白能够与乳胶过敏原发生交叉反应。cDNA序列分析结果表明,Prs a 1的cDNA能够编码326个氨基酸组成的内切几丁质酶。采用毕赤酵母(Pichia pastoris)重组表达的Prs a 1蛋白具有内切几丁质酶活性并与IgE发生结合。氨基酸序列分析表明,Prs a 1与主要乳胶过敏原-橡胶蛋白的前体(Prohevein)在CtBM区的氨基酸序列相似度为70%[29]。Mikkola等从香蕉中分离得到的I类几丁质酶(分子量分别为32ku和33ku)均能与15份来自对天然乳胶过敏患者血清中的9份发生免疫反应,其N-末端序列与橡胶蛋白(Hevein)氨基酸序列相似度约为80%,其中33 ku蛋白中内含肽(20个氨基酸)与水稻(Oryza sativa)内切性几丁质酶的相似度为95%[32-33]。I类致敏性植物几丁质酶的来源和特点见表1。
3.2Ⅰ类植物几丁质酶
与I类植物几丁质酶相比,II类植物几丁质酶仅缺乏CtBM结构。但由于早期研究认为几丁质酶的致敏性与CtBM结构有关,因此在早期研究中,II类几丁质酶并未被归为食品过敏原。例如,Diaz-Perales等的研究表明,鳄梨和欧洲栗中II类几丁质酶因缺乏CtBM而不能与患者血清发生IgE结合反应[30]。但最新研究表明,存在于小麦粉中的II类几丁质酶(分子量约为26ku)能与过敏性皮肤炎患者血清IgE发生结合反应[38]。此外,从大麦[39]、番木瓜[40]、黑麦[41]、感染晚疫病的马铃薯[42]等样品中分离得到了多种II类几丁质酶,但目前尚不能确定上述II类几丁质酶是否具有致敏性。
3.3ⅠⅠ类植物几丁质酶
大量研究表明,来源于滇刺枣(Zizyphusmauritiana)[43-44]、橡胶(Hevea brasiliensis)[45]、树莓(Rubus ideaus)[46]和小果咖啡(Coffea arabica)[47]的III类几丁质酶可能具有致敏性。滇刺枣又名毛叶枣、印度枣和台湾青枣等,是一种主产于印度、泰国和中国台湾的新兴热带水果[48]。Lee等先后从滇刺枣中分离得到分子量分别为42ku和30ku的两种蛋白质,均能与滇刺枣过敏者血清IgE发生结合反应[43-44]。30ku蛋白(被命名为Ziz m 1)的cDNA克隆能够编码330个氨基酸,分子量为33.86ku,并推测含有25个氨基酸组成的前导肽。重组表达的Ziz m 1蛋白具有几丁质酶活力,其皮肤过敏测试阳性率为87.5%。Ziz m 1蛋白与来源于橡胶(Hevea brasiliensis)、红豆(Vigna angularis)、辣椒(Capsicum annuum)、水稻(Oryza sativa)的III类几丁质酶的氨基酸序列相似度分别为45.2%、45.3%、44.7%和41.2%[44]。此外,Marzban等从树莓(Rubus ideaus)分离得到的III类几丁质酶与血清IgE结合率超过80%[46]。Manavski等从小果咖啡分离得到一种III类几丁质酶(Cof a1),发现重组表达的Cof a 1能与2名咖啡过敏患者的血清发生免疫反应,并在两人血清中均发现了抗Cof a1的IgE抗体[47]。

表1 I类致敏性植物几丁质酶的来源和特点Table 1 Sources and characteristics of class I allergenic plant chitinases
3.4ⅠV类植物几丁质酶
来源于日本柳杉(Cryptomeria japonica)的IV类植物几丁质酶被证实具有过敏原性[49]。日本柳杉是导致日本国内超过13%的人口出现花粉症的主要原因[50-51]。Fujimura等从日本柳杉花粉中分离得到一种具有高IgE结合能力的蛋白(CJP-4),其cDNA克隆能够编码281个氨基酸,天然状态下能与31名患者血清中的IgE全部结合,并能与乳胶过敏原发生交叉反应[49]。同时,CJP-4蛋白具有内切性几丁质酶活性,与IV类几丁质酶具有很高的同源性,序列相似度可以达到43.1%~65.6%[49]。此外,从酿酒葡萄(Vitis vinifera)、美洲葡萄(Vitis labrusca)及其所制葡萄酒[52]、玉米粉[53-54]等样品中分离鉴定了具致敏性的IV类几丁质酶,但尚未确定其结构。同时,Ubhayasekera等分析了一种挪威云杉IV类几丁质酶的x-射线晶体结构但未报到其是否具有致敏性[20]。针对上述IV类几丁质酶,应继续开展其结构和过敏原性评价等方面的研究。
3.5V类植物几丁质酶
目前,从苏铁(Cycas revoluta)、拟南芥(Arabidopsis thaliana)和烟草(Nicotiana tabacum)中分离鉴定了多种V型几丁质酶,但尚未证实其是否具有过敏原性[55-57]。Taira等从苏铁中分离得到一种分子量为40ku、等电点为5.6的V型几丁质酶[55]。Ohnuma等先后从拟南芥和烟草中分离得到两种V型几丁质酶并测定了其晶体结构[56-57]。因此,针对V类植物几丁质酶,研究工作的重点应为采用IgE检测技术、皮肤过敏测试等方法评价其过敏原性。
4 几丁质酶相关过敏原蛋白
与植物几丁质酶结构类似的一些蛋白质也具有致敏性,其中最常见的是橡胶蛋白(Hevein)。橡胶蛋白是由巴西橡胶树(Hevea brasilensis)乳管细胞特化的液泡-黄色体分泌的小分子酸性凝集素[58]。橡胶蛋白(Hev b 6.02)是主要的乳胶过敏原,能与80%以上的乳胶过敏患者血清中的IgE发生结合反应[59]。橡胶蛋白由43个氨基酸残基组成,分子量为4.7ku。橡胶蛋白的氨基酸序列与I类和IV类几丁质酶的CtBM区、小麦胚凝集素(Wheat germ agglutinin,WGA)等具有高度的同源性,说明橡胶蛋白与它们有着极其相似的生理功能[59]。Diaz-Perales等的研究表明,来源于鳄梨和欧洲栗的I类几丁质酶与橡胶蛋白N-末端氨基酸序列相似度很高,均能与患者血清IgE发生结合反应,这与其CtBM氨基酸序列与橡胶蛋白类似有关[30]。Broekaert等获得了橡胶蛋白的cDNA克隆(HEV1)。HEV1包括编码204个氨基酸的开放阅读框架,其中分为由17个氨基酸组成的信号序列、C-端区域(144个氨基酸残基)和高度保守的N-端区域(Hevein,43个氨基酸残基)三个组成部分[60]。橡胶蛋白与几丁质酶、小麦胚凝集素的结构比较见图2[61]。
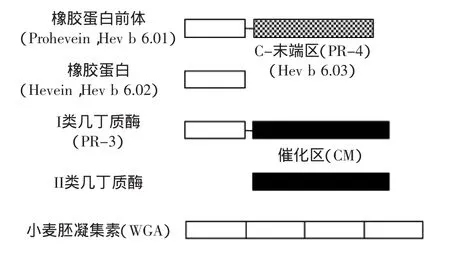
图2 橡胶蛋白,几丁质酶和小麦胚凝集素结构比较Fig.2 Comparison of structures among hevein,chitinases,and wheat germ agglutinin
5 结论与展望
食物过敏已成为全球关注的食品安全问题和公共健康问题。作为一类重要的食品过敏原,植物几丁质酶广泛存在于香蕉、猕猴桃、滇刺枣、四季豆、小麦、玉米等蔬菜、水果和食品原料中,其潜在的健康危害引起了广泛的关注。国外学者对致敏性植物几丁质酶进行了分离纯化、结构鉴定和过敏原性评价等诸多方面的研究。然而,国内针对食品中致敏性植物几丁质酶的研究尚处于起步阶段。鉴于植物几丁质酶在蔬菜水果、食品原料中的广泛分布和潜在的健康危害,国内研究机构非常有必要加强该领域的研究工作。首先应加强基础研究工作,开展食品中几丁质酶过敏原性评价方面的研究,并对食品中致敏性植物几丁质酶进行分离、纯化和结构鉴定。针对目前多采用血清IgE检测食品中致敏性植物几丁质酶检测的现状,还应建立快速、高效、高通量、高灵敏度的食品中致敏性植物几丁质酶检测方法。此外,还应系统研究热加工、辐照、高压、高压脉冲电场、超声波等食品物理加工方式对几丁质酶过敏原结构和致敏性的影响,开发降低或脱除食品中致敏性植物几丁质酶过敏原的新型加工技术,以期保障消费者的饮食安全和身体健康。
[1]Sicherer SH,Sampson HA.Food allergy:Epidemiology,pathogenesis,diagnosis,and treatment[J].J Allergy Clin Immunol,2014,133(2):291-307.
[2]Van Hengel AJ.Food allergen detection methods and the challenge to protect food-allergic consumers[J].Anal Bioanal Chem,2007,389(1):111-118.
[3]Ho MHK,Wong WHS,Chang C.Clinical spectrum of food allergies:a comprehensive review[J].Clin Rev Allergy Immunol,2014,46(3):198-210.
[4]Ebo DG,Stevens WJ.IgE-mediated food allergy-extensive review of the literature[J].Acta Clin Belg,2001,56(4):234-247.
[5]Sampson HA,Mendelson L,Rosen JP.Fatal and near-fatal anaphylactic reactions to food in children and adolescents[J].N Engl J Med,1992,327(6):380-384.
[6]金莹,房保海.食品过敏原的分类及安全管理[J].食品安全质量检测学报,2012,3(4):240-244.
[7]Breiteneder H,Radauer C.A classification of plant food allergens[J].J Allergy Clin Immunol,2004,113(5):821-830.
[8]Zuidmeer L,Goldhahn K,Rona RJ,et al.The prevalence of plant food allergies:a systematic review[J].J Allergy Clin Immuno,2008,121(5):1210-1218.
[9]Hamid R,Khan MA,Ahmad M,et al.Chitinases:an update[J].J Pharm Bioallied Sci,2013,5(1):21-29.
[10]Volpicella M,Leoni C,Fanizza I,et al.Overview of plant chitinases identified as food allergens[J].J Agric Food Chem,2014,62(25):5734-5742.
[11]Lee CG,Da Silva CA,Dela Cruz CS,et al.Role of chitin and chitinase/chitinase-like proteins in inflammation,tissue remodeling,and injury[J].Annu Rev Physiol,2011,73:479-501.
[12]Duru S,Yüceege M,Ardıç S.Chitinases and lung diseases[J].Tuberk Toraks,2013,61(1):71-75.
[13]赵晓琴,李冠.几丁质酶研究进展[J].新疆大学学报:自然科学版,2003,20(4):1210-1218.
[14]Henrissat B.A classification of glycosyl hydrolases based on amino acid sequence similarities[J].Biochem J,1991,280(2):309-316.
[15]Henrissat B,Bairoch A.New families in the classification of glycosyl hydrolases based on amino acid sequence similarities[J]. Biochem J,1993,293(3):781-788.
[16]Henrissat B,Bairoch A.Updating the sequenee-based classification of glycosyl hydrolases[J].Bioehem J,1996,316(2):695-696.
[17]Passarinho PA,de Vries SC.Arabidopsis chitinases:a genomic survey[J].Arabidopsis Book,2002,1:e0023.
[18]Watanabe T,Kanai R,Kawase T,et al.Family 19 chitinases of Streptomyces species:characterization and distribution[J]. Microbiology,1999,1451(1):3353-3363.
[19]Neuhaus JM,Fritig B,Linthorst HJM,et al.A revised nomenclature for chitinase genes[J].Plant Mol Biol Rep,1996,141(1):102-104.
[20]Ubhayasekera W,Rawat R,Ho SW,et al.The first crystal structures of a family 19 class IV chitinase:the enzyme from Norway spruce[J].Plant Mol Biol,2009,71(3):277-289.
[21]Raikhel NV,Lee HI,Broekaert WF.Structure and function of chitin-binding proteins[J].Ann Rev of Plant Physiol Plant Mol Biol,1993,44:591-615.
[22]欧阳石文,赵开军,冯兰香,等.植物几丁质酶的研究进展[J].生物工程进展,2001,21(4):30-34.
[24]Herrera-Estrella A,Chet I.Chitinases in biological control[J].EXS,1999,87:171-184.
[23]Collinge DB,Kragh KM,Mikkelsen JD,et al.Plant chitinases[J].Plant J,1993,3(1):31-40.
[25]Adrangi S,Faramarzi MA.From bacteria to human:a journey into the world of chitinases[J].Biotechnol Adv,2013,31(8):1786-1795.
[26]Sels J,Mathys J,De Coninck BM,et al.Plant pathogenesisrelated(PR)proteins:a focus on PR peptides[J].Plant PhysiolBiochem,2008,46(11):941-950.
[27]Sharma N,Sharma KP,Gaur RK,et al.Role of Chitinase in Plant Defense[J].Asian J Biochem,2011,6(1):29-37.
[28]Cletus J,Balasubramanian V,Vashisht D,et al.Transgenic expression of plant chitinases to enhance disease resistance[J]. Biotechnol Lett,2013,35(11):1719-1732.
[29]Sowka S,Hsieh LS,Krebitz M,et al.Identification and cloning of prs a 1,a 32-kua endochitinase and major allergen of avocado,and its expression in the yeast Pichia pastoris[J].J Biol Chem,1998,273(43):28091-28097.
[30]Díaz-Perales A,Collada C,Blanco C,et al.Class I chitinases with hevein-like domain,but not class II enzymes,are relevant chestnut and avocado allergens[J].J Allergy Clin Immunol,1998,102(1):127-133.
[31]Allona I,Collada C,Casado R,et al.Bacterial expression of an active class Ib chitinase from Castanea sativa cotyledons. Plant Mol Biol,1996,32(6):1171-1176.
[32]Sánchez-Monge R,Blanco C,Díaz-Perales A,et al.Isolation and characterization of major banana allergens:identification as fruit class I chitinases[J].Clin Exp Allergy,1999,29(5):673-680.
[33]Mikkola JH,Alenius H,Kalkkinen N,et al.Hevein-like protein domains as a possible cause for allergen cross-reactivity between latex and banana[J].J Allergy Clin Immunol,1998,102(6):1005-1012.
[34]Bublin M,Mari A,Ebner C,et al.IgE sensitization profiles toward green and gold kiwifruits differ among patients allergic to kiwifruit from 3 European countries[J].J Allergy Clin Immunol,2004,114(5):1169-1175.
[35]Hanninen AR,Mikkola JH,Kalkkinen N,et al.Increased allergen production in turnip(Brassica rapa) by treatments activating defense mechanisms[J].J Allergy Clin Immunol,1999,104(1):194-201.
[36]Díaz-Perales A,Collada C,Blanco C,et al.Cross-reactions in the latex-fruit syndrome:A relevant role of chitinases but not of complex asparagine-linkedglycans[J].J Allergy Clin Immunol,1999,104(3):681-687.
[37]Sánchez-Monge R,Blanco C,Perales AD,et al.Class I chitinases,the panallergens responsible for the latex-fruit syndrome,are induced by ethylene treatment and inactivated by heating[J].J Allergy Clin Immunol,2000,106(1 Pt 1):190-195.
[38]Sotkovsky P,SklenárˇJ,Halada P,et al.A new approach to the isolation and characterization of wheat flour allergens[J].Clin Exp Allergy,2011,41(7):1031-1043.
[39]Hart PJ,Monzingo AF,Ready MP,et al.Crystal structure of an endochitinase from Hordeum vulgare L.seeds[J].J Mol Biol,1993,229(1):189-193.
[40]Huet J,Rucktooa P,Clantin B,et al.X-ray structure of papaya chitinase reveals the substrate binding mode of glycosyl hydrolase family 19 chitinases[J].Biochemistry,2008,47(32):8283-8291.
[41]Ohnuma T,Numata T,Osawa T,et al.Crystal structure and chitin oligosaccharide-binding mode of a‘loopful’family GH19 chitinase from rye,Secale cereale,seeds[J].FEBS J,2012,279(19):3639-3651.
[42]Büchter R,Strömberg A,Schmelzer E,et al.Primary structure and expression of acidic(class II)chitinase in potato[J].Plant Mol Biol,1997,35(6):749-761.
[43]Lee MF,Hwang GY,Chen YH,et al.Molecular cloning of Indian jujube(Zizyphus mauritiana)allergen Ziz m 1 with sequence similarity to plant class III chitinases[J].Mol Immunol,2006,43(8):1144-1151.
[44]Lee MF,Chen YH,Lan JL,et al.Allergenic components of Indian jujube(Zizyphus mauritiana)show IgE cross-reactivity with latex allergen[J].Int Arch Allergy Immunol,2004,133(3):211-216.
[45]Alenius H,Kalkkinen N,Lukka M,et al.Prohevein from the rubber tree(Hevea brasiliensis)is a major latex allergen[J].Clin Exp Allergy,1995,25(7):659-665.
[46]Marzban G,Herndl A,Kolarich D,et al.Identification of four IgE-reactive proteins in raspberry(Rubus ideaeus L.)[J]. Mol Nutr Food Res,2008,52(2):1497-1506.
[47]Manavski N,Peters U,Brettschneider R,et al.Cof a 1:identification,expression and immunoreactivity of the first coffee allergen[J].Int Arch Allergy Immunol,2012,159(3):235-242.
[48]何秋香,潘琪,陈新来,等.浸钙、壳聚糖浸泡和温烫处理对台湾青枣果皮褐变的影响[J].中国农学通报,2013,29(19):120-124.
[49]Fujimura T,Shigeta S,Suwa T,et al.Molecular cloning of a class IV chitinase allergen from Japanese cedar(Cryptomeria japonica)pollenandcompetitiveinhibitionofits immunoglobulin E-binding capacity by latex C-serum[J]. Clin Exp Allergy,2005,35(2):234-243.
[50]Wang Q,Gong X,Suzuki M,et al.Size-segregated allergenic particles released from airborne Cryptomeria japonica pollen grains during the Yellow Sand Events within the pollen scattering seasons[J].Asian J Atmos Environ,2013,7(4):191-198.
[51]李文静,刘光辉.日本柳杉花粉变应原致敏蛋白组分[J].中华临床免疫和变态反应杂志,2013,7(3):93-98.
[52]Pastorello EA,Farioli L,Pravettoni V,et al.Identification of grape and wine allergens as an endochitinase 4,a lipid-transfer protein,and a thaumatin[J].J Allergy Clin Immunol,2003,111(2):350-359.
[53]Pastorello EA,Farioli L,Pravettoni V,et al.Maize food allergy:lipid-transfer proteins,endochitinases,and alpha-zein precursor are relevant maize allergens in double-blind placebocontrolled maize-challenge-positive patients[J].Anal Bioanal Chem,2009,395(1):93-102.
[54]Fasoli E,Pastorello EA,Farioli L,et al.Searching for allergens in maize kernels via proteomic tools[J].J Proteomics,2009,72(3):501-510.
[55]Taira T,Hayashi H,Tajiri Y,et al.A plant class V chitinase from a cycad(Cycas revoluta):biochemical characterization,cDNA isolation,and posttranslational modification[J].Glycobiology,2009,19(12):1452-1461.
[56]Ohnuma T,Numata T,Osawa T,et al.A class V chitinase from Arabidopsis thaliana:gene responses,enzymatic properties,and crystallographic analysis[J].Planta,2011,234(1):123-137.
[57]Ohnuma T,Numata T,Osawa T,et al.Crystal structure and mode of action of a class V chitinase from Nicotiana tabacum[J]. Plant Mol Biol,2011,75(3):291-304.
[58]Van Parijs J,Broekaert WF,Goldstein IJ,et al.Hevein:an antifungal protein from rubber-tree(Hevea brasiliensis)latex[J]. Planta,1991,183(2):258-264.
[59]Sánchez-Monge R,Díaz-Perales A,Blanco C,et al.Class I chitinases and the latex-fruit syndrome[J].Internet Symposium on Food Allergens,2000,2(3):137-144.
[60]Broekaert I,Lee HI,Kush A,et al.Wound-induced accumulation of mRNA containing a hevein sequence in laticifers of rubber tree(Hevea brasiliensis)[J].Proc Natl Acad Sci USA,1990,87(19):7633-7637.
[61]Division of Medical Devices,National Institute of Health Sciences.Latex-fruit syndrome and class 2 food allergy[EB/OL]. http://dmd.nihs.go.jp/latex/cross-e.html.
Research progress on allergenic plant chitinases
XIANG Qi-sen,SHI Guo-qing,MA Yun-fang,DONG Ji-lin,SHEN Rui-ling*
(College of Food and Biological Engineering,Zhengzhou University of Light Industry,Henan Collaborative Innovation Center for Food Production and Safety,Zhengzhou 450002,China)
Chitinases(EC 3.2.1.14)are hydrolytic enzymes that catalyse the random cleavage of internal β-1,4 glycosidic linkages in the chitin polymer,and they occur in a wide range of organisms including plants,animals,and microorganisms.High-level expression of chitinases could enhance the resistance of plants to pest infestation,pathogens,and other environmental challenges.Recent studies have revealed that chitinases isolated from many common plant foods were allergens involved in the latex-fruit syndrome.The potential adverse health effects of allergenic plant chitinases have attracted much attention.Advances in researches on the distributions,structures,classifications of allergenic plant chitinases and their influence on food safety were summarized in this paper to provide guidelines for the isolation,structural identification,allergenicity assessment,and control of allergenic plant chitinases in foods.
food allergy;plant chitinases;allergens
TS201.6
A
1002-0306(2015)10-0389-06
10.13386/j.issn1002-0306.2015.10.074
2014-08-04
相启森(1984-),博士,讲师,研究方向:食品化学与营养。
申瑞玲(1967-),博士,教授,研究方向:食品化学与营养。
郑州轻工业学院博士科研基金资助项目(2013BSJJ079)。

