基于语义关系的疾病知识提取系统
吴晓芳,杨志豪,林鸿飞,王 健
(大连理工大学计算机科学与技术学院,辽宁大连116024)
基于语义关系的疾病知识提取系统
吴晓芳,杨志豪,林鸿飞,王 健
(大连理工大学计算机科学与技术学院,辽宁大连116024)
在生物医学领域,通过知识提取过程从海量的生物医学文献中提取疾病、基因和药物之间的关系并可视化显示,可以为临床医学实验提供有效的假设检验,推动生物医学科技的发展。为此,提出一种基于语义关系的以疾病为中心的疾病、基因和药物间的知识提取系统。利用SemRep得到特定主题Medline文献的语义输出,通过显著信息提取算法提取SemRep的语义输出关系。对照OMIM和GHR在线数据库进行评估,实验结果显示该显著信息提取系统的准确率可达0.76。
知识提取;语义关系提取;显著信息提取算法;SemRep工具;语义输出;网络图可视化
1 概述
生物医学文献持续不断的增长给传统的信息检索技术带来极大的挑战。有效的医学文献检索,尤其是从海量的生物医学文献中发现显著的疾病、基因、药物之间的关联信息对生物医学工作者在临床试验和病患诊疗方面有极大的帮助。传统的人工阅读大量文献费时费力且效果甚微,在如今的数字化信息时代已经不再适用。虽然早先的信息检索技术已经应用到生物医学领域的知识提取,但是信息检索算法的有效性并没有得到很好的评估[1]。文献[2]从Medline中抽取有用的关系,简洁地概括出原始文献的主要信息。文献[3]提出了一个自动从文献集中提取摘要的算法Combo,该算法提取了与某一特定疾病相关的基因。文献[4]在之前实验基础上又提出了一个用于决策支持的文本摘要生成方法。为了跟踪最新的医学研究领域的工作进展,生物医学文献的高效检索,有效关系的提取和展示对临床决策支持[5]来说尤为重要。自动摘要方法[6]在信息提取中有较好的效果,但是自动摘要最终形成的依然是文本形式,不够直观。因此,需要有效基于语义关系抽取的算法来从大量的生物医学文献中提取出重要的实体关联信息,并用可视化的方法将该关联信息呈现给医学工作者。
与以往研究不同,本文提出一个基于语义关系的以疾病为中心的疾病、基因和药物间的知识提取系统。该系统利用从Medline生物医学数据库检索到的相关疾病的语料集,运用SemRep工具处理得到相关疾病语料集的语义输出。通过显著信息提取算法筛选出以疾病为中心的疾病、基因和药物三者之间重要的关联信息,并以网络关系图的形式呈现给生物医学工作者。
2 系统方法
2.1 系统流程
系统流程如图1所示。

图1 系统流程
对于特定的疾病,从PubMed上检索到2003年-2013年与疾病相关的文献集。针对疾病和基因、疾病和药物给出不同的检索语句,检索得到相应的文献集。
通过SemRep工具处理文献集得到相应的语义输出。SemRep能够从Medline语料的句子中抽取出2个实体之间的关联关系。如果一个句子中存在多个实体词和关系连接词,那么SemRep通过算法给每个关系打分,取分数最高的连接关系作为语义输出。
用KL散度、RlogF矩阵显著信息评价算法分别对谓词关系、谓词关系连接的实体语义类型进行筛选,利用PredScal平衡前2种算法间的数值差,综合3种算法共同完成对疾病和基因、疾病和药物显著信息的提取。
最后将提取得到的以疾病为中心的显著信息网络图可视化,在系统界面中呈现给用户。
2.2 文献语料处理工具SemRep
SemRep[7]是一个基于规则自动从文献中识别关系预测的自然语言处理系统。SemRep集成了MetaMap规范化的概念实体,并通过谓词关系将不同的实体概念连接起来。此外,SemRep为每个实体词定义了相关的语义类型,方便特征选取和语义类型过滤。SemRep提取的关系是根据UMLS的规则进行输出的,其原始结果中包含有很多条目,主要用到其中的实体名、语义类型和谓词关系部分。
例如,对于句子:
Expression levels of CBX7 inversely correlate with the progression of tumor stage and grade in urothelial carcinomas of the bladder,suggesting that downregulation of CBX7 indicates aggressive urothelial carcinoma phenotype.
SemRep可以得到如下的语义输出:
SE|18984978|RESULTS|ab|5|relation|5|1||| gngm,aapp|gngm|23492|CBX7|CBX7||||1000|53 |56|VERB|PART_OF||71|79|2|1|C0007138| Carcinoma,Transitional Cell|neop|neop|||urothelial carcinomas||||981|84|104
这里主要关注的是关联信息[8]:
CBX7|gngm|PART_OF|urothelial carcinomas| neop
CBX7是一种参与调控细胞增殖衰老的转录抑制因子。从得到的输出可以看出,CBX7转录抑制因子是癌细胞病变因子的组成部分。
关联信息是一个三元组(概念1|语义类型, Predication,概念2|语义类型)[9],概念1和概念2是UMLS的超级叙词表中定义的概念,每个概念包含该概念的标准化表示、概念标示符(Concept Unique Identifier,CUI)和语义类型。UMLS的语义网络中共定义了54中谓词关系(PART_OF是其中之一)。利用SemRep可以从一个句子中得到出一个或多个语义输出,通过一定的算法,对得到的语义输出进行打分,选取得分高的语义输出作为该句的关联信息。从文献中所有的句子里抽取出关联信息集,进一步运用显著信息提取算法进行筛选。
2.3 实验数据
以膀胱癌(Carcinoma of bladder)为例,介绍实验中用到的数据集以及显著信息提取算法的实现。
(1)与Carcinoma of bladder相关的基因方面的文献集A
(“2003/01/01”[Publication Date]:“2013/07/31”[Publication Date])AND(Urinary Bladder Neoplasms/ genetics[majr] AND Urinary Bladder Neoplasms/ etiology[majr])AND English[la]AND humans[mh]
(2)与Carcinoma of bladder相关的药物方面的文献集B
(“2003/01/01”[Publication Date]:“2013/07/31”[Publication Date])AND Urinary Bladder Neoplasms [mh noexp]AND drug therapy[sh]AND Clinical Trial [pt]AND English[Lang]AND humans[mh]
这2组查询语句检索了从2003年-2013年的Medline文献。与基因相关的文献集A设定了基因和膀胱病因学等限制词,检索得到与膀胱癌相关的基因类的文献。与药物相关的文献集B设定了药物、临床治疗和膀胱病因学等限制词,检索得到与膀胱癌相关的药物类文献。通过上面2组查询语句,从PubMed上下载对应的Medline文献集。
2.4 显著信息评价算法
为了实现有用信息的提取,本文实验中使用了3种显著信息提取算法,自动地从SemRep的输出结果中筛选出查询的疾病与基因、药物之间的关联关系,排除掉繁多的相关性弱的关系。这3种显著信息提取算法介绍如下:
(1)KL散度
KL散度[10],又叫相对熵,在信息论中用于衡量2个概率分布的相对距离。在这里对关系谓词在疾病数据集A中的概率P和关系谓词在所有数据集B中的概率Q作为要衡量的2个概率。相对距离大的关系谓词表示在该疾病数据集中有比较突出的作用,从而通过得到的KLD(Kullback-Leibler Divergence)得分值对关系谓词进行排名,得到关系谓词的筛选结果。
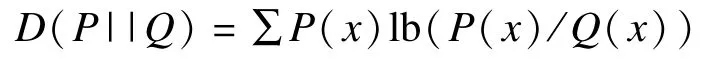
其中,x代表一个关系谓词;P(x)代表关系谓词x在分布P中的概率;Q(x)代表关系谓词x在分布Q中的概率。例如,关系谓词ASSOCIATED_WITH在分布P中的概率为0.290,在分布Q中的概率为0.076,那么关系谓词ASSOCIATED_WITH的KLD值为0.560 3。
KLD算法中分布Q的统计数据选取了2003年1月1日-2013年7月31日之间所有的Medline文献集。
(2)RlogF
RlogF矩阵[11]旨在得到SemRep输出中同一个关系谓词相关度较高的语义类型,用函数R表示。关系谓词在做统计的时候受限于它在SemRep中的语义类型。
把检索词Carcinoma of bladder的语义类型neop作为种子语义类型。因为数据集是跟Carcinoma of bladder直接相关的,所以得到的语义类型中定有很多的neop,排除掉该语义类型的影响,从而能更好地筛选出与该语义类型相关联的非种子语义类型。

其中,条件概率(P(relevant|patterni))是在语料A中出现的与关系谓词直接相关的实体的语义类型的个数(包含重复的部分)与所有出现的语义类型个数的比例。
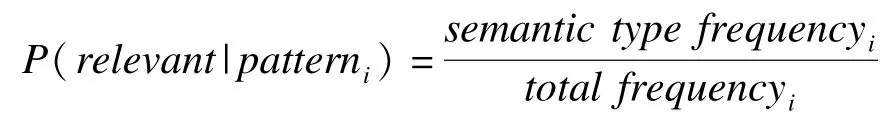
例如,如果与关系谓词ASSOCIATED_WITH共现的非种子语义类型 gngm在文献集 A中出现107次,所有与关系谓词ASSOCIATED_WITH共现的非种子语义类型共有171个(包含重复的部分),那么关系谓词ASSOCIATED_WITH的RlogF值为4.22。
(3)PredScal
RlogF算法得到的值会远远超过KLD算法得到的值,在衡量一个关系的时候,RlogF的结果占很大的比例。为了共同引用2种算法的思想,引入一个尺度函数p作为平衡因子来调整2个函数在同一数据集中的计算结果。
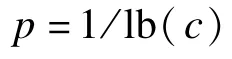
在这个计算中,c代表不同的关系谓词个数。例如,如果数据集中有16个不同的关系谓词,那么PredScal的平衡因子值0.25。
以上3种算法结合起来共同完成对SemRep的输出结果的信息提取,用Summa算法来表示每个谓词关系的分值,运算结果表示为summa。

对于SemRep输出中的每一个关系,将谓词和语义类型分别通过算法KLD和RlogF筛选出来,通过算法PredScal来矫正2个结果数值间的成倍差距。这样每个关系都有一个Summa值来作为它们的显著程度的量化。
3 实验与评价
实验中基因部分的信息提取共得到与疾病Carcinoma of bladder相关的基因54个。参照Online Mendelian Inheritance in Man(OMIM)和 Genetics Home Reference(GHR)中的基因文献记录进行标注,在得到的54个基因中有41个与疾病Carcinoma of bladder在OMIM和GHR里有关联关系。由此计算得出,实验提取结果的准确率为0.76。而SemRep语料中抽取的实体之间的关系准确率为0.73,召回率为0.55,综合分类率F值为0.63[12],本文显著信息提取算法的准确率有所提升。
3.1 疾病与基因的关系
运用KLD算法得到了与Carcinoma of bladder相关的关系谓词,通过对关系谓词排序筛选出前5个得分最高的实验结果,见表1。从表1可以看出关系谓词ASSOCIATED_WITH得分最高,这说明在疾病Carcinoma of bladder与基因的关系中,它们之间的相互作用关系,由ASSOCIATED_WITH关系词所连接的关系尤其重要。生物医学工作者可以从这个关系中寻找到与该疾病相关的基因,从而更有效地找到治疗该疾病的基因方法。

表1 KLD算法得到的前5个关系谓词(与基因相关)
运用RlogF算法得到了与Carcinoma of bladder相关的谓词以及语义类型之间的关系排名,筛选出前5个得分最高的实验结果,见表2。从表2可以看出语义类型gngm与关系谓词ASSOCIATED_WITH得分最高,这说明在疾病Carcinoma of bladder与基因之间的相互作用关系中,由谓词ASSOCIATED_ WITH所连接的实体类型为gngm的关系最为突出。语义类型gngm是Gene or Genome的缩写,代表基因类。从结果中可以看出,运用显著信息提取算法有效地筛选出了跟疾病相关的基因。

表2 RlogF算法得到的数据(与基因相关)
以上2种算法,用PredScal算法做权衡后,得到疾病与基因相关的Summa的排名结果,见表3。

表3 Summa信息提取的前5个结果(与基因相关)
3.2 疾病与药物的关系
运用KLD算法得到了与Carcinoma of bladder相关的谓词,通过对关系谓词排序筛选出前5个得分最高的实验结果,见表4。从表中4可以看出关系谓词TREATS得分最高,这说明在疾病Carcinoma of bladder与药物之间的相互作用中,由谓词TREATS所连接的关系尤其重要,通过KLD算法有效地找到了治疗疾病的相关药物。

表4 KLD算法得到的前5个关系谓词(与药物相关)
运用RlogF算法得到了与Carcinoma of bladder相关的谓词以及语义类型之间的关系排名,筛选出前5个得分最高的实验结果,如表5所示。从表5中可以看出语义类型phsu与关系谓词TREATS得分最高。这说明,在疾病与基因之间的相互作用关系中,由谓词TREATS所连接的实体类型为phsu的关系最为突出。语义类型 phsu是 Pharmacologic Substance的缩写,代表药物学物质。结果表明,显著信息提取算法有效地筛选出了能治疗疾病Carcinoma of bladder的药物。

表5 RlogF算法得到的数据(与药物相关)
以上2种算法,用PredScal算法做权衡后,得到疾病与药物相关的 Summa的排名结果,如表6所示。

表6 Summa信息提取的前5个结果(与药物相关)
3.3 基因与药物的关系
通过 Summa算法得到了疾病 Carcinoma of bladder分别与基因、药物的相关关系实体集合。对得到的基因和疾病词对依次在SemRep数据库中进行检索,得到了基因和药物之间的关联关系。表7为选取的部分相关的基因和药物。
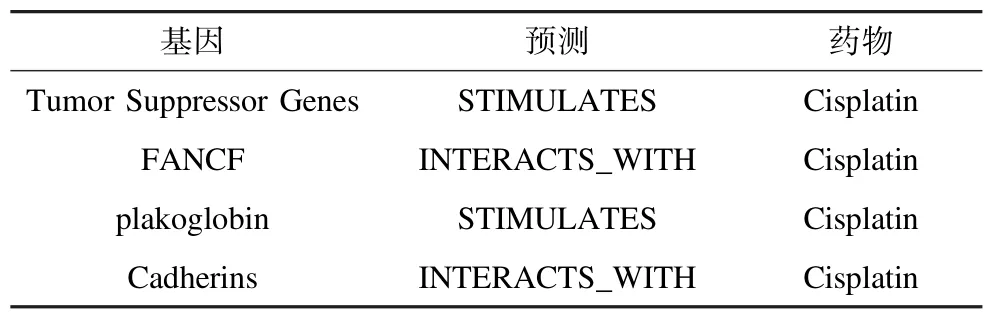
表7 部分基因和药物的关联关系
4 系统描述
4.1 JUNG工具包
系统可视化用到的 JUNG[13](Java Universal Network/Graph framework)是一个Java开源项目,其目的在于为开发关于图或网络结构的应用程序提供一个易用、通用的基础架构。在系统实现过程中,使用JUNG功能调用,可以方便地构造图或网络的数据结构。应用经典算法如聚类、最短路径、最大流量等,编写和测试用户自己的算法,以及可视化的显示数据的网络图。
4.2 系统界面
图2中的网络图是以疾病Carcinoma of bladder为中心的疾病和基因、药物的关联信息。
浅色的结点表示的是跟疾病相关的基因,深色的结点表示的是跟疾病相关的药物。同时,部分基因和药物的关联关系也在图中展示出。
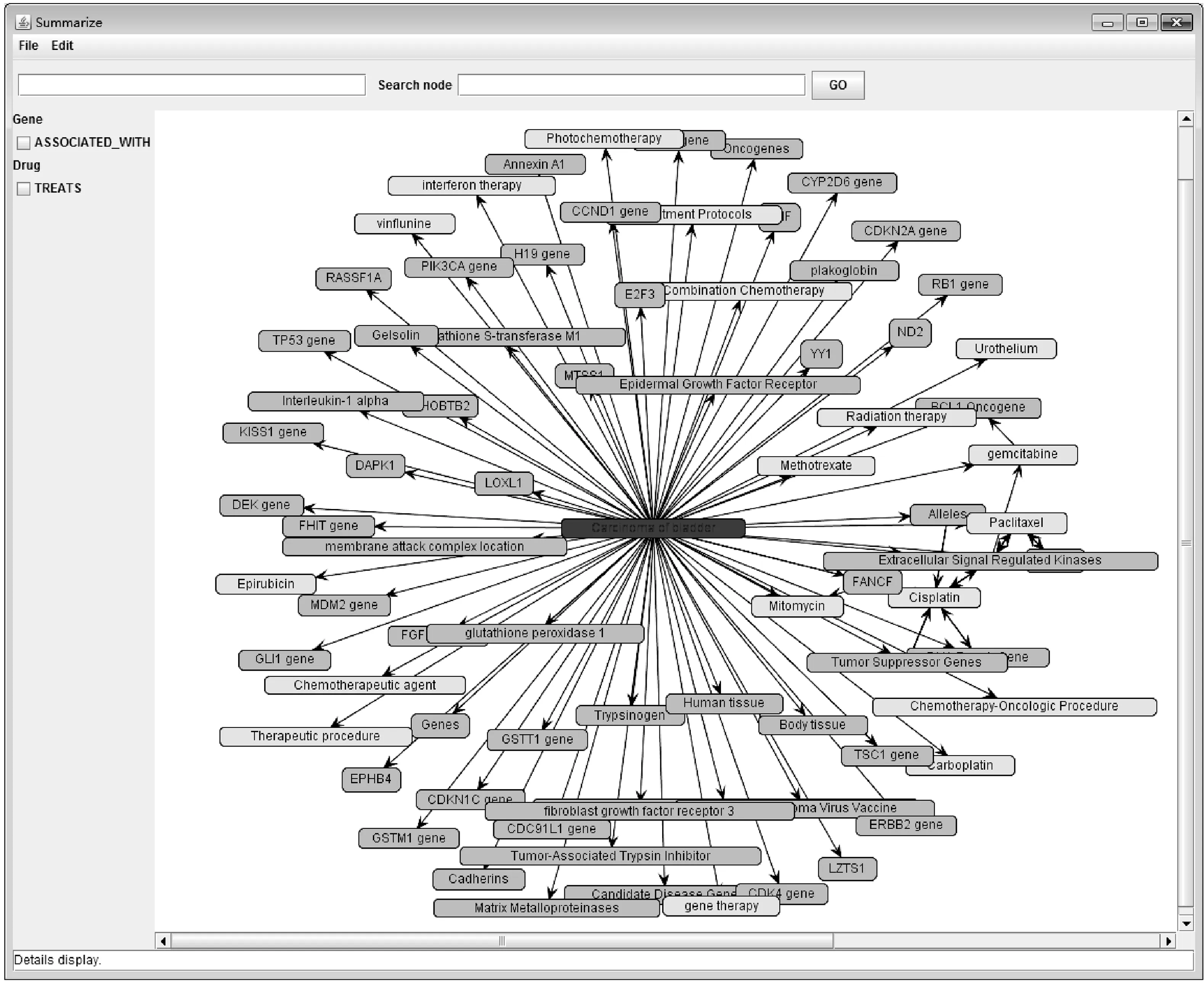
图2 系统初始化显示及结点详细信息显示
对于整个网络图,编辑栏可以选择整体移动(TRANSFORMING)和部分选取(PICKING)功能。在选择(PICKING)功能,选择图中的任何一个结点,在底部面板的Details display栏显示该结点的详细信息,包括实体所在的PubMed文档号和包含该实体的句子。在Search node搜索框,输入一个疾病,便可手动检索疾病,并将该结点移至面板中心,在底部显示该结点的详细信息。左边的复选框用于单独显示某个模块、关系的单独子图。例如,选择Gene模块中的ASSOCIATED_WITH就可以单独显示与疾病相关的基因,这些基因跟疾病之间的谓词关系为ASSOCIATED_WITH。单个关系的子图可以更方便用户找到与疾病有显著关系的基因和药物,有针对性地对得到的关系进行分析,提高生物医学工作者的查询效率。
5 结束语
信息提取在生物医学领域发展迅速,信息时代的科技发展需要高效的工具作为辅助。本文在提出信息提取算法的基础上,以疾病为中心,将疾病、基因和药物三者信息集成在可视化系统中。该系统有利于医学工作者快速了解跟疾病相关的基因信息,并能根据得到的药物信息对病情进行有效的分析和诊断。在算法方面,结果的准确率还有欠缺,下一步将研究改进方向并应用到信息提取中,完善系统功能。
[1] Hersh W R,Hickam D H.How Well Do Physicians Use Electronic Information Retrieval Systems?[J].The Journal of the American Medical Association,1998, 280(15):1347-1352.
[2] Kilicoglu H,Fiszman M,Rodriguez A,et al.Semantic MEDLINE:A WebApplicationforManagingthe Results of Pub Med Searches[C]//Proceedings of the 3rd International Symposium for Semantic Mining in Biomedicine.[S.l.]:IEEE Press,2008:69-76.
[3] Workman T E,Hurdle J F.Dynamic Summarization of Bibliographic-based Data[J].BMC Medical Informatics and Decision Making,2011,11(1).
[4] Workman T E,Fiszman M,Hurdle J F.Text Summarization as a Decision Support Aid[J].BMC Medical Informatics and Decision Making,2012,12(1).
[5] Fraser C,Murray A,Burr J.Identifying Observational Studies of Surgical Interventions in Medline and Embase[J].BMC Medical Research Methodology,2006,6(1).
[6] 廖 涛,刘宗田,王 利.多主题文本摘要抽取的研究与实现[J].计算机工程,2011,37(6):21-23.
[7] Rindflesch T C,Fiszman M,Libbus B.Semantic Interpretation for the Biomedical Research Lite-rature[M].[S.l.]: Springer,2005.
[8] Fiszman M,Rindflesch T C,Kilicoglu H.Abstraction Summarization for Managing the Biomedical Research Literature[C]//Proceedings of Workshop on Computational Lexical Semantics.[S.l.]:Springer,2004:76-83.
[9] 商 玥,林鸿飞,杨志豪.利用语义关系抽取生成生物医学文摘的算法[J].计算机科学与探索,2011, 5(11):1027-1036.
[10] Kullback S,Leibler R A.On Information and Sufficiency[J].The Annals of Mathematical Statistics, 1951,22(1):79-86.
[11] Riloff E.Automatically Generating Extraction Patterns from Untagged Text[C]//Proceedings of National Conference on Artificial Intelligence.[S.l.]:Springer, 1996:1044-1049.
[12] Ahlers C B,Fiszman M,Demner F D,et al.Extracting Semantic Predications from Medline Citations for Pharmacogenomics[C]//Proceedings of Pacific Symposium on Biocomputing.[S.l.]:Springer,2006:209-210.
[13] O’Madadhain J,Fisher D,White S,et al.The Jung(Java Universal Network/Graph)Framework[D].Irvine, USA:University of California,2003.
编辑 顾逸斐
Disease Knowledge Extraction System Based on Semantic Relation
WU Xiaofang,YANG Zhihao,LIN Hongfei,WANG Jian
(School of Computer Science and Technology,Dalian University of Technology,Dalian 116024,China)
In the biomedical field,knowledge summarization can greatly promote the innovation of biomedical science and technology.Dynamic summarization can provide novel clinical experimental hypothesis by extracting the links among diseases,genes,drugs from the mass of biomedical literature and visualizing it.This paper presents a system which summarizes the salient relations by the salient extraction algorithm using the specific subject Medline corpus by SemRep semantic output.Experimental results show that the precise of experimental result is 0.76 referring to OMIM and GHR online databases.
knowledge extraction;semantic relation extraction;significant information extraction algorithm;SemRep tool;semantic output;network diagram visualization
1000-3428(2015)01-0284-05
A
TP311
10.3969/j.issn.1000-3428.2015.01.054
国家自然科学基金资助项目(61070098,61272373,61340020);中央高校基本科研业务费专项基金资助项目(DUT13JB09);国家社会科学基金资助项目(08BTQ025)。
吴晓芳(1989-),女,硕士研究生,主研方向:知识发现,文本挖掘;杨志豪,副教授、博士、博士生导师;林鸿飞,教授、博士、博士生导师;王 健,副教授。
2013-12-30
2014-03-14 E-mail:xfwu@mail.dlut.edu.cn
中文引用格式:吴晓芳,杨志豪,林鸿飞,等.基于语义关系的疾病知识提取系统[J].计算机工程,2015,41(1): 284-288.
英文引用格式:Wu Xiaofang,Yang Zhihao,Lin Hongfei,et al.Disease Knowledge Extraction System Based on Semantic Relation[J].Computer Engineering,2015,41(1):284-288.
——论胡好对逻辑谓词的误读

