高内涵筛选技术的原理及其在生态毒理学的应用
黄超,言野,李娜,马梅,王子健
中国科学院生态环境研究中心 中国科学院饮用水科学与技术重点实验室,北京 100085
高内涵筛选技术的原理及其在生态毒理学的应用
黄超,言野,李娜,马梅*,王子健
中国科学院生态环境研究中心 中国科学院饮用水科学与技术重点实验室,北京 100085
大量存在于环境中的有毒污染物仍然缺乏足够的毒理学数据来对其进行有效的监管,为了满足海量化合物毒性评价的需要,基于离体生物测试的高通量毒性筛选方法在近些年得到了迅猛的发展。高内涵筛选技术是新型的高通量化合物毒性筛选方法,该方法最显著的特点是能够在保持细胞结构和功能完整的基础上同时获取多种毒性指标。因此在简介高内涵筛选技术原理的基础上,综述了其在生态毒理学领域已有的应用,并针对性地对高内涵筛选技术的发展和挑战进行了展望。
高内涵筛选;高通量筛选;离体毒性测试;生态毒理学
随着现代工业进步,人类合成、使用和间接产生的化合物的数量和种类在不断增长,其中包括了化工原料、阻燃剂、农药、增塑剂、食品添加剂、药物、天然化合物及衍生物、饮用水消毒副产物和化学合成副产物等多个类别[1-2]。然而由于在对化合物毒性作用方面认识的不足,绝大多数的化合物缺乏有效监管,部分化合物因此能够以直接或间接的方式进入环境,成为环境污染物。事实上,根据美国国家毒理规划处(NTP)生物分子筛选部负责人Tice等[2]在2013年的估计,至少有数万种存在于环境中的污染物仍然缺乏足够的毒理学数据来预测其对人类和生态系统的影响。所以在生态毒理学领域,对这些化合物展开环境危害性和毒理特性鉴定,进行风险管理显得尤为必要而迫切。为此,美国环境保护局(EPA)在2011年已开始推进新一代的健康风险评估(NexGen)项目,NexGen项目强调了体外高通量毒性筛选在揭示化合物毒性作用机制方面的重要性[3],而该项目的实施对中国的毒理学发展具有借鉴意义。
模式动物的活体毒性测试由于通量低、成本高和周期长已不能满足当前巨量化合物毒性评价的需要,为了应对毒理学领域所面临的新挑战,2007年加拿大国家研究委员会(NRC)的“21世纪毒性测试:一种远见与策略”报告[4]为毒理学领域新世纪的发展奠定了基石。该报告不仅提出了21世纪毒性测试应当从活体生物检测向高通量的离体检测转变,更期望采用离体生物检测去揭示化合物毒性作用机制,从而使在这个框架下所进行的风险科学具有充分的科学依据。毒性作用机制的研究是以毒性途径紊乱(perturbations of toxicity pathways)为观察指标[4],这一特点与传统离体或活体生物检测中,以死亡、突变、肿瘤形成等细胞或动物终点事件(apical end points)为观察指标所不同。而所谓的毒性通路在NRC的报告中被定义为:正常细胞受外来化合物干扰后所产生的,能导致损害健康效应的细胞内应答通路[4]。为了对这一观察指标进行全面的衡量,新的高通量生物检测方法仍然有待进一步的发展。
高内涵筛选(High Content Screening, HCS)是新型的高通量化合物毒性检测方法,能够在保持细胞结构和功能完整的基础上,运用多种荧光标记物标记细胞,自动化地对细胞内多靶点的复杂表型(phenotype)进行筛选[5-7]。不同筛选条件下产生的表型特征既包括了完整细胞所表现的凋亡、坏死、增殖、迁移等形式,还包括了细胞内的细胞器损伤、信号转导、代谢途径以及遗传损伤等[8]。不同于传统离体生物检测中以细胞内单靶点的作用为检测端点,HCS以高内涵(high content)的方式呈现化合物暴露下所产生的多维表型信息[9],从而系统的对化合物的毒性作用机制展开研究[10],更好的对化学污染物进行风险评估。
当前随着HCS设备及分析技术的长足的进步,HCS技术已在毒理学、药理学和医学领域有了极大的发展,在生态毒理学领域,HCS的应用亦在积极的展开。本文系统介绍了HCS技术原理,在此基础上总结了HCS在当前生态毒理学领域的应用,并对其发展前景和所面临的挑战进行了展望。
1 HCS技术原理
1.1 HCS概念
光学显微镜设备在自动化程度和图像收集速率的提高促进了HCS的发展,研究人员已可以利用图像分析方法在单细胞水平展开研究。因为HCS以完整细胞为研究对象,细胞结构和功能完整,真实反应了细胞内的毒性作用机制[11]。细胞荧光图像是HCS获取数据的主要手段,特异性荧光探针或荧光蛋白的采用使细胞内不同结构被多种荧光信号同时标记,从而反应细胞表型。能够被HCS检测得到的表型变化除包括标记点的荧光强度改变外,还有细胞或细胞内结构形态变化。图像分析技术使得表型变化的定量分析成为可能,每个细胞的荧光和形态学特征得以被聚类和分类等数据挖掘手段所整合分析,从而转换为可解释的多维细胞表型信息,这种多生物学信息呈现方式拓宽了我们对化合物毒性作用机制的理解和认识。
1.2 HCS实验流程
HCS的试验流程包括了细胞培养、样品制备和暴露、图像获取、图像分析和数据挖掘5个部分[5],同时,基于高性能工作站和服务器建立起来的数据库系统对于HCS数据的存储和调用分析也至关重要[12],以下对HCS过程中几个关键步骤进行相关介绍。
1.3 样品制备和暴露
常规的HCS过程同样依赖于实验员的操作,需要进行细胞培养、化合物暴露和细胞染色等一系列样品前处理过程,这在大规模高通量化合物的HCS中会造成了2方面的影响,一方面384、1536等多孔板的应用增加了实验员的操作难度;另一方面,实验员在操作的过程中容易引入人为误差,整个实验的质量控制难以保证[13]。因此在大规模高通量的HCS中,样品制备和暴露更依赖于自动化的液体处理设备,尽管当前自动化液体处理设备价格较高,但其引入使得实验的整体精度、重复性和可靠性被极大的增强,并满足了大部分液体处理的需要。目前,已有多个生物公司,如Tecan,PerkinElmer等都开发了自己的液体处理设备。
1.4 图像获取
随着近几年显微镜领域的发展,HCS设备已日趋成熟,主要在检测速度、图像质量、共聚焦和厚组织扫描这4个方面有了极大的进步。高速转盘的应用使得HCS设备能够在短时间内扫描多通道的荧光信号[14],降低了检测环境对细胞的影响。新型的HCS设备已采用了基于新一代CMOS图像传感器(CIS)设计的科学级CMOS(sCMOS)。相比于传统的CCD图像传感器,sCMOS不仅能以4倍的视场范围在短时间内完成高质量的图像扫描,更能细致的展现细胞内微观结构[15]。光路系统的改进也增强了HCS设备共聚焦能力,满足了以三维细胞培养为概念的一些新的细胞及组织培养技术的发展要求。表1总结当前厂商所提供的商品化高内涵设备。
1.5 图像分析与数据挖掘
为了从HCS所获得图像中提取所需的生物学信息,需要对其进行图像分析和数据挖掘[16-17]。图像分析的主要目的是对每个细胞都进行定量化分析,主要包括:(1) 细胞及细胞内结构定位;(2) 细胞结构分割;(3) 线性结构提取(针对神经细胞的神经突);(4) 细胞追踪(针对活细胞检测);(5) 特征提取共5个部分[16]。经过图像分析,得到了与图像信息关联的单细胞特征,包括了荧光强度、形态、表面纹理(texture)和位置等多维数据。将多维数据正态化后再对其进行数据挖掘,最广泛的应用是聚类和分类算法,目的是将不同处理下所呈现不同表型特征的细胞归入所属类型[17],例如根据DNA含量与细胞形态对处于不同细胞周期下的细胞进行划分[18-19]。图像分析和数据挖掘是HCS研究的热点,只有通过该过程才能阐述图像背后的生物学问题。商品化的HCS分析软件的使用极大的降低了HCS应用的难度,是HCS图像分析和数据处理过程中的主流策略,但其使用成本较高,通用性相对较差。现也有一些较为成熟的高内涵分析开源软件,如由美国哈佛-麻省理工的博德研究所主导开发的cellprofiler[20],可以通过模块化定制来对HCS图像进行分析,分析得到的数据可以进一步采用基于随机森林数的机器学习方法来进行分选。除此之外,结合R[21-22]和Weka[23]等数据分析软件,来对HCS图像分析经过进行数据处理、数据挖掘和数据可视化的应用也越来越多。
1.6 生物数据库系统
Keefer等估计,一个HCS平台每年数据产出量在500 GB~6 TB左右[10]。如此高的数据产出,不仅要求在数据分析过程中采用并行甚至集群计算,数据的储存和调用也依赖于服务器。但是,几乎每个HCS设备提供商都以各自格式储存数据,这导致显微实验中图像格式冗余,制约了HCS数据库的管理和维护[12]。OME以增强HCS分析软件之间的互通性为目的而开发的“Bio-format”已得到各个软硬件的广泛支持,使HCS中统一的数据管理成为可能[24]。HCS的数据库系统除了有传统的商业数据库Oracle[25],MDCStoreTM和ColumbusTM等,HCS开源数据库SIDB[26]和OME的OMERO[27]也有了广泛的应用。
2 HCS在生态毒理学的应用
对环境中有毒有害化合物进行毒性筛查,分析其毒性作用机制是化学物质风险评估中必要的过程,也正是生态毒理学领域研究的重点。而HCS最大的优势是在一次实验中对观测对象,如离体培养细胞,进行多维表型分析,因此也可以同时得到多组反应化合物毒性的指标[9]。荧光图像中毒性指标的提取依赖于图像分析和数据挖掘,该过程的目的是对荧光图像中的对象进行分割和分类。所以HCS的毒性指标既来源于荧光标记靶点所在位置和荧光强度,也能在细胞和亚细胞的形态结构上反应[9]。正因如此,HCS对于其他毒性评价方法中难表征的毒性指标,如凋亡小体的形成,信号通路中转录因子的核转位,细胞趋化和集落形成等也能定量化的测定。Zock[28]已在其综述中对HCS所能得到的毒性指标进行了较为完整的综述,总结在表2中。由于HCS中毒性指标的多产出,需要对其并行分析,这已在生态毒理学领域展开了众多实际的应用。而在HCS应用较为成熟的药物筛查过程中,其在化合物作用机制的研究也对生态毒理学领域的应用具有借鉴意义。
2.1 遗传毒性
遗传毒性用于描述某化合物能够直接或间接的方式引起DNA或染色体损伤的性质,具有这种性质的化合物被称为遗传毒物[29]。工业污染使众多潜在的遗传毒物进入环境介质,而遗传毒物对生物胚胎细胞和体细胞的损害可能引起多种疾病,包括了新生儿畸形和癌症生成等[30]。因此为了对这些化合物进行监管,需要对其遗传毒性定量测定,而相比于传统遗传毒性评价方法,HCS在具有较高的灵敏度和特异性同时,能够高通量的检测多个遗传毒性指标,因此应用广泛。
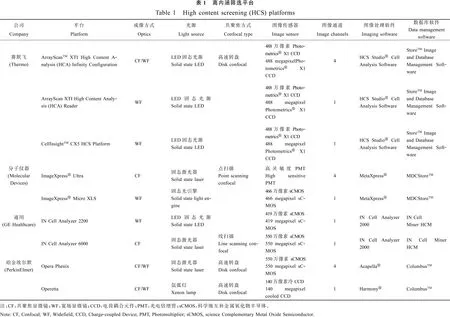
表2 HCS主要的应用领域和研究对象[28]

应用领域Feild研究对象Object应用领域Feild研究对象Object应用领域Feild研究对象Object细胞信号通路Cellsignalingpathways靶点验证TargetvalidationNF-κBSTATWnt/fzdPKBNFATP38TGF-betaSmad2/3GPCR糖皮质激素受体(Glucocorticoidreceptor)FOXO1α酵母(yeast)shRNA干扰文库(shRNAinterferencelibrary)siRNA干扰文库(siRNAinterferencelibrary)干细胞自我更新(Stemcellself-renewal)干细胞分化(Stemcelldifferentiation)细胞生理学Cellphysiology增殖(Proliferation)磷酸化(Phosphorylation)吞噬作用(Phagocytosis)自噬作用(Autophagy)间隙连接诱导(Gapjunctioninduction)线粒体健康(Mitochondrialhealth)核形态(Nuclearmorphology)凋亡(Apoptosis)细胞膜通透性(Membranepermeability)细胞周期(Cellcycle)细胞迁移(Motility/migration)细胞骨架重拍(Cytoskeletalrearrangement)神经轴突生长(Neuiteoutgrowth)转化(Transformation)机体生理学Organismphysiology离体毒性测试(InVitroToxicology)心脏衰竭(Cardiacfailure)生物节律(Circadianrhythms)免疫抑制(Immunosupression)骨质疏松(Osteoporosis)真菌致病性(Fungalpathogensis)病毒中和(Virusneutralization)寄生物感染(Parasiteinfection)血管生成(Angiogenesis)阿兹海默综合征(Alzheimer'sdisease)帕金森综合征(Parkinson'sdisease)亨廷顿综合症(Huntington'sdisease)肌萎缩性侧素硬化(Amyotrophiclateralsclerosis)微核生成(Micronucleusinduction)磷脂沉积(Phospholipidosis)神经毒性(Neurotoxicity)细胞器健康(Organellehealth)肝毒性(Hepatotoxicity)
注:NF-κB,核因子活化B细胞κ轻链增强子;STAT信号转导及转录激活蛋白;Wnt/fzd,Wnt/卷曲受体;PKB,蛋白激酶B;NFAT,激活T细胞核因子;p38,促分裂原活化蛋白激酶;TGF-beta,转化生长因子β;Smad2/3,母亲DPP同源物2;GPCR,G蛋白偶联受体;FOXO1α,叉头基因O1。
Note: NF-κB, nuclear factor kappa-light-chain-enhancer of activated B cells; STAT, signal transducer and activator of transcription; Wnt/fzd, Wnt/frizzled; PKB, protein kinase B; NFAT, nuclear factor of activated T-cells; p38, p38 mitogen-activated protein kinases; TGF-beta, transforming growth factor beta; Smad2/3, mothers against decapentaplegic homolog 2; GPCR, G protein-coupled receptor; FOXO1α, Forkhead box O1α.
2.1.1 细胞周期阻滞
生物的生长和繁衍依赖于遗传信息在细胞中准确而稳定的复制,为了满足遗传信息在复制过程中的高保真度,细胞进化拥有了一套高效的监控机制—DNA损伤应答(DNA Damage Response, DDR)系统[31]。DDR信号通路与多个细胞内信号通路相互关联,其中,一旦DNA发生了结构性损伤,DDR信号通路被激活,可能根据不同的损伤类型和时间点首先将细胞阻滞于不同的细胞周期阶段,主要是G1、S和G2期[32],并在这些阶段中尝试对DNA损伤进行修复,因此细胞周期阻滞与DNA损伤具有较强的关联性。HCS不仅能够进行DNA含量分析[18],采用数学模型来对细胞周期进行划分,还能充分利用细胞形态特征,采用机器学习的方法来判别更为细致的细胞周期状态[33]。另外多种荧光探针也能在HCS被用来表征不同的细胞周期状态,例如BrdU[34]和Edu[35]是HCS过程常用的S期荧光标记物[19,33],它们分别是胸腺嘧啶核苷衍生物和类似物,能够代替胸腺嘧啶(T)渗入进S期细胞的DNA分子中,并通过荧光标记识别。
2.1.2 特定DNA损伤检测
DNA损伤具有多种类别,包括了氧化性DNA损伤、DNA加合物、DNA甲基化、双链DNA损伤等[29],其中8-OHdG[36]、DNA甲基化结合蛋白[37]和磷酸化组蛋白H2AX(γH2AX)[38]分别是氧化性DNA损伤、DNA甲基化和双链DNA损伤特异性标志物。采用免疫荧光方法,能标记特定的DNA损伤标志物,并通过HCS高通量来对化合物DNA损伤类型进行判定。Barber[39]和Kim等[40]以抗体标记γH2AX,并采用HCS来对生成的γH2AX foci进行定量分析,通过该方法可以对遗传毒物的作用机制有一定了解。
2.1.3 微核试验
微核实验是在细胞核水平对遗传毒性进行评价,微核的生成存在2种机制,一种是由遗传毒物干扰细胞分裂器所引起的[41],这使得生成的绝大部分微核中含有着丝点或着丝粒,这类遗传毒物被称为非整倍体断裂剂,能间接的造成遗传毒性。另一种微核生成是由高水平的DNA损伤引起的[32],这种机制与Cyclin B1浓度含量有关[42]。微核生成前,细胞核内已发生严重的基因突变和染色体损伤断裂,然而过低的Cyclin B1浓度使得纺锤体组装检验点(spindle assembly checkpoint, SAC)并不会被激活,因此DNA严重损伤的细胞被迅速退出M期,导致部分脱核的染色体形成微核[42-45]。基于这一机制生成微核的遗传毒物被称为染色体断裂剂[41],其能直接的对DNA造成高水平的损伤。这2种微核生成的机制是细胞微核实验的基础。
通过图像特征提取算法的优化,HCS可以自动化高通量的定位存在于细胞质中的微核,微核的生成率是遗传毒性可靠的衡量指标。例如,Yan等[46]通过HCS对9种苯并噻唑类化合物进行了遗传毒性评价,其中以4-NQQ和B(α)P为阳性对照,确保了实验的灵敏度,同时细胞急性毒性采用细胞减少率表示并在实验中加以控制。在此基础上对9种苯并噻唑类暴露MGC803和A549后微核的生成率进行衡量,HCS的检测结果表明2-氨基苯并噻唑(ABT)、2-羟基苯并噻唑(OHBT)、2-氯苯并噻唑(CBT)、2-溴苯并噻唑(BrBT)和2-甲硫基苯并噻唑(MTBT)的微核率能够呈现出剂量响应关系。HCS也能根据上述2种微核生成机制,利用微核形态区分非整倍体毒剂和染色体断裂剂。Kiyohiro等[41]发现通过区分微核的大小可以和FISH方法一样有效的区分非整倍体毒剂和染色体断裂剂,因为非整倍体毒剂所造成的微核相比于染色体断裂剂的尺寸更大。Walter等[47]又通过HCS分析,以MMS和紫杉醇为指标化合物,确定了不同机制下产生的2种微核面积区分阀值,并采用这一阀值去测定了62种指标化合物,该方法不仅检测过程更为简单快速,且具有较高的灵敏度和特异性。
2.2 器官毒性评价
生物在环境中所暴露的化合物会对体内特定的器官产生各异的毒性作用,对化合物展开特定器官毒性评价能够发现该化合物在体内所影响的主要靶器官,部分揭示其所具有的毒性作用机制。同时随着毒理学研究的进步,替代动物实验的体外模型研究是发展主流[2]。利用HCS能够高通量对不同器官来源的细胞系和原代细胞进行多参数高灵敏的毒性评价,从而使器官毒性评价的方式从活体实验向离体实验转变。以肝毒性为例,肝是外源化合物主要的代谢器官,外源化合物进入体内后首先被哺乳动物肝脏中I相和II相代谢酶代谢,尽管对于大多数化合物来说,代谢过程是一个解毒的过程[48],但有些化合物在代谢后会产生毒性更强的代谢产物,例如B(α)P[49]。因此在毒理学中对化合物肝毒性评价是研究的重点,肝损伤的类型包括肝细胞的坏死、凋亡、致癌作用和氧化应激等[48]。HepG2是在离体细胞实验中评价肝毒性常用的细胞系,属于人源肝癌上皮细胞[50]。O'Brien等[51]采用HepG2对243种来源于药品的化合物进行HCS,以评估该方法对肝毒性评价的灵敏度和特异性。细胞采用4种染料分别标记了细胞内钙离子浓度、线粒体膜电和细胞膜通透性和DNA含量,结果表明,相比另外7种在离体实验中常用的细胞毒性评价手段,基于HCS肝毒性评价方法不仅具有类似的特异性,灵敏度也高达93%。通过机器学习分析HCS数据,Edward等[52]只通过PI和hoechst对细胞染色,即根据细胞核形态和细胞膜完整性这2个方面的参数,采用有监督的分类方法来自动区分了健康、凋亡和坏死细胞,从而评价了金属纳米材料所能导致的肝毒性。除了HepG2细胞,其他细胞系和原代培养细胞也广泛的应用于化合物肝毒性评价。而对于其他器官毒性的评价,如肾[53]和心脏[54]毒性等,HCS也应用广泛。
2.3 神经毒性评价
神经毒性适用于描述毒性化合物对神经系统的结构和功能造成不良影响的能力[55]。众多疾病,如阿尔茨海默病[56],都是由于神经系统的损伤而引起的。已有多种环境污染物被发现对神经系统会造成损伤,尽管神经毒性也是器官毒性的一种表现,但是其毒性指标的获取过程更为复杂,而采用离体神经细胞结合HCS来评价化合物的神经毒性已是一种有效的毒性测试手段。评价的指标包括了细胞毒性、神经细胞间信号传递、神经纤维生长和突触形成。Joseph等[57]采用来源于人神经前体细胞系ReNcell CX细胞结合HCS表明,结合神经细胞增殖和活力指标可以用来评价化合物的神经毒性。Nicholas等[57]也发现HCS能高效的提取和分析神经突的线性结构,化合物的神经毒性大小能以对神经突生长抑制的能力和影响细胞活力的大小来进行衡量。随着HCS图形分析技术的提高,更为细致的神经细胞结构能够被提取和分析。Joshua等[58]确认了在正常培养的啮齿动物原代培养的混合皮质细胞中,抗体标记的点状突触蛋白在树突处具有更高的密度,表明突触主要形成于该区域。而神经毒性化合物暴露后可以通过HCS来自动的判断在树突处突触的减少量,结合神经细胞其他的形态特征,这些参数能够有效的来判断化合物的神经毒性。
化合物在完整的生命体中的神经毒性作用也能利用HCS进行高通量的评价。Tara等[59]采用EGFP标记斑马鱼,评价了斑马鱼胚胎在受精后19 h到29 h内会产生自发性尾部收缩现象。这种现象被认为是斑马鱼胚胎的第一次肌动活动,潜在的发育神经毒性化合物能够显著的干扰该现象的产生。来源于EPA的ToxCast一期化学品文库中的16种化合物在斑马鱼胚胎受精后5 h到25 h内进行静态暴露,再用HCS设备在随后大约1.4 h的视频获取时间内对不同化合物暴露浓度下的自发性尾部收缩现象进行统计,结果表明阿维菌素(abamectin)和埃玛菌素(emamectin benzoate)的暴露下,斑马鱼胚胎虽然没有严重的畸形,但完全丧失自发性尾部收缩能力,从而通过HCS可以间接证明这两种化合物具有发育神经毒性。
2.4 化合物作用机制
当前,HCS在对海量化合物作用机制(mode of action, MoA)的研究已经极大的促进了药物筛选的发展[5],尽管药物筛查和生态毒理学这2个领域中对化合物的毒性测试的出发点和目的性都不尽相同,但是药筛过程中利用HCS展开海量化合物的MoA研究方法对生态毒理学中HCS的应用提供了有益的参考。药物筛查的目的是在药物研发的初期,在大型化合物文库(chemical library)中寻找针对特定药物作用靶点(target)具有生物活性的化合物。而对于同一靶点有效的化合物也可能会发生脱靶效应(off-target effect),即化合物会同时对特定靶点以外的体内生物大分子结合,这可能导致该化合物在药物开发后期被发现具有严重的副作用而造成巨大经济损失[60-62]。正因如此在药物筛查的过程中应尽早发现化合物的脱靶效应,所以需要在这一过程中对化合物的MoA展开研究[63]。尽管生物组学技术的发展,如基因芯片、转录组测序等已能够有效的寻找化合物的MoA,但是高昂的成本使其难以在大规模的药物筛查中展开,而利用HCS对产生的细胞多维表型进行挖掘,能够了解化合物的MoA,寻找到目标化合物[33]。
药物筛查过程中,HCS对MoA的研究主要通过2个方向来进行,一是对细胞内信号转导通路中关键的蛋白进行荧光标记,通过对该蛋白的表达和转位来直接了解暴露化合物的MoA。例如对于核转录因子-κB(NF-κB)[64-65],NF-κB的核转位是调控细胞生命活动的一个重要的分子途径,也是药物筛查中一个重要的靶点。类似的HCS筛查也对细胞骨架[66-67]、蛋白酶体[68]和细胞凋亡相关蛋白[66]等展开。另一个方向是结合计算毒理学来对HCS的多维细胞表型筛选来进行化合物MoA的研究。Wawera等[69]认为HCS中所得到的表型是一种无偏的,多维度的“通用性语言(univeral language)”,可以最大限度的反应生物活性,有利于化合物的MoA研究。Young等[33]在这一个方向上展开了一些探索性的工作,结合了HCS的表型分析和配体-预测模型来对化合物的作用机制进行了研究,结果揭示HCS所得到的细胞表型更应该去预测化合物的作用靶点,而不是去反映化合物的结构特征。得益于生物数据的积累和计算机水平的进步,在此方向上对化合物的MoA研究能在更大的药物筛查规模上进行,近些年来已有众多的MoA分析方法被建立和应用[63,69]。
2.5 RNA干扰和cDNA过表达
RNA干扰[70]和cDNA过表达[71]技术的应用使我们得以以“loss of fuction”和“gain of function”的遗传学手段来研究化合物的MoA,在药理学领域提出了化学遗传学(chemical genetic)的概念[72],其目的是对化合物库中的小分子化合物进行筛查来研究这些化合物对生物大分子和信号转导通路的影响,也是为了发现新的药物作用靶点。HCS能够同时对基因表达变化和化合物处理所引起的细胞表型的微小改变做出响应,化学遗传学中通常采用RNA干扰文库和化合物文库并行HCS(parallel HCS)[66,73],来探索两者细胞表型之间的关系。两种文库中筛选产生的细胞表型首先被聚类,在同一个聚类类别中,筛选出的化合物可能和这个类别下所沉默的基因导致相同的表型,因此可以认为这些化合物能够干扰该沉默基因原本的表达,从而找到潜在的药物靶标和生物标记物。
在生态毒理学领域,更多的应用是通过RNA干扰和cDNA过表达技术来构建基因低表达和过表达系统,HCS也能灵敏的检测到构建前后的表型差异。例如,尽管HepG2细胞被广泛的应用于肝毒性检测,但是多种CYPs在这种细胞模型中表达量较低[74],不利于模拟体内肝脏的代谢功能, Tolosa等[75]利用cDNA过表达技术在HepG2细胞中过表达多个CYPs,该细胞模型与原代肝细胞中CYPs表达量相类似,因此显著提高了HCS对肝毒性检测的灵敏度。
3 展 望
当前HCS已发展到一个新的阶段,性能日益强大的设备拓展了HCS在生态毒理学的应用,但HCS并不仅仅依赖于设备本身,更需要利用海量数据去回答亟待解决的生物学问题,因此数据库系统和数据挖掘已成为了HCS应用所要着力加强的地方。
在面对从活体向离体毒性实验转换的过程中出现的新问题,一些新的生物学技术也应当在HCS中得到进一步的应用。例如三维细胞培养技术,因为尽管单层细胞培养具有众多优点,但是单层细胞不能很好的模拟细胞在体内真实的生理状态,缺乏细胞间通讯机制,培养周期短且只适合单种细胞类型细胞培养[76]。在药物毒理学领域,已有多项证据表明了这种差异性对特定器官毒性评价时会产生不利影响[77]。例如曲伐沙星(trovafloxacin)在临床使用过程中首先被发现有很强的肝毒性[78],而在对人源肝细胞(hepatocyte)药物筛查期间并没有明显的毒性作用[79],最近的研究表明,可能是体内肝脏细胞与单层细胞培养模型间的差异造成的[80]。而三维细胞培养模型[81]能够部分弥补单层细胞培养模型的不足,模拟更真实的体内细胞生长环境。得益于HCS设备性能的提高,利用HCS基于三维培养细胞对化合物进行毒性评价也展开了探索性的研究[82]
HCS在生态毒理学的应用仍然需要结合其他学科的发展,例如结合生物组学和计算毒理学的相关应用,来研究海量化合物的毒性作用机制。因为在当前化合物的毒性评价中,对其毒性作用机制的研究已越来越为重要,作用机制的研究能够提供从化合物暴露到毒性效应之间一系列有证据支持的、具有因果关系的关键事件(key events),从而提高评价效率,减少由毒性资料外推到人的不确定因素[4,83],来更好的对化合物的生产和使用展开监管。
[1] Schmidt C W. TOX 21: New dimensions of toxicity testing [J]. Environmental Health Perspectives, 2009, 117(8): A348-A353
[2] Tice R R, Austin C P, Kavlock R J, et al. Improving the human hazard characterization of chemicals: A Tox21 update [J]. Environmental Health Perspectives, 2013, 121(7): 756-765
[3] Krewski D, Westphal M, Andersen M E, et al. A Framework for the next generation of risk science [J]. Environmental Health Perspectives, 2014, 122(8): 796-805
[4] Krewski D, Acosta Jr D, Andersen M, et al. Toxicity testing in the 21st Century: A vision and a strategy [J]. Journal of Toxicology and Environmental Health, Part B, 2010, 13(2-4): 51-138
[5] Zanella F, Lorens J B, Link W. High content screening: Seeing is believing [J]. Trends in Biotechnology, 2010, 28(5): 237-245
[6] Taylor D, Giuliano K A. High Content Screening [M]. Wiley Online Library, 2007: 1-39
[7] Comley J. High content screening [J]. Drug Discovery World, 2005, 6(3): 31-53
[8] Zheng W, Thorne N, Mckew J C. Phenotypic screens as a renewed approach for drug discovery [J]. Drug Discovery Today, 2013, 18(21): 1067-1073
[9] Singh S, Carpenter A E, Genovesio A. Increasing the content of high-content screening: An overview [J]. Journal of Biomolecular Screening, 2014, 19(5): 640-650
[10] Keefer S, Zock J. Approaching high content screening and analysis: Practical advice for users [M] // High Content Screening: Science, Techniques and Applications, 2008: 1-24, DOI: 10.1002/9780470229866.ch1
[11] Starkuviene V, Pepperkok R. The potential of high-content high-throughput microscopy in drug discovery [J]. British Journal of Pharmacology, 2007, 152(1): 62-71
[12] Schiffmann D A, Dikovskaya D, Appleton P L, et al. Open microscopy environment and findspots: Integrating image informatics with quantitative multidimensional image analysis [J]. BioTechniques, 2006, 41(2): 199-208
[13] Huang D, Ou B, Hampsch-Woodill M, et al. High-throughput assay of oxygen radical absorbance capacity (ORAC) using a multichannel liquid handling system coupled with a microplate fluorescence reader in 96-well format [J]. Journal of Agricultural Food Chemistry, 2002, 50(16): 4437-4444
[14] Wang E, Babbey C, Dunn K. Performance comparison between the high-speed Yokogawa spinning disc confocal system and single-point scanning confocal systems [J]. Journal of Microscopy, 2005, 218(2): 148-159
[15] Coates C. New sCMOS vs. current microscopy cameras [J]. Biophotonics International, 2011, 18(5): 24-27
[16] Zhou X, Wong S T. A primer on image informatics of high content screening [M]// High Content Screening: Science, Techniques and Applications, 2008: 43-84, DOI: 10.1002/9780470229866.ch3
[17] Zhou X, Wong S T. Informatics challenges of high-throughput microscopy [J]. Signal Processing Magazine, IEEE, 2006, 23(3): 63-72
[18] Lee J A, Cox K, Kriauciunas A, et al. The roles of high content cellular imaging in lead optimization [M]// High Content Screening: Science, Techniques and Applications, 2008: 249-268, DOI: 10.1002/9780470229866.ch11
[19] Lyman S K, Crawley S C, Gong R, et al. High-content, high-throughput analysis of cell cycle perturbations induced by the HSP90 inhibitor XL888 [J]. PloS one, 2011, 6(3): e17692
[20] Carpenter A E, Jones T R, Lamprecht M R, et al. CellProfiler: Image analysis software for identifying and quantifying cell phenotypes [J]. Genome Biology, 2006, 7(10): R100
[21] Team R C. R: A Language and Environment for Statistical Computing [M]. R Foundation for Statistical Computing, 2012: 1-3551
[22] Liu C. OperaMate: An R package of Data Importing, Processing and Analysis for Opera High Content Screening System. R package version 0.99.6 [CP]. Bioconductor, 2015
[23] Horvath P, Wild T, Kutay U, et al. Machine learning improves the precision and robustness of High-Content Screens using nonlinear multiparametric methods to analyze screening results [J]. Journal of Biomolecular Screening, 2011, 16(9): 1059-1067
[24] Goldberg I. Open microscopy environment [C]// Computational Systems Bioinformatics Conference, 2005, Workshops and Poster Abstracts, IEEE
[25] Lee S, Howell B J. High-content screening: Emerging hardware and software technologies [J]. Methods in Enzymology, 2006, 414: 468-483
[26] Stuurman N, Swedlow J R. Software tools, data structures, and interfaces for microscope imaging [J]. Cold Spring Harbor Protocols, 2012, 2012(1): 50-61
[27] Allan C, Burel J M, Moore J, et al. OMERO: Flexible, model-driven data management for experimental biology [J]. Nature Methods, 2012, 9(3): 245-253
[28] Zock J M. Applications of high content screening in life science research [J]. Combinatorial Chemistry & High Throughput Screening, 2009, 12(9): 870-876
[29] Phillips D H, Arlt V M. Genotoxicity: Damage to DNA and its consequences [J]. Molecular, Clinical and Environmental Toxicology, 2009, 99: 87-110
[30] Cooke M S, Evans M D, Dizdaroglu M, et al. Oxidative DNA damage: Mechanisms, mutation, and disease [J]. The FASEB Journal, 2003, 17(10): 1195-1214
[31] Zhou B B S, Elledge S J. The DNA damage response: Putting checkpoints in perspective [J]. Nature, 2000, 408(6811): 433-439
[32] Heijink A M, Krajewska M, Van Vugt M A. The DNA damage response during mitosis [J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 2013, 750(1-2): 45-55
[33] Young D W, Bender A, Hoyt J, et al. Integrating high-content screening and ligand-target prediction to identify mechanism of action [J]. Nature Chemical Biology, 2007, 4(1): 59-68
[34] Eldridge S R, Tilbury L F, Goldsworthy T L, et al. Measurement of chemically induced cell proliferation in rodent liver and kidney: A comparison of 5-bromo-2′-deoxyuridine and [3H] thymidine administered by injection or osmotic pump [J]. Carcinogenesis, 1990, 11(12): 2245-2251
[35] Buck S B, Bradford J, Gee K R, et al. Detection of S-phase cell cycle progression using 5-ethynyl-2'-deoxyuridine incorporation with click chemistry, an alternative to using 5-bromo-2'-deoxyuridine antibodies [J]. Biotechniques, 2008, 44(7): 927-929
[36] Wu L L, Chiou C-C, Chang P-Y, et al. Urinary 8-OHdG: A marker of oxidative stress to DNA and a risk factor for cancer, atherosclerosis and diabetics [J]. Clinica Chimica Acta, 2004, 339(1-2): 1-9
[37] Fuks F, Hurd P J, Wolf D, et al. The methyl-CpG-binding protein MeCP2 links DNA methylation to histone methylation [J]. Journal of Biological Chemistry, 2003, 278(6): 4035-4040
[38] Bonner W M, Redon C E, Dickey J S, et al. γH2AX and cancer [J]. Nature Reviews Cancer, 2008, 8(12): 957-967
[39] Barber P, Locke R, Pierce G, et al. Gamma-H2AX foci counting: Image processing and control software for high-content screening [C]. Progress in Biomedical Optics and Imaging - Proceedings of SPIE, 6441, 2007
[40] Kim S, Jun D H, Kim H J, et al. Development of a high-content screening method for chemicals modulating DNA damage response [J]. Journal of Biomolecular Screening, 2011, 16(2): 259-265
[41] Aardema M, Albertini S, Arni P, et al. Aneuploidy: A report of an ECETOC task force [J]. Mutation Research:Reviews in Mutation Research, 1998, 410(1): 3-79
[42] Gascoigne K E, Taylor S S. Cancer cells display profound intra-and interline variation following prolonged exposure to antimitotic drugs [J]. Cancer Cell, 2008, 14(2): 111-122
[43] Crasta K, Ganem N J, Dagher R, et al. DNA breaks and chromosome pulverization from errors in mitosis [J]. Nature, 2012, 482(7383): 53-58
[44] Dotiwala F, Harrison J C, Jain S, et al. Mad2 prolongs DNA damage checkpoint arrest caused by a double-strand break via a centromere-dependent mechanism [J]. Current Biology, 2010, 20(4): 328-332
[45] Nitta M, Kobayashi O, Honda S, et al. Spindle checkpoint function is required for mitotic catastrophe induced by DNA-damaging agents [J]. Oncogene, 2004, 23(39): 6548-6558
[46] Yan Y, Jiang W W, Li N, et al. Application of the SOS/umu test and high‐content in vitro micronucleus test to determine genotoxicity and cytotoxicity of nine benzothiazoles [J]. Journal of Applied Toxicology, 2014, 34(12): 1400-1408
[47] Westerink W, Schirris T J, Horbach G J, et al. Development and validation of a high-content screening in vitro micronucleus assay in CHO-k1 and HepG2 cells [J]. Mutation Research/Genetic Toxicology and Environmental Mutagenesis, 2011, 724(1-2): 7-21
[48] Hodgson E, Levi P E. A Textbook of Modern Toxicology [M]. Wiley Online Library, 2004: 111-148
[49] Buening M, Wislocki P, Levin W, et al. Tumorigenicity of the optical enantiomers of the diastereomeric benzo[a]pyrene 7,8-diol-9,10-epoxides in newborn mice: Exceptional activity of (+)-7beta, 8alpha-dihydroxy-9alpha, 10alpha-epoxy-7,8,9,10-tetrahydrobenzo[a]pyrene [J]. Proceedings of the National Academy of Sciences, 1978, 75(11): 5358-5361
[50] Wilkening S, Stahl F, Bader A. Comparison of primary human hepatocytes and hepatoma cell line Hepg2 with regard to their biotransformation properties [J]. Drug Metabolism and Disposition, 2003, 31(8): 1035-1042
[51] O’brien P, Irwin W, Diaz D, et al. High concordance of drug-induced human hepatotoxicity with in vitro cytotoxicity measured in a novel cell-based model using high content screening [J]. Archives of Toxicology, 2006, 80(9): 580-604
[52] Jan E, Byrne S J, Cuddihy M, et al. High-content screening as a universal tool for fingerprinting of cytotoxicity of nanoparticles [J]. ACS Nano, 2008, 2(5): 928-938
[53] Ross S, Chen T, Yu V, et al. High-Content Screening Analysis of the p38 pathway: Profiling of structurally related p38 α kinase inhibitors using cell-based assays [J]. Assay and Drug Development Technologies, 2006, 4(4): 397-409
[54] Kim M J, Lee S C, Pal S, et al. High-content screening of drug-induced cardiotoxicity using quantitative single cell imaging cytometry on microfluidic device [J]. Lab on a Chip, 2011, 11(1): 104-114
[55] Blake B L. Toxicology of the nervous system [M]// A Textbook of Modern Toxicology. Third Edition. 2004: 279-297
[56] Bard F, Cannon C, Barbour R, et al. Peripherally administered antibodies against amyloid β-peptide enter the central nervous system and reduce pathology in a mouse model of Alzheimer disease [J]. Nature Medicine, 2000, 6(8): 916-919
[57] Breier J M, Radio N M, Mundy W R, et al. Development of a high-throughput screening assay for chemical effects on proliferation and viability of immortalized human neural progenitor cells [J]. Toxicological Sciences, 2008, 105(1): 119-133
[58] Harrill J A, Robinette B L, Mundy W R. Use of high content image analysis to detect chemical-induced changes in synaptogenesis in vitro [J]. Toxicology in Vitro, 2011, 25(1): 368-387
[59] Raftery T D, Isales G M, Yozzo K L, et al. High-content screening assay for identification of chemicals impacting spontaneous activity in zebrafish embryos [J]. Environmental Science & Technology, 2014, 48(1): 804-810
[60] Macdonald M L, Lamerdin J, Owens S, et al. Identifying off-target effects and hidden phenotypes of drugs in human cells [J]. Nature Chemical Biology, 2006, 2(6): 329-337
[61] Abassi Y A, Xi B, Zhang W, et al. Kinetic cell-based morphological screening: Prediction of mechanism of compound action and off-target effects [J]. Chemistry Biology, 2009, 16(7): 712-723
[62] Bender A, Scheiber J, Glick M, et al. Cover Picture: Analysis of pharmacology data and the prediction of adverse drug reactions and off-target effects from chemical structure [J]. ChemMedChem, 2007, 2(6): 861-873
[63] Ravindranath A C, Perualila-Tan N, Kasim A, et al. Connecting gene expression data from connectivity map and in silico target predictions for small molecule mechanism-of-action analysis [J]. Molecular BioSystems, 2015, 11(1): 86-96
[64] 穆蕊, 李腾, 高彦飞, 等. 高内涵筛选NF-κB信号通路的技术体系建立[J]. 科学技术与工程, 2011, 11(14): 3162-3164
[65] Ghosh S, Baltimore D. Activation in vitro of NF-κB “by phosphorylation of it’s inhibitor IκB” [J]. Nature, 1990, 344(6267): 678-682
[66] Giuliano K A, Chen Y T, Taylor D L. High-content screening with siRNA optimizes a cell biological approach to drug discovery: Defining the role of P53 activation in the cellular response to anticancer drugs [J]. Journal of Biomolecular Screening, 2004, 9(7): 557-568
[67] Wang J, Zhou X, Bradley P L, et al. Cellular phenotype recognition for high-content RNA interference genome-wide screening [J]. Journal of Biomolecular Screening, 2008, 13(1): 29-39
[68] Dragunow M. High-content analysis in neuroscience [J]. Nature Reviews Neuroscience, 2008, 9(10): 779-788
[69] Wawer M J, Li K, Gustafsdottir S M, et al. Toward performance-diverse small-molecule libraries for cell-based phenotypic screening using multiplexed high-dimensional profiling [J]. Proceedings of the National Academy of Sciences, 2014, 111(30): 10911-10916
[70] Zamore P D, Tuschl T, Sharp P A, et al. RNAi: Double-stranded RNA directs the ATP-dependent cleavage of mRNA at 21 to 23 nucleotide intervals [J]. Cell, 2000, 101(1): 25-33
[71] Papdi C, brahám E, Joseph M P, et al. Functional identification of Arabidopsis stress regulatory genes using the controlled cDNA overexpression system[J]. Plant physiology, 2008, 147(2): 528-542.
[72] Walsh D P, Chang Y-T. Chemical genetics [J]. Chemical Reviews, 2006, 106(6): 2476-2530
[73] Eggert U S, Kiger A A, Richter C, et al. Parallel chemical genetic and genome-wide RNAi screens identify cytokinesis inhibitors and targets [J]. PLoS Biology, 2004, 2(12): e379
[74] Westerink W, Schoonen W G. Cytochrome P450 enzyme levels in HepG2 cells and cryopreserved primary human hepatocytes and their induction in HepG2 cells [J]. Toxicology in Vitro, 2007, 21(8): 1581-1591
[75] Tolosa L, Donato M T, Pérez-Cataldo G, et al. Upgrading cytochrome P450 activity in HepG2 cells co-transfected with adenoviral vectors for drug hepatotoxicity assessment [J]. Toxicology in Vitro, 2012, 26(8): 1272-1277
[76] Astashkina A, Mann B, Grainger D W. A critical evaluation of in vitro cell culture models for high-throughput drug screening and toxicity [J]. Pharmacology & Therapeutics, 2012, 134(1): 82-106
[77] Lin Z, Will Y. Evaluation of drugs with specific organ toxicities in organ specific cell lines [J]. Toxicological Sciences, 2012, 126(1): 114-127
[78] Bertino Jr J, Fish D. The safety profile of the fluoroquinolones [J]. Clinical Therapeutics, 2000, 22(7): 798-817
[79] Liguori M J, Blomme E A, Waring J F. Trovafloxacin-induced gene expression changes in liver-derived in vitro systems: Comparison of primary human hepatocytes to HepG2 cells [J]. Drug Metabolism and Disposition, 2008, 36(2): 223-233
[80] Shaw P J, Ganey P E, Roth R A. Tumor necrosis factorα is a proximal mediator of synergistic hepatotoxicity from trovafloxacin/lipopolysaccharide coexposure [J]. The Journal of Pharmacology and Experimental Therapeutics, 2009, 328(1): 62-68
[81] Roth A, Singer T. The application of 3D cell models to support drug safety assessment: Opportunities & challenges [J]. Advanced Drug Delivery Reviews, 2014, 69: 179-189
[82] Wenzel C, Riefke B, Gründemann S, et al. 3D high-content screening for the identification of compounds that target cells in dormant tumor spheroid regions [J]. Experimental Cell Research, 2014, 323(1): 131-143
[83] 张波, 杨萍, 陈雯. 基于毒性通路的毒理学危险度评价方法[J]. 中华预防医学杂志, 2010, 44(7): 587-590
◆
Principles and Applications of High Content Screening Technology in Ecotoxicology
Huang Chao, Yan Ye, Li Na, Ma Mei*, Wang Zijian
Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China
15 November 2014 accepted 7 January 2015
As a large amount of environmental pollutants may pose hazardous effects on the environment and human beings at low concentrations, evaluating the toxic effects of these chemicals and understanding their mechanisms of action (MOA) are essential for management of chemical environmental risks. Therefore, high-throughputinvitrobioassay methods have been rapidly developed to satisfy the toxicity assessment of countless chemicals. High-content screening (HCS), as a new cell-based screening approach, allows multi-endpoint determination simultaneously in intact cells. This review introduced the principles of HCS and summarized its application in ecotoxicology. Finally, the new developments and challenges of HCS are proposed.
high-content screening; high-throughput screening; in vitro bioassay; ecotoxicology
自然科学基金重点课题(No. 51290283);自然科学基金重点基金(No. 21437006)
黄超(1989-),男,硕士生,研究方向为水生态毒理学,E-mail: hch-app@163.com;
*通讯作者(Corresponding author), E-mail: mamei@rcees.ac.cn
10.7524/AJE.1673-5897.20141115001
2014-11-15 录用日期:2015-1-7
1673-5897(2015)2-2-11
X171.5
A
马梅(1967-),女,博士,研究员,博士生导师,主要从事水生态毒理学研究。
黄超, 言野, 李娜, 等. 高内涵筛选技术的原理及其在生态毒理学的应用[J]. 生态毒理学报, 2015, 10(2): 2-12
Huang C, Yan Y, Li N, et al. Principles and applications of high content screening technology in ecotoxicology [J]. Asian Journal of Ecotoxicology, 2015, 10(2): 2-12 (in Chinese)

