构建基于荧光多重cPCR的小鼠基因拷贝数检测方法
晁天柱,李鹏翔,徐福意,李凯,周宇荀,肖君华
(东华大学生物科学与技术研究所,上海 201620)
研究报告
构建基于荧光多重cPCR的小鼠基因拷贝数检测方法
晁天柱,李鹏翔,徐福意,李凯,周宇荀,肖君华*
(东华大学生物科学与技术研究所,上海 201620)
目的建立基于竞争性聚合酶链式反应(competitive polymerase chain reaction,cPCR)小鼠基因拷贝数变异(copy number variations,CNVs)的检测方法,用于检测野生小家鼠来源一号染色体替换系群体(population of specific chromosome 1 substitution strains,PCSSs)的CNVs。方法选取小鼠一号染色体上11个CNVs位点,及7、9和X染色体上各1个内对照位点,分别构建克隆质粒为竞争性粒模板,应用cPCR技术,建立荧光通用引物多重cPCR检测方法。结果多重cPCR方案适用于小鼠一号染色体上11个CNV位点的拷贝数检测,且能准确检测X染色体的拷贝数。结论实现小鼠快速、高通量的CNVs检测,可准确检测小鼠1号染色体中11个CNV位点的拷贝数变异。
拷贝数变异;竞争性模板;通用荧光引物;cPCR;小鼠
小鼠是多基因复杂性状研究的重要模式动物,但利用遗传多样性缺乏的近交系小鼠只鉴定出小部分基因[1-3]。以具有丰富遗传多样性野生小家鼠[4]为供体建立的PCSSs群体,是QTL精确定位、基因鉴定的新策略[5],是近交系品系的有益补充。但迄今,野生小家鼠和PCSSs群体的CNVs遗传多态性研究甚少。拷贝数变异 (CNVs)是人[6]和小鼠[7]基因组中一种丰富、重要的的遗传多态性[8-10]。广泛研究发现,CNV不仅影响哺乳动物表型差异[11],同时与许多人类疾病相关[12,13]。在41个近交系小鼠中,已鉴定2096个CNVs,共发现53个与人类疾病相关的基因[14]。且通过解析CNVs多样性,能揭示近交系小鼠种群历史,基因功能,指导建立人类疾病模型,并理解CNV发生机制和其潜在的生物功能[14]。
以芯片技术为基础的CNV检测方法,虽适用于CNVs的高通量检测,但其费用高,敏感性、操作性和重复性上存在较大问题[15]。简单多重荧光PCR方法成功检测FCGR3A和FCGR3B的拷贝数,却需要与靶基因相似的内源 DNA重复序列为内对照[16]。结合外源竞争模板的cPCR技术,能够精确定量DNA,检测DNA拷贝数[17,18]。质谱与cPCR技术相结合成功定量mRNA,具有极佳的准确性和重复性[19]。而单碱基延伸与cPCR结合的方法,适用于所有序列和基因及基因内相似CNVs,甚至是微小缺失和重复的检测,但操作繁琐[15,20]。因此,廉价、简单的cPCR技术是检测PCSSs群体CNV多样性的合适选择。
本研究以“野生小家鼠来源一号染色体替换系”群体构建过程中的N7代染色体工程小鼠为样本,利用cPCR技术,根据aCGH检测结果选择11个CNVs位点,构建克隆质粒竞争模板及荧光多重cPCR检测方案,以期实现野生小家鼠来源1号染色体上CNVs的高通量检测。
1 材料与方法
8周龄SPF级C57BL6/J(B6)小鼠(雌、雄各10只),体重18~26g,购自上海斯莱克实验动物有限公司[SCXK(沪)2012-0002]。动物实验遵守1988年动物管理条例。实验在东华大学生物科学与技术研究所屏障动物实验设施进行[SYXK(沪) 2014-0022]。收集14个PCSSs品系构建过程中的F1和N7代鼠尾组织,-20℃保存备用。
1.2 DNA提取和酶切
DNA提取采用Axygen(爱思进生物技术有限公司)基因组DNA抽提试剂盒。以0.8%琼脂糖凝胶电泳,全自动紫外与可见光分析仪FR-200A(上海复日科技实验技术研究所)和NanoDrop 2000c超微量分光光度计(Thermo Fisher Scientific,美国)确定DNA质量和浓度。基因组DNA经限制性内切酶BamHⅠ(NEB)酶切。
1.3 引物设计
利用棋盘法设计通用荧光引物:上游 FAMTTCATCCGTTCGTCCTAC; 下 游: ACAGCGTCAATCTCGTTC。14个位点的DNA序列信息来源于NCBI。所有引物用Oligo6设计,并由上海生物工程技术有限公司合成。图1A示意引物设计原则,所有cPCR特异引物和质粒构建用特异引物的序列信息,分别见表1和表2。
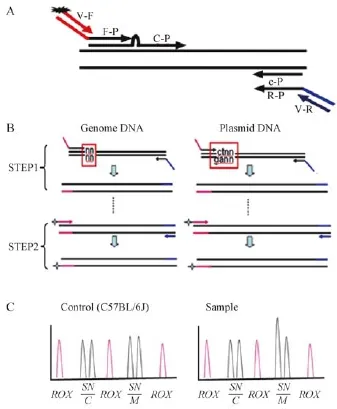
图1 检测位点侧翼引物和cPCR原理示意图Fig.1 Schematic diagram of the primers and cPCR principle
1.4 制备竞争性模板
将嵌合体引物的 PCR产物连接到pMD18-T Vector载体[宝生物工程(大连)有限公司]并转化到大肠埃希菌。质粒抽提采用SanPrep柱式质粒小量抽提试剂盒(上海生工生物工程技术服务有限公司)。提取的质粒经限制性内切酶HindIII和EcoR I [宝生物工程(大连)有限公司]酶切为线性DNA,以1%的琼脂糖凝胶电泳和FR-200A确定质量。采用Tiagen(天根生化科技有限公司)质粒小提试剂盒纯化酶切质粒,使用NanoDrop 2000c确定酶切质粒的质量和浓度。

表1 11个CNV位点和3个内对照位点的cPCR特异引物Tab.1 The specific primers of 11 CNVs and 3 internal control loci for cPCR

表2 构建竞争性质粒模板的特异引物Tab.2 The primers for the constrution of cloned plasmid competitors
1.5 aCGH分析
周妈的遗像摆在矮柜上,前面还放着一个玻璃瓶,周泽赡(小)踮起脚尖正要把鲜花插在玻璃瓶里,手不小心挡了一下瓶子,瓶子啪的一声,连同鲜花掉在了地上。
选用SurePrint G3 Bovine CGH Microarray(Agilent)芯片,对样本进行CNV检测。用 Agilent Genomic Workbench软件采集并分析数据。所有微阵列比较基因组杂交(Array-CGH)检测反应由复旦大学生命科学学院张峰课题组完成。
1.6 两步法cPCR
特异引物PCR体系(20μL)包括10×PCR buffer(30 mmol/L Mg2+)2μL,dNTP mix(2.5 mmol/L)3μL,上下游引物0.05μm/L,BSA(10 mg/mL)0.28μL,竞争性质粒模板,DNA模板10 ng/μL,Taq DNA Polymerase 0.5 unit。反应条件: 95℃ 2 min;94℃ 30 s,57℃ 90 s,65℃ 60 s,28个循环;68℃ 2 min(图1B-Step1)。
通用荧光引物PCR体系(5μL)包括10×PCR buffer(30 mmol/L Mg2+)0.5μL,dNTP mix(2.5 mmol/L)0.75μL,上下游引物 0.05μm/L,BSA(10 mg/mL)0.07μL,Taq DNA polymerase 0.5 unit。反应条件:94℃ 30 s,57℃ 90 s,65℃ 60 s,5个循环;68℃ 20 min(图1B-Step2)。
1.7 毛细管电泳
PCR产物1μL,加入8.6μL的Hi-Di和0.4 μL的DNA分子标准,振荡混匀变性。通过ABI测序仪3730(Applied Biosystems)分析样本。用GeneScan Analysis®3.0和 GeneMapper®ID software v3.2软件分析数据(图1C)。
1.8 CNV位点拷贝数
每个CNV位点的R值(样本峰/质粒峰),通过内对照(7号和19号染色体上的检测位点)和质控样本(B6)纠正,最终反映每个CNV位点的拷贝数变异。例图1C,C位点为内对照,M为检测位点,相对拷贝数[RR=(SC/NC)/(SM/NM)],所有值均为3730电泳峰高。质控样本RR=1.0,检测样本RR=1.5。
2 结果
2.1 CNV位点验证
aCGH检测结果表明,11个CNV位点全部存在于染色体工程小鼠中。例如,1qE2.1-1qE2.位点,在大新和三门峡 F1代中为重复(图2A和 B),1qH2.3-1qH4位点在大新样本是缺失(图2C)。cPCR结果显示,重复为样本峰高于质粒峰(图2D),缺失为样本峰低于质粒峰(图2E)。
2.2 多重cPCR检测方案
11个CNV位点的通用荧光引物多重cPCR检测方案结果表明,相对质控样本B6小鼠DNA在目标区段位置cPCR产物样本峰和质粒峰高度比值,能检测样本拷贝数变异。例如图3中1qE2.1-1qE2.3和1qH2.3-1qH4位点(N1和N2)RR为1,在大新F1样本中RR分别为0.5和1.5(图2)。
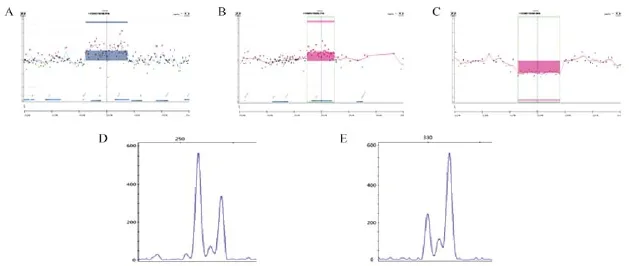
图2 aCGH和cPCR结果Fig.2 The results of aCGH and cPCR assays
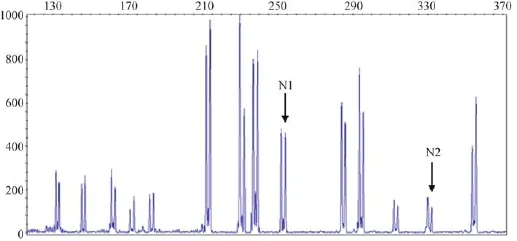
图3 通用cPCR方案的分离胶电泳Fig.3 Electrophoretogram of POP-6 products of the versatile cPCR
2.3 N7代样本检测
13个品系的N7代样本检测结果显示,有5个CNV位点在60%以上野生小家鼠中具有拷贝数差异,4个 CNV位点具有拷贝数变异的比例小于46%,1个CNV位点和2个内对照位点无拷贝数变异。在44个样本的 cPCR检测结果中,Chr.X: 103039160-103039780位点拷贝数差异能准确反映样本性别。
3 讨论
模式动物小鼠是研究遗传致病因素CNV的重要资源。且在近交系小鼠中,已发现与精神病[21,22]、焦虑[23]等多种复杂疾病相关的 CNVs,促进了小鼠基因组的CNV变异情况的研究。目前CNV检测方法包括芯片技术、测序技术和PCR技术[24-26]。CGH和SNP芯片技术具有快速、高通量和自动化的优点,但CGH技术难以检测平衡相互异位及倒位,aCGH技术的敏感性和操作性亟待提高,SNP芯片技术不适用于重复序列和复杂CNV区域。基于测序技术的CNV检测方法,其速度快、分辨率高、重复性高等优点,但费用高昂。以PCR为基础的CNV检测方法中:实时荧光定量PCR技术,通量低,稳定性和准确性不高;多重连接探针扩增技术(multiplex ligation-dependent probe amplification,MLPA),灵敏度和特异性较高,重复性强,检测不到平衡异位和倒位突变。针对染色体工程小鼠一号染色体上少量CNV位点的检测,需要一简便、准确的技术方法。
本研究结果揭示,利用根据低TM值,二级结构的自由能数值△G(单位kcal/mol)> -4等原则获得的通用荧光引物,成功构建的基于荧光通用引物多重cPCR方案,适用于染色体工程小鼠一号染色体11个CNVs的拷贝数检测工作。染色体工程小鼠在2个位点Chr.19:27915739-27916361和Chr.7:99758205-99758827无拷贝数变异,是合适的内对照位点。
相比于近交系C57BL/6J小鼠,N7代染色体工程小鼠的一号染色体在10个CNV位点具有拷贝数变异,如CNV位点Chr 1:141896999-141897619在13个品系中具有较大拷贝数差异(1-4个重复),CNV位点Chr 1:175354538-175355160在群体中无拷贝数变异。单一亲本起源,导致染色体工程小鼠群体中CNV位点的突变类型一致。而因亲本来源不同,致使Chr.1:173496351-173496972和Chr. 1:168146833-168147454位点分别在枣庄1品系N7代群体内和三门峡的F1代与N7代样本间存在拷贝数差异。三门峡F1代和N7代个体间其余10个CNV位点拷贝数一致无突变发生,说明一般性传代并不会改变CNV拷贝数。
总之,利用竞争性质粒模板结合通用荧光引物的多重cPCR方法是一种廉价、准确的CNV检测方案。能准确检测小家鼠一号染色体上11个CNV位点的拷贝数变异,适用于少量小家鼠样本的快速、高效的CNV检测工作。
[1] Mott R,Flint J.Prospects for complex trait ananlysis in the mouse[J].Mamm Genome,2008,19(5):306-308.
[2] Flint J,ValdarW,Shifman S,et al.Strategies formapping and cloning quantitative trait genes in rodents[J].Nat Rev Genet,2005,6(4):271-286.
[3] Churchill GA,Airey DC,Allayee H,et al.The collaborative cross,a community resource for the genetic analysis of complex traits[J].Nat Genet,2004,36(11):1133-1137.
[4] Laurie CC,Nicker son DA,Nachman MW,et al.Linkage disequilibrium in wild mice[J].PLoSGenet,2007,3(8):e144.
[5] Xiao J,Liang Y,Li K,etal.A novel strategy for genetic dissection of complex traits:the population of specific chromosome substitution strains from laboratory and wild mice[J].Mamm Genome,2010,21(7-8):370-376.
[6] Redon R,Ishikawa S,Fith KR,et al.Global variation in copy number in the human genome[J].Nature,2006,444:444-454.
[7] Egan C,Sridhar S,Wigler M,et al.Recurrent DNA copy number variation in the laboratorymouse[J].NatGenet,2007,39: 1384-1389.
[8] Jakobsson C,Ozaki H,Nakajima T,et al.Genotype,haplotype and copy number variation in worldwide human populations[J]. Nature,2008,451:998-1003.
[9] She X,Cheng Z,Zollner S,et al.Mouse segment duplication and copy number variation[J].Nat Genet,2008,40:909-941.
[10] Watkins-Chow DE,Pavan WJ.Genomic copy number and expression variation within the C57BL/6J inbred mouse strains [J].Genome Res,2008,18:60.
[11] Freeman JL,Perry GH,Feuk L,et al.Copy number variation: new insights in genome diversity[J].Genome Res,2006,16: 949-961.
[12] Gonzalez E,Kulkarni H,Bolivar H,et al.The influence of CCL3L1 gene-containing segment duplications on HIV-1/AIDS susceptibility[J].Science,2005,307:1434-1440.
[13] Aitman TJ,Dong R,Vyse TJ,etal.Copy number polymorphism in Fcgr3 predisposes to glomerulonephritis in rats and humans[J].Nature,2006,439(7078):851-855.
[14] Culter G,kassner PD.Copy number variation in the mouse genome:implications for themouse as amodel organizm for human disease[J].Cytogenet Genome Res,2008,123:297-306.
[15] Kim HK,Hwang HL,Park SY,et al.Simple and versatilemolecularmethod of copy-number measurement using cloned competitors[J].PLoSONE,2013,8(7):e69414.
[16] Hollox EJ,Detering JC,Dehnugara T,et al.An integrated approach for measuring copy number variation at the FCGR3(CD16)locus[J].Hum Mutat,2009,30:477-483.
[17] Sestini R,Orlando C,Zentilin L,et al.Measuring c-erbB-2 oncogene amplification in fresh and paraffin-embedded tumors by competitive polymerase chain reaction[J].Clin Chem,1994,40:630-636.
[18] Okuyama N,Hatano Y,Park Y,et al.Quantitation of c-erbB-2 gene amplification in breast cancer tissue by competitive PCR [J].Tumour Biol,1999,20:153-161.
[19] Ding C,Cantor CR.A high-throughput gene expression analysis technique using competitive PCR and matrix-assisted laser desorption ionization time-of-flightMS[J].Proc Natl Acad SciU S A,2003,100:3059-3064.
[20] Stofanko M,Han JC,Elsea SH,et al.Rapid and inexpensive screening of genomic copy number variationsusing a novelquantitative fluorescent PCRmethod[J].Dis Markers,2013,35(6): 589-596.
[21] Avrabos C,Sotnikov SV,Dine J,et al.Real-time imaging of amygdalar network dynamics in vitro reveals a neurophysiological link to behavior in a mouse model of extremes in trait anxiety [J].JNeurosci,2013,33(41):16262-16267.
[22] Brenndörfer J,Altmann A,Widner-AndräR,et al.Connecting anxiety and genomic copy number variation:a genome-wide analysis in CD-1 mice[J].Plos ONE,2015,10(5):e0128465.
[23] Gonik M,Frank E,KeBler MS,et al.The endocrine stress response is linked to one specific locuson chromosome3 in amouse model based on extremes in trait anxiety[J].BMC genomics. 2012,13:579.
[24] TakahashiN,Tsuyama N,Sasaki K,etal.Segmental copy-number variation observed in Japanese by array-CGH[J].Ann Human Genet,2008,72:193-204.
[25] Kamath BM,Thiel BD,Gai X,et al.SNP array mapping of chromosome 20p deletions:genotypes,phenotypes,and copy number variation[J].Human Mutat,2008,30:371-378.
[26] Goossens D,Moens LN,Nelis E,et al.Simultaneousmutation and copy number variation(CNV)detection by multiplex PCR-based GS-FLX sequencing[J].Human Mutat,2009,30:472 -476.
Establishment of a universal fluorescentmultiplex cPCR method for detection of copj number variations in m ice
CHAO Tian-zhu,LIPeng-xiang,XU Fu-yi,LIKai,ZHOU Yu-xun,XIAO Jun-hua*
(Institute of Biological Sciences and Biotechnology,Donghua University,Shanghai201620,China)
ObjectiveTo establish a high throughput general multiple competitive polymerase chain reaction(cPCR)detectingmethod of copy number variations(CNVs)for the population of chromosome 1 substitution strains from wild mice.MethodThe selected 14 loci,including 11 CNVs on chromosome 1 and internal control loci on other three chromosmes(Chr 7,Chr19 and Chr X),were detected based on the universal fluorescentprimermultiple competitive polymerase chain reaction.All specific cloned plasmidswere constructed as competitors.ResultsAltogether 11 CNVswere designed in one panel,and the copy of Chr X accurately reflects the gender.ConclusionsA rapid and high-throughput fluorescentmultiplex cPCR assay is established which can be used for detection of copy number variations on chromosome 1 in mice.
Copy number variations,CNVs;Competitive template;Universal fluorescence primers;cPCR;Mice
Q95-33
A
1005-4847(2015)06-0591-06
10.3969/j.issn.1005-4847.2015.06.009
2015-06-19
国家自然科学基金面上项目(编号:31371257);上海市科委关键项目(编号:12140900404)。
晁天柱(1982-),男,博士研究生,研究方向:医学分子遗传学,E-mail:chaotianzhu@126.com
肖君华(1968-),男,教授,研究方向:医学分子遗传学,E-mail:xiaojunhua@dhu.edu.cn

