施马伦贝格病毒RT-LAMP检测方法的建立
袁向芬,吴绍强,张永宁,林祥梅
(中国检验检疫科学研究院动物检疫研究所,北京 100029)
2011年秋季,德国北部和荷兰西部等地一些奶牛场暴发了一种以发热、产奶量下降、腹泻、严重时流产等为临床症状的新型动物疫病。德国弗里德里希洛福勒研究所(Friedrich-Loeffler-Institut,FLI)通过实验室诊断与研究,确定导致此次疫病的病原体为一种新型布尼亚病毒,并将其命名为施马伦贝格病毒(Schmallenberg virus,SBV),将其引起的疫病称为施马伦贝格病[1]。该病主要感染绵羊、牛、山羊以及鹿等反刍动物,截至2013年10月,其已迅速蔓延至28个国家,引起了世界动物卫生组织(OIE)、欧盟和国际社会的高度重视。
鉴于SBV可通过精液等遗传物质传播,加之我国与欧盟国家之间的反刍动物遗传物质贸易频繁,建立一种敏感、快速、特异及临床诊断可行的检测方法尤为迫切,目前,可用于施马伦贝格病诊断的技术主要包括:病毒分离[2,3]、中和试验[4]、免疫荧光[5]、ELISA[5]及Real-Time RT-PCR[6]等方法。然而,以上几种检测方法或需借助特殊仪器,或需较长周期,对试验环境的要求也较为严谨,因此并不完全适用于养殖场、野外及口岸的现场检疫。环介导等温扩增技术(loop-medieated isothermal amplif i cation,LAMP)自2000年首次报道以来,因其高效、快速、高特异性等优点被广泛应用于动物疫病的检测工作[7-10]。该技术依赖于能够识别靶序列上6个特异性区域的引物和一种具有链置换特性的DNA聚合酶,在简单的等温条件下即可实现靶序列的高效扩增[11,12],整个过程中对环境、仪器等条件的要求均极其简单,特别适用于动物疫病的现场检疫。本研究针对SBV的S基因节段建立了其RTLAMP方法,并通过一系列试验证明该方法与实时荧光RT-PCR方法相比具有良好的敏感性和特异性,同时对于临床样品的快速检测也具有高度适用性。
1 材料和方法
1.1 样品
SBV阳性RNA样品由德国FLI实验室提供,用于提取RNA的病毒培养液浓度为6×107TCID50/mL。由于与SBV同属、且亲缘关系很近[13]的赤羽病病毒(Akabane virus,AKAV)、沙门达病毒(Shamonda virus,SHAV)、艾罗病毒(Aino virus,AINOV)、道格拉斯病毒(Douglas virus,DOUV)Australia 株、道格拉斯病毒(Douglas virus)Texas株和辛波病毒(Simbu virus)多为国外动物疫病,因此特异性试验所需的相关病毒S基因节段核酸均由宝生物工程(大连)有限公司全基因合成获得,合成序列参考表1。
103份牛、羊临床全血样品采集自内蒙、天津等地养殖场。
1.2 引物的设计与合成
本研究对比了GenBank中SBV及其同属各病毒的基因序列,针对SBV的S基因节段序列,利用Primer Explorer version 4在线引物设计软件进行引物设计,设计了一套用于LAMP反应的引物(表2),引物交宝生物工程(大连)有限公司合成。

表1 病毒参考序列

表2 引物设计
1.3 RNA提取
阳性RNA样品由德国FLI实验室提供,其提取按照Trizol法进行,取1mLSBV病毒培养 液(6×107TCID50/mL) 进 行RNA的 提 取,用30μLDEPC水进行RNA的溶解,经换算可知RNA浓度相当于2×106TCID50/μL。临床血液样品RNA的提取,有本实验室按照Trizol法提取,具体操作参照说明书进行。提取的RNA均置于-80℃冰箱内保存备用。
1.4 RT-LAMP条件优化与方法建立
为实时监测不同反应条件下RT-LAMP的进展情况,反应条件的优化借助环介导等温扩增实时浊度仪LA-320c进行,在Loopamp RNA Amplification Kit(RT-LAMP)试剂盒(Eiken Chemical Co.,Ltd,Tokyo,Japan)基础上,主要对反应的引物浓度、退火温度(60-65℃)、反应时间进行优化(表3)。
1.5 RT-LAMP方法敏感性及特异性分析
用优化后的RT-LAMP检测方法对SBV同属的 AKAV、SHAV、AINOV、DOUV Australia株、DOUV Texas株及Simbu病毒的S节段核酸样品进行扩增,评估本方法的特异性。设置SBV RNA样品为阳性对照,灭菌水为阴性对照。另外,按照优化后的条件和程序对本方法灵敏度进行评估,采用10倍系列稀释的SBV RNA样品(2×106~2×10-1TCID50/μL)进行,来确定本方法的最低检测限。反应结果可通过肉眼观察判定:当反应液中加入荧光染料钙黄绿素(Calcein)时,可根据反应后溶液的颜色变化进行鉴定,阳性反应溶液会由原来的橙色变为亮绿色,阴性仍保持橙色;还可利用电泳分析进行判定:取5μL反应液于2.5%的琼脂糖凝胶中进行电泳,在紫外下观察电泳条带并拍照记录。

表3 温度和内引物浓度优化
1.6 SBV与SHAV的鉴别诊断
SBV和SHAV S基因节段序列同源性极高,可达97%,因此基于S节段的常规核酸扩增技术无法实现两者的准确鉴别。通过对GenBank中SBV和SHAV的S节段基因序列进行酶切位点分析发现,引物F1和F2对应序列之间的靶序列上,SHAV存在一特异性酶切位点Af l II(5'C^TTAAG3')(表4),而SBV并不存在该位点,因此,可通过对RT-LAMP反应产物进行酶切来进一步鉴别SBV与SHAV。

表4 酶切位点序列分析
1.7 Real-time RT-PCR方法
德国FLI实验室针对SBV的S基因节段建立了检测施马伦贝格病的荧光定量RT-PCR方法[6]。该方法的商品化试剂盒已由Qiagen公司进行生产与销售(货号FLI B585)。参照该试剂盒说明书,对10倍系列稀释的SBV阳性RNA及制备的其同属6种病毒S节段核酸样品进行扩增,反应结束后,与RT-LAMP检测方法的灵敏度及特异性进行比较。其反应程序为:45℃ 10min(反转录);95℃ 10min(预变性);95℃ 15s,56℃ 30,72℃30s,42个循环(PCR反应),在56℃退火阶段进行荧光信号的收集,全程108min。
1.8 临床样品的检测
用建立的RT-LAMP方法检测103份临床样品,并与荧光定量RT-PCR的检测结果进行比较。
2 结果
2.1 RT-LAMP方法优化
经过一系列优化,确定了SBV RT-LAMP方法的反应体系(25μL)为:FIP和BIP各40pmol,F3和B3各5pmol,LF和LP各20pmol,2×Reaction Mix 12.5μL(Tris-HCl(pH 8.8)40 mM,KCl 20mM,MgSO416mM,(NH4)2SO420mM,Tween 200.2%,Betaine 1.6 M,dNTPs 2.8mM each),Enzyme Mix 1.0μL,核酸RNA 5μL,另外,反应液中还加入1μL荧光试剂FDR(Eiken Chemical Co.,Ltd,Tokyo,Japan)来监测扩增产物。反应程序为:63℃恒温下扩增50min;80℃酶灭活5min,该程序既可在恒温水浴锅中进行,也可在金属浴及各种核酸扩增仪等仪器中进行。反应后,通过肉眼观察颜色变化即可判定反应阴阳性。
2.2 RT-LAMP方法的敏感性和特异性
用建立的RT-LAMP方法对10倍系列稀释的SBV阳性RNA样品进行扩增,结果如图所示(图1A,1B),本方法可成功检测到2×100TCID50/μL的阳性RNA,染料法与琼脂糖凝胶电泳法的结果一致,结合加入反应体系的RNA体积,灵敏度可达10TCID50。特异性试验显示(图1D),仅SBV及SHAV发生扩增,反应管颜色由原来的橙色转变为亮绿色,与电泳结果一致(图1E),说明该方法与SHAV存在交叉反应。荧光定量RTPCR方法的灵敏度同为10TCID50(图1C),特异性试验显示其可与SHAV、DOUV Australia株和DOUV Texas株发生交叉反应(图1F)。相比之下,本研究建立的RT-LAMP方法兼具了良好的敏感性和特异性。
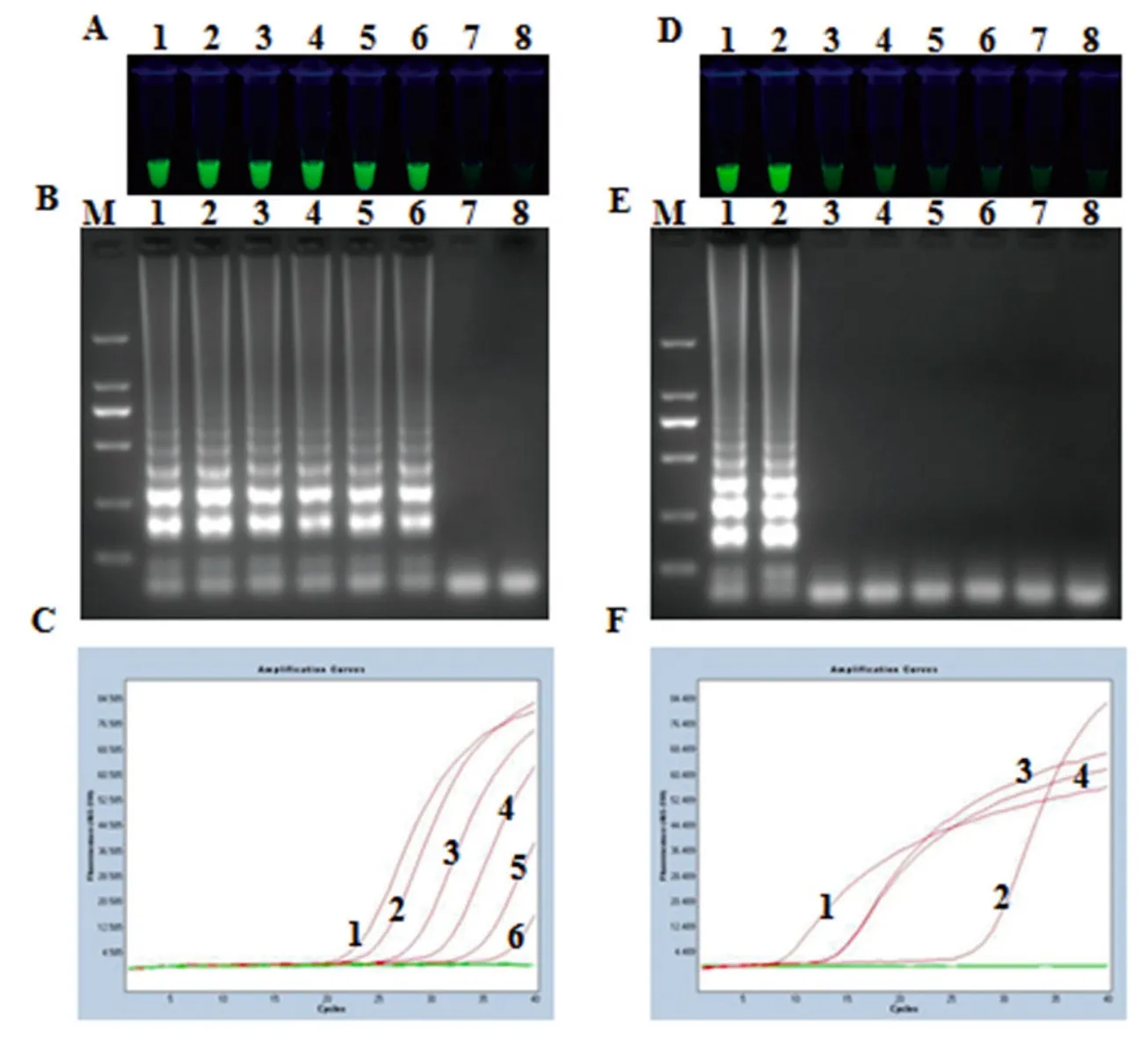
图1 灵敏度与特异性试验
2.3 SBV与SHAV的鉴别诊断
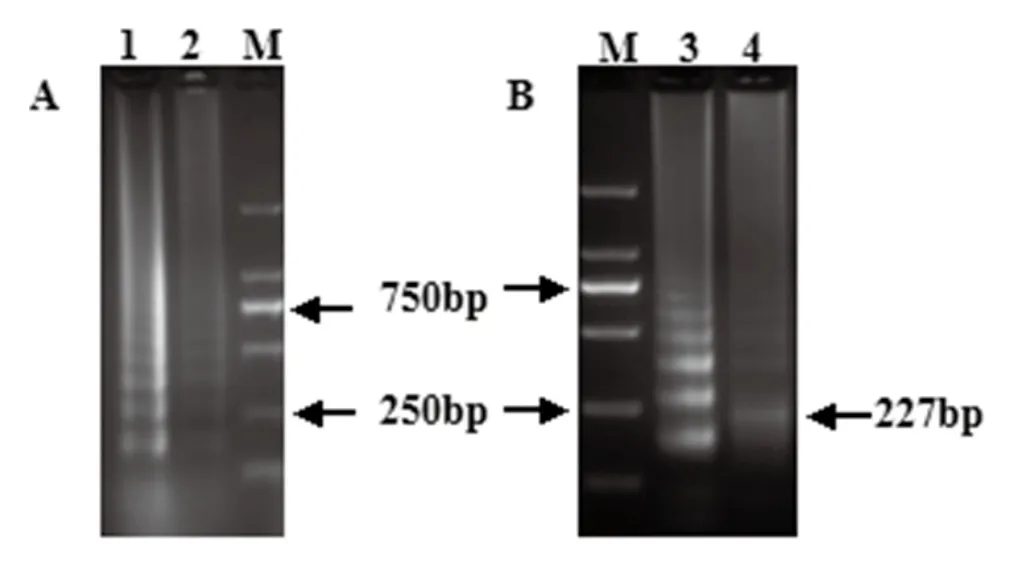
图2 酶切鉴定试验
25μL酶切反应体系中包含5μL RT-LAMP产物,2.5μL cutsmart buffer,8U Af l II酶(New England Biolabs,USA)。37℃孵育过夜后,取10μL酶切产物于2.5%的琼脂糖凝胶中进行电泳,在紫外显像仪中观察酶切后条带的变化。结果与预期结果相符,SBV与SHAV呈现出不同的酶切结果,如图2所示,SBV LAMP产物经酶切后,与SBV LAMP产物的电泳条带一致,未发生变化,说明未发生酶切反应;而SHAV则在227bp位置有一明显新增条带产生,借此即可实现SBV与SHAV的鉴别性诊断。
2.4 临床样品检测
为评估本研究建立的RT-LAMP方法的临床适用性,本课题组采集了内蒙、天津等地的103份牛、羊全血样品,分别用本研究建立的RTLAMP方法及荧光定量RT-PCR方法对样品进行检测。结果显示,两种方法检测的阳性率均为0%。说明所采集的103份样品均为SBV阴性。
3 讨论
本研究建立了一种快速检测SBV的RT-LAMP方法,并利用酶切可鉴别诊断SBV及SHAV。环引物的应用大大加速了反应速率[14],可在50分钟内完成扩增。同时,本方法具有高度的敏感性,可达10TCID50,与荧光定量RTPCR方法的敏感性相同。而特异性试验显示,本研究建立的RT-LAMP方法与德国FLI实验室所建立的qRT-PCR检测方法[6]相同。本法可与SHAV发生交叉反应,因为SBV和的SHAV两者S节段核酸序列同源性高达97%[15]。基于S节段的常规核酸检测方法极不易对两者进行区分,本类方法更适用于田间、养殖场及口岸等大量样品的初检。另外,本研究建立了基于Af l II酶切位点的酶切鉴定方法,可实现两者的鉴别诊断,增加本方法的特异性,为实验室确诊提供了可信的鉴别诊断方法。本方法的另外一个优点还在于,相比于荧光RT-PCR要借助昂贵精密的仪器而言,RT-LAMP可利用简单的恒温装置进行扩增,并可通过多种方法进行结果的判定,如通过肉眼判定、染料法以及传统的电泳法。研究显示,本方法采用的染料法完全可以满足常规检测的需要[16],特别适用于大规模、野外或口岸的临床样品检测。相比之下,RT-LAMP方法兼具了良好的敏感性、特异性、快捷性及便利性,更适于口岸、养殖场等的现场检疫,为SBV的防控提供技术储备。
采集内蒙古、天津等地的牛、羊全血样品103份,统一处理后进行RT-LAMP检测。其结果与荧光定量RT-PCR方法的结果完全符合,样品检测结果均为SBV阴性,与中国境内目前无SBV存在的相关报道相符[17,18]。但考虑到SBV传播速度快、涉及范围广,该结果同时提示我国继续加强对SBV的口岸防控,同时制定相关防控政策,防止SBV跨境传入中国。
然而,LAMP技术也存在不足之处,这些不足也是限制LAMP技术发展的主要障碍。LAMP方法具有高效的反应速率,单个目的基因可于短时间内迅速扩增至109拷贝数,此时极易产生气溶胶,导致环境污染以及假阳性的出现,为控制该污染,实验人员在操作过程中应严格注意空间、仪器耗材等的分隔使用,同时反应后尽量利用染料法、比浊法等闭管方法进行判定[8,19-20],防止反应产物的扩散。另外,有研究者通过试验发现LAMP反应对培养基及生物物质干扰具有较强抗性,因此对病毒核酸的提取质量要求较低[21];且目前对于核酸提取技术的研究也有了很大的进展[22-25],国内外已有公司 推出了一管式核酸提取试剂,在核酸提取时间及操作上提供了很大便利,然而探索一种可与LAMP技术配套的经济、快速、便捷、成熟的核酸提取技术,仍是LAMP技术发展及应用的迫切需要。
[1]Hoffmann B,Scheuch M,Hoper D,et al.Novel orthobunyavirus in Cattle,Europe,2011[J].Emerg Infect Dis,2012,18(3):469-472.
[2]Hoffmann B,Schulz C,Beer M.First detection of Schmallenberg virus RNA in bovine semen,Germany,2012[J].Vet Microbiol,2013,167(3):289-295.
[3]van der Heijden H M,Boustra R J,Mars M H,et al.Development and validation of an indirect enzyme-linked immunosorbent assay for the detection of antibodies against Schmallenberg virus in blood samples from ruminants[J].Res Vet Sci,2013,95(2):731-735.
[4]Leoffen W,Quak S,de Boer-Luijtze E,et al.Development of a virus neutralisation test to detect antibodies against Schmallenberg virus and serological results in suspect and infected herds[J].Acta Vet Scand,2012,54(1):44.
[5]Breard E,Lara E,Comtet L,et al.Validation of a commercially available indirect ELISA using a nucleocapside recombinant protein for detection of Schmallenberg virus antibodies[J].PLoS One,2013,8(1):e53446.
[6]Bilk S,Schulze C,Fischer M,Beer M,et al.Organ distribution of Schmallenberg virus RNA in malformed newborns[J].Vet Microbiol,2012,159(1/2):236-238.
[7]鑫 婷,侯绍华,贾 红,等.猪呼吸与繁殖综合症病毒RT-LAMP 检测方法的建立[J].中国农业科学,2010,43(1):185-191.
[8]Tomita N,Mori Y,Kanda H,et al.Loop-mediated isothermal amplification(LAMP)of gene sequences and simple visual detection of products[J].Nature Protocols,2008,3(5):877-882.
[9]Yin S,Shang Y,Zhou G,et al.Development and evaluation of rapid detection of classical swine fever virus by reverse transcription loop-mediated isothermal amplification(RT-LAMP)[J].Journal of Biotechnology,2010,146(4):147-150.
[10]Reddy V,Ravi V,Desai A,et al.Utility of IgM ELISA,TaqMan real-time PCR,reverse transcription PCR,and RT-LAMP assay for the diagnosis of Chikungunya fever[J].Journal of Medical Virology,2012,84(11):1771-1778.
[11]Notomi T,Okayama H,Masubuchi H,et al.Loopmediated-isothermal amplification of DNA[J].Nucleic Acids Research,2000,28:E63.
[12]罗力涵,张波.环介导等温扩增技术及其在传染性疾病检测中的应用[J].中国国境卫生检疫杂志,2014,37(1):68-71.
[13]Goller K V,Hoper D,Schirrmeier H,et al.Schmallenberg virus as possible ancestor of Shamonda virus[J].Emerge Infect Dis.2012,18(10):1644-1646.
[14]Nagamine K,Hse T,Notomi T.Accelerated reaction by loop-mediated isotherml amplification using loop primers[J].Mol Cell Probes, 2002, 16(3):223-9.
[15]Yanase T,Kato T,Aizawa M,et al.Genetic reassortment between Sathuperi and Shamonda viruses of the genus Orthobunyavirus in nature:Implications for their genetic relationship to Schmallenberg virus[J].ArchVirol,2012,157(8):1611-1616.
[16]Higashimoto Y,Ihira M,Ohta A,et al.Discriminating between Varicella-Zoster virus vaccine and wild-type strains by loop-mediated isothermal amplif i cation[J].J Clin Microbiol 2008,46:2665-2670.
[17]张永宁,吴绍强,刘 建,等.欧洲暴发施马伦贝格病疫情及我国应采取的对策[J].中国兽医杂志,2012,48(4):92-95.
[18]张永宁,吴绍强,吕继洲,等.施马伦贝格病毒核衣壳蛋白的原核表达、纯化及多克隆抗体的制备[J].畜牧兽医学报,2013,44(10):1629-1236.
[19]Mori Y,Kitao M,Tomita N,et al.Real-time turbidimetry of LAMP reaction for quantifying template DNA[J].J Biochem Bioph Meth,2004,59:145-157.
[20]Mori Y,Nagamine K,Tomita N,et al.Detection of loop-mediated isothermal amplification reaction by turbidity derived from magnesium pyrophosphate formation[J].Biochem Bioph Res Co,2001,289:150-154.
[21]Kaneko H,Kawana T,Fukushima E,et al.Tolerance of loop-mediated isothermal amplif i cation to a culture medium and biological substances[J].J Biochem Bioph Meth,2007,70:499-501.
[22]Kiefer E,Heller W,Ernst D.A simple and efficient protocol for isolation of functional RNA from plant tissues rich in secondary metabolites[J].Plant Molecular Biology Reporter,2000,18(1):33-39.
[23]Cho Y K,Lee J G,Park J M,et al.One-step pathogen specific DNA extraction from whole blood on a centrifugal microf l uidic device[J].Lab Chip,2007,7(5):565-573.
[24]Rohland N,Hofreiter M.Comparison and optimization of ancient DNA extraction[J].Biotechniques,2007,42(3):343-52.
[25]Chomczynski P,Sacchi N.The single-step method of RNA isolation by acid guanidinium thiocyanate-phenolchloroform extraction:twenty-something years on[J].Nat Protoc.2006,1(2):581-585.

