代谢工程中途径和菌株改造的几种新技术
孙新晓,唐秋雅,袁其朋
(北京化工大学化工资源有效利用国家重点实验室,北京 100029)
引言
从 1991年首次提出代谢工程的概念,已经有20多年的发展历史。代谢工程通过对菌株进行遗传改造,重新配置细胞的生化代谢网络,将可再生原料转化成有附加值的化合物。然而,通过操纵内源基因和引进外源途径,改造微生物长期进化的代谢网络,经常会导致途径通量的显著不平衡,进一步产生多种不良后果[1]。例如,中间代谢产物的积累可能对上游酶产生反馈抑制,导致副产物的形成,降低了产品的生产效率[2]。因此解决代谢失衡问题,可以大大改善细胞的生长状况、产品产量及产率。其中,找到最佳途径平衡点是菌株改造过程的重要一步。
代谢工程的早期研究通过解除酶的反馈抑制、简单的基因敲除或过表达等理性方法解决途径中存在的瓶颈。例如,Liao课题组[3]通过对途径中的酶进行筛选,并采用汽提的方式不断分离发酵液中产物,最终发酵罐中异丙醇的产量可达143 g·L-1。然而,仅仅对菌株进行局部改造经常会引入新的、意想不到的瓶颈。由于代谢网络各支路及基因之间的相互关联性,导致需要从全局水平进行代谢调控。DNA测序技术、组学技术以及基因组水平的计算机模拟技术的进步,使得全局水平的菌株及途径优化成为可能。例如,Becker等[4]通过计算机模拟对代谢网络进行分析,对参与中心代谢的12个基因靶点进行调节,将代谢流导向产物的合成,最终谷氨酸棒杆菌中赖氨酸产量达到了120 g·L-1。除了以上的分析及调控技术,最近几年又出现了一些新的代谢途径的调控技术,比较有代表性的包括多变量的模块化优化技术、蛋白质支架技术、代谢流量的动态调控技术以及大规模基因编辑技术。
1 多变量的模块化优化技术
多变量的模块化优化技术将代谢途径中的酶按照途径节点、酶的催化效率等分成几个模块,通过在转录(如启动子,基因拷贝数)、翻译(如核糖体结合位点)或酶的催化特性等水平对这些途径模块进行调整,对少量条件进行摸索,不需要高通量筛选就可实现途径的优化。Ajikumar等[5]利用该技术成功提高了紫杉醇前体紫杉烯的产量。他们将紫杉烯合成途经分成上游2 -甲基-D-赤藓糖醇-4-磷酸(MEP)模块和下游模块。然后通过改变启动子强度和质粒拷贝数调节酶的表达水平,限制副产物吲哚的积累。最终紫杉烯产量与对照菌株相比提高了15000倍,达到1 g·L-1。
认识到该方法的应用潜力后,许多课题组也运用这一方法,进行各种生物过程的开发和优化。Koffas等[6]利用模块化优化改造大肠杆菌,将大肠杆菌的中心代谢途径分成3个模块:乙酰辅酶A的合成(GLY模块),乙酰辅酶A活化(ACA模块),丙二酰ACP消耗(FAS模块)。通过改变质粒拷贝数和核糖体结合位点的强度,能够平衡这3个模块,为后续的下游反应提供适量的乙酰辅酶A,最终脂肪酸产量达到8.6 g·L-1(图1)。Liu等[7]将N-乙酰葡萄糖胺(GlcNAc)合成途径分为外源GlcNAc的合成模块、GlcNAc糖酵解模块和肽聚糖合成模块。通过同时改变内源和外源基因的表达,优化菌株可以产生36.35 g·L-1的GlcNAc,比对照菌株提高3.8倍。本课题组[8]也利用该技术优化了大肠杆菌的黏糠酸合成途径,最终黏糠酸产量提高了275倍,达到 1.5 g·L-1。
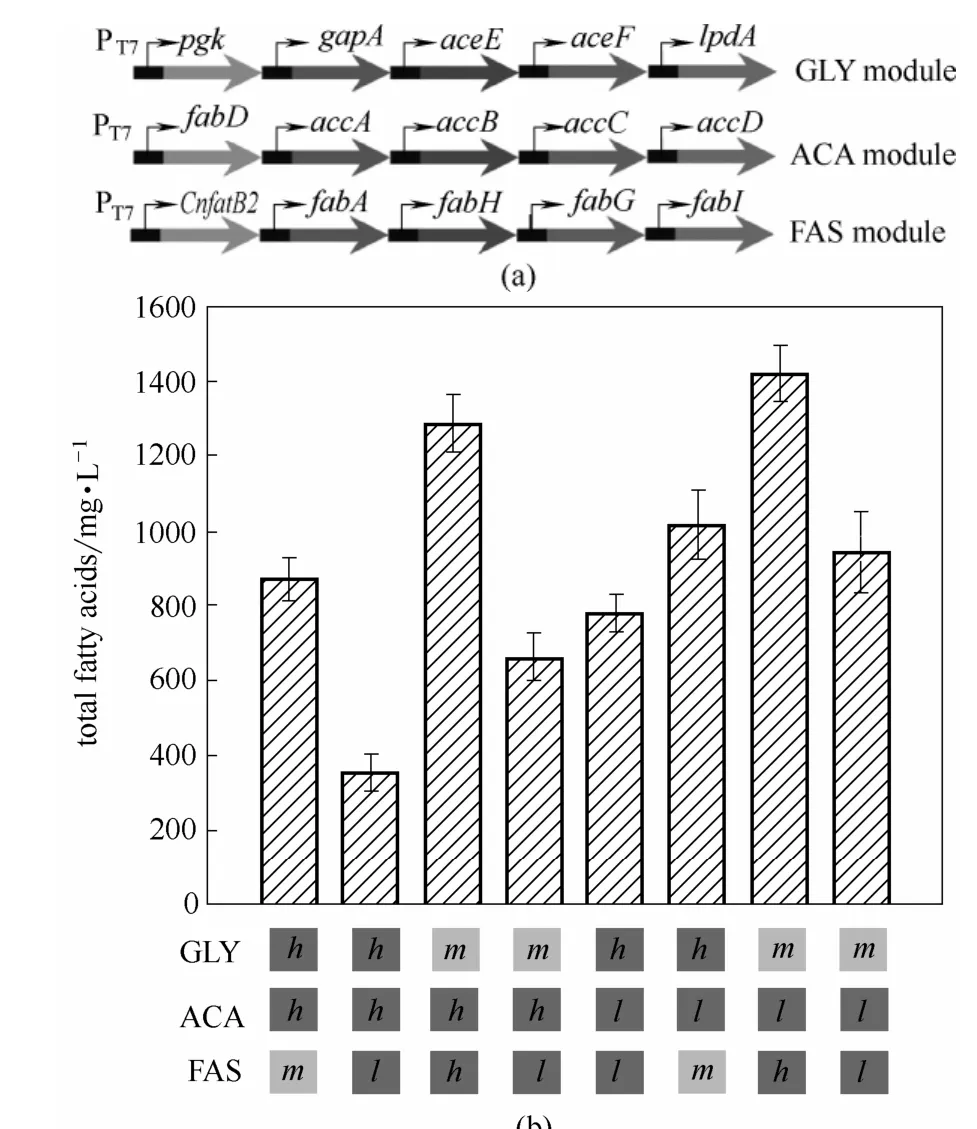
图1 模块化优化大肠杆菌脂肪酸生产[6]Fig.1 Modular optimization for fatty acids production in E. coli[6]
Wu等[9]将白藜芦醇的合成途径分成3个模块,分别为香豆酰辅酶A合成模块、丙二酸辅酶A合成模块以及二苯基乙烯(stilbene)合成酶模块。通过调整3个模块的表达水平,平衡了途径的通量,避免香豆酰辅酶A的毒性积累,直接从葡萄糖产生35.03 mg·L-1的白藜芦醇。类似地,他们还建立了四模块系统用于从葡萄糖生产(2S)-松属素。通过简单地改变每一个模块质粒的拷贝数,减少了肉桂酰辅酶A的毒性积累,产生了40.02 mg·L-1的(2S)-松属素[10]。Zhao等[11]进一步将该技术扩展到五模块系统,用于β-胡萝卜素的生产。其途径包括MEP和下游合成酶模块,以及 ATP的合成、TCA循环和磷酸戊糖生产3个模块。通过调换启动子和mRNA稳定区域来调节表达,提供了更多的前体进入外源β-胡萝卜素的途径,最终产量达到2.1 g·L-1。
除了在原核系统优化中的成功应用,多变量的模块化优化技术也成功应用于真核系统。Dai等[12]将次丹参酮二烯(miltiradiene)合成的上游前体供应途径分为两个模块:法尼基焦磷酸的合成和异戊二烯焦磷酸合成。通过在含有染色体整合的次丹参酮二烯合成酶的菌株中用不同的质粒表达每个模块,成功地增加了前体供应,将次丹参酮二烯产量提高到 488 mg·L-1。
以上这些例子说明,模块的结构、目标化合物的种类以及宿主的范围呈现出多样性,显示了该方法广泛的适用性。总的来说,该技术为代谢工程提供了一个通用的、简单有效的半组合菌株优化工具。
2 酶的支架技术
代谢途径的中间产物可能对宿主产生毒性,被竞争途径消耗,或通过分泌丢失。解决这些问题的一个新兴的策略是将合成途径的酶组合成多酶复合物。该策略将途经中的酶共定位形成最优比例的复合物,可以增加途径代谢产物和酶的局部浓度,限制途径中间体的积累,减少与其他细胞成分之间不必要反应的发生。多酶复合物在自然界中是常见的。例如,催化色氨酸合成的最后两个步骤的酶可以形成复合物,中间产物吲哚通过一个通道由一个活性位点到达另一个[13]。聚酮和脂肪酸合成相关酶是多酶复合物形成的另外的例子[14],中间体在酶之间穿梭最终形成特定长度和化学结构的终产物。
将酶组合成复合物的第一种方法是将催化连续反应的酶进行融合。酶-酶融合策略在某些情况下能够增强途径通量,然而,该策略有3个明显的缺点:①不适用于包含超过两个酶的途径;②融合之后经常会导致一个或者两个酶活性的降低;③不能轻易地改变复合物中酶的比例。酶的合成支架技术为途径中两个或多个酶的共定位提供了新的方法。蛋白质、DNA和RNA都可以作为支架用于酶复合物的形成。
许多信号蛋白包含模块化的蛋白质相互作用域,可以特异性地与其他域或短肽结合。当这些域融合到其他蛋白的N端或者C端时,还可以保持结合功能。携带多个蛋白质相互作用域的支架蛋白可以和带有多肽配体标签的酶相互作用,用于共定位途径中连续的酶。通过改变蛋白支架上结构域的数量,可以控制不同酶的相对比例。因为只需要在每个酶上加一条短肽,因此蛋白质支架技术对酶活性的影响要低很多。甲羟戊酸的生物合成途径包括乙酰乙酰辅酶 A转移酶(AtoB)、羟甲戊二酰辅酶 A(HMG-CoA)合成酶(HMGS)和 HMG-CoA 还原酶(HMGR)。利用该途径,由乙酰辅酶A生产甲羟戊酸,存在 HMGS和HMGR之间流量不平衡的问题,导致中间产物HMG-CoA的积累。HMG-CoA对宿主大肠杆菌细胞具有很强的毒性。通过蛋白质支架将 HMG-CoA产生和消耗酶之间的共定位(HMGS和 HMGR),调节两个酶的比例,使甲羟戊酸的产量提高了10倍。通过另外一个支架分子将途经的第一个酶(AtoB)引入到复合物中,使甲羟戊酸的产量较无蛋白支架的对照提高了77倍[15](图2)。相同的策略被应用于含有3个酶的葡糖二酸合成途径。在该途径中,肌醇氧化酶(MIOX)可以被其底物肌醇激活。通过蛋白支架,增加了MIOX周围肌醇的浓度,进而使葡糖二酸产量提高5倍[16]。
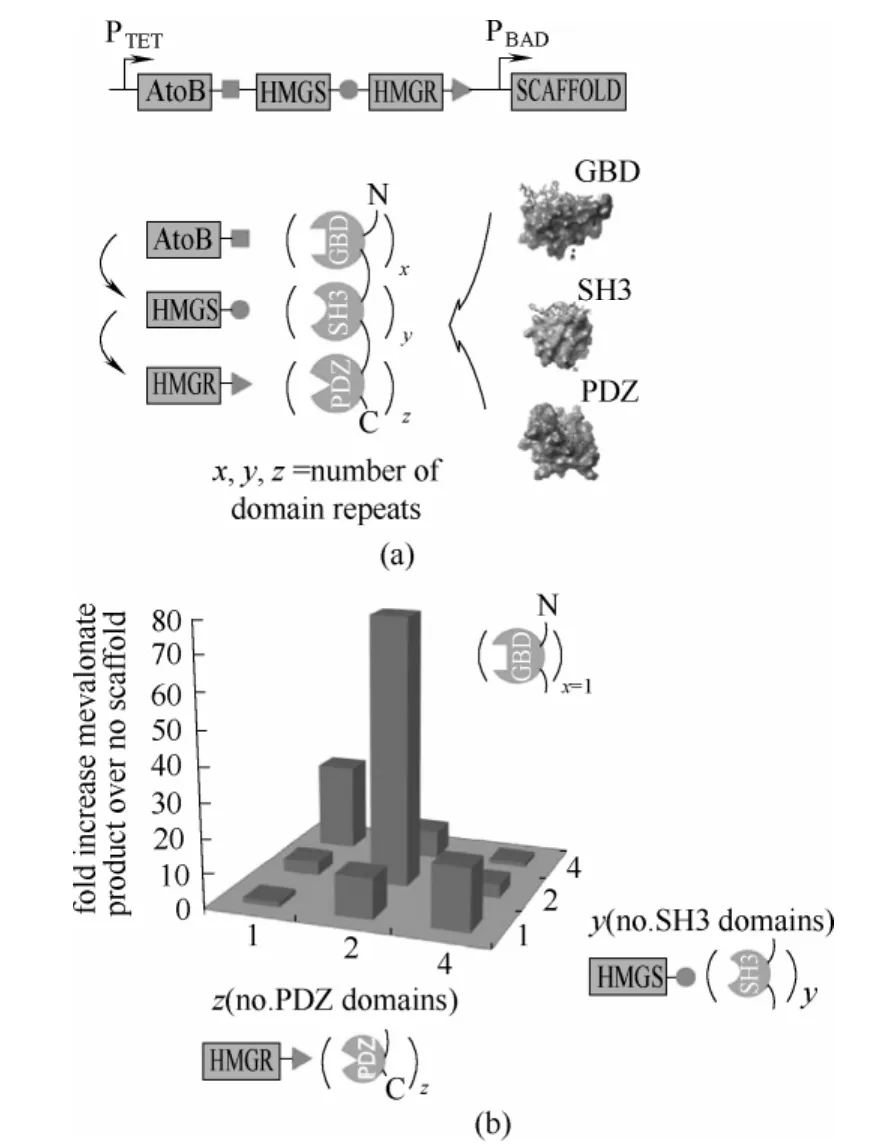
图2 运用蛋白质支架技术优化甲羟戊酸生物合成[15]Fig. 2 Optimization of mevalonate production using protein scaffold technology[15]
类似地,研究者对 DNA分子作为支架用于酶的聚合进行了研究。DNA之间可以通过杂交进行结合,DNA和蛋白质之间可以通过锌指DNA结合域实现相互作用。在一项研究中,葡萄糖氧化酶和辣根过氧化物酶通过赖氨酸残基与 DNA寡核苷酸共价交联,特异性地与多聚六面体 DNA纳米结构杂交。当酶的间距由从4个六面体 (约33 nm)缩短到2个六面体(约13 nm),产物的产量有了显著的增加,表明途径酶彼此靠近确实有利于提高产物的形成[17]。然而,将寡核苷酸共价结合到酶上以及组装DNA纳米结构,操作烦琐,使该策略难以实际应用。Delebecque等[18]构建了序列对称的RNA结构模块,允许聚合成一维或两维RNA支架。每个RNA结构模块中包含PP7和MS2适配体域(aptamer domain),用于结合PP7和MS2融合蛋白。荧光互补实验表明,二维RNA支架在细胞中形成稳定的约100 nm的球形结构,使融合蛋白能够近距离聚集。利用RNA支架进行氢酶和铁氧还蛋白(ferrodoxin)的共定位,使氢气产量提高了约48倍。
3 代谢流量的动态调控技术
在菌株的代谢工程改造过程中,大多数提高产量(yield)的策略往往会减慢菌株的生长速率,进而会导致低容积生产率(productivity),增加工业设备投资费用。在接近最佳理论产量的菌株中,大部分代谢流量被导向产物合成,因此单独优化代谢网络不足以显著提高生长速度。两阶段培养策略将发酵过程分为前期生长阶段和后期产物生成阶段。该方法可以显著改善细胞生长,增加最终产物产量。例如,将乳酸生产过程分成需氧生长阶段和厌氧生产阶段。与单级厌氧策略相比,两阶段策略的生产率可以提高约10倍,达到3.32 g·L-1·h-1[19]。该策略还用于 1,4-丁二醇生产,细胞需氧生长至 OD600达到10,然后切换到微好氧条件并使用IPTG诱导途径基因表达,1,4-丁二醇产量可达18 g·L-1[20]。两阶段发酵已经证明可以成功用于厌氧产品和高价值蛋白的生产;然而,有些产品的途径难以与过程水平参数耦合,可能导致无法使用氧浓度或pH作为触发来切换培养状态,而且使用诱导剂,如IPTG,成本过高。为了解决这些问题,研究者提出了动态控制策略,以允许两阶段发酵和基因表达的动态控制。动态代谢调控主要依靠合成生物学的进展,创造遗传传感器(sensor)和执行器(actuator)。Williams等[21]利用信息素和 RNA干扰技术建立了一个群体感应系统,用于在酿酒酵母中生产对羟基苯甲酸(PHBA)。在发酵初期,通过抑制生产途径基因,表达分支酸变位酶(ARO7)基因,实现菌株正常生长。当达到合适的细胞密度,使用RNA干扰抑制ARO7基因的表达,同时开启生产途径基因的表达,最终PHBA的产量达到1.1 mmol·L-1,是目前报道的酿酒酵母中的最高产量。开关基因控制也可以用温度或诱导剂作为触发器来实现。利用噬菌体Pr和PL启动子控制乳酸脱氢酶基因(ldhA),使得在 33℃生长时ldhA表达受到抑制,与静态策略相比生物量增加了10%。切换到42℃诱导ldhA基因的表达,最终的乳酸产量提高了30%[22]。类似的开关被设计用于生产异丙醇。生长阶段是通过表达柠檬酸合成酶(gltA),抑制通路基因实现高增长率。通过添加IPTG,抑制gltA表达和诱导途径基因表达使流量导向异丙醇生产[23]。
上面的例子都是简单的开-关控制策略,在整个发酵过程只开关一次。连续控制能够动态地感测环境和代谢流的变化,并使细胞对这些变化作出反应。Farmer等[24]利用受乙酰磷酸激活的传感器感应过量的糖酵解流量,动态地控制番茄红素合成基因的表达,提高了番茄红素的产量和生产率。最近,通过动态控制,在检测到毒性代谢物时上调外排泵相关基因的表达,利用合成反馈回路提高产量。Xu等[25]将动态调控策略运用到大肠杆菌脂肪酸的生产中。来自枯草芽孢杆菌的 FapR蛋白能够感知脂肪酸合成前体丙二酸辅酶A的浓度,激活pGAP启动子的转录,抑制 T7启动子的转录。含有代谢开关的工程菌株能够动态地调节上下游基因的表达,使代谢流量有效地导向脂肪酸的合成。通过动态调控,平衡细胞生长和产物合成,与野生菌株相比脂肪酸产量提高了15.7倍(图3)。
通过转录组分析可以寻找和鉴定动态感应器,识别对途径中间体敏感的启动子并利用它们来控制生物合成路径。虽然动态调控策略已有许多成功应用的例子,但是为目标产物寻找合适传感器和执行器可能费时费力。
4 大规模基因编辑技术
在基因组范围内高度特异性的消除、替换或修饰序列的能力具有重要的基础和实际应用,包括关键基因的发现,植物和微生物生物工程和基因治疗等。体内靶基因的编辑可以通过人工蛋白核酸酶异位表达来实现,例如,锌指核酸酶(ZFN)或转录活化剂样的效应核酸酶(TALENS),用于识别特定的目标 DNA位点和引入双链 DNA断裂[26-27]。不同的DNA修复途径可以产生不同类型的突变。在有同源模板存在的条件下,可以通过高保真的同源重组途径进行基因替换、删除或校正。如果不存在同源模板,细胞通常通过相对易错的非同源末端连接来修复双链DNA断裂。这通常会导致基因插入或缺失,因此,可以产生基因的失活突变。
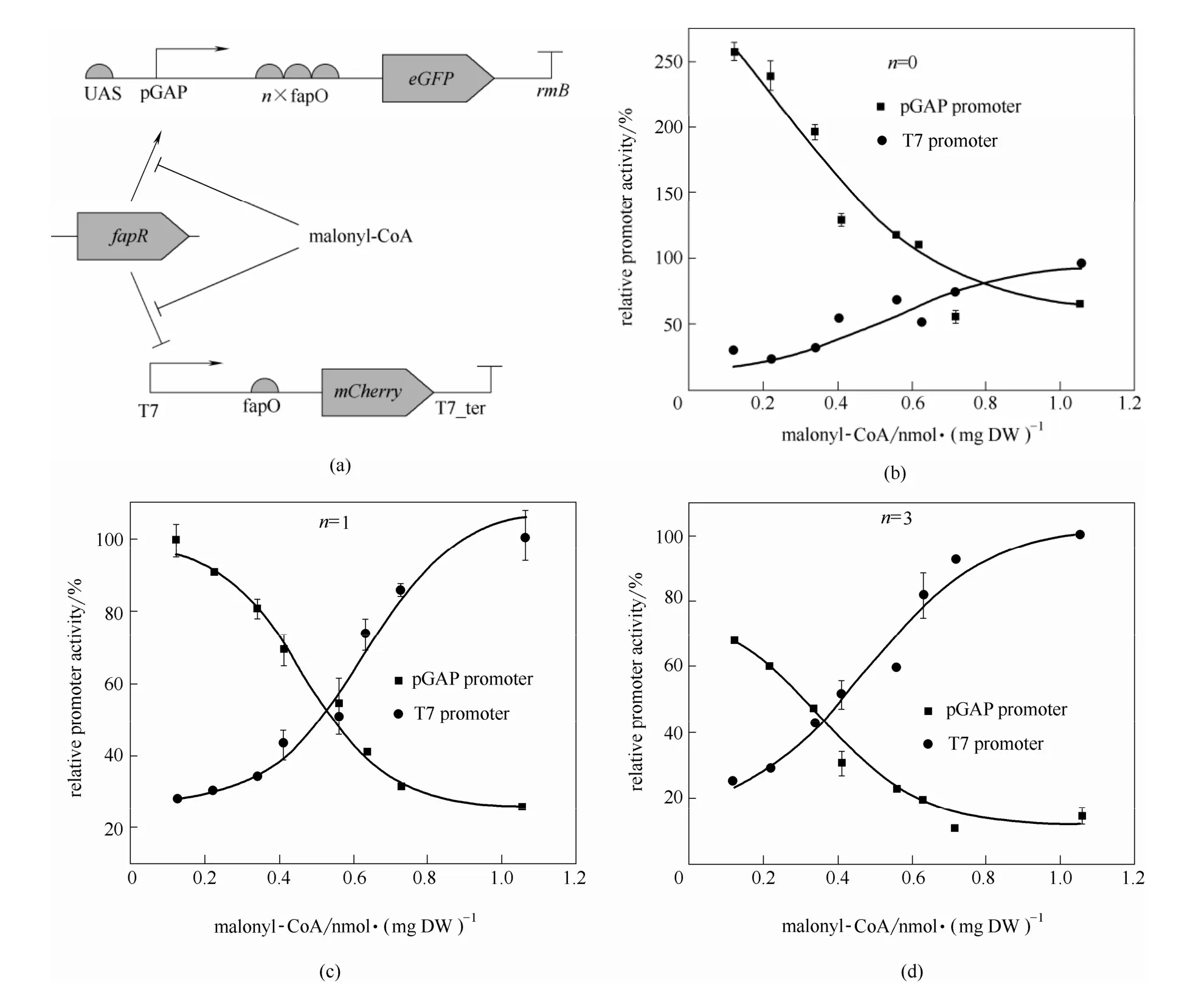
图3 利用动态调控技术优化大肠杆菌脂肪酸合成[25]Fig. 3 Optimization of fatty acid production inE. coliusing dynamic pathway regulation[25]
CRISPR-Cas9系统是最近出现的、可调节的下一代基因组编辑工具[28]。最近几年大量的研究已经成功应用该系统在不同类型的细胞和模式生物中进行RNA指导的基因组编辑。CRISPR-Cas9系统包含单一蛋白质Cas9,是一种RNA指导的核酸内切酶,其催化平末端双链 DNA断裂。该系统需要两种 CRISPR RNA(crRNA):一种通过碱基配对引导Cas9到达 DNA靶序列;另一种反式激活crRNA(tracrRNA),与crRNA配对,在靶DNA裂解过程中具有尚不明确但很重要的作用。这两种RNA可以通过 RNA环连接形成单个指导RNA(sgRNA),使系统简化成只有一个蛋白质(Cas9)和一个RNA(sgRNA)[29](图4)。该系统要求目标位点下游具有一段很短的 PAM 序列。最广泛使用的Cas9酶来自化脓性链球菌。它所需的PAM序列为5′-GG-3′(或 5′-AG-3′,活性略低)并且位于靶位点下游1个碱基处。
Cas9蛋白需要经过密码子优化以促进其在异源细胞或生物体中的表达,而且在真核物种中还需使用核定位信号。与ZFN或TALENS相比,RNA指导的 Cas9基因组编辑在多个系统具有相近的或者更高的效率。此外,基于Cas9的基因组编辑相比蛋白介导的 ZFN和 TALEN系统具有若干优点:Cas9靶向新的DNA位点只需设计与靶DNA互补的sgRNA,使该方法更快捷、更便宜,并且更容易实现,相比之下需要为每个目标位点开发新的ZNF和TALEN蛋白;通过表达多个sgRNA,Cas9平台允许对多个位点同时进行编辑。Jiang等[30]利用RNA指导的 Cas9基因组编辑技术能够在Streptococcus pneumoniae和大肠杆菌引入特定突变,成功率分别为 100%和 65%。通过引入两条crRNAs还可实现多位点同时突变。Keasling课题组[31]利用该技术同时对5个基因位点进行修饰。不需要过表达任何基因,仅通过组合基因敲除,使甲羟戊酸的产量提高了41倍。这些例子充分证明了该技术在原核及真核生物里的普遍适用性及高度位点特异性。
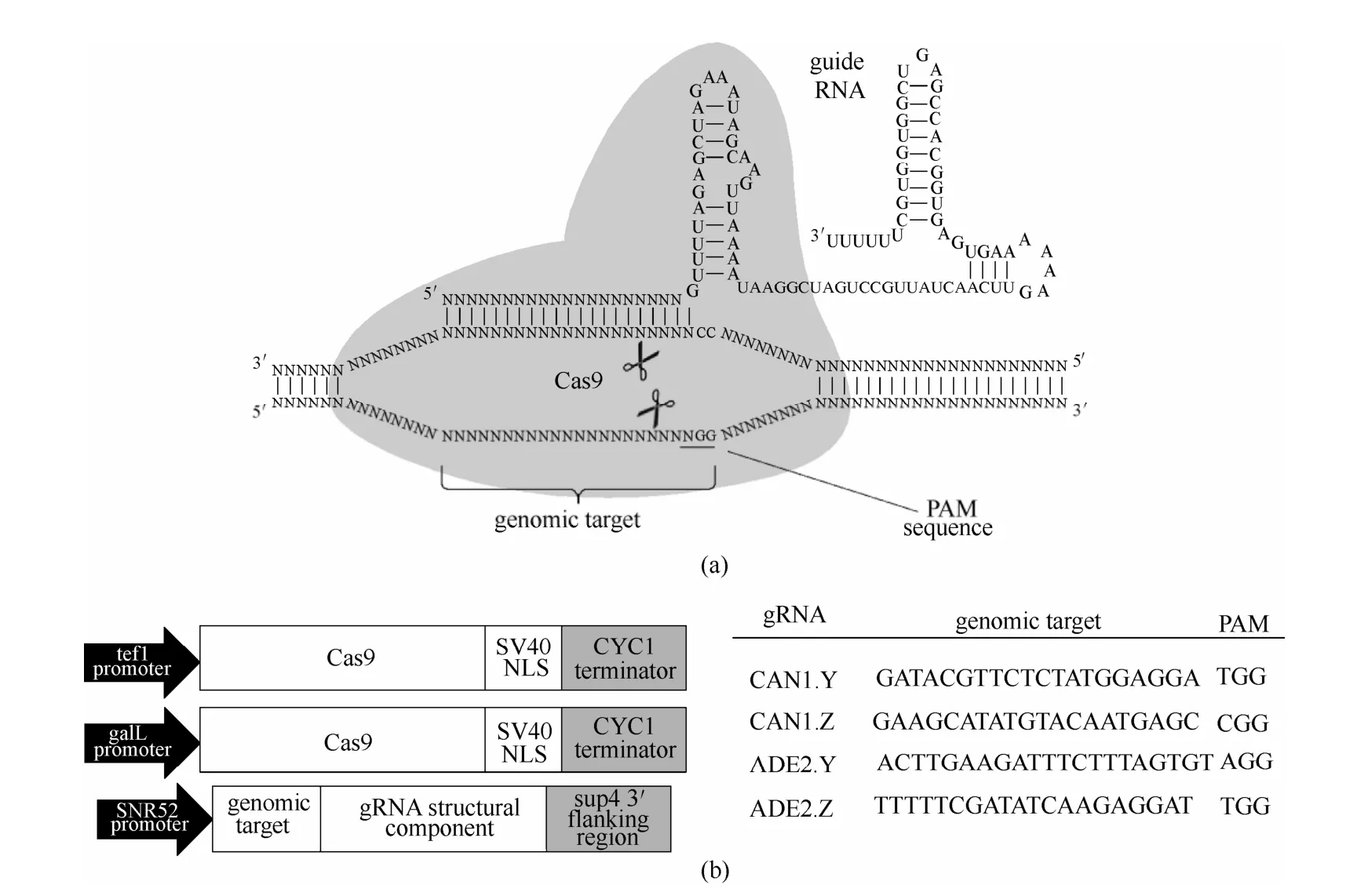
图4 CRISPR-Cas9系统原理示意图[29]Fig. 4 Schematic diagram of CRISPR-Cas9 system[29]
Cas9采用不同的活性位点(RuvC和HNH核酸酶结构域)切割靶 DNA的两条链。通过突变可以使一个位点失去活性,产生只切割一条链的突变Cas9。因此,野生型Cas9可用于双链DNA裂解,而活性位点突变体(RuvC或 HNH)仅切割单链。这将为向基因组中引入可预测的突变提供可靠的方法。如果Cas9的两个核酸酶结构域同时失活,则产生了仅具有 DNA结合功能的突变蛋白。利用这种核酸酶缺陷的 Cas9靶向启动子元件会导致基因转录的明显下降。这种新颖的平台被称为CRISPR干扰(CRISPRi)[32]。核酸酶缺陷Cas9也能与转录抑制子结合位点作用激活基因的表达。Cas9复合物还可以通过与特异性转录激活因子或抑制因子融合,用于激活或沉默基因的表达[33-34]。这些CRISPR转录因子可与sgRNAs一起识别特定的转录控制元件,对目标位点内源基因的表达产生可预测的影响。
5 结 论
本文总结介绍了4种代谢工程的新技术,可以从不同层次水平对菌株和途经进行调节和优化。基因元件的模块化、标准化不仅可以有效地简化途经优化的工作量,而且通过相关模块的简单组合可以组建新的合成途径。目前标准生物部件登记处(Standard Biological Parts registry)数据库已经收集了超过5000个标准化的部件。该数据库的进一步丰富和发展将会极大地推动人工代谢途径的设计和构建。酶的支架技术从翻译后水平调节酶之间的空间组织顺序和比例,以较低的表达量实现较高的催化效率,还可以减少与细胞其他成分不必要的相互作用。代谢流量的动态调控技术通过感应及效应元件,根据细胞的不同生长阶段及代谢物水平,调节基因的转录表达,平衡细胞的生长和产物合成,可以有效地提高产量和产率。为了进一步提高其有效性和适用性,还需要寻找具有更高鲁棒性(robustness)和快速反应性的调控元件。CRISPR-Cas9是一种强大的基因组编辑和基因表达调控工具。然而,对该系统的认识还很不完整,该技术的其他工具和应用还会不断出现。总之,这些新技术的进一步发展必将为代谢工程增添新的活力,降低发酵成本,提高产品的多样性,为各种化合物的绿色生产奠定基础。
[1] Lo T M, Teo W S, Ling H, Chen B, Kang A, Chang M W. Microbial engineering strategies to improve cell viability for biochemical production [J].Biotechnol. Adv., 2013, 31:903-914.
[2] Yadav V G, De Mey M, Lim C G, Ajikumar P K, Stephanopoulos G.The future of metabolic engineering and synthetic biology:towards a systematic practice [J].Metab. Eng., 2012, 14:233-241.
[3] Inokuma K, Liao J C, Okamoto M, Hanai T. Improvement of isopropanol production by metabolically engineeredEscherichia coliusing gas stripping [J].J. Biosci. Bioeng., 2010, 110:696-701.
[4] Becker J, Zelder O, Häfner S, Schröder H, Wittmann C. From zero to hero—design-based systems metabolic engineering ofCorynebacterium glutamicumfor L-lysine production [J].Metab.Eng., 2011, 13:159-168.
[5] Ajikumar P K, Xiao W H, Tyo K E J, Wang Y, Simeon F, Leonard E,Mucha O, Phon T H, Pfeifer B, Stephanopoulos G. Isoprenoid pathway optimization for taxol precursor overproduction inEscherichia coli[J].Science, 2010, 330:70-74.
[6] Xu P, Gu Q, Wang W, Wong L, Bower A G, Collins C H, Koffas M A.Modular optimization of multi-gene pathways for fatty acids production inE. coli[J].Nat. Commun., 2013, 4:1409.
[7] Liu Y, Zhu Y, Li J, Shin H D, Chen R R, Du G, Liu L, Chen J.Modular pathway engineering ofBacillus subtilisfor improvedN-acetylglucosamine production [J].Metab. Eng., 2014, 23:42-52.
[8] Lin Y, Sun X, Yuan Q, Yan Y. Extending shikimate pathway for the production of muconic acid and its precursor salicylic acid inEscherichia coli[J].Metab. Eng., 2014, 23:62-69.
[9] Wu J, Liu P, Fan Y, Bao H, Du G, Zhou J, Chen J. Multivariate modular metabolic engineering ofEscherichia colito produce resveratrol from L-tyrosine [J].J. Biotechnol., 2013, 167:404-411.
[10] Wu J, Du G, Zhou J, Chen J. Metabolic engineering ofEscherichia colifor (2S)-pinocembrin production from glucose by a modular metabolic strategy [J].Metab. Eng., 2013, 16:48-55.
[11] Zhao J, Li Q, Sun T, Zhu X, Xu H, Tang J, Zhang X, Ma Y.Engineering central metabolic modules ofEscherichia colifor improving beta-carotene production [J].Metab. Eng., 2013, 17:42-50.
[12] Dai Zhubo, Liu Yi, Huang Luqi, Zhang Xueli. Production of miltiradiene by metabolically engineeredSaccharomyces cerevisiae[J].Biotechnology and Bioengineering, 2012, 109(11):2845-2853.
[13] Miles E W. Tryptophan synthase:a multienzyme complex with an intramolecular tunnel [J].Chem. Rec., 2001, 1:140-151.
[14] Smith S, Tsai S C. The type Ⅰ fatty acid and polyketide synthases:a tale of two megasynthases [J].Nat. Prod. Rep., 2007, 24:1041-1072.
[15] Dueber J E, Wu G C, Malmirchegini G R, Moon T S, Petzold C J,Ullal A V, Prather K L, Keasling J D. Synthetic protein scaffolds provide modular control over metabolic flux [J].Nat. Biotechnol.,2009, 27:753-759.
[16] Moon T S, Dueber J E, Shiue E, Prather K L J. Use of modular,synthetic scaffolds for improved production of glucaric acid in engineeredE. coli[J].Metab. Eng., 2010, 12:298-305.
[17] Wilner O I, Weizmann Y, Gill R, Lioubashevski O, Freeman R,Willner I. Enzyme cascades activated on topologically programmed DNA scaffolds [J].Nat. Nanotechnol., 2009, 4:249-254.
[18] Delebecque C J, Lindner A B, Silver P A, Aldaye F A. Organization of intracellular reactions with rationally designed RNA assemblies [J].Science, 2011, 333:470-474.
[19] Zhou L, Zuo Z R, Chen X Z, Niu D D, Tian K M, Prior B A, Shen W,Shi G Y, Singh S, Wang Z X. Evaluation of genetic manipulation strategies on D-lactate production byEscherichia coli[J].Curr.Microbiol., 2011, 62:981-989.
[20] Yim H, Haselbeck R, Niu W, Pujol-Baxley C, Burgard A, Boldt J,Khandurina J, Trawick J D, Osterhout R E, Stephen R, Estadilla J,Teisan S, Schreyer H B, Andrae S, Yang T H, Lee S Y, Burk M J, Van Dien S. Metabolic engineering ofEscherichia colifor direct production of 1,4-butanediol [J].Nat. Chem. Biol., 2011, 7:445-452.
[21] Williams T C, Averesch N J H, Winter G, Plan M R, Vickers C E,Nielsen L K, Krömer J O. Quorum-sensing linked RNA interference for dynamic metabolic pathway control inSaccharomyces cerevisiae[J].Metab. Eng., 2015, 29:124-134.
[22] Zhou L, Niu D D, Tian K M, Chen X Z, Prior B A, Shen W, Shi G Y,Singh S, Wang Z X. Genetically switched D-lactate production inEscherichia coli[J].Metab. Eng., 2012, 14:560-568.
[23] Soma Y, Tsuruno K, Wada M, Yokota A, Hanai T. Metabolic flux redirection from a central metabolic pathway toward a synthetic pathway using a metabolic toggle switch [J].Metab. Eng., 2014, 23:175-184.
[24] Farmer W R, Liao J C. Improving lycopene production inEscherichia coliby engineering metabolic control [J].Nat. Biotechnol., 2000, 18:533-537.
[25] Xu P, Li L, Zhang F, Stephanopoulos G, Koffas M. Improving fatty acids production by engineering dynamic pathway regulation and metabolic control [J].Proc. Natl. Acad. Sci. U. S. A., 2014, 111:11299-11304.
[26] Urnov F D, Rebar E J, Holmes M C, Zhang H S, Gregory P D.Genome editing with engineered zinc finger nucleases [J].Nat. Rev.Genet., 2010, 11:636-646.
[27] Joung J K, Sander J D. TALENs:a widely applicable technology for targeted genome editing [J].Nat. Rev. Mol. Cell Biol., 2013, 14:49-55.
[28] Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea:versatile small RNAs for adaptive defense and regulation [J].Annu. Rev. Genet., 2011, 45:273-297.
[29] DiCarlo J E, Norville J E, Mali P, Rios X, Aach J, Church G M.Genome engineering inSaccharomyces cerevisiaeusing CRISPR-Cas systems [J].Nucleic Acids Res., 2013, 41:4336-4343.
[30] Jiang W, Bikard D, Cox D, Zhang F, Marraffini L A. RNA-guided editing of bacterial genomes using CRISPR-Cas systems [J].Nat.Biotech., 2013, 31:233-239.
[31] Jakočiūnas T, Bonde I, Herrgård M, Harrison S J, Kristensen M,Pedersen L E, Jensen M K, Keasling J D. Multiplex metabolic pathway engineering using CRISPR/Cas9 inSaccharomyces cerevisiae[J].Metab. Eng., 2015, 28:213-222.
[32] Qi Lei S, Larson Matthew H, Gilbert Luke A, Doudna Jennifer A,Weissman Jonathan S, Arkin Adam P, Lim Wendell A. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression [J].Cell, 152:1173-1183.
[33] Cheng A W, Wang H, Yang H, Shi L, Katz Y, Theunissen T W,Rangarajan S, Shivalila C S, Dadon D B, Jaenisch R. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system [J].Cell Res., 2013, 23:1163-1171.
[34] Gilbert L A, Larson M H, Morsut L, Liu Z, Brar G A, Torres S E,Stern-Ginossar N, Brandman O, Whitehead E H, Doudna J A, Lim W A, Weissman J S, Qi L S. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes [J].Cell, 2013, 154:442-451.

