HRM分析方法中不同扩增片段、染料和仪器的比较
饶 丹,嘎利兵嘎,郭小权,张建峰,张春红,沈海燕,
朱余军2,刘志成2 ,孙俊颖2,郭鹏举2*
(1.江西农业大学动物科学技术学院,江西南昌330045;2.广东农科院动物卫生研究所,广东广州510640)
HRM分析方法中不同扩增片段、染料和仪器的比较
饶丹1,2,嘎利兵嘎2,郭小权1*,张建峰2,张春红2,沈海燕2,
朱余军2,刘志成2,孙俊颖2,郭鹏举2*
(1.江西农业大学动物科学技术学院,江西南昌330045;2.广东农科院动物卫生研究所,广东广州510640)
摘要:高分辨率熔解曲线技术(High resolution melting,HRM)是近年发展起来的基于实时荧光定量技术分析核苷酸突变而进行基因分型的一种新型技术。比较了几种动物疫病病原(猪瘟病毒、禽白血病病毒、鸡艾美耳球虫株)的不同扩增片段(C202、ALV450、En573和C965)、不同荧光染料(LC Green、SYTO9 和SYBR GreenⅠ)和不同仪器[LightScanner96(Idaho), LightCycler480(Roche)和Rotor-Gene Q(Qiagen)]在HRM基因分型上的异同,揭示了HRM技术对病原体进行分型时,不同扩增片段和不同性质荧光染料对其分型结果影响较大:拥有多个熔解区间的核酸序列比单独1个SNP位点特异性高,更适合于分型;核酸相邻熔解区间Tm差值影响实际HRM分型结果;饱和荧光染料(LC Green和SYTO9)比非饱和染料(SYBR GreenⅠ)分辨率更高,更适合用于HRM分析,并且不同浓度荧光染料对Tm值有一定的影响;不同HRM仪器之间HRM结果无明显差异。
关键词:高分辨率熔解曲线;单核苷酸多态性;荧光染料;熔解Tm;熔解曲线模拟
The Comparison of Amplified Fragments,Different Dyes
高分辨率熔解曲线技术(HRM)是美国Utah大学Wittwer等人在2003年首次提出的基于新型饱和荧光染料LC Green的发明而进行基因突变检测的新技术[1]。HRM技术最初应用于单核苷酸多态(single nucleotide polymorphism,SNP)检测分析,HRM-SNP检测原理是根据熔解温度的微弱变化来区分野生型和纯合子突变单碱基差异,根据熔解曲线的形态变化来区分纯合子和杂合子之间的单碱基差[2]。由于大多数病原体尤其病毒核苷酸不存在纯合子和杂合子之分,单独1个SNP位点又往往无法提供足够多的分型信息。因此,在利用HRM技术对病原体进行分型时,选取合适大小的扩增片段使其含有足够多的分型所需序列非常关键;如所选取的扩增片段太小,只出现一个熔解峰不利于基因的分型和诊断;而扩增片段太大时,因GC分布不均匀出现多个熔解峰使基因分型复杂化。目前已有HRM在线模拟软件可为不同的用户提供HRM分析服务,利用HRM模拟软件对所要扩增的目的片段进行熔解曲线预测可极大地提高基因分型的成功率。
在利用HRM技术对病原体进行分型时除了要选取合适大小的目的片段外也要考虑不同仪器设备和不同荧光染料对分型结果的影响。目前,用于HRM检测的仪器主要 LightScanner96/384(Idaho),LightCycler480(Roche)和Rotor-Gene Q(Qiagen);常用的商品化饱和染料有Eva Green、LC Green和SYTO9等。近年来虽然有文献报道不同操作平台间、不同饱和染料间,HRM检测性能存在一定差异[3-4],但这些都是关于不同仪器设备和不同饱和染料对SNP检测方面的影响。国内尚无有关HRM在病原体分型方面的比较报告。本文拟利用HRM在线模拟软件对所扩增目的片段进行HRM分型评估,比较不同扩增片段、仪器和荧光染料在HRM病原体分型方面的差异,为HRM技术在病原基因分型和兽医分子诊断方面的应用提供参考依据。
1材料与方法
1.1主要试剂样品与仪器
RNA/DNA提取试剂盒、Premix ExTaq酶、pMD-18T载体、DNA Marker DL 1 000、反转录酶M-MLV等,购自大连TaKaRa公司;SYBR GreenⅠ、LC Green、SYTO9分别来自Sigama公司、美国Idaho公司、美国Invitrogen公司。主要仪器:Light Scanner96(Idaho)、LightCycler480(Roche)和Rotor-Gene Q(Qiagen)。
猪瘟野毒株和禽白血病毒株(本研究室保存)、艾美耳球虫株(本单位寄生生物研究室)。
1.2RNA病毒(CSFV和ALV)反转录
扩增的四个片段中,扩增猪瘟病毒两个片段(202 bp、965 bp)和禽白血病病毒片段(450 bp)前需用相应引物先进行反转录。艾美耳球虫株片段(573 bp)PCR反应直接获得。合成cDNA的反转录程序是:加有5 μL RNA、1.5 μL下游引物(10 μmol/L)的体系在70 ℃保温10 min后迅速在冰上急冷2 min以上;在体系中加入5×M-MLV Buffer 2 μL、dNTP(2.5 mmol/L)1 μL、0.25 μL RNase Inhibitor(40 U/μL)和M-MLV(200 U/μL),42 ℃保温1 h、95 ℃ 5 min。
1.3质粒构建
构建插入片段大小为202 bp(C202)和965 bp(C965)的猪瘟野毒株片段质粒、573 bp(En573)的艾美耳球虫片段质粒和450 bp(ALV450)的禽白血病病毒(ALV450)片段质粒,将鉴定为阳性的每份质粒样品送测序(北京奥科鼎盛生物科技有限公司测序部)。
1.4溶解曲线在线模拟
分别上传C202、C965、En573和ALV450目的片段序列至HRM在线模拟窗口,预测每个目的片段熔解曲线形状和相应Tm值(https://www.dna.utah.edu/umelt/um.php)。
1.5不同染料、扩增片段和不同仪器间的HRM比较
本研究选取不同荧光染料(SYBR Green Ⅰ、LC Green和SYTO9),不同扩增片段(C202、C905、En573和ALV450)和不同仪器(LightScanner 96,LightCycler480和Rotor-Gene Q)来进行HRM比较。具体步骤如下:PCR反应体系(20 μL):dH2O 5.4 μL,Premix ExTaq10 μL,分别加相应的上游引物F(10 μmol/L)0.8 μL和下游引物R(10 μmol/L)0.8 μL,荧光染料1 μL,cDNA或DNA模板2 μL(浓度已优化)。C202 PCR程序:94 ℃预变性5 min;94 ℃变性30 s、54 ℃退火30 s、72 ℃延伸30 s,循环35次;72 ℃终延伸8 min。En573/ALV450 PCR程序:94 ℃预变性5 min;94 ℃变性35 s、54 ℃退火35 s、72 ℃延伸40 s,循环35次;72 ℃终延伸8 min。C965 PCR程序:94 ℃预变性5 min;94 ℃变性35 s、54 ℃退火35 s、72 ℃延伸1 min,循环35次;72 ℃终延伸8 min。SYBR GreenⅠ(Sigma USA)是由Sigma公司生产的10 000×的溶液(DMSO),LC Green(Idaho USA)提供的是10×的溶液,而SYTO9(Invitrogen USA)为5 mmol/L的溶液(DMSO)。为了使实验数据具有可比性,分别优化三种荧光染料浓度使其荧光信号大小一致,即三种荧光染料原液稀释后在PCR反应液的终浓度分别为SYBR GreenⅠ1×、LC Green 0.2×、SYTO9 1.25 μmol/L(浓度表示方式参照说明书)。PCR扩增后将PCR产物移至LightScanner 96(Idaho)仪器进行HRM分析。质粒样品ALV450在PCR扩增后依次用LightScanner 96(Idaho),LightCycler480(Roche)和Rotor-Gene Q(Qiagen)仪器进行HRM分析,比较不同设备对实验结果的影响。
2结果与分析
2.1PCR扩增产物凝胶电泳结果
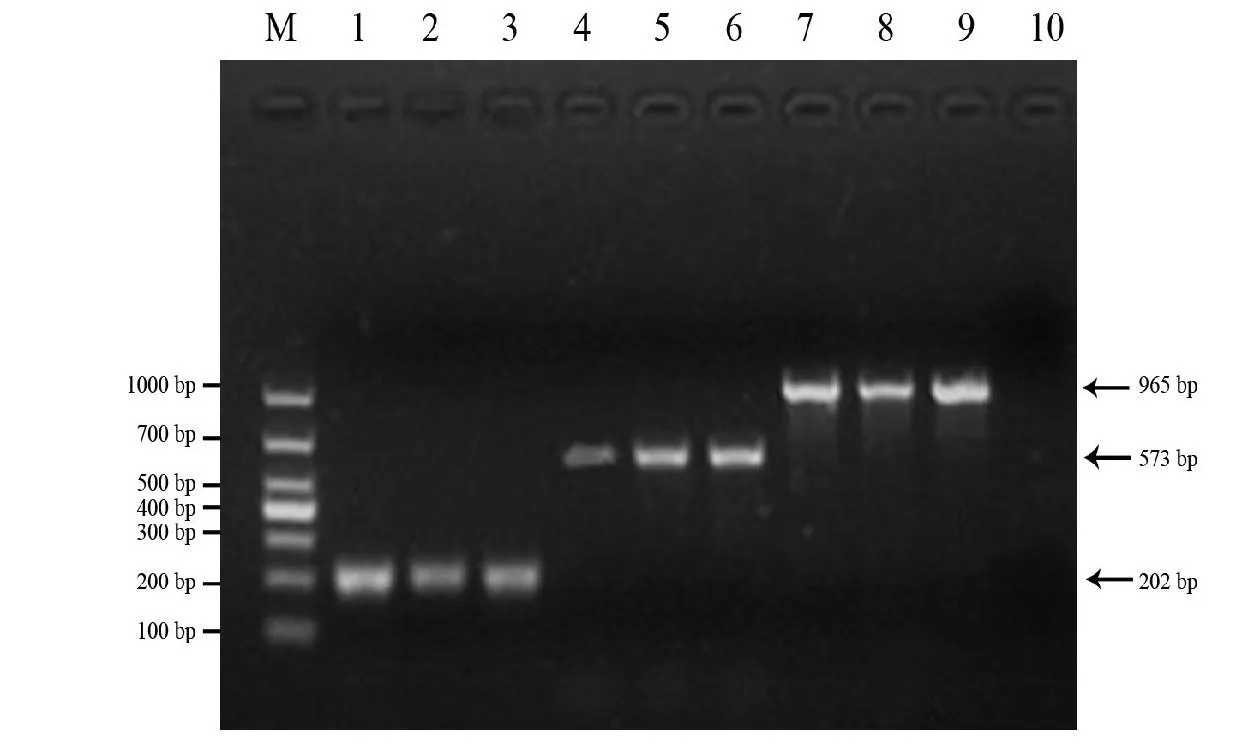
M:DL 1 000;1-3:C202(SYBR Green Ⅰ、LC Green、SYTO9);4-6:En573(SYBR Green Ⅰ、LC Green、SYTO9);7-9:C965(SYBR Green Ⅰ、LC Green、SYTO9);10:阴性对照。图1 加有核酸染料的3种DNA模板的 PCR产物电泳图Fig.1 Agarose gel electrophoresis of three different DNA amplicons with the addition of nucleonic acid dye
为了消除非特异性扩增对HRM分析的影响,对每个PCR扩增片段进行凝胶电泳分析,如图1。电泳分析结果表明,3组片段均扩增出预期目的片段,无非特异性扩增,良好的PCR扩增为后续HRM分析提供了保证。
2.2核酸序列熔解曲线模拟结果
C202、C905、En573和ALV450扩增序列HRM模拟结果如图2。HRM模拟图中,不同序列之间熔解曲线差异明显;C202具有熔解温度差值较大的两个熔解峰,En573、C965和ALV450均有3个熔解峰,其中En573在高温区有更高的熔解峰,而ALV450高温区熔解峰相对较低。
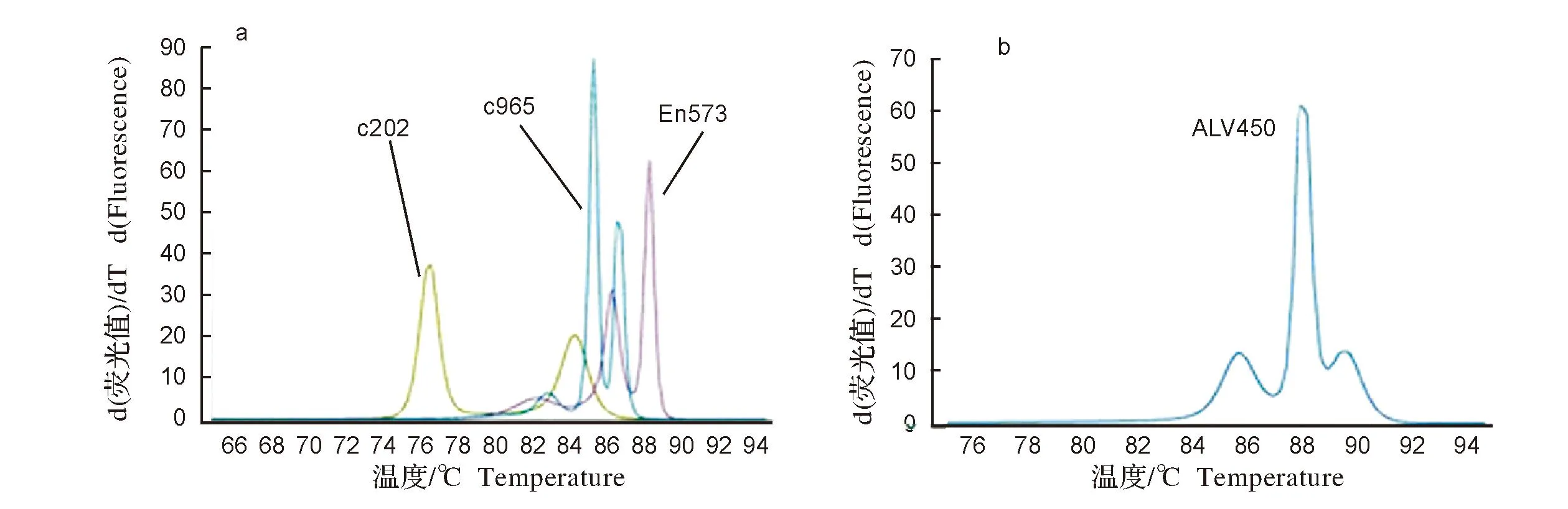
图2 4组DNA片段测序序列在线模拟软件熔解曲线峰型图Fig.2 Conventional melting curves by online simulation software for sequence of four DNA fragments
2.3不同染料和扩增片段对HRM结果的影响
研究结果(图3)表明加有饱和染料的C202和En573 HRM实验结果与理论模拟相似,而加有非饱和染料SYBR GreenⅠ的反应体系HRM结果与理论模拟具有明显差异,这种差异主要是由SYBR GreenⅠ染料在低温区熔解曲线分辨率不高的因素导致。而扩增片段大小为965 bp的片段,无论是在饱和荧光染料还是非饱和荧光染料条件下,其HRM实验结果与理论模拟均有差异,即理论模拟时C965序列有3个熔解峰,而实际HRM结果只有2个熔解峰且第1个峰不明显。不同荧光染料除了对熔解曲线形状有影响外,对熔解温度(Tm值)也有一定的影响。标准化熔解曲线图(图3.a1、a2和a3)中,同一样品分别在3种不同荧光染料下的熔解温度有微弱变化;使用SYBR GreenⅠ时熔解温度为最高,其次为LC Green,最低为SYTO9。此外,随着荧光染料浓度的升高,扩增片段的熔解温度也有相应升高的趋势。
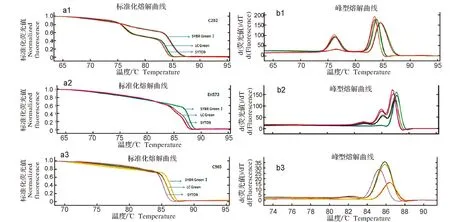
图3 C202 、En573和C965 PCR产物的标准熔解曲线和峰型熔解曲线Fig.3 Normalized and conventional melting curves of PCR amplifications of C202/ En573/ C965
本试验中用LC Green和SYTO9进行比较,两者都是饱和染料因而更具可比性,扩增模板为En573。如在LC Green不同浓度实验中染料浓度最高(1×)时其熔解温度变化最大。在SYTO9不同浓度实验中,染料浓度最高(5 μmol/L)时其溶解曲线信号与第2浓度(2.5 μmol/L)荧光信号相当但Tm值依旧最大,推测是由于染料浓度过高降低了PCR扩增效率使其荧光信号受到影响(图4)。
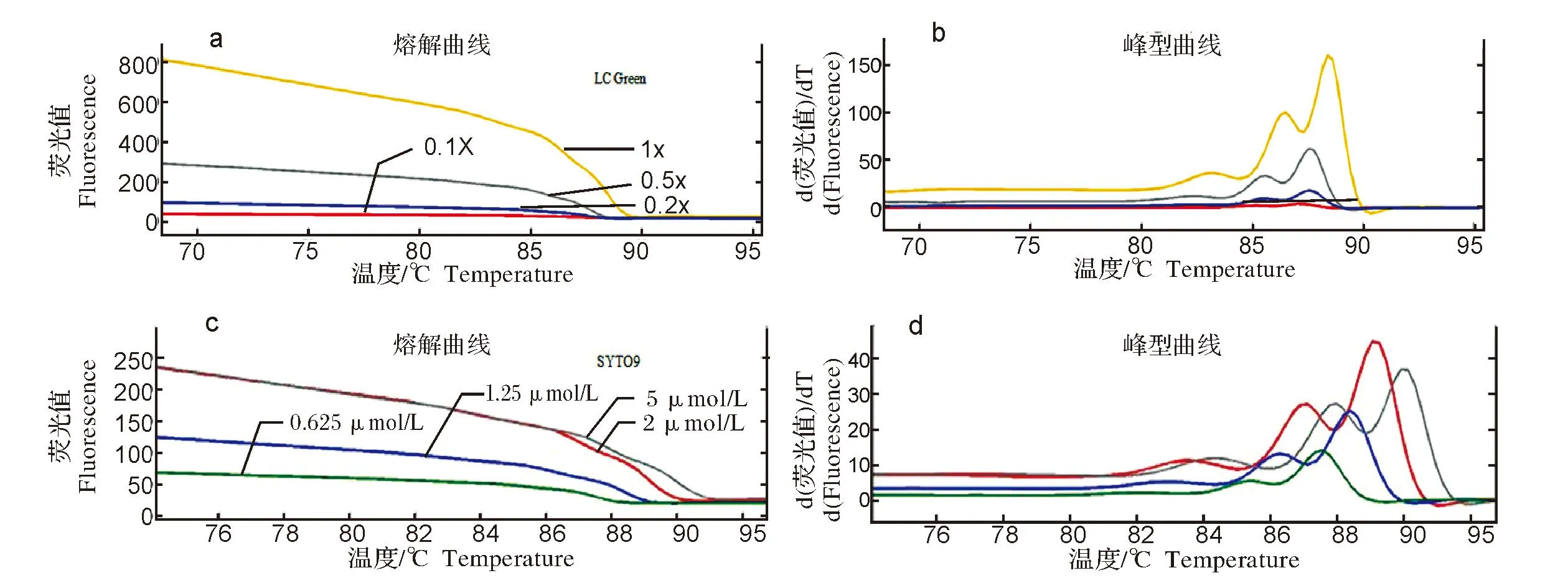
图4 不同荧光染料浓度下熔解曲线的比较Fig.4 The comparison of melting curves under fluorescent dyes with different concentrations
2.4不同仪器对HRM结果的影响
不同仪器HRM结果如图5, LightScanner96(Idaho),LightCycler480(Roche) 和 Rotor-Gene Q(Qiagen)3种不同仪器之间HRM结果无明显差别。样品ALV450在3种不同仪器上均出现两个熔解峰,其中高温区熔解峰明显变宽。而HRM模拟结果显示,ALV450序列在高温区有两个熔解峰(图2),表明ALV450序列高温区熔解峰的变化可能导致了其HRM理论模拟与实际结果的不同。
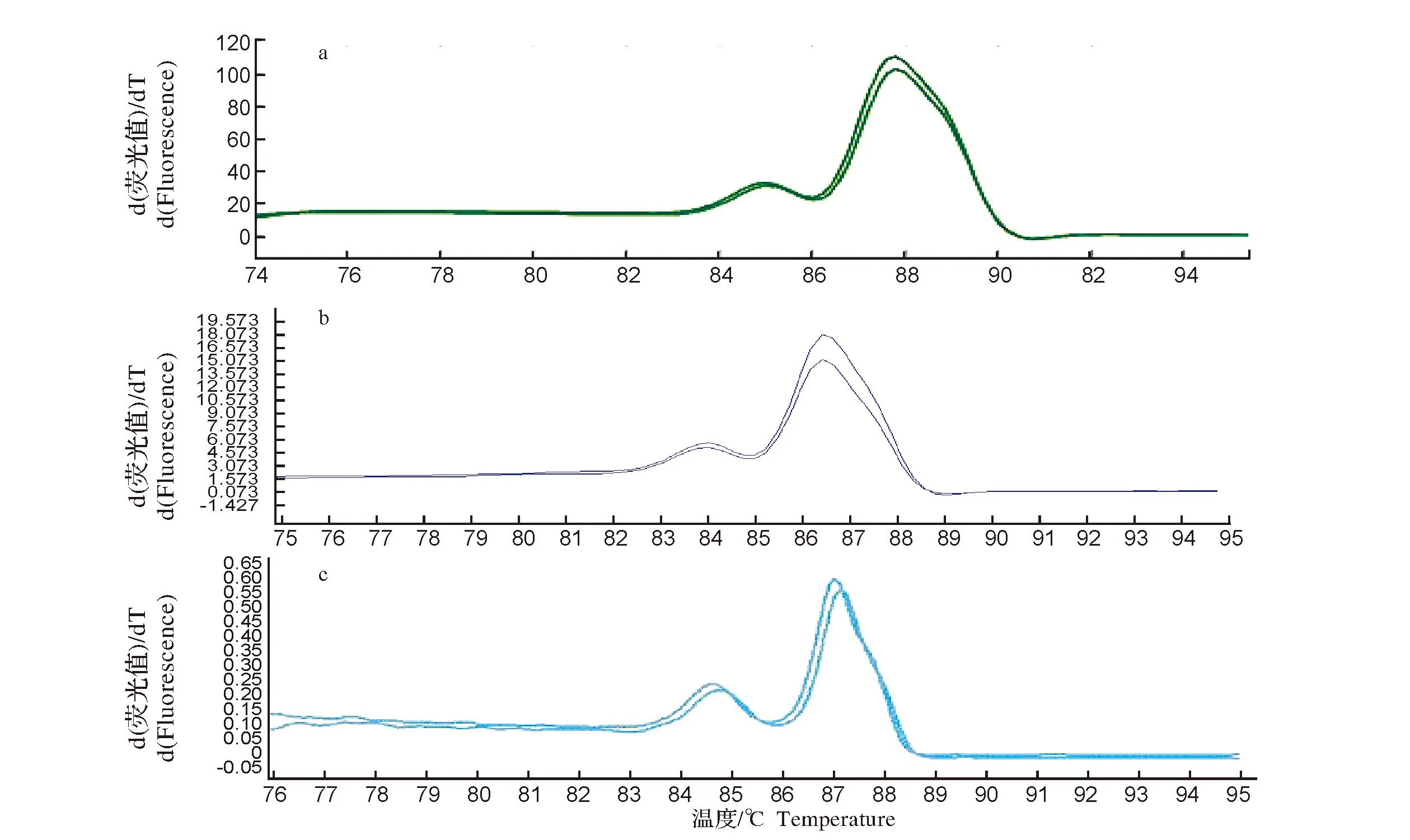
图5 3种仪器下峰型熔解曲线的比较Fig.5 A comparison of conventional melting curves generated with three HRM analyzers
3讨论
近年来随着附带HRM检测功能的 real-time PCR仪的普及,HRM基因分型技术在分子诊断领域受到普遍的关注。研究者利用HRM技术建立了鸡传染性支气管炎病毒、鸡传染性囊病病毒、禽腺病毒和鸡毒支原体的 PCR-HRM 检测方法[5-9],检测牛奶样品中葡萄球菌的PCR-HRM方法[10],通过对细菌16S rRNA基因序列进行HRM分析建立了快速鉴定奶牛乳房炎病原菌PCR-HRM技术[11]。与HRM技术应用于单个SNP位点检测相比,利用HRM技术对病原体进行分型时选取合适大小的扩增片段最为关键。扩增片段过小时,由于只出现单个熔解峰不利于分型,扩增片段过大时由于GC分布不均匀出现多个熔解峰使基因分型复杂化。为了提高HRM技术在病原体分型上的成功率,使这一技术更好应用于动物疫病分型及检测,本文利用HRM在线模拟软件对所要扩增的目的片段进行了熔解曲线预测。实际扩增片段HRM结果(如图3)表明,在线模拟软件对不同大小扩增片段具有较好的预测能力。其中片段大小为202 bp和573 bp的两个片段的HRM结果与预测的一致,而片段大小为965 bp时HRM实验结果与模拟结果有一定的差异。从C965 HRM图上可看出,溶解曲线只出现两个熔解峰,而不是理论模拟时的3个熔解峰。这种现象可能是由于965 bp序列高温区2个熔解峰之间的Tm差值过小所导致。如573 bp序列高温区2个熔解峰之间的Tm差值为2.1 ℃,而965 bp序列熔解峰Tm差值只有1.35 ℃(图2)。为了验证熔解峰之间Tm值差异会影响实际样品HRM结果,本研究设计了熔解峰Tm差值为1.75 ℃的DNA片段ALV450。实验结果显示,由于ALV450熔解峰Tm差值在1.35~2.1 ℃之间,高温区出现了较宽的溶解峰(如图5)。为了进一步排除不同仪器设备对这一实验结果的影响,比较了LightScanner 96(Idaho),LightCycler480(Roche)和Rotor-Gene Q(Qiagen)三种常用的HRM分析仪器对实验结果的影响。不同仪器结果显示ALV450 HRM结果在3种仪器之间无明显差异,如图5。因此,理论熔解峰之间的Tm差值是影响实际HRM结果的主要因素。
除了比较扩增片段大小对HRM分型结果的影响外,本研究分析了3种不同荧光染料(LC Green、SYTO9 和SYBR GreenⅠ)在HRM基因分型结果上的异同。实验结果表明,在熔解曲线形态变化上饱和荧光染料(LC green和SYTO9)HRM图形更接近理论模拟图,而非饱和荧光染料(SYBR GreenⅠ)HRM结果与理论模拟图之间有明显差异(如图3);不同荧光染料之间的这种差异是和染料本身特有的性质有关。非饱和染料SYBR GreenⅠ对PCR反应存在抑制作用[12-14],故在实验中的使用浓度很低,远低于将DNA双螺旋结构中的小沟饱和的浓度,由于使用浓度未达到饱和,在DNA双链解链的过程中,SYBR GreenⅠ分子会发生重排,那些从已经解链的DNA片段上脱离下来的染料分子又与尚未解链的双链DNA结合,造成结果失真。此外,SYBR GreenⅠ易于GC含量高的双链区域结合,从而它对高温区具有较高的特异性[15],而对低温区熔解曲线分辨率较差(如图3)。由于饱和染料如LC green和SYTO9对PCR反应抑制作用极低[1,16-17],在DNA解链过程中不会发生重排,这使得用这些染料的熔解曲线有了更高的分辨率。不同染料对熔解曲线形状有影响外,对熔解温度也有一定的影响。如在本研究中,不同染料扩增片段Tm值之间存在差异,使用SYBR GreenⅠ时Tm值最高,其次为LC green,使用SYTO9时Tm值最低,表明不同染料结合DNA双链能力存在差异。除了染料性质对Tm有影响外,染料不同浓度对Tm值的影响也应当考虑。如在本研究中,随着染料浓度的升高,Tm值也有相应升高的趋势(如图4);这与Paul T、Monis等[18]比较SYTO9和SYBR GreenⅠ染料结果一致。
本研究利用HRM技术比较了不同扩增片段、染料和仪器在HRM病原体分型方面的异同。实验结果表明,利用HRM技术对病原体进行分型时片段大小和染料性质对其影响较高,因此实际应用时应考虑以下几点:①拥有多个熔解区间的核酸序列比单独1个SNP位点特异性高,更适合于分型(HRM分析推荐片段长度200~600 bp);②HRM模拟分析可以提高分型成功率,但应考虑多个熔解区间Tm差值对实际结果的影响,如溶解峰之间的△Tm不宜过小(<1.35 ℃);③饱和荧光染料之间分型结果无明显差异,但不同浓度染料对Tm值影响较大(在适宜范围内随染料浓度升高,溶解曲线峰Tm值升高;LC Green推荐使用1×浓度以获得高的信号;SYTO9推荐使用量为2.5 μmol/L);④不同HRM仪器之间分型结果无明显差异,比较的3种仪器[(Light Scanner96(Idaho)、LightCycler480(Roche)和Rotor-Gene Q(Qiagen)]都适合运用于HRM病原体基因分型。
致谢:感谢广东省农科院动物卫生研究所生物技术室提供的研究平台和技术指导,嘎利兵博士及老师们提供的实验帮助。
参考文献:
[1]Wittwer C T,Reed G H,Gundry C N,et al.High-resolution genotyping by amplicon melting analysis using LC Green[J].Clinical Chemistry,2003,49(6):853-860.
[2]Reed G H,Wittwer C T.Sensitivity and specificity of single-nucleotide polymorphism scanning by high-resolution melting analysis[J].Clinical Chemistry,2004,50(10):1748-1754.
[3]Herrmann M G,Durtschi J D,Bromley L K,et al.Amplicon DNA melting analysis for mutation scanning and genotyping:Cross-platform comparison of instruments and dyes[J].Clinical Chemistry,2006,52(3):494-503.
[4]Herrmann M G,Durtschi J D,Wittwer C T,et al.Expanded instrument comparison of amplicon DNA melting analysis for mutation scanning and genotyping[J].Clinical Chemistry,2007,53(8):1544-1548.
[5]Hewson K A,Browning G F,Devlin J M,et al.Application of high-resolution melt curve analysis for classification of infectious bronchitis viruses in field specimens[J].Australian Veterinary Journal,2010,88(10):408-413.
[6]Steer P A,O’Rourke D,Ghorashi S A,et al.Application of high-resolution melting curve analysis for typing of fowl adenoviruses in field cases of inclusion body hepatitis[J].Australian Veterinary Journal,2011,89(5):184-192.
[7]Steer P A,Kirkpatrick N C,O’Rourke D,et al.Classification of fowl adenovirus serotypes by use of high-resolution melting curve analysis of the hexon gene region[J].Journal of Clinical Microbiology,2009,47(2):311-321.
[8]Jeffery N,Gasser R B,Steer P A,et al.Classification of mycoplasma synoviae strains using single-strand conformation polymorphism and high-resolution melting curve analysis of thevlhA gene single-copy region[J].Microbiology,2007,153(8):2679-2688.
[9]Ghorashi S A,O’Rourke D,Ignjatovic J,et al.Differentiation of infectious bursal disease virus strains using real-time RT-PCR and high resolution melt curve analysis[J].Journal of Virological Methods,2011,171(1):264-271.
[10]Ajitkumar P,Barkema H W,Zadoks R N,et al.High-resolution melt analysis for species identification of coagulase-negative staphylococci derived from bovine milk[J].Diagn Microbiol Infect Dis,2013,75(3):227-234.
[11]Ajitkumar P,Barkema H W,De Buck J.Rapid identification of bovine mastitis pathogens by high-resolution melt analysis of 16S rDNA sequences[J].Veterinary Microbiology,2012,155(2-4):332-340.
[12]Pirulli D,Boniotto M,Puzzer D,et al.Flexibility of melting temperature assay for rapid detection of insertions,deletions,and single-point mutations of theAGXTgene responsible for type 1 primary hyperoxaluria[J].Clinical Chemistry,2000,46(11):1842-1844.
[13]Lipsky R H,Mazzanti C M,Rudolph J G,et al.DNA melting analysis for detection of single nucleotide polymorphisms[J].Clinical Chemistry,2001,47(4):635-644.
[14]Dufresne S D,Belloni D R,Wells W A,et al.BRCA1 and BRCA2 mutation screening using SmartCycler II high-resolution melt curve analysis[J].Arch Pathol Lab Med,2006,130(2):185-187.
[15]Rasmussen J P,Saint C P,Monis P T.Use of DNA melting simulation software for in silico diagnostic assay design:targeting regions with complex melting curves and confirmation by real-time PCR using intercalating dyes[J].BMC Bioinformatics,2007,8:107.
[16]Krypuy M,Newnham G M,Thomas D M,et al.High resolution melting analysis for the rapid and sensitive detection of mutations in clinical samples:KRAS codon 12 and 13 mutations in non-small cell lung cancer[J].BMC Cancer,2008,8:142.
[17]Wojdacz T K,Dobrovic A.Methylation-sensitive high resolution melting(MS-HRM):a new approach for sensitive and high-throughput assessment of methylation[J].Nucleic Acids Res,2007,35(6):e41.
[18]Paul T,Monis,Steven Giglio.Comparison of SYTO9 and SYBR Green I for real-time polymerase chain reaction and investigation of the effect of dye concentration on amplication and DNA melting curve analysis [J].Analytical Biochemistry,2005,340(1):24-34.

陈文波,陇灵宇,刘宇琼.基于两种方法结果差异的市域土地整治功能分区研究[J].江西农业大学学报,2015,37(1):176-182.
and Instruments in the Analysis of HRM
RAO Dan1,2,GALI Bing-ga2,GUO Xiao-quan1*,ZHANG Jian-feng2,
ZHANG Chun-hong2,SHEN Hai-yan2,ZHU Yu-jun2,
LIU Zhi-cheng2,SUN Jun-yin2,GUO Peng-ju2*
(1.College of Animal Science and Technology,Jiangxi Agricultural University,Nanchang 330045 China; 2.Laboratory of Veterinary Biotechnology,Institute of Animal Health,Guangdong Academy of Agricultural Sciences,Guangzhou 510640,China)
Abstract:High resolution melting curve(HRM) technology is a new type of technology for genotyping and mutation scanning developing in recent years based on the technology of real-time fluorescence quantitative PCR.This study compared several causative agents of animal diseases,including Classical swine fever virus/Eimeria species/Avain leukemia virus.The effects of different amplified regions of pathogens’ genomes(C202/
ALV450/En573 /C965),different fluorescent dyes(SYBR Green Ⅰ/LC Green /SYTO9)and different instruments(LightScanner96(Idaho)/LightCycler480(Roche)/Rotor-Gene Q(Qiagen)) on the HRM-based genotyping.The experimental data showed that HRM analysis was mainly affected by different amplified fragments and fluorescent dyes with different properties:nucleic acids with multiple melting range had higher specificity than that of single nucleotide polymorphism(SNP)and were more suitable for differentiation analysis; difference inTmon multiple melting range influenced HRM results; saturated fluorescent dyes(LC Green and SYTO9) had higher resolution than unsaturated dye(SYBR GreenⅠ); concentration of fluorescent dye had a greater influence on the value ofTm; there was no significant difference in the HRM results from different PCR-HRM analyzers.
Key words:HRM;SNP;fluorescent dye;melting Tm;imitating melting curve
作者简介:饶丹(1988—),女,硕士生,研究方向为兽医临床应用分子生物学,E-mail:raodan2007@126.com;*通信作者:郭小权,教授,E-mail:xqguo20720@aliyun.com;郭鹏举,研究员,E-mail:guopengju@hotmail.com。
基金项目:国家自然科学基金资助项目(31260627)、广东省科技计划项目(2011B050700007,2012B050500013)、广东省重大专项(2012A020100001)、广东省动物防疫专项资金项目(粤农计[2013]31号)、江西省青年科学家培养对象项目(20122BCB23022)、广东省科技计划(星火计划)项目(2012A020602094)和国家国际科技合作专项(2014DFA31730)
收稿日期:2014-03-19修回日期:2014-11-07
中图分类号:S854.4+3
文献标志码:A
文章编号:1000-2286(2015)01-0169-07

