EPAS1基因SNPrs13419896多态性与HiHiLo低氧训练适应效果关联性研究
汪洪波 刘海平 毛颖颖

摘要:目的:选取EPAS1基因序列第1内含子序列上rs13419896 G/A单核苷酸多态(single nucleotide polymorphism, SNP)位点,研究其与HiHiLo低氧训练后生理表型指标关联性,探寻预测低氧训练效果的遗传学标记。方法:选取35名北方汉族健康受试者(平均年龄19.54±2.55岁,平均身高177.94±4.27 cm,平均体重68.07±5.579 kg)在模拟海拔2 500 m高度(低氧环境O2浓度为15.4%)进行4 周高住-高练-低训(living high-exercise high-training low, HiHiLo)。采用关联研究方法(Association-Study),运用聚合酶链反应和基因测序实验技术分析EPAS1基因SNP rs13419896位点,研究该位点 与4周HiHiLo低氧训练后最大摄氧量(maximal oxygen uptake,VO2max)、血红蛋白(hemoglobin, Hb)、红细胞数(red blood cell, RBC)等生理表型指标变化的关联性。结果:1)EPAS1基因SNP rs13419896位点中GG、GA、AA基因型频率分别为40%、51.43%和8.57%,符合Hardy-weinberg遗传平衡定律(x2=0.70,P=0.40)。2)4周低氧训练后,EPAS1基因SNPrs13419896基因型组间ΔVO2max、ΔrVO2max、ΔRBC及ΔHb均无显著性差异。合并AA(AA基因型只有3例)与GA基因型统计发现,GA基因型受试者经4周低氧训练后,VO2max绝对值、相对值增长幅度有好于GG基因型趋势(76.17±245.31 ml/min vs -5.29±276.42 ml/min,P=0.09;1.22±3.37 ml/min·kg vs 0.13±4.18 ml/min·kg,P=0.09)。结论:平原人群4周 HiHiLo低氧训练后,EPAS1基因第1内含子序列上SNP rs13419896多态位点与VO2max指标变化可能存在着一定关联性。
关键词:内皮PAS结构;域蛋白1;高住-高练-低训;单核苷酸多态性
中图分类号:G804.7 文献标识码:A 文章编号:1006-2076(2015)01-0075-05
Abstract:Objective: This paper researched association between a single nucleotide polymorphisms rs13419896 (SNP rs13419896) in EPAS1 gene and physiological phenotype parameters response to hypoxia training. Methods: 35 healthy subjects completed 4 weeks living high-exercise high
低氧训练作为提高耐力项目运动员运动能力的手段一直是体育界的研究热点。近十几年来,低氧训练的有效性已被国内外学者所认同。但由于个体对低氧环境适应的生理反应差异性,会产生不同的低氧训练适应效果[1]。Friedmann B等的研究证实,低氧训练后运动员红细胞生成增多存在显著的个体间差异[2]。大量研究表明,由于低氧训练适应的生理反应差异性,会导致个体低氧训练效果的差异性,且产生不同低氧训练效果差异性与遗传学因素有着密切关联[3-5]。SCIENCE杂志2010年报道,人类对低氧环境适应与遗传因素有着高度紧密关联[6]。
内皮PAS结构域蛋白1(endothelial PAS domain protein 1, EPAS1) 又称低氧诱导因子-2α(hypoxia inducible factor-2α, HIF2α)。EPAS1在哺乳动物各种组织的内皮细胞中广泛表达,是慢性缺氧过程中的关键调节因子[7-8]。低氧环境下,机体内EPAS1蛋白的稳定性、表达量明显增加[9],并通过识别靶基因,如促红细胞生成素基因(erythropoietin, EPO)、血管内皮生长因子基因(vascular endothelial growth factor, VEGF)、血管内皮生长因子受体基因(vascular endothelial growth factor receptor, VEGFR)、乳酸脱氢酶基因-A(lactate dehydrogenase-A, LDHA)的核心序列5`-TACGTGCT-3`与低氧反应元件(hypoxia reaction element, HRE)结合,诱导靶基因的转录,介导细胞对缺氧应答,在蛋白水平发挥其低氧适应的调节作用[10]。Yi 等在研究影响藏族与汉族人群低氧适应遗传因素差异中发现,众多低氧反应基因频率都有差异,其中EPAS1 差别最大,并提示EPAS1 参与了藏族的高原低氧适应过程[11]。同时,Beall 等采用Illumina Veracode 平台以及610-Quad SNP 分型芯片分析藏族基因组SNP 变化情况发现,与藏族低氧适应相关联的EPAS1基因多态位点大多位于该基因的内含子序列上,最远的SNP 位点位于基因下游235kb 处[12]。
鉴于此,本研究选取EPAS1基因第1内含子序列上一重要单核苷酸多态位点(single nucleotide polymorphism, SNP)rs13419896[13],研究其与4周高住-高训-低氧(Living high-exercise high-training low, HiHiLo)低氧训练后生理表型指标变化的关联性,探究影响低氧训练期间生理表型指标敏感变化的遗传学标记。
1 研究方法
1.1 受试对象
本研究选取35名健康受试者(男性23名,女性12名),平均年龄19.54±2.55岁,平均身高177.94±4.27 cm,平均体重68.07±5.579 kg,均系中国北方汉族健康人群,无低氧暴露经历。
1.2 研究方案
所有受试者进行4周高住-高训-低氧(Living high-exercise high-training low, HiHiLo)方案。具体包括:每天在氧浓度为15.4%常压低氧环境(相当于海拔2 500 m高度,低氧制造设备:Hypoxic Tent System TM”和“CAT Hatch TM”,美国)休息和睡眠,低氧暴露时间≥10 h/天(pm 8:00-am 6:00),白天在海拔50 m的常氧环境下生活、训练,并且在低氧实验期间,每周进行3次、每次30分钟的低氧环境下蹬功率自行车定量负荷运动(以基础70%VO.2max强度为运动负荷,功率自行车转速为60转/分)。
生理表型指标测试:低氧实验前后分别进行1次最大摄氧量(maximal oxygen uptake,VO.2max)、血象指标(hematological parameters)、低氧定量负荷实验下血氧饱和度(oxygen saturation, SpO.2)的测试。VO.2max测试采用递增负荷实验进行(MedGraphicsVO.2000便携式气体代谢分析仪,美国),起始负荷60 W,功率车转速为60 RPM,每3分钟递增30 W,直至力竭,VO.2max判定标准以心率超过180 b/min,呼吸商超过1.1,VO.2不再增加,受试者不能保持蹬车频率等因素综合确定;血象指标测试要求受试者晨起后,空腹取静脉血0.5 ml,采用BECKMAN STKS型全自动血细胞分析仪(日本)进行测试分析,主要包括红细胞(RBC)、血红蛋白(Hb)等。
基因多态性分析:取静脉血2 ml,2% EDTA抗凝,用promega公司全血总DNA提取专用试剂盒提取受试者DNA。EPAS1基因内含子序列上SNP rs13419896多态性分析,依据人类EPAS1基因序列,用Primer 5. 0设计引物,引物由上海生工生物工程有限公司合成,上引物:5CTTCAGAGCAAATCAGAGGT 3,下引物:5CTCGCACCCAGATGACA 3。该位点应用LifePro Thermal Cycler PCR仪(中国)进行扩增(20μLPCR反应体系扩增,包括:10×PCR 2μL, 25 mM MgCl2 1.2 μL,dNTP各10 mM 1μL,上、下引物各10 pmol 0.5 μL,Taq酶2U,模板100 ng。各种药品均为上海生工产品。扩增方案是:(1)94℃预变性5 min;(2)94 °C变性40 s,50°C退火40 s,72°C延伸40 s,35个循环;(3)72°C延伸10 min。扩增产物为460 bp 片段。扩增PCR产物由上海生工公司测序,进行基因分型。
1.3 数据处理
数据统计学处理采用SPSS 11.5软件统计包(SPSS software for Windows 11.5 package)完成,所有数据以[AKX-]±SD表示。采用卡方检验(chi-square test)验证受试者人群的基因型频率是否符合Hardy-weinberg遗传平衡定律;低氧训练前后生理指标数据先用K-S检验是否符合正态分布,如符合进行下面统计学处理:低氧训练前基因型之间的生理指标差异,采用单因素方差分析处理,基因型之间生理指标的变化量采用协方差处理,以低氧训练前的初始值作为协变量进行分析。显著水平设为P<0.05。
2 结果
2.1 EPAS1基因SNPrs13419896分析
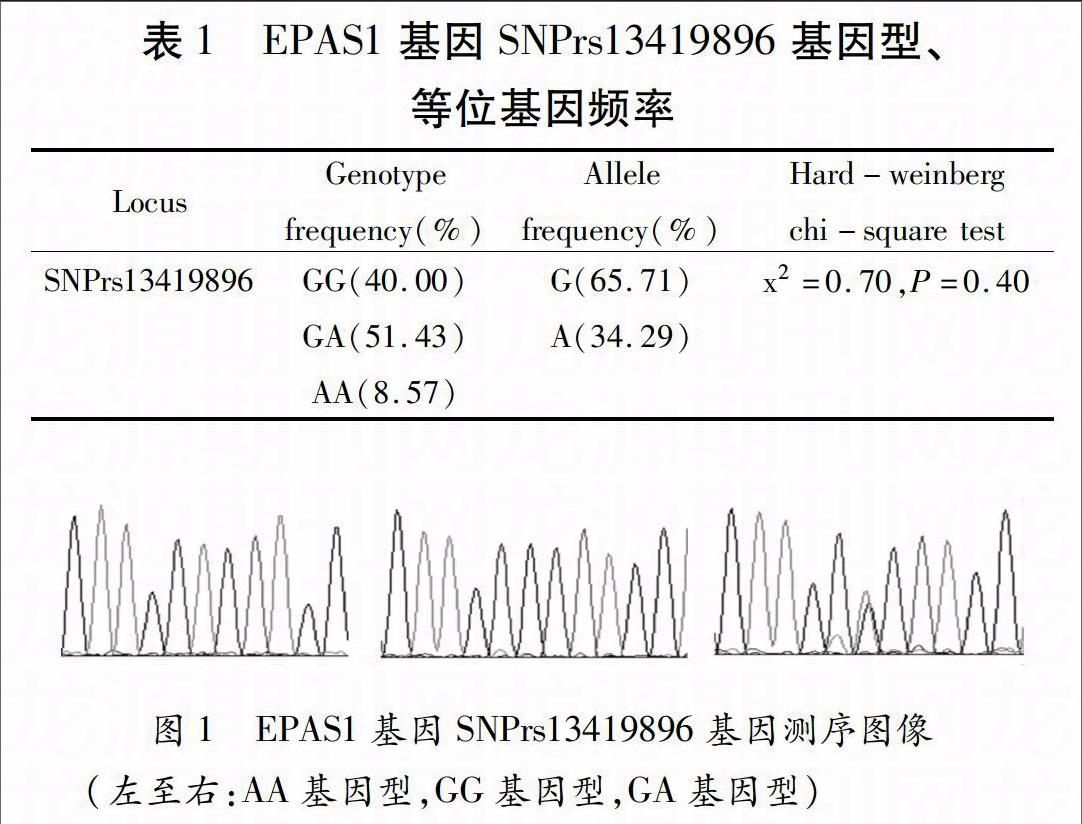
EPAS1基因内含子序列上SNPrs13419896位点分析结果见表1、图1。SNPrs13419896位点中GG、GA、AA基因型频率分别为40%、51.43%和8.57%,其中G、A等位基因频率分别为65.71%、34.29%; SNPrs13419896经卡方检验(x2=0.70,P=0.40),符合Hardy-weinberg遗传平衡定律,表明所选人群具有群体代表性。
2.2 EPAS1基因SNPrs13419896与VO2max和血象指标关联结果
低氧训练前,EPAS1基因SNPrs13419896多态位点各基因型组间的生理表型指标VO.2max、rVO.2max、RBC及Hb 经单因素方差分析显示无统计学显著性差异(表2)。
4周低氧训练后,EPAS1基因SNPrs13419896的各基因型组间与生理表型指标变化量分析结果见表2。结果显示,SNPrs13419896基因型组间ΔVO.2max、ΔrVO.2max、ΔRBC及ΔHb均无显著性差异。鉴于AA基因型只有3例,故将此基因型与其性质相近GA基因型合并后统计发现(见图2),GA基因型受试者经4周低氧训练后,VO2max绝对值、相对值增长幅度有好于GG基因型趋势(76.17±245.31 ml/min vs -5.29±276.42 ml/min,P=0.09;1.22±3.37 ml/min·kg vs 0.13±4.18 ml/min·kg,P=0.09)。
3 讨论
EPAS1是HIF家族一重要成员,对哺乳动物低氧适应过程中发挥着重要调节作用。人类EPAS1基因全长约120 kb,位于第2号染色体(2p16-21),包含15 个外显子和14 个内含子,cDNA 开放阅读框2607 bp,编码869个氨基酸,约为96.5 kD。本研究选取该基因序列第1内含子上SNPrs13419896位点研究其与低氧训练过程中生理适应效果关联性。
从本研究对SNPrs13419896位点分析发现,GG、GA、AA基因型频率分别为40%、51.43%和8.57%,即AA基因型较少(仅为3例),这与NCBI的SNP数据库中汉族人群基因型分布频率基本一致。EPAS1基因SNPrs13419896的研究对于揭示人类适应低氧环境的遗传因素是非常重要的。已有研究证实,低氧适应良好的夏尔巴和中国藏族人群,通过长期进化,其EPAS1基因SNPrs13419896中A等位基因频率分别为77.2%和79%,明显高于欧洲人群(0.009%)、日本人(33.6%)和中国北方汉族人群(30.3%)[13],柯金坤等研究显示,对低氧环境适应能力越好的藏族人群,SNPrs13419896位点中AA基因型、A等位基因频率越高[14]。提示我们,是否具有SNPrs13419896位点A等位基因的平原人群进行低氧训练后会产生较好的生理适应效果呢,值得研究。
本研究通过平原人群4周低氧训练后生理表型指标变化与EPAS1基因SNPrs13419896关联研究并未发现SNPrs13419896各基因型试者低氧训练后VO.2max、RBC及Hb变化差异性,但通过合并性质相近的AA基因型(AA基因型例数仅为3例)和GA基因型进一步分析低氧训练后基因型组间生理表型指标变化发现,GA基因型受试者低氧训练后,VO.2max绝对值、相对值与训练前相比分别提高2.2%和2.4%,而GG基因型受试者低氧训练后,VO.2max绝对值降低0.2%,相对值仅提高0.3%,GA基因型受试者低氧训练后,在VO.2max改善程度表现出优于GG基因型的趋势,但在Hb和RBC指标变化上未发现基因型组间差异性。从EPAS1生物学功能分析,低氧环境下,EPAS1蛋白结构上的氧依赖降解区(oxygen dependent degradation, ODD)不能被脯氨酸羟化酶识别并羟化,使其不能与VHL泛素2蛋白酶复合体结合,导致EPAS1经蛋白水解酶而降解的途径被抑制,从而使EPAS1蛋白稳定增强、含量增加,充分发挥其诱导靶基因转录表达及其生物学功能[15]。EPAS1通过识别靶基因核心序列5-TACGTGCT-3与其HRE结合,诱导靶基因的转录。在EPAS1众多靶基因中,其对VEGF及血管内皮生长因子受体的转录调节尤为明显。研究发现,与HIF-1α相比,EPAS1(HIF-2α)与VEGF的结合更容易,同时在表达丰富的血管内皮中,与VEGF 的表达非常一致[16],同时,通过诱导表达其他一系列与血管生成相关的基因,如VEGF受体(Flk-1 )以及Dll4 和Notch 信号途径,诱导血管生成,并发挥着对血管的稳定和正常功能的作用[17]。当平原人群进入低氧环境中,EPAS1在骨骼肌中的表达量同样明显提高[18]。由此推测平原人进行低氧训练过程中,SNPrs13419896位点的GA基因型或携带A等位基因受试者骨骼肌中EPAS1表达量较多,从而促进骨骼肌毛细血管增生,增加了骨骼肌细胞氧的运输量,导致VO2max明显改善。
此外,从EPAS1基因SNPrs13419896位点与红细胞生成关联研究发现[19],高原红细胞增多症人群和高原健康人群间SNPrs13419896位点基因型、等位基因频率无显著性差异,提示,该位点可能与低氧环境适应过程中血象指标改善无明显关联,这与本研究结果是一致的。
4 结论
平原人群4周 HiHiLo低氧训练后,EPAS1基因第1内含子序列上SNPrs13419896多态位点与VO.2max指标变化可能存在着一定关联性,表现为A等位基因携带者具有一定优势;该位点与血象指标变化无关联。
参考文献:
[1]Suzuki K, Kizaki T, Hitomi Y,et al.Genetic variation in hypoxia-inducible factor 1alpha and its possible association with high altitude adaptation in Sherpas [J]. Med Hypotheses, 2003, 61(3): 385-389.
[2]Friedmann B, Frese F, Menold E,et al.Individual variation in the erythropoietic response to altitude training in elite junior swimmers [J]. Br J Sports Med, 2005, 39(3): 148-153.
[3]刘海平, 胡扬. 低氧诱导因子-1α基因C958G多态性与个体低氧训练效果的关联性研究[J]. 体育科学,2007, 27(2): 38-41,93.
[4]刘海平, 胡扬. HIF-1α和eNOS基因多态性与个体低氧训练效果的关联性研究[J]. 中国应用生理学杂志, 2006,22(4): 434-438.
[5]周多奇, 龚莉, 胡扬,等. HiHiLo对心功能的影响与ACE基因I/D多态性的关联研究[J]. 体育科学,2011, 31(11): 52
-55,76.
[6]Simonson TS, Yang Y, Huff CD,et al.Genetic evidence for high-altitude adaptation in Tibet [J]. Science, 2010, 329(5987): 72-75.
[7]Tian H, McKnight SL, Russell DW. Endothelial PAS domain protein 1 (EPAS1), a transcription factor selectively expressed in endothelial cells [J]. Genes Dev, 1997, 11(1): 72-82.
[8]Holmquist-Mengelbier L, Fredlund E, Lofstedt T,et al.Recruitment of HIF-1alpha and HIF-2alpha to common target genes is differentially regulated in neuroblastoma: HIF-2alpha promotes an aggressive phenotype [J]. Cancer Cell, 2006, 10(5): 413-423.
[9]陈伟. 一种新的转录因子——EPAS1[J]. 生理科学进展, 2000, 31(4): 334-336.
[10]Asikainen TM, Ahmad A, Schneider BK,et al.Stimulation of HIF-1alpha, HIF-2alpha, and VEGF by prolyl 4-hydroxylase inhibition in human lung endothelial and epithelial cells [J]. Free Radic Biol Med, 2005, 38(8): 1002-1013.
[11]Yi X, Liang Y, Huerta-Sanchez E,et al.Sequencing of 50 human exomes reveals adaptation to high altitude [J]. Science, 2010, 329(5987): 75-78.
[12]Beall CM, Cavalleri GL, Deng L,et al.Natural selection on EPAS1 (HIF2alpha) associated with low hemoglobin concentration in Tibetan highlanders [J]. Proc Natl Acad Sci U S A, 2010, 107(25): 11459-11464.
[13]Hanaoka M, Droma Y, Basnyat B,et al.Genetic variants in EPAS1 contribute to adaptation to high-altitude hypoxia in Sherpas [J]. PLoS One, 2012, 7(12): e50566.
[14]柯金坤, 姚宇峰, 刘舒媛,等. 不同海拔高度低氧环境差异对EPAS1基因多态性的影响[J]. 中华医学遗传学杂志, 2011, 28(5): 583-588.
[15]Hu CJ, Wang LY, Chodosh LA,et al.Differential roles of hypoxia-inducible factor 1alpha (HIF-1alpha) and HIF-2alpha in hypoxic gene regulation [J]. Mol Cell Biol, 2003, 23(24): 9361-9374.
[16]Aprelikova O, Wood M, Tackett S,et al.Role of ETS transcription factors in the hypoxia-inducible factor-2 target gene selection [J]. Cancer Res, 2006, 66(11): 5641-5647.
[17]Skuli N, Majmundar AJ, Krock BL,et al.Endothelial HIF-2alpha regulates murine pathological angiogenesis and revascularization processes [J]. J Clin Invest, 2012, 122(4): 1427-1443.
[18]Morita M, Ohneda O, Yamashita T,et al.HLF/HIF-2alpha is a key factor in retinopathy of prematurity in association with erythropoietin [J]. EMBO J, 2003, 22(5): 1134-1146.
[19]陈郁. EPAS1 基因单核苷酸多态性与男性汉族高原红细胞增多症的相关性研究[D].重庆:第三军医大学,2013.

