四翅滨藜盐胁迫应答基因AcPsbQ1的克隆及其在酵母中的功能分析
孙新华,贾攀,张继超,潘洪玉
吉林大学植物科学学院,长春 130062
四翅滨藜盐胁迫应答基因AcPsbQ1的克隆及其在酵母中的功能分析
孙新华,贾攀,张继超,潘洪玉
吉林大学植物科学学院,长春 130062
光系统II亚基蛋白Q(Photosystem II subunit Q, PsbQ)是组成光系统II(PSII)复合体的重要外周蛋白之一,对维持PSII放氧活力起重要作用。在盐胁迫下,高浓度的Na+和Cl-在叶绿体中积累会严重破坏叶绿体的结构,抑制光合作用,使植物生长缓慢。文章通过筛选盐生植物四翅滨藜(Atriplex canescens) cDNA文库,获得一个在PSII中与放氧关系密切的放氧复合体蛋白基因PsbQ,命名为AcPsbQ1(GenBank登录号:KJ027025)。AcPsbQ1基因开放阅读框为699 bp,编码由233个氨基酸组成的前体蛋白。为了研究该基因的功能,文章克隆了AcPsbQ1基因并转化至酵母中,高盐和干旱胁迫处理实验表明转 AcPsbQ1基因酵母相比于野生型酵母对高盐和干旱有更强的耐受力。为了进一步研究AcPsbQ1是否参与植物抗逆应答反应,利用实时荧光定量PCR法测定AcPsbQ1在胁迫处理条件下的表达变化。结果表明,用0.4 mol/L NaCl处理四翅滨藜12 h后,AcPsbQ1基因的表达量明显高于未处理的对照,表明AcPsbQ1基因的表达受盐胁迫诱导,同时,干旱胁迫也会提高AcPsbQ1基因的表达水平。上述结果表明该基因可能参与盐生植物四翅滨藜的抗逆胁迫应答反应。
四翅滨藜;AcPsbQ1;盐诱导;酵母表达;qRT-PCR
盐胁迫是植物生长发育的主要限制因子之一,会同时造成渗透胁迫和离子胁迫,二者联合起来使植物体发生多种生理生化变化。大多数植物对盐胁迫非常敏感,在盐胁迫下,植物生长缓慢,高浓度的Na+和Cl-在叶绿体中积累,严重破坏叶绿体的结构,大大抑制光合作用,最终引起作物减产。植物通过调节相关的代谢途径、激发自身体内的分子信号传递从而形成新的离子平衡和渗透压进而提高植物自身对外界盐胁迫的耐受力。在分子水平,高盐胁迫会诱导植物中耐盐及其他胁迫信号通路中相关基因表达水平发生变化,提高植物体内参与抗盐胁迫的蛋白质或其他小分子含量进而增强其抗盐性。近年来,将挖掘出的植物抗盐基因通过现代分子生物学技术转入植物从而提高植物的抗逆性已取得了很大的进展。
四翅滨藜(Atriplex canescens)又称灰毛滨藜,属藜科滨藜属,是世界干旱、半干旱地区的典型植物,有极强的耐盐、干旱、低温、碱等优良特性;适生范围广泛,可作为优良的盐碱地改良品种,有生物吸盐器之称[1,2]。在盐胁迫条件下,四翅滨藜能将吸收的盐分积累到叶片表面形成一层白色屑状物体,从而提高对高盐的抗性。此外,SaiKachout等[3]报道由于四翅滨藜对Cu、Pb、Ni和Zn等重金属离子具有较强的吸附能力,故可作为矿井和重金属污染较严重地区良好的生物修复树种。
光系统Ⅱ(PSⅡ)在高等植物完成光合作用的过程中发挥重要作用,其核心功能是将光能转化为化学能。PSⅡ复合体可分为两个功能域:(1)电子传递功能域,由PSⅡ核心、内周捕光色素蛋白和外周捕光色素蛋白组成;(2)放氧复合体(Oxygen evolving complex,OEC),PSⅡ的放氧复合体催化水分子的裂解,释放出氧气和质子。在高等植物中,核基因PsbO、PsbP和PsbQ分别编码PSⅡ的33 kDa、24 kDa和17 kDa放氧复合体蛋白(这3个蛋白也被称为OEE1、OEE2和OEE3),这3个外周蛋白位于PSⅡ囊腔侧面,紧密结合在PSⅡ的锰簇周围,起维持PSⅡ放氧活力的关键作用[4,5]。其中PsbQ编码的17 kDa蛋白已在菠菜、小麦、水稻、玉米、拟南芥等多种植物中发现。研究表明,PsbQ蛋白通过调节放氧复合体与Ca2+、Cl-的结合从而优化光氧化水释放氧的过程[6~8]。最近,RNAi技术应用到 PsbQ的功能研究。Yi等[9]证明当光照不足时,拟南芥PsbQ蛋白对维持光系统Ⅱ的组装和稳定是必须的。拟南芥缺失PsbQ蛋白后,锰簇更容易从PSⅡ解离下来,同时不能有效的重新组装到PSⅡ上,这导致了低光照条件下PSⅡ的有效数量不足进而使光合作用受阻。本实验室以盐生灌木四翅滨藜(A.canescens)为研究材料,前期构建了盐(0.4 mol/L NaCl)胁迫下四翅滨藜的全长cDNA文库,通过测序获得300余个ESTs[10],其中分离得到一个PSⅡ中与放氧关系密切的放氧复合体蛋白AcPsbQ1的全长cDNA。本研究对四翅滨藜AcPsbQ1基因序列与表达特性等进行了分析,同时模拟盐胁迫和干旱胁迫,分析了转AcPsbQ1基因酵母菌株对盐和干旱胁迫的耐受性,初步探讨了AcPsbQ1在四翅滨藜耐盐过程中的功能。
1 材料和方法
1.1 材料
大肠杆菌E.coli DH5α(本实验室保存);酿酒酵母菌株INVSc1与表达载体pYES-DEST52(Invitrogen公司);由实验室构建并保存的文库大肠杆菌菌株DH10B;Trizol试剂盒、反转录试剂盒、限制性内切酶购自TaKaRa公司。
1.2 方法
1.2.1 四翅滨藜AcPsbQ1基因全长cDNA的获得及序列分析
提取用盐(0.4 mol/L NaCl)胁迫处理2月龄的四翅滨藜茎叶RNA,进而构建全长cDNA文库,通过Gateway重组到载体pYES-DEST52,以载体pYESDEST52上游引物 T7、下游引物 R为测序引物[10],将片段长度超过1 000 bp的文库克隆测序,其中获得一个四翅滨藜编码PsbQ蛋白的cDNA全长序列。
1.2.2 AcPsbQ1基因转化酵母并进行胁迫处理
将载体pYES-AcPsbQ1和空载体pYES-DEST52通过醋酸锂转化法转至酵母菌株INVSc1,详细的酵母转化及检测方法参考文献[11]。
配制NaCl终浓度为1 mol/L、1.5 mol/L的盐模拟胁迫固体培养基和山梨醇终浓度为 1.5 mol/L、2 mol/L的干旱模拟胁迫固体培养基。对验证正确的转基因重组酵母INVSc1(pYES2-AcPsbQ1)和转空载的对照酵母 INVSc1(pYES-DEST52)进行盐胁迫和干旱胁迫。将转基因酵母和对照一起接种于葡萄糖终浓度为2%的SC-U液体培养基,200 r/min、30℃培养24 h,利用紫外分光光度计测定菌液在600 nm波长处的吸光值OD600。取等量菌体离心,用含2%半乳糖的诱导培养基重悬菌体,并将OD600调为0.4,200 r/min、30℃振荡培养24 h后调整两种重组酵母的OD600至2,将上述菌液分别稀释0、10、100、1000、10000倍后,取2 μL菌液点在含胁迫剂的固体培养基(2%半乳糖)上,30℃培养3 d后,比较盐胁迫和干旱胁迫条件下菌落的差异,每组实验重复3次。
1.2.3 qRT-PCR检测AcPsbQ1基因表达水平
四翅滨藜在Hoagland(pH 5.8)培养液中水培2月后,用0.4 mol/L NaCl、20% PEG6000、0.2 mol/L NaHCO3和低温(4℃)分别处理0、6、12、24和48 h后收集样品,用Trizol 提取四翅滨藜总RNA。参照TaKaRa公司反转录试剂盒(PrimeScript® RT reagent Kit With gDNA Eraser)说明书进行cDNA合成。以四翅滨藜管家基因Eukaryotic elongation factor1A(eEF1A)为内参,引物序列为:

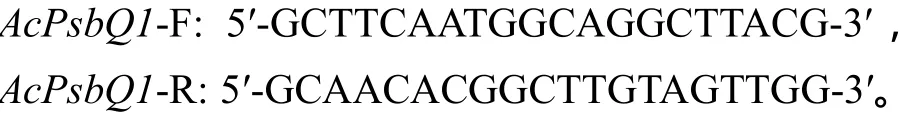
以合成的cDNA为模板进行qRT-PCR分析。采用 2−ΔΔCT法分析数据,确定基因的相对表达量。每个样本设3次重复。
2 结果与分析
2.1 AcPsbQ1基因全长cDNA的获得及序列分析
经测序,获得一个编码四翅滨藜光系统PSII亚基Q的cDNA序列,故命名为AcPsbQ1。序列分析表明AcPsbQ1基因含有完整的开放阅读框,为699 bp。该基因5′非编码区为48 bp,3′非编码区为115 bp,其中包括一个可能为多聚腺苷酸化信号的特征序列AATGAA序列,Poly A尾长23 bp。开放阅读框起始密码子为ATG,终止密码子为TAA,编码233个氨基酸。在ExPASY网站和NCBI在线分析可知,该氨基酸序列包括2个结构域,N端第1~84氨基酸为转运肽,第85~233氨基酸为成熟蛋白。用蛋白亚细胞定位预测软件WoLFPSORT(http://wolfpsort.seq.cbrc. jp/) 在线预测AcPsbQ1蛋白定位在叶绿体上。
AcPsbQ1转运肽由84个氨基酸组成,分为两个结构域(图 1):第 1~52氨基酸为叶绿体导向结构域(Chloroplast import domain, CID),可以形成双性β折叠(β-sheet forming domain 46~52);第53~84氨基酸为包含疏水域(h domain 64~79)的类囊体转移结构域(thylakoid transfer domain, TTD)。蛋白质疏水性分析分别表明该蛋白N端转运肽疏水性较强,其切割位点前(第60~80氨基酸)有一段疏水性急剧变化的区域,成熟蛋白区域亲水氨基酸分布均匀,属亲水性蛋白。
利用BioEdit和ClustalW程序对5种不同植物的 PsbQ基因所编码的前体蛋白进行多序列比对分析(图 1),结果表明 AcPsbQ1成熟蛋白与其他物种PsbQ成熟蛋白的同源性很高,但N端的转运肽保守性较低。将AcPsbQ1前体蛋白与已深入研究的植物PsbQ前体蛋白进行进化分析和同源性分析,并用ClustalX和 MEGA4软件绘制进化树,AcPsbQ1前体蛋白的氨基酸序列与菠菜(P12301.1)、海蓬子(ABQ23675.1)、拟南芥(At4g05180)、烟草(AAW80967.1) PsbQ前体蛋白序列相似性分别为87%、81%、73%、72%(图1、图2)。

图2 AcPsbQ1与其他植物PsbQ蛋白构建系统进化树
2.2 转AcPsbQ基因重组酵母的鉴定与抗盐、抗干旱性分析
酿酒酵母菌株 INVSc1是尿嘧啶营养缺陷型菌株,故可用 SC-U缺陷型培养基初步筛选获得的转化子,再将挑取的INVSc1(pYES2- AcPsbQ1)阳性克隆,以特异引物和载体上的通用引物进行菌液 PCR鉴定,进一步鉴定重组质粒已成功转化酵母细胞。
分别用高浓度的 NaCl和山梨醇模拟盐胁迫和干旱胁迫,对阳性的重组酵母(pYES- AcPsbQ1)与对照酵母(pYES-DEST52)进行盐胁迫和干旱处理(图3)。在含 1 mol/L NaCl的培养基上,重组酵母(pYESAcPsbQ1)与转空载体(pYES-DEST52)酵母生长都受到抑制,当培养基中的NaCl浓度升高至1.5 mol/L,两种酵母生长被显著抑制,但转AcPsbQ1基因重组酵母长势明显优于转空载体酵母,表明AcPsbQ1基因的转入表达使转基因重组酵母细胞对盐胁迫的抗性明显提高。转基因酵母对干旱胁迫的耐受力强于对高盐的耐受力,干旱胁迫结果表明1.5 mol/L的山梨醇已显著抑制了对照酵母的生长,而转 AcPsbQ基因酵母可以在胁迫培养基上正常生长。在2 mol/L山梨醇培养基上,对照酵母的存活率为零,而转AcPsbQ1基因重组酵母仍有较高的存活率,表明AcPsbQ1基因表达后可以明显提高酵母对干旱的抗性。
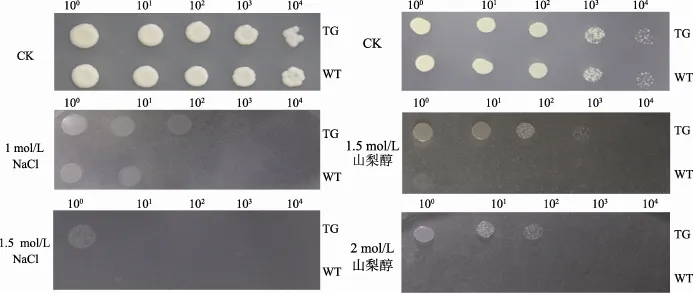
图3 转化酵母INVSc1(pYES-AcPsbQ1)和INVSc1 (pYES-DEST52)在盐和干旱胁迫条件下的生长状况
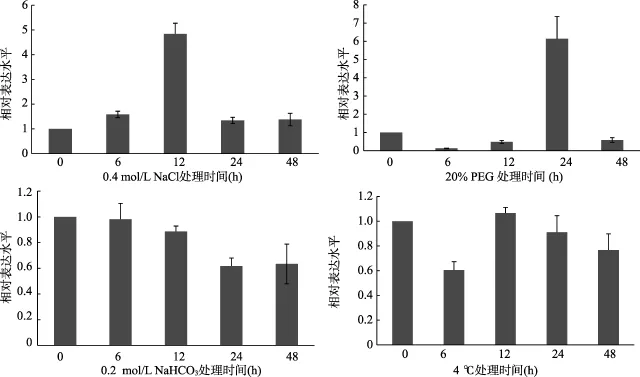
图4 AcPsbQ1在逆境胁迫下的表达情况
2.3 AcPsbQ1基因的诱导表达特性
为了探究AcPsbQ1基因对盐胁迫和干旱胁迫的应答响应,对四翅滨藜幼苗用NaCl和PEG6000模拟盐和干旱胁迫进行了处理,通过实时荧光定量PCR分析了表达动态。结果表明(图4),在0.4 mol/L 的NaCl胁迫下,AcPsbQ1基因在四翅滨藜中的表达明显受盐诱导。在NaCl胁迫初期,AcPsbQ1基因表达量增加,随后有所下降,但仍明显高于对照,胁迫12 h后基因表达量达到最大,表明AcPsbQ1基因的表达受盐胁迫诱导。20%PEG诱导初期,AcPsbQ1基因略有下降,但胁迫24 h后基因表达量明显升高。碱(0.2 mol/L NaHCO3)诱导初期AcPsbQ1基因表达水平没有明显变化,胁迫12 h后其表达量开始下降,低温(4℃)刺激四翅滨藜6 h后,AcPsbQ1基因相对表达水平下降0.6倍,但12 h后基因表达水平又恢复至胁迫处理前水平,此后基本保持不变。
3 讨 论
通过筛选四翅滨藜的全长cDNA文库,获得四翅滨藜抗性cDNA全长序列AcPsbQ1,该基因编码PSⅡ亚基Q的前体蛋白,许多叶绿体蛋白都是在细胞质中合成的较大的前体蛋白,利用N末端的转运肽,跨膜运输进入叶绿体内行使功能[12],PsbQ前体蛋白作为叶绿体蛋白也是如此。其前体的N端带有一段含有84个氨基酸的转运肽,该转运肽包含叶绿体导向结构域CID和类囊体转移结构域TTD。PsbQ蛋白作为高等植物放氧复合体的一部分,但不是PSⅡ放氧功能的必需蛋白[13]。最近的研究表明 PsbQ蛋白在维持PSⅡ的结构完整性、增强PSⅡ对环境适应性方面发挥重要作用。研究表明,PsbQ蛋白可以稳定其他外周蛋白与PSⅡ的结合,Kakiuchi等[14]证明PsbQ与PsbO和PsbP存在互作关系,并且可以恢复N端缺失的 PsbP蛋白与 PSⅡ的结合能力。同时,PsbQ、PsbO和PsbP一起参与和维持外周捕光色素蛋白复合体(主要指LHCII复合体)的完整性,确保PSⅡ的光能吸收过程顺利进行[15]。Allahverdiyeva等[16]发现拟南芥 PsbQ缺失突变体通过影响短期协调机制而改变光合机构对变化环境的适应能力,如PsbQ蛋白缺失植株的非光化学猝灭过程和状态转换的速率发生明显了变化。
盐害是植物生长过程中一种重要的非生物胁迫,高浓度的直接破坏PSⅡ的结构,使其光合作用受阻。同时,盐胁迫降低PSⅡ光反应中心D1蛋白及其他相关蛋白的合成速率进而抑制植物自身对PSⅡ的修复[17,18]。研究表明盐胁迫能提高PsbQ基因的转录水平。Sugihara等[19]用凝胶双向电泳技术研究红树盐胁迫下蛋白的差异表达时,检测出一些盐胁迫应答蛋白,其中就包括PsbQ,该研究认为PsbQ和PsbP一起维持NaCl胁迫下PSⅡ的活性。在高浓度的盐胁迫条件下(150 mmol/L NaCl),PsbQ和PsbP亚基很容易与 PSⅡ发生解离,PsbQ基因转录水平的升高被认为是可能参与PSⅡ的修复与重装进而保证放氧复合体放氧能力[20]。当受到高盐和干旱刺激后,四翅滨藜AcPsbQ1基因的表达量明显上调,表明该基因参与四翅滨藜的盐、干旱胁迫应答过程。qRTPCR结果表明,AcPsbQ1基因对碱(0.2 mol/L NaHCO3)和低温(4℃)刺激的应答比较微弱,基因表达水平略有下降。
真核生物的一些耐盐相关基因具有功能保守性,高等植物的抗逆胁迫应答及其信号分子传递的分子机制与酵母细胞具有一定的相似性[21]。同时,很多从植物中分离出来的的抗逆相关基因在酵母中表达后可以明显提高酵母细胞对盐胁迫的耐受力[22]。因此利用简单的酵母系统来分离鉴定高等植物功能保守的抗性基因是行之有效的快捷途径。PsbQ蛋白作为光系统Ⅱ的主要成分可以维持高盐胁迫下PSⅡ的完整性,通过提高光合作用和加强代谢来增强植物抗逆性[23]。Banieka等[24]利用酵母表达系统筛选马铃薯高温胁迫相关的抗逆基因,其中包括11个与光合作用密切相关的基因,如光系统Ⅱ亚基蛋白 R(PsbR)、叶绿素 a/b结合蛋白(Cab)等,这些从马铃薯中筛选出来的与光合作用相关的基因在酵母中过表达均提高了酵母细胞对高温的抗性。本研究中,AcPsbQ1在酵母中直接表达提高了酵母对盐和干旱胁迫的抗性,表明PsbQ可能直接参与植物对逆境胁迫的响应,但其在植物抗逆反应过程的具体作用和机制还需要进一步验证。
[1]李敬涛, 余刚, 陈宣明, 范臻, 刘金亮, 潘洪玉. 四翅滨藜 AcDHN基因的克隆及其抗逆功能分析. 植物生理学报, 2012, 48(7): 676-682.
[2]余刚, 李敬涛, 孙新华, 刘金亮, 潘洪玉. 四翅滨藜LRR类受体蛋白激酶基因在酵母中的表达及抗逆分析.吉林大学学报(理学版), 2012, 5(6): 1257-1263.
[3]Kachout SS, Leclerc JC, Mansoura AB, Rejeb MN, Ouerghi Z. Effects of heavy metals on growth and bioaccumulation of the annual halophytes: Atriplex hortensis and A. rosea. J Appl Sci Res, 2009, 5(7): 746-756.
[4]Ido K, Nield J, Fukao Y, Nishimura T, Sato F, Ifuku K. Cross-linking evidence for multiple interactions of the PsbP and PsbQ proteins in a higher plant photosystem Ⅱsupercomplex. J Biol Chem, 2014, 289(29): 20150-20157.
[5]Liu HJ, Zhang H, Weisz D A, Vidavsky I, Gross ML,Pakrasi HB. MS-based cross-linking analysis reveals the location of the PsbQ protein in cyanobacterial photosystem Ⅱ. Proc Natl Acad Sci USA, 2014, 111(12): 4638-4643.
[6]Åkerlund HE, Jansson C, Andersson B. Reconstitution of photosynthetic water splitting in inside-out thylakoid vesicles and identification of a participating polypeptide. Biochim Biophys Acta, 1982, 681(1): 1-10.
[7]Kuwabara T, Murata T, Miyao M, Murata N. Partial degradation of the 18-kDa protein of the photosynthetic oxygen-evolving complex: a study of a binding site. Biochim Biophys Acta, 1986, 850(1): 146-155.
[8]Ghanotakis DF, Babcock GT, Yocum CF. Calcium reconstitutes high rates of oxygen evolution in polypeptide depleted photosystem Ⅱ preparations. FEBS Lett, 1984, 167(1): 127-130.
[9]Yi XP, Hargett SR, Frankel LK, Bricker TM. The PsbQ protein is required in Arabidopsis for photosystem II assembly/stability and photoautotrophy under low light conditions. J Biol Chem, 2006, 281(36): 26260-26267.
[10]Li JT, Sun XH, Yu G, Jia CG, Liu JL, Pan HY. Generation and analysis of expressed sequence tags(ESTs) from halophyte Atriplex canescens to explore salt-responsive related genes. Int J Mol Sci, 2014, 15(6): 11172-11189.
[11]孙新华, 李敬涛, 刘言志, 贾承国, 潘洪玉. 四翅滨藜AcERF1基因的克隆及其在酵母中的抗逆分析. 植物生理学报, 2013, 49(8): 811-816.
[12]Robinson C, Klösgen RB. Targeting of proteins into and across the thylakoid membrane: a multitude of mechanisms. Plant Mol Biol, 1994, 26(1): 15-24.
[13]Ifuku K, Sato F. A truncated mutant of the extrinsic 23-kDa protein that absolutely requires the extrinsic 17-kDa protein for Ca2 +retention in photosystem Ⅱ. Plant Cell Physiol, 2002, 43(10): 1244-1249.
[14]Kakiuchi S, Uno C, Ido K, Nishimura T, Noguchi T, Ifuku K, Sato F. The PsbQ protein stabilizes the functional binding of the PsbP protein to photosystem Ⅱ in higher plants. Biochimica et Biophysica Acta, 2012, 1817(8): 1346-1351.
[15]Allahverdiyeva Y, Suorsa M, Rossi F, Pavesi A, Kater MM, Antonacci A, Tadini L, Pribil M, Schneider A, Wanner G, Leister D, Aro EM, Barbato R, Pesaresi P. Arabidopsis plants lacking PsbQ and PsbR subunits of the oxygen-evolving complex show altered PSⅡ super-complex organization and short-term adaptive mechanisms. Plant J, 2013, 75(4): 671-684.
[16]Ifuku K. The PsbP and PsbQ family proteins in the photosynthetic machinery of chloroplasts. Plant Physiol Biochem, 2014, 81: 108-114.
[17]Allakhverdiev SI, Nishiyama Y, Miyairi S, Yamamoto H, Inagaki N, Kanesaki Y, Murata N. Salt stress inhibits the repair of photodamaged photosystem Ⅱ by suppressing the transcription and translation of psbA genes in Synechocystis. Plant Physiol, 2002, 130(3): 1443-1453.
[18]Murata N, Takahashi S, Nishiyama Y, Allakhverdiev SI. Photoinhibition of photosystem Ⅱ under environmental stress. Biochimica Biophysica Acta, 2007, 1767(6): 414-421.
[19]Sugihara K, Hanagata N, Dubinsky Z, Baba S, Karube I. Molecular characterization of cDNA encoding oxygen evolving enhancer protein 1 increased by salt treatment in the mangrove Bruguiera gymnorrhiza. Plant Cell Physiol, 2000, 41(11): 1279-1285.
[20]Tada Y, Kashimura T. Proteomic analysis of salt-responsive proteins in the mangrove plant, Bruguiera gymnorhiza. Plant Cell Physiol, 2009, 50(3): 439-446.
[21]Nylander M, Heino P, Helenius E, Palva ET, Ronne H, Welin BV. The low temperature and salt induced RCI2A gene of Arabidopsis complements the sodium sensitivity caused by a deletion of the homologous yeast gene SNA1. Plant Mol Biol, 2001, 45(3): 341-352.
[22]Kanhonou R, Serrano R, Palau RR. A catalytic subunit of the sugar beet protein kinase CK2 is induced by salt stress and increases NaCl tolerance in Saccharomyces cerevisiae. Plant Mol Biol, 2001, 47(5): 571-579.
[23]Roose JL, Wegener KM, Pakrasi HB. The extrinsic proteins of Photosystem Ⅱ. Photosynth Res, 2007, 92(3): 369-387.
[24]Gangadhar BH, Yu JW, Sajeesh K, Park SW. A systematic exploration of high-temperature stress-responsive genes in potato using large-scale yeast functional screening. Mol Genet Genomics, 2014, 289(2): 185-201.
(责任编委: 赖锦盛)
Cloning and characterization of a salt responsive gene AcPsbQ1 from Atriplex canescens
Xinhua Sun, Pan Jia, Jichao Zhang, Hongyu Pan
College of Plant Science, Jilin University, Changchun 130062, China
PsbQ is an extrinsic subunit of the photosystem II in eukaryotic photosynthetic organisms. Numerous studies have demonstrated that PsbQ can stabilize the inorganic cofactors and enhance the oxygen release in PSII. The decrease of photosynthesis rate under salinity condition is normally attributed to the high concentration of injurious ions, such as Na+and Cl-, which accumulate in the chloroplast and damage thylakoid membrane under salinity stress. In this study, AcPsbQ1 was isolated from a halophyte Atriplex canescens cDNA library. The AcPsbQ1 contains anopen reading frame of 699 bp encoding a 233 amino acid protein. In order to investigate its function, AcPsbQ1 was cloned and transformed into Saccharomyces cerevisiae INVSc1. The heterologous expression of AcPsbQ1 in transgenic yeast significantly helped to increase the adapting and recovery ability of yeast cells under the salt and drought. Quantitative real-time PCR assay was performed to reveal the expression pattern of AcPsbQ1 under different abiotic stresses. On exposure to NaCl stress, the transcript level of AcPsbQ1 was significantly enhanced. AcPsbQ1 expression level was also up-regulated under drought stress. These results indicated that AcPsbQ1 might involve in the response to salt stress in A. canescens.
Atriplex canescens; AcPsbQ1; salt stress response; yeast expression; qRT-PCR
2014-07-15;
2014-10-26
吉林省发改委产业研发项目(编号:2013C001),农业部抗病虫转基因大豆新品种培育项目(编号:2013ZX08004004)和国家科技支撑计划课题项目(编号:2014BAD14B02)资助
孙新华,硕士研究生,专业方向:真菌分子生物学与植物抗逆及抗病基因工程。E-mail: xinhua_sun@126.com
潘洪玉,教授,研究方向:植物病原真菌分子生物学、抗病基因工程及抗逆基因工程以及植物病害生物防治。E-mail: panhongyu@jlu.edu.cn
10.16288/j.yczz.2015.01.012
时间: 2014-11-5 10:17:55
URL: http://www.cnki.net/kcms/detail/11.1913.R.20141105.1017.002.html

