5株发酵剂对熏马肠成熟过程中内源菌的影响*
李蕊婷,卢士玲,李开雄,马宇霞,邓红梅
(石河子大学食品学院,新疆石河子,832000)
目前,肉制品的发酵剂研究在我国属于一个比较热门的领域。在肉制品成熟的过程中添加合适的发酵剂,对实现发酵肉工业化生产,缩短产品成熟期,对使产品特征标准化和安全化将起重要的作用[1]。但是随着肉制品的发酵和成熟,其内源菌群不断变化,加入不同的发酵剂可能对内源菌群产生一定影响(拮抗或协同)。本研究在不采取化学添加物的前提下,在熏马肠生产过程中添加有抑菌作用并可产生物胺氧化酶的菌株作发酵剂,以不同的添加组合,采用PCR-DGGE技术研究发酵剂对内源微生物在熏马肠发酵和成熟过程中动态变化的影响,为熏马肠安全生产提供技术支持。
1 材料与主要试剂
1.1 试验材料
马肉及其他辅料均购自石河子爱家超市。
发酵剂:本实验室分离提取到的4株葡萄球菌和1株植物乳杆菌(Staphylococcus epidermidis;Staphylococcussimulans.001;Staphylococcussimulans.002;Staphylococcus simulans.003;Lactobacillus plantarum)注:Staphylococcus simulans有3个不同种,分别编号为 001、002、003。
1.2 主要仪器和试剂
恒温恒湿培养箱,德国 BINDER公司;灌肠机(MM12型绞肉机);海尔冰箱;电子天平;高速离心机;小型离心机(Anke TLG-16G);超净工作台(SWCF-1F);PCR仪,美国 Bio-Rad公司;DGGE电泳仪,美国Bio-Rad公司;凝胶成像仪(GelDoc 2000 system Bio-Rad,美国);微型振荡器;海尔控温冰箱;Eppendorf移液枪(1 ~10μL、1 ~20 μL、20 ~ 200 μL、10 ~1 000 μL)。
细菌总DNA提取试剂盒(天根);溶菌酶(sigma);PCR相关试剂盒,MarkⅢ,去离子水,丙烯酰胺,甲叉双丙烯酰胺(Sigma),尿素(Sigma),TRIS(Sigma),EDTA-2Na(Sigma),TE(Sigma),过硫酸铵(Sigma),SYBR Green I(Bio-V)。
2 试验内容及方法
2.1 熏马肠基本加工工艺
原料肉进行处理,将肥膘(20%)切丁进行漂洗(40℃),瘦肉(80%)绞肉,混合腌制接种发酵剂;灌入肠衣发酵2 d,(发酵条件:18 ±0.5℃,RH 90% ~95%);成熟过程控制不同条件3~4 d[(14±0.5)℃,RH 80% ~85%],5 ~7 d[(12 ± 0.5)℃,RH 75% ~80%],8 ~28 d[(10 ± 0.5)℃,RH 70% ~75%]。
2.2 试验设计
按照熏马肠的加工工艺,利用分离纯化得到的4株葡萄球菌及1株植物乳杆菌,制作9种不同类型熏马肠(见表1),其中发酵剂的接种量为107CFU/g,复合发酵剂以1∶1添加。在熏马肠的发酵和成熟的过程中,分别对 9 组熏马肠 A、B、C、D、E、F、G、H、I于0、2、7、14、28 d 进行无菌取样并冷冻保藏,分别对其进行菌相测定。
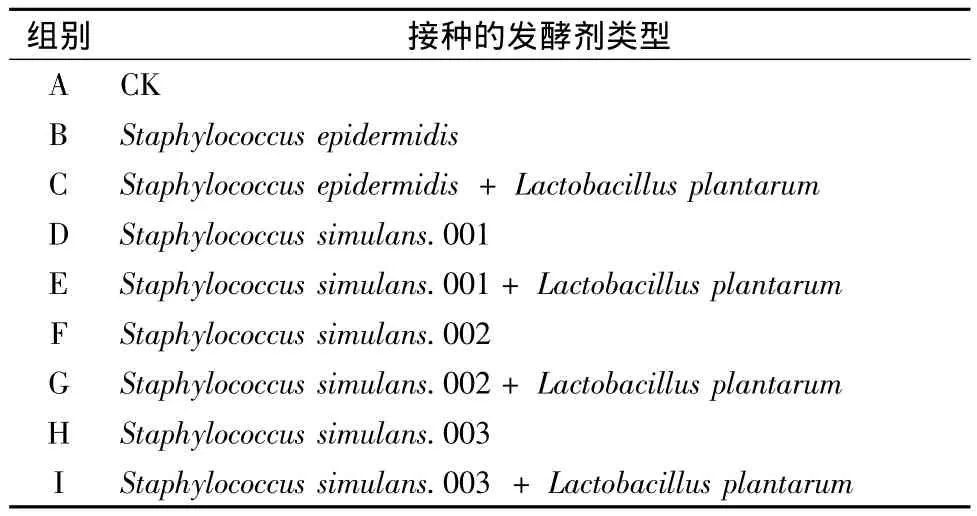
表1 接种不同发酵剂类型的熏马肠Table 1 The smoked horsemeat sausage of inoculating with different starter culture
2.3 实验方法
2.3.1 细菌总DNA的提取
取待测样品20g于180 mL生理盐水中,摇床振荡30 min;4 000重力加速度的条件下4℃离心10 min;再取上清液1 mL于1.5 mL离心管在10 000重力加速度条件下4℃离心10 min,弃去上清液,反复几次。参照DNA提取试剂盒(天根)的说明,提取样品总细菌的DNA。所提取的DNA溶于TE缓冲液,经1.0%的琼脂糖凝胶电泳检测后,于-20℃条件下贮藏以备PCR扩增使用。
2.3.2 PCR扩增
所选用的引物为[2]:上游引物为带GC夹子的U968(5'-CGC CCG GGG CGC GCC CCG GGC GGG GCG GGG GCA CGG GGG GAA CGC GAA GAA CCT TAC-3'),下游引物为 L1401(5'-CGG TGT GTA CAA GAC CC-3')。对细菌的16S rDNA的 V6-V8区段进行PCR扩增,扩增出的片段为400bp。
PCR 反应为25 μL体系:包括1μL模板DNA,上游引物和下游引物各0.5μL,HS MIX(东盛)12.5μL,无菌水10.5μL。
PCR反应程序:95℃预变性10 min,35个循环(95℃变性30s;56℃退火30s;72℃延伸30s),最终72℃延伸5 min。PCR产物经1.0%琼脂糖凝胶电泳检测后,-20℃冰箱保存以备DGGE使用。
2.3.3 变性梯度凝胶电泳(DGGE)
DGGE采用Bio-rad电泳仪,聚丙烯酰胺凝胶浓度为8%(丙烯酰胺:甲叉双丙烯酰胺37.5:1)。变性梯度为40%~60%(40%变性剂凝胶溶液:9.6 mL 0%变性剂+14.4 mL 100%变性剂 +50μL 20%APS溶液+5μL TEMED;60%变性剂凝胶溶液:14.4 mL 0%变性剂 +9.6 mL 100%变性剂 +50μL 20%APS溶液 +5μL TEMED),浓缩胶为:6 mL 0%变性剂 +30μL 20%APS溶液 +6μL TEMED。在 pH 为7.4的1×TAE缓冲液中,将PCR产物加入胶孔,温度设定为60℃,然后开始电泳,电泳条件为:200V电压预跑10 min,之后85V电压16 h。
2.3.4 SYBR Green I染色
待电泳结束后,小心取下胶片,放入50 mL pH为7.0~8.5的1×TAE(含5μL SYBR Green I)中室温避光振荡染色30 min,之后再将胶片放入超纯水中漂洗几次。
2.3.5 凝胶成像及DNA的回收
待染色后,小心地将DGGE胶片转移到凝胶成像系统下拍照并割胶(要尽快操作,否则在紫外光的作用下,很快图像的效果就会降低),图像分析采用Quantity one软件。割胶回收是用无菌手术刀切下DGGE胶上相应位置的条带,分别放入已经灭菌的1.5 mL离心管里,加入20μL无菌的TE溶液,置于4℃条件下过夜以备DNA测序的使用。
2.3.6 DNA的测序
取2μL回收的DNA为模板进行16S rDNA的V6-V8区域扩增,细菌上游引物为U968(5'-AAC GCG AAG AAC CTT AC-3'),下游引物 L1401(5'-CGG TGT GTA CAA GAC CC-3'),PCR 扩增程序同2.3.2。扩增后的PCR产物经1.0%琼脂糖凝胶电泳检验,条带的位置在400bp就可以证明该回收DNA的存在,之后将剩余PCR产物送到华大基因测序,登录NCBI将所得序列与数据库中已知序列进行相似性比对[3]。
3 结果与分析
3.1 细菌总DNA提取结果
选取9组不同处理的样品,分别在其成熟过程中第0,2,7,14,28天的取样。用细菌总 DNA 提取试剂盒,对样品进行无菌操作并提取样品中的总DNA,之后将溶于TE缓冲液的DNA经1.0%琼脂糖电泳检测,得到明显的条带,说明在实验过程中对样品的细菌总DNA提取效果很好,提取方法合适,所得到总DNA适合后续PCR扩增实验。
3.2 细菌的16S rDNA PCR扩增
以不同处理的熏马肠细菌总DNA为模板,用引物GC-U968和L1401对16S rDNA的V6-V8可变区进行PCR扩增,扩增产物经1.0%琼脂糖电泳检测,获得约400bp的特异性片段,所有样品均有较亮的扩增条带,说明本实验的PCR扩增条件是适合的,可以满足后续DGGE电泳分析实验的条件。
3.3 熏马肠成熟过程中细菌DGGE图谱主要条带的DNA序列相似性比较
由图1和图2可看出,因2张图中均有对照组A,将2张图片进行位置比对后,发现图2中条带在图1中有相互对应的条带。DGGE指纹图上的1个条带就代表1个微生物类群,因此,图2中部分条带割胶后没有进行测序。经比对后,对应的位置为:m1→M1,m2→M2,m3→M3,m4→M6,m6→M8,m7→M9,m8→M10,m10→M11,m11→M13,m12→M14,m13→M15。其他 M4,M5,M7,M12,m5,m9 没有相对应的条带。
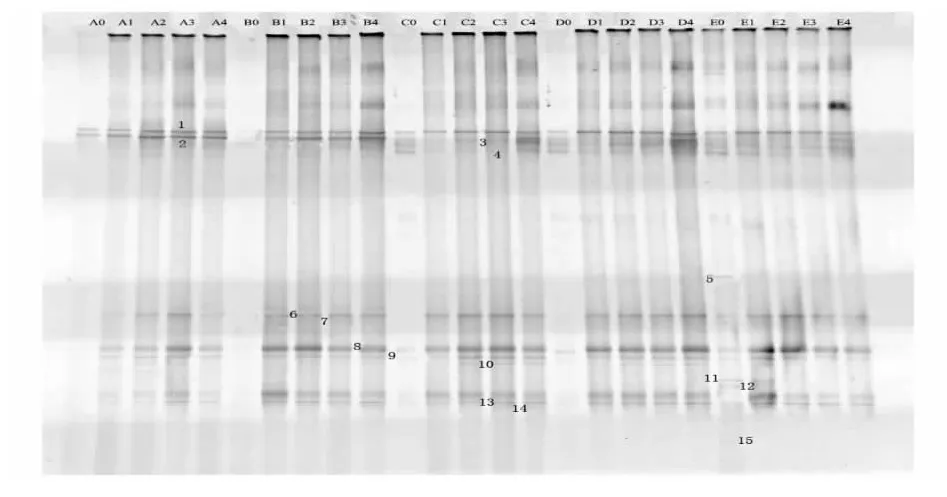
图1 熏马肠成熟过程中细菌PCR-DGGE图谱Fig.1 DGGE fingerprinting of PCR products of smoked horsemeat sausage during ripening
3.4 细菌DGGE图谱主要条带的DNA序列分析
通过PCR-DGGE电泳照相后,把条带割胶用不带GC夹子的引物扩增序列(同2.3.6)后,其中m9和M12于条带太弱,菌活力弱,菌液中浓度过低,没有测出相应的序列,其余可进行后续分析。将测序结果与Gene bank数据库中序列进行比对,确定结果(见表2)。

图2 熏马肠成熟过程中细菌PCR-DGGE图谱Fig.2 DGGE fingerprinting of PCR products of smoked horsemeat sausage during ripening
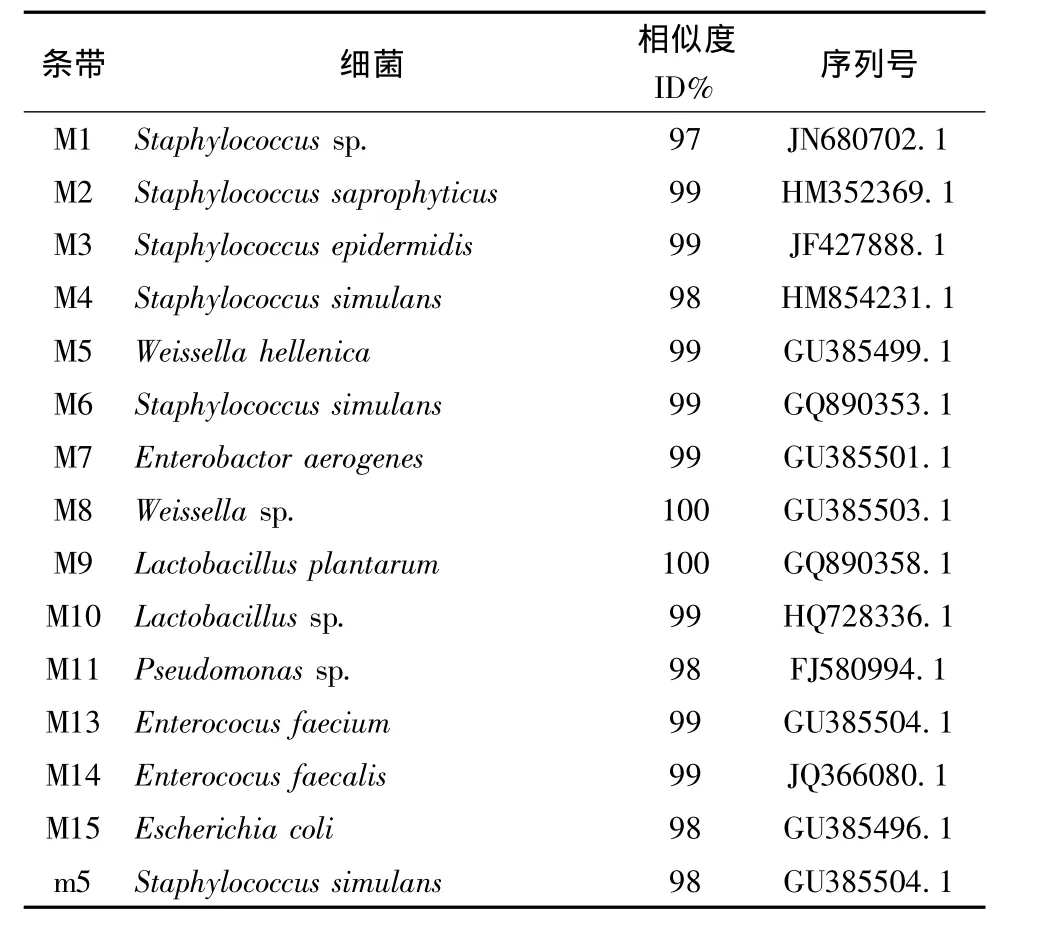
表2 熏马肠成熟过程中细菌PCR-DGGE图谱上条带系列分析(图1中的条带)Table 2 Identities of bands obtained from DGGE analysis of bactrial communities in smoked horsemeat sausage during ripening(bands obtained from Fig.1)
4 讨论
如图1、图2可看出,空白组第14天的菌相最丰富,且数量最大,第0天使M1、M2数量多,其他的菌浓度都低。第2天时,随着发酵时间的增长,菌相开始丰富,M6、M7、M8、M9、M10 均出现。第 7 天时,种类基本无变化,数量增多。第14天时,M10消失,出现M13、M14。第28天时,菌数量减少,菌群达到动态稳定状态,M1、M2为自然发酵剂,且一直是优势菌种,几乎不受添加发酵剂的影响。M9为植物乳杆菌,在C、E、G、I四组复合添加植物乳杆菌中,数量比不复合的相应组数量要多,同时其他条带的亮度也都降低,说明此株植物乳杆菌能够在一定程度上抑制内源菌的生长。
B组和C组中,M3、M9为发酵剂,B组中M3随着发酵时间的增加越来越少,C组中M3增强,2组中的M9数量越来越大。与空白组对照,增加了内源菌的生长;D组和E组中,M4、M9为加入的发酵剂,D组中M4一直未变,M9逐步增多,内源菌增多。E组中M4越来越少,M9到第2天时增多后又下降。E组的发酵剂的内源菌的数量减少,种类变多;F组和G组中,m4、m7为发酵剂,2组的M7都逐渐增多,F组中的m4增大到28 d时降低且稳定,G组的m4持续增多。内源菌的种类和数量都降低;H组和I组中,m5、m7为发酵剂,m5在0 d时出现,后被抑制。m7越来越多,发酵结束后并没有明显影响内源菌的种类和数量。
在熏马肠中,实际菌相可能比检测到的更加复杂,存在一些在本研究过程中没有检测到的其他属菌或乳酸菌、肠细菌、微球菌和假单胞菌属中的其他种类。在很多相关的报道中,对主要腐败菌进行初步分离和鉴定,发现假单胞菌(Pseudomonas sp.)是主要的腐败优势菌之一[4-5]。有研究表明,当该种微生物占整个微生物种群数小于1%时,DGGE将检测不到该种微生物的存在[6]。
研究表明,粪肠球菌、屎肠球菌、阴沟肠杆菌、大肠埃希氏菌和产气肠杆菌可产生大量的生物胺[7],一些乳酸菌也可产生生物胺[8],控制它们的生长能够更好的保障发酵香肠的品质。Cocolin等人[9]将DGGE用于自然发酵意大利香肠成熟过程中菌相的分析,实验结果表明乳酸菌是其香肠发酵过程中的优势菌,其中 Lactobacillus sake和 Lactobacillus curvatus对产品的酸化和蛋白质的水解有重要作用,决定了发酵香肠的感官质量。在本研究中,Weissella sp.是优势菌,但Weissella sp.不产生物胺已经得到证实[10];微球菌属中的一些菌也能产生生物胺,但产生物胺的能力极其有限[11],它们的存在对香肠的品质影响不明显。
5 结论
本研究采用传统PCR-DGGE的方法揭示了熏马肠成熟过程中优势菌相的变化。结果表明,添加不同发酵剂的熏马肠菌相分布呈现不同变化,但是随着成熟时间的变化,优势菌趋于明显。添加发酵剂所有组合对内源菌Escherichia coli有较强的抑制作用;腐败菌条带 M8,M10,M13,M14从成熟初期到结束一直存在,添加发酵剂所有组合对内源菌Escherichia coli有较强的抑制作用;其中C,D,E组中细菌种类在发酵初期较多,但到发酵结束时,其菌含量下降,尤其对内源菌 Pseudomonas sp.,Staphylococcus carnosus和Staphylococcus xylosus有较强抑制作用;G组中内源的Pseudomonas sp.一直存在;与E组对应的添加单一发酵剂的D组在发酵初期并没有E组的微生物种类多,但其抑菌效果较好;内源的Weissella sp.和Staphylococcus sp.在成熟过程中一直存在,不受发酵剂的抑制,但Weissella sp.不产生物胺。F组在28 d发酵结束时,菌群的种类和数量为最低,抑菌效果最好。
[1] 贾士芳,陈美玲,钟瑾,等.肉制品发酵剂木糖葡萄球菌I2的研究[J].食品与发酵工业,2006,32(8):72-74.
[2] Ercolini D.PCR-DGGE fingerprinting:novel strategies for detection of microbes in food[J].Journal of Microbiological Methods,2004,56(3):297-314.
[3] Altschul S F,Madden T L,Schaffer A A,et al.Gapped BLAST and PSIBLAST:a new generation of protein database search programs [J].Nucleic Acidas Research,1997,25(17):3 389-3 402.
[4] Ols son C,Ahrné S,Pettersson B,et al.The bacterial flora of fresh and chill-stored pork:analysis by cloning and sequencing of 16S rRNA genes[J].International Journal of Food Microbiology,2003,83(3):245-252.
[5] Gill C O,Dus sault F,Holley R A A,et al.Evaluation of the hygienic perform ances of the processes for cleaning,dressing and cooling pig carcasses at eight packing plants[J].International Journal of Food Microbiology,2000,58(1-2):65-72.
[6] Muyzer G,E C de Waa,Uitterlinden A G.Profiling of comples microbial population by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rDNA[J].Appl Environ Microbial,1993,59(3):695-700.
[7] 卢士玲,樊庆鲁,李开雄.猪肉自然发酵香肠中细菌和乳酸菌对生物胺积累的作用[J].食品研究与开发,2006,27(2):37-40.
[8] Montel M C,F.M,R.T.Comparison of biogenic amine content in traditional and industrial French dry sausages[J].Sciences des Aliments,1999,19(2):247-254.
[9] Cocolin L,Manzano M,Cantoni C,et al.Denaturing gradient gel electrophoresis analysis of the 16S rRNA gene V1 region to Monitor dynamic changes in the bacterial population during fermentation of Italian sausages[J].Appl Environ Microbiol,2001,67(11):5 113-5 121.
[10] Bover-Cid S,Holzapfel W H.Improved screening procedure for biogenic amines production by lactic acid bacteria[J].International Journal of Food Microbiology,1999,53(1):33-41.
[11] Martuscelli M,Gardini F,Torriani S,et al.Production of biogenic amines during the ripening of Pecorino Abruzzese cheese[J].International Dairy Journal,2005,15(6-9):571-578.

