芽胞杆菌T61的辐射抗性和伽玛射线辐照后蛋白质组的变化分析
宣慧娟 李 利 白明艳 孙云峰 万 平 杨志伟
(首都师范大学生命科学学院 北京 100048)
生物体辐射抗性的机理一直是人们关注的热点。耐辐射奇异球菌(Deinococcus radiodurans)是迄今发现的具有极强辐射抗性的微生物之一,对其辐射抗性机制的研究也最为透彻[1-5]。迄今为止,大量研究主要集中在DNA修复能力方面[6-8]。近年来,Daly 等[9-10]认为抗氧化保护是辐射抗性的关键因素。这一假说提出电离辐射可以导致水光解,产生的活性氧自由基(ROS)会损伤核酸、蛋白质和脂类等生物大分子。因此推测有机体抗氧化能力的高低决定了其辐射生存率。在耐辐射奇异球菌中,研究表明Mn复合物是ROS的有效清除剂,但不同生物体可能具有不同的辐射抗性机制[11]。
芽胞杆菌也存在辐射抗性现象。蜡样芽胞杆菌(Bacillus cereus)的表面S-层蛋白可赋予细胞更强的辐射抗性[12]。枯草芽胞杆菌(Bacillus subtilis)的非同源末端连接(NHEJ)参与了辐射后DNA的修复[13]。在巨大芽胞杆菌(Bacillus megaterium)等其它芽胞杆菌中,电离辐射可以影响多个细胞代谢途径,包括DNA重组、蛋白质更新和胁迫响应机制等[14-18]。但目前对芽胞杆菌辐射抗性机理的研究还十分有限。
芽胞杆菌T61(Bacillus sp. T61)是从西藏土样中分离得到的一株具有较强辐射抗性的菌株。本研究测定了该菌株对γ-射线的辐射抗性,并研究了γ-射线辐照后芽胞杆菌T61在蛋白质组水平发生的变化,以期发现细菌辐射抗性的新机制,拓展对生物体辐射抗性的认识。
1 材料与方法
1.1 实验菌株和培养条件
本研究所用菌株为芽胞杆菌 T61,来自西藏冈巴拉山土样,由本实验室分离保存。对照菌株 D.radiodurans (CGMCC 1.633) 购自中国普通微生物菌种保藏管理中心,E. coli K12由中国农业科学院张维研究员馈赠。芽胞杆菌T61和耐辐射奇异球菌用TGY培养基(0.5%胰蛋白胨、0.3%酵母提取物、0.1%葡萄糖)30 ℃培养,E. coli K12用LB培养基(1%胰蛋白胨、0.5%酵母提取物、1%NaCl) 37 ℃培养。
1.2 主要试剂和仪器
B-PER 细菌总蛋白抽提试剂盒(Thermo scientific);双向电泳相关试剂(GE healthcare);Ultrospec 3100 pro 紫外可见分光光度计(GE healthcare);Ettan IPGphor 3 等电聚焦仪(GE healthcare);垂直电泳仪(BIO-RAD);ImageScanner III 扫描仪(GE healthcare);4800 Plus MALDI TOF/TOFTMAnalyzer (ABI);60Co γ-射线射源(北京大学化学与分子工程学院应用化学系钴源室)。
1.3 芽胞杆菌T61的γ-射线辐射抗性的分析
将芽胞杆菌T61菌株接种于TGY培养基中,于30 ℃振荡培养至对数早期(A600=0.3-0.4)。离心收集菌体,用0.1 mol·L-1的磷酸钾缓冲液(pH7.0)洗一次,并重新悬浮于等体积的磷酸钾缓冲液中。取1.5mL 菌液加到1.5mL EP 管中,室温下(10- 20 ℃)进行60Co γ-射线辐照(剂量率 10 Gy·min-1),辐射剂量分别为2、4、6、8和10 kGy。辐射后,分别用0.1 mol·L-1磷酸钾缓冲液适当稀释,取稀释液涂布于TGY平板,30 ℃培养。计算辐射存活率。以对数早期D. radioduran 和E. coli K12细胞为对照。
1.4 辐射处理和细胞可溶性蛋白的提取
将培养至对数早期(A600=0.3-0.4)的芽胞杆菌T61菌液分为两部分,一部分进行10 kGy60Co γ-射线辐射,另一部分作为对照,同样条件下放置,未经辐射。辐射后离心收集菌体,-20 ℃储存。
处理组(γ-射线辐射)和对照组(未辐射)菌体各3g,分别悬浮于适当体积的B-PER® Reagent(Pierce)中,补加 0.2 μmol·mL-1PMSF 和 200 μg·mL-1溶菌酶,轻微摇动 20 min。4 ℃ 15000g离心15 min,上清液转移至新管,加入1.2倍体积的Tris饱和酚,冰上涡旋20 min。4 ℃ 20000g离心20 min,酚相(下相)转移至新管,加入5倍体积0.1 mol·L-1乙酸铵(甲醇配制),混匀,-20 ℃静置2 h。4 ℃ 20000g离心10 min,沉淀用无水乙醇和丙酮(含0.07% β-巯基乙醇)各洗一次,冷冻干燥,-20℃保存。
1.5 双向电泳分离蛋白质
将蛋白质样品溶于适量上样缓冲液(8 mol·L-1Urea, 2% CHAPS, 50 mmol·L-1DTT 和 0.5% IPG buffer pH=4-7)中,采用 Bradford法测定蛋白质浓度。按照生产厂商(GE Healthcare)的使用说明进行双向电泳分析。首先取340 μL样品(含800 μg蛋白)加入水化盘中,放入18 cm的胶条 (18 cm, pH 4-7),水化18 h。水化结束后,采用Ettan IPGphor III进行等电聚焦,聚焦程序为250 V,3 h;500 V,2 h;1000 V,1 h;10000 V,3 h;10000 V,100000 Vh;500 V,20 h。聚焦完毕,平衡后进行第二向SDS-PAGE,分离胶浓度为 12.5%。用考马斯亮蓝G-250染液染色过夜,次日进行脱色。将脱色后的凝胶用ddH2O清洗,采用ImageScanner III 扫描仪进行图像采集,用BIO-RAD PDQuestTM2-D Analysis Software 8.0.1对图像进行分析。
处理组和对照组分别提取了3次蛋白质样品,每个样品至少进行2次双向电泳分离。从凝胶上切取表达量发生2倍以上变化的蛋白质样点进行质谱分析。
1.6 质谱分析和差异蛋白的数据库查询
差异蛋白送交上海中科新生命生物科技有限公司进行MALDI-TOF/TOF质谱分析。采用MASCOT搜索软件在NCBI非冗余库 (NCBInr) 中进行肽谱比对,根据蛋白质得分(C. I. % >95%)、肽段的匹配数目(≥5)和序列覆盖度(>15%)等评价蛋白质鉴定的可靠性。
2 结果与讨论
2.1 芽胞杆菌T61的γ-射线辐射抗性分析
芽胞杆菌T61对γ-射线的辐射抗性远高于对照菌株大肠杆菌K12。在本实验条件下,当辐射剂量为 1 kGy时,大肠杆菌 K12的生存率接近于0.0001%,而T61的生存率约为40%。随着辐射剂量的增加,T61存活率下降,D10为4 kGy。当辐射剂量为10 kGy时,存活率为0.002%,比耐辐射奇异球菌低100倍(见图1)。
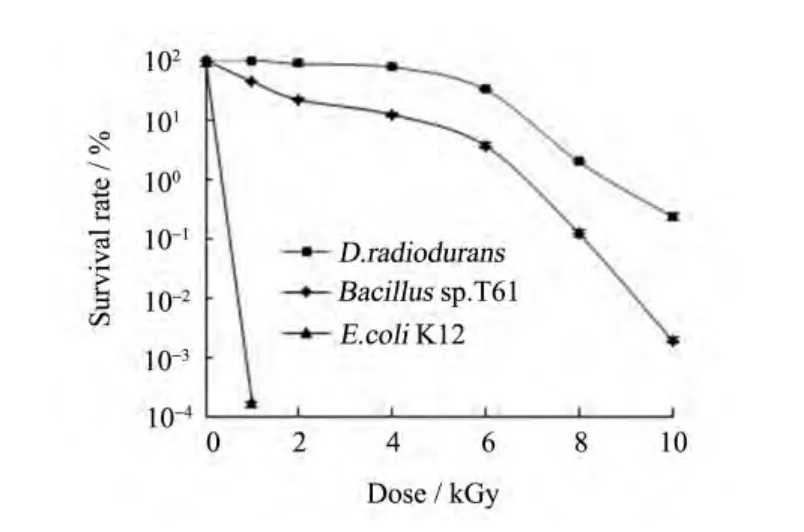
图1 芽胞杆菌T61 γ-射线辐照后的存活曲线Fig.1 Survival curves of strain Bacillus sp. T61 after γ-ray irradiation
2.2 γ-射线辐射后芽胞杆菌T61蛋白组的变化
对处理组(γ-射线辐射)和对照组(未辐射)可溶性蛋白进行双向电泳分析,代表性电泳图谱如图2所示。在每块胶上可分离得到900多个蛋白点,这些蛋白点主要集中在分子量20-100 kDa,pH4-7的范围内。与未经辐射的蛋白质样品相比,10kGy60Co γ-射线辐射后至少有 30个蛋白质点出现显著变化。其中22个蛋白点上调为辐射前的两倍以上,8个蛋白点下调为辐射前的二分之一以下。
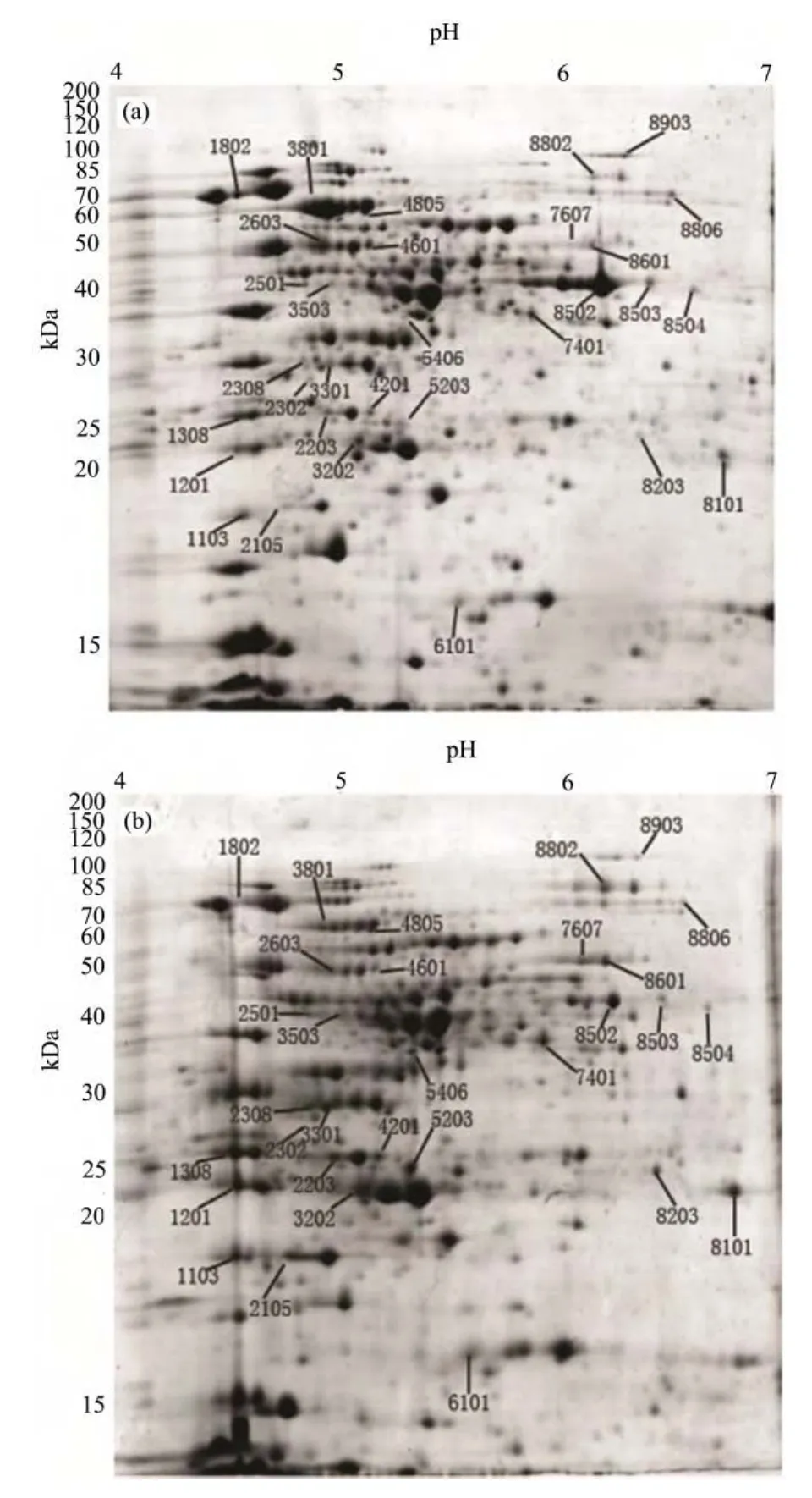
图2 辐照前(a)后(b)芽胞杆菌T61的蛋白质组图谱Fig.2 Protein profile of Bacillus sp. T61 cells before (a) and after (b) irradiation
根据双向电泳的分析结果,对辐射后发生显著变化的 30个蛋白点进行 MALDI-TOF-MS质谱分析,鉴定结果如表1所示。其中有4个蛋白点(2203,3202, 4601, 6101)含有两种蛋白,因此30个蛋白点包括34种蛋白质,其中25个蛋白质上调,9个蛋白质下调。进一步根据NCBI 蛋白相邻类的聚簇分析(Cluster of orthologous groups of proteins, COGs)对这些差异蛋白质进行了分类,它们分属信息贮存和加工、细胞过程和信号、代谢和未鉴定蛋白四大类,主要涉及细胞的抗氧化防御、核苷酸代谢、能量代谢、转录以及蛋白质的翻译和更新等过程。
在这些差异表达的蛋白质中,引人注目的是,一大组抗氧化酶类在辐射后上调表达,包括醛-酮还原酶(蛋白点2308和5406)、巯基过氧化物酶(蛋白点2105)、烷基过氧化氢还原酶(蛋白点1201)、Mn-超氧化物歧化酶(蛋白点3202)和硝基还原酶家族蛋白(蛋白点3202)等(图3)。在耐辐射奇异球菌中也观察到超氧化物歧化酶(SOD)等在辐射后表达量增加[19]。本研究首次发现醛-酮还原酶(AKR)在辐射后显著上调表达。由于醛-酮还原酶是一种NAD(P)H依赖型的氧化还原酶类,可以催化多种脂肪族和芳香族醛、酮类物质的还原[20],我们推测醛-酮还原酶可能参与了辐射后脂类过氧化物的解毒过程,但具体机制有待进一步研究。
在对抗辐射导致的氧化胁迫方面,除抗氧化酶类上调之外,一些可以减少活性氧自由基生成的酶类也发生了变化。琥珀酸脱氢酶(蛋白点8802)在辐射后上调5.2倍, 该酶氧化还原中心的有效排列可以使电子快速转移给泛醌,从而减少了电子传递过程中自由基的产生[21]。亚硫酸还原酶β-亚基(蛋白点 8806)和 4-羟基-3-甲丁基-2-烯-1-基-二磷酸合成酶(GcpE,蛋白点 8504)分别具有一个[4Fe-4S]簇,该铁硫簇容易受到O2·-和 H2O2的攻击,继而产生羟基自由基(·OH)[22-23]。辐射后亚硫酸还原酶β-亚基和GcpE下调,从而可以进一步减少辐射后自由基的生成。
辐射后芽胞杆菌T61下调有氧氧化途径,而以酵解为获得能量的主要方式,这一点与耐辐射奇异球菌十分相似[3]。辐照后,可以观察到参与酵解途径的一些酶类,例如磷酸丙糖异构酶(蛋白点1308)和果糖-1,6-二磷酸醛缩酶(蛋白点3301)和乳酸脱氢酶(蛋白点7401)上调表达,而三羧酸循环(TCA)酶类,例如丙酮酸脱氢酶复合体E3(蛋白点4805)和丙酮酸脱氢酶E1α亚基(蛋白点8503)下调表达。此外,芽胞杆菌还可能利用细胞内其它代谢物获得能量,例如丁醇脱氢酶(蛋白点3503)和聚羟基烷酸合酶(蛋白点2203)在辐射后上调,二者分别参与丁醇[24]和聚羟基烷酸(PHAs)[25]的代谢。
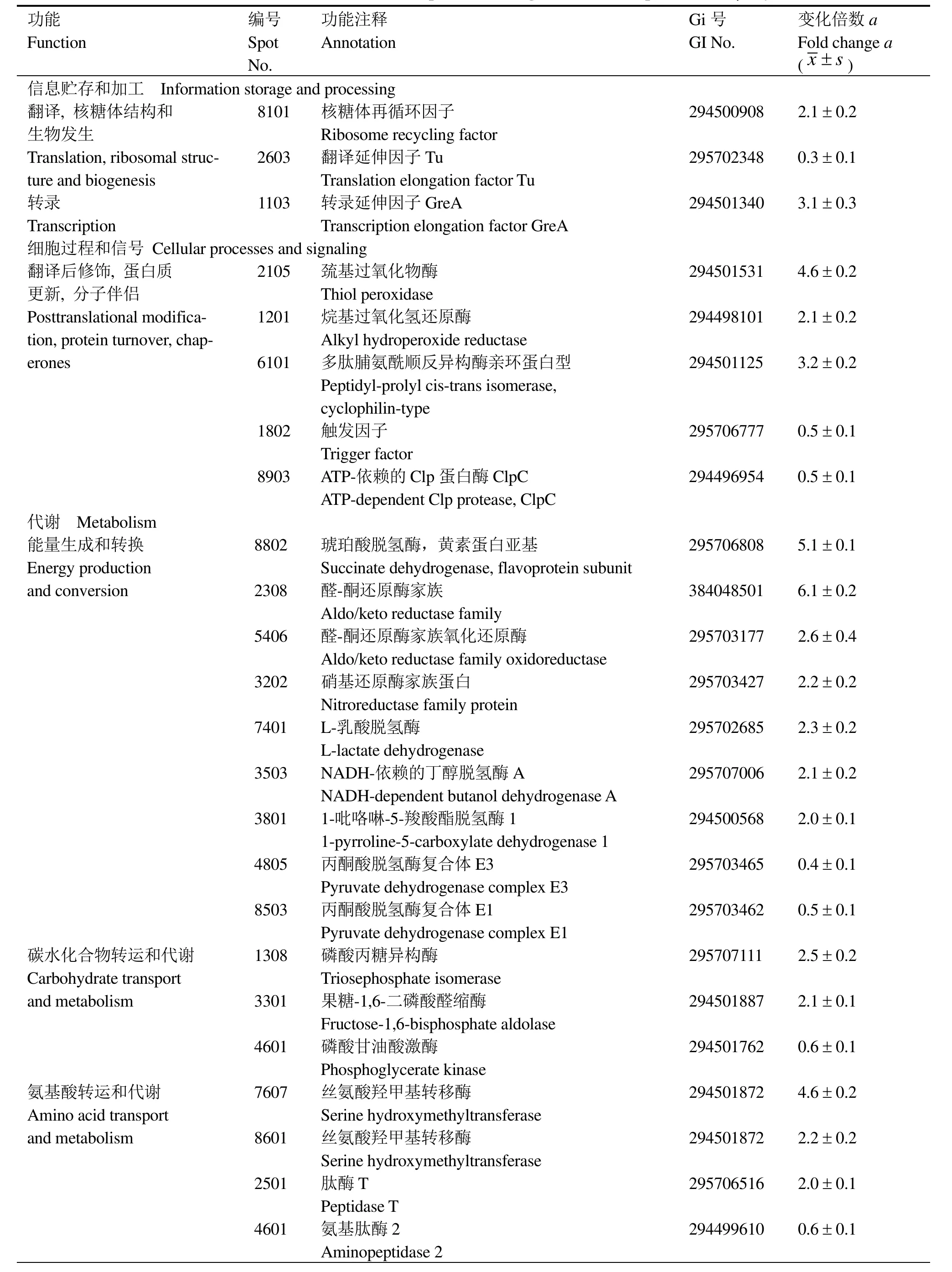
表1 γ-射线辐射后芽胞杆菌T61差异蛋白质的功能注释Table 1 Functional annotation of the cellular proteins changed in Bacillus sp. T61 after γ-ray irradiation
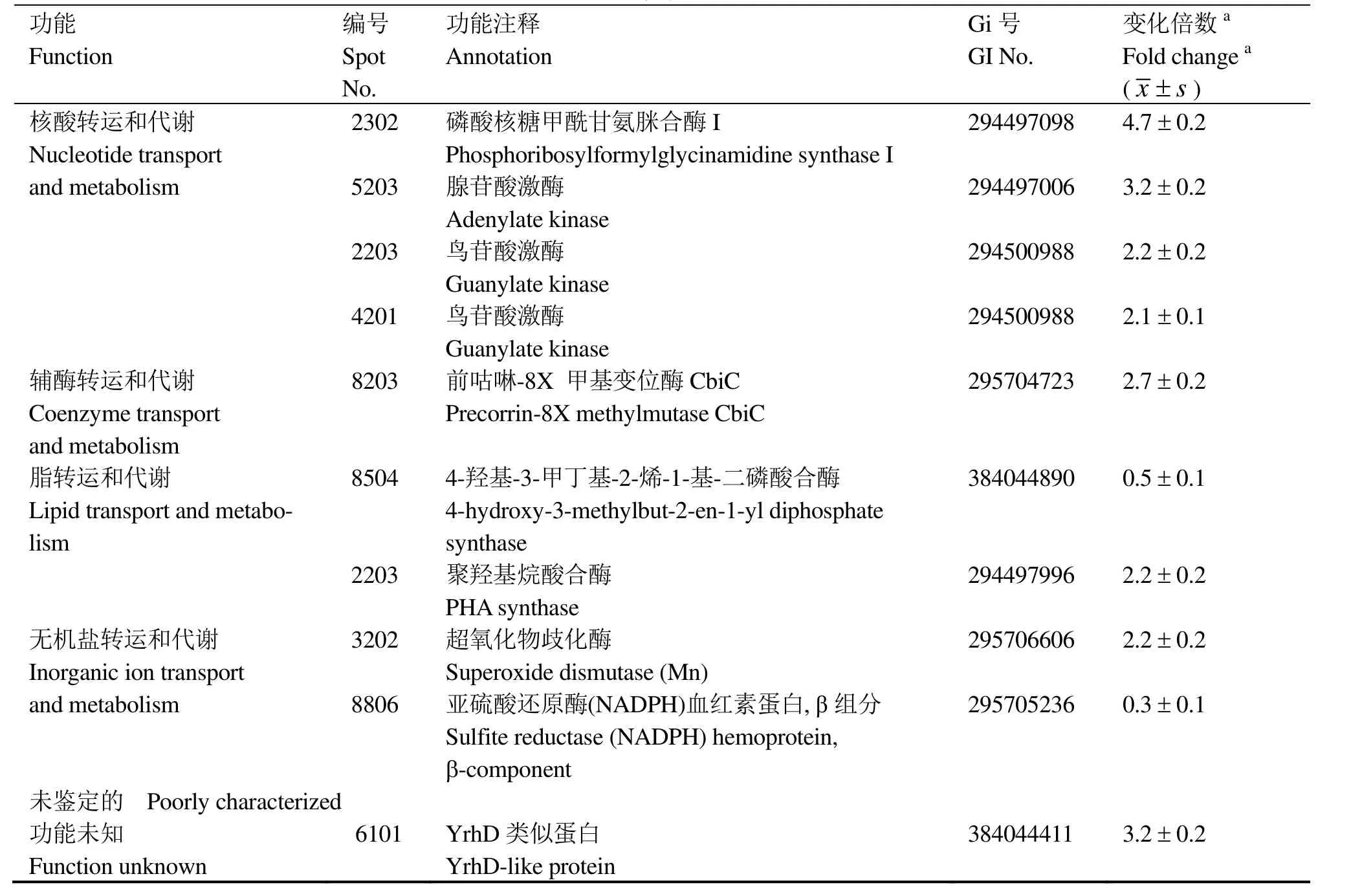
续表

图3 γ-射线辐照后表达量增加的抗氧化酶类(局部放大图)未辐照(a);辐照后(b);从左至右:蛋白点1201, 2105, 2308, 3202, 5406Fig.3 Zoomed view of antioxidation enzymes enhanced by γ-ray irradiation
辐射后细胞除抵抗氧化胁迫外,还需要修复DNA损伤。本研究发现5种参与核苷酸代谢的酶类在辐照后上调,包括磷酸核糖甲酰甘氨脒合酶(蛋白点2302)、丝氨酸羟甲基转移酶(蛋白点7607和8601)、前咕啉-8X甲基变位酶(蛋白点8203)、腺苷酸激酶(蛋白点5203)和鸟苷酸激酶(蛋白点2203和 4201)。其中丝氨酸羟甲基转移酶和鸟苷酸激酶分别具有两种亚型,其分子量相同,但等电点发生变化,暗示出辐射过程中可能发生了蛋白质修饰。
辐射后转录和翻译系统也发生了一些变化。转录延伸因子GreA(蛋白点1103)、核糖体再循环因子(蛋白点 8101)和多肽脯氨酰顺反异构酶(PPI)亲环蛋白型(蛋白点 6101)在辐射后上调。在D.radiodurans也观察到转录延伸因子和PPI在辐射后表达量增加[19,26-27]。触发因子(蛋白点 1802)是PPI家族的成员[28],在辐射后下调。翻译延伸因子EFTu(蛋白点2603)也出现下调。但在D.radiodurans和其它Bacillus的辐射后恢复生长中,EFTu是上调表达的[29-30],推测EFTu 在辐射过程中被降解,但可能在细胞恢复生长中被重新合成。
在蛋白质的降解方面,研究表明 Clp 蛋白酶(蛋白点8903)下调,而肽酶T(蛋白点2501)上调,推测不同类型的蛋白酶可能参与了辐射后受损蛋白质的降解。
无机离子和有机小分子在辐射抗性中的作用日益受到重视。在D.radiodurans中,Mn(II)-复合物可以清除辐射产生的自由基[11]。在 Halobacterium NRC-1中,高浓度的Br-可以增强细胞辐射抗性[31]。本研究发现,一种功能未知的蛋白,YrhD-类似蛋白(蛋白点6101)在辐射后显著上调表达。在巨大芽胞杆菌中,这一基因两侧是 modA (编码钼 ABC转运蛋白)和yrhE(编码钼氧化还原酶家族蛋白)[32],暗示出 YrhD-类似蛋白可能参与钼代谢,其与辐射抗性的关系尚需进一步研究。
3 结论
芽胞杆菌 T61是一种具有较强辐射抗性的菌株,在10 kGy γ-射线辐射后,芽胞杆菌T61蛋白质组发生了以下变化:(1)多种类型抗氧化酶在辐射后上调,推测抗氧化保护在T61辐射抗性中发挥关键作用;(2)辐射过程中,细胞主要通过酵解或利用其它代谢物获得能量;(3)核苷酸从头合成途径的上调有利于促进DNA的修复;(4)转录和翻译系统的启动以保证受损蛋白质的修复和新蛋白质的再合成。图4概括了芽胞杆菌T61细胞蛋白质组在60Co γ-辐射过程中所发生的主要变化。
本研究首次发现醛-酮还原酶在辐射后显著上调。由于醛-酮还原酶超家族可以催化脂类物质过氧化形成的醛类物质的还原,因而在辐射后的抗氧化保护中发挥重要作用。我们目前正在进行醛-酮还原酶基因缺失突变体的构建和功能分析,初步研究结果表明醛-酮还原酶基因的缺失可使菌株的辐射抗性降低,相关生物学功能的分析正在进行中。
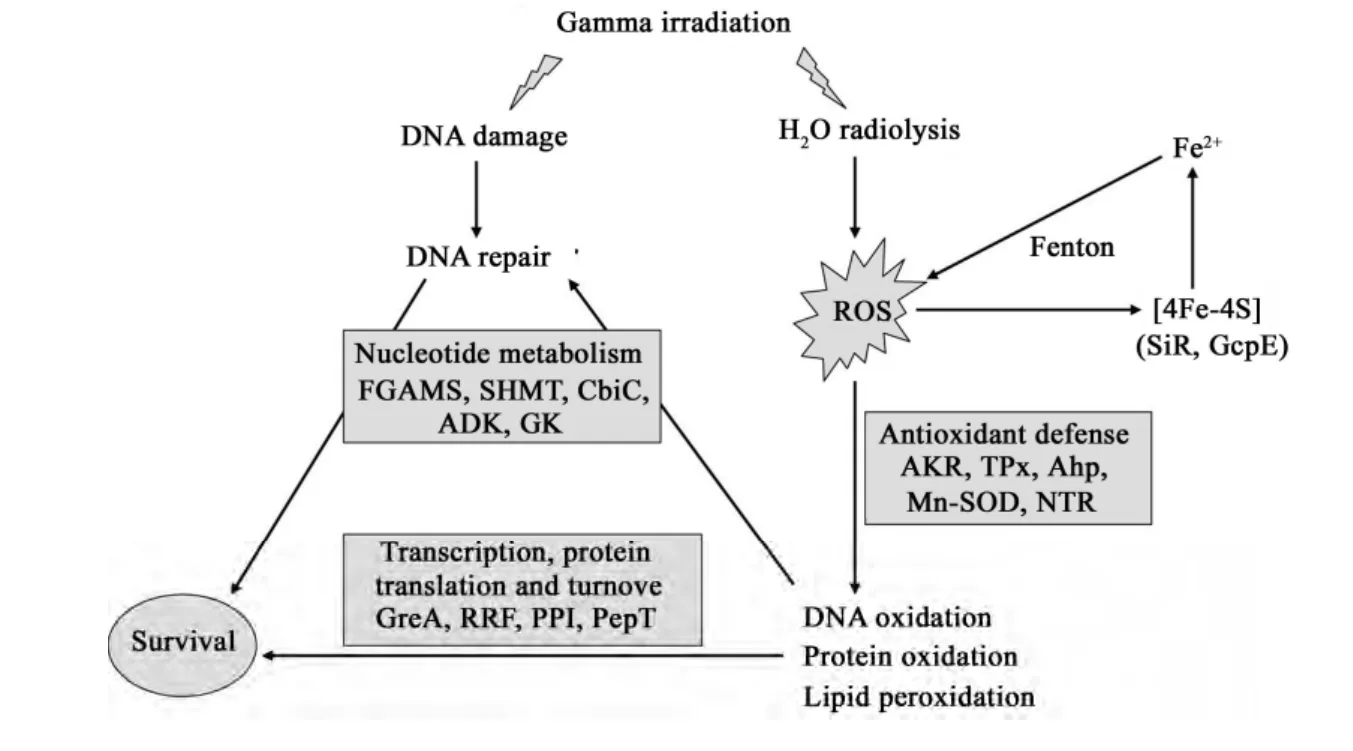
图4 γ-射线辐照后芽胞杆菌T61细胞蛋白质组变化图解Fig.4 Main change scheme of cellular proteins in Bacillus sp. T61 after γ-ray irradiation
致谢感谢崔素霞教授在蛋白质组学分析方面给予的建议和指导。感谢北京大学化学与分子工程学院应用化学系钴源室为60Co γ-射线辐照处理提供的帮助。
1 Krisko A, Radman M. Biology of extreme radiation resistance: the way of Deinococcus radiodurans [J]. Cold Spring Harbor Perspectives in Biology, 2013, 5(7):a012765.
2 Cox M M, Battista J R. Deinococcus radiodurans-the consummate survivor [J]. Nature Review Microbiology,2005, 3(11): 882-892.
3 Slade D, Radman M. Oxidative stress resistance in Deinococcus radiodurans [J]. Microbiology and Molecular Biology Reviews, 2011, 75(1): 133-191.
4 Misra H S, Rajpurohit Y S, Kota S. Physiological and molecular basis of extreme radioresistance in Deinococcus radiodurans [J]. Current Science, 2013, 104(2): 194-205.
5 Luan H, Meng N, Fu J, et al. Genome-wide transcriptome and antioxidant analyses on gamma-irradiated phases of Deinococcus radiodurans R1 [J]. PLoS One, 2014, 9(1):e85649.
6 Zahradka K, Slade D, Bailone A, et al. Reassembly of shattered chromosomes in Deinococcus radiodurans [J].Nature, 2006, 443(7111): 569-573.
7 Slade D, Lindner A B, Paulg, et al. Recombination and replication in DNA repair of heavily irradiated Deinococcus radiodurans [J]. Cell, 2009, 136(6): 1044-1055.
8 de la Tour C B, Passot F M, Toueille M et al.Comparative proteomics reveals key proteins recruited at the nucleoid of Deinococcus after irradiation-induced DNA damage [J]. Proteomics, 2013, 13(23-24): 3457-3469.
9 Daly M J. A new perspective on radiation resistance based on Deinococcus radiodurans [J]. Nature Review Microbiology, 2009, 7(3):237-245.
10 Daly MJ. Death by protein damage in irradiated cells [J].DNA Repair, 2012, 11(1):12-21.
11 Culotta V C, Daly M J. Manganese complexes: diverse metabolic routes to oxidative stress resistance in prokaryotes and yeast [J]. Antioxidants & Redox Signaling, 2013, 19(9): 933-944.
12 Kotiranta A K, Ito H, Haapasalo M P, et al. Radiation sensitivity of Bacillus cereus with and without a crystalline surface protein layer [J]. FEMS Microbiology Letters, 1999, 179(2): 275-280.
13 Moeller R, Stackebrandt E, Reitzg, et al. Role of DNA repair by nonhomologous-end joining in Bacillus subtilis spore resistance to extreme dryness, mono- and polychromatic UV, and ionizing radiation [J]. Journal of Bacteriology, 2007, 189(8): 3306-3311.
14 Granger A C, Gaidamakova E K, Matrosova V Y, et al.Effects of Mn and Fe levels on Bacillus subtilis spore resistance and effects of Mn2+, other divalent cations,orthophosphate, and dipicolinic acid on protein resistance to ionizing radiation [J]. Applied and Environmental Microbiology, 2011, 77(1): 32-40.
15 Ghosh S, Ramirez-Peralta A, Gaidamakova E, et al.Effects of Mn levels on resistance of Bacillus megaterium spores to heat, radiation and hydrogen peroxide [J].Journal of Applied Microbiology, 2011, 111(3): 663-670.
16 Gupta A K, Pathak R, Singh B, et al. Proteomic analysis of global changes in protein expression during exposure of gamma radiation in Bacillus sp HKG 112 isolated from saline soil [J]. Journal of Microbiology and Biotechnology,2011, 21(6): 574-581.
17 Yazdani M, Naderi-Manesh H, Khajeh K, et al. Isolation and characterization of a novel gamma-radiation-resistant bacterium from hot spring in Iran [J]. Journal of Basic Microbiology, 2009, 49(1): 119-127.
18 Yazdani M, Naderi-Manesh H. Comparative proteomics analysis of a novel γ-radiation-resistant bacterium wild-type Bacillus megaterium strain WHO DQ973298 recovering from 5 kGy γ-irradiation [J]. Iranian Journal of Biotechnology, 2012, 10(2): 96-105.
19 Basu B, Apte S K. Gamma radiation induced proteome of D radiodurans primarily targets DNA repair and oxidative stress alleviation [J]. Molecular and Cellular Proteomics,2012, 11(1): M111. 011734
20 Mindnich R D, Penning T M. Aldo-keto reductase (AKR)superfamily, genomics and annotation [J]. Human Genomics, 2009, 3(4): 362-370.
21 Yankovskaya V, Horsefield R, Törnroth S, et al.Architecture of succinate dehydrogenase and reactive oxygen Species generation [J]. Science, 2003, 299(5607):700-704.
22 Crane B R, Siegel L M, Getzoff E D. Structures of the siroheme- and Fe4S4-containing active center of sulfite reductase in different states of oxidation, heme activation via reduction-gated exogenous ligand exchange [J].Biochemistry, 1997, 36(40): 12101-12119.
23 Rivasseau C, Seemann M, Boisson A M, et al.Accumulation of 2-C-methyl-D-erythritol 2,4-cyclodiphosphate in illuminated plant leaves at supraoptimal temperatures reveals a bottleneck of the prokaryotic methylerythritol 4-phosphate pathway of isoprenoid biosynthesis [J]. Plant, Cell & Environment,2009, 32(1): 82-92.
24 Veeranagouda Y, Benndorf D, Heipieper H J, et al.Purification and characterization of NAD-dependent n-butanol dehydrogenase from solvent-tolerant n-butanol-degrading Enterobacter sp VKGH12 [J].Journal of Microbiology and Biotechnology, 2008, 18(4):663-669.
25 McCoolg J, Cannon M C. PhaC and PhaR are required for polyhydroxyalkanoic acid synthase activity in Bacillus megaterium [J]. Journal of Bacteriology, 2001, 183(14):4235-4243.
26 Zhang C, Wei J, Zheng Z, et al. Proteomic analysis of Deinococcus radiodurans recovering from gamma-irradiation [J]. Proteomics, 2005, 5(1): 138-143.
27 Lu H, Gaog, Xug, et al. Deinococcus radiodurans PprI switches on DNA damage-response and cellular survival networks after radiation damage [J]. Molecular and Cellular Proteomics, 2009, 8(3): 481-494.
28 Bigot A, Botton E, Dubail I. A homolog of Bacillus subtilis trigger factor in Listeria monocytogenes is involved in stress tolerance and bacterial virulence [J].Applied and Environmental Microbiology, 2006, 72(10):6623-6631.
29 Tanaka A, Hirano H, Kikuchi M, et al. Changes in cellular proteins of Deinococcus radiodurans following gamma irradiation [J]. Radiation and Environmental Biophysics,1996, 35(2): 95-99.
30 Joshi B, Schmid R, Altendorf K, et al. Protein recycling is a major component of post-irradiation recovery in Deinococcus radiodurans strain R1 [J]. Biochemical Biophysical Research Communications, 2004, 320(4):1112-1117.
31 Kish A, Kirkalig, Robinson C, et al. Salt shield:intracellular salts provide cellular protection against ionizing radiation in the halophilic archaeon,Halobacterium salinarum NRC-1 [J]. Environmental Microbiology, 2009, 11(5): 1066-1078.
32 Liu L, Li Y, Zhang J, et al. Complete genome sequence of the industrial strain Bacillus megaterium WSH-002 [J].Journal of Bacteriology, 2011, 193(22): 6389-6390.

