番茄细菌性叶斑病菌超分支滚环扩增快速检测技术
王念武,王 婷,沈建国,胡方平*
(1.福建农林大学植物保护学院,福州 350002; 2.福建出入境检验检疫局,福州 350001)
实验方法与技术
番茄细菌性叶斑病菌超分支滚环扩增快速检测技术
王念武1,2,王 婷1,沈建国2,胡方平1*
(1.福建农林大学植物保护学院,福州 350002; 2.福建出入境检验检疫局,福州 350001)
根据番茄细菌性叶斑病菌(Pseudomonassyringaepv.tomato,Pst)的一段特异蛋白基因序列,按照锁式探针的设计原理设计探针和扩增引物,优化系列反应条件,建立了特异性的Pst超分支滚环扩增技术。试验结果表明:该检测技术能够从供试的10种不同的病原菌中特异性地检测出番茄细菌性叶斑病菌,DNA检测的最低浓度为500fg/μL,检测灵敏度高于常规PCR。
番茄细菌性叶斑病菌; 超分支滚环扩增; 锁式探针
番茄细菌性叶斑病(bacterial speck of tomato)是番茄上重要的病害之一,病原菌为Pseudomonassyringaepv.tomato(Okabe) Young ,Dye & Wilkie(Pst)。该病害最早于1933年在美国的布雷登顿市被发现,但因为这种病害很容易和其他细菌性病害混淆,致使在其他地方很少被报道,直到1977-1978年在霍姆斯特德地区大暴发之后才被重视[1]。该病菌仅危害番茄,可造成叶片斑点、坏死,导致植株生长不良而减产;在果实上危害形成瑕疵症状,影响番茄的感官品质而导致难以销售[2]。该病害因缺少典型的诊断鉴别症状极易和其他病害的症状混淆,另外,病原菌又可由种子携带,所以对该病害的快速诊断和病原菌的快速检测是植病工作者的研究内容之一。
传统的方法可用于诊断和鉴定P.syringaepv.tomato病原,但费力费时;用非放射性元素标记DNA探针可用于检测番茄植株上的叶斑病菌,但因菌株自身产生毒素,检测结果常受到影响[3];Zaccardelli等[4]利用hrpZ基因特异性序列通过常规PCR方法来检测受感染的植株和幼苗,获得了一定的成功。但常规PCR的方法有一定的局限性,容易受污染形成假阳性结果,而且不能高通量检测病原物。自锁式探针被报道以来[5],它以灵敏、特异、稳定等特点被广泛应用到分子检测中。锁式探针是由3′和5′端两段靶标序列和中间的一段对检测结果没有影响的连接序列构成的一种较长的单链寡核苷酸片段。其工作原理是当检测体系中存在靶标DNA时,锁式探针两端的靶标序列就会与靶标DNA完全互补配对,线型锁式探针在连接酶的作用下被有效地连接成环型,而在没有相应靶标DNA存在时锁式探针不被连接酶连接,仅以线性形式存在。在核酸外切酶的作用下,多余的线性锁式探针被消化水解,连接成环状的锁式探针在BstDNA聚合酶和通用引物的作用下恒温扩增[6],其扩增效率在1h之内就可以达到109或更多拷贝[7]。锁式探针的恒温扩增具有极好的检测灵敏度、特异性和实用性,近几年已被广泛地应用于细胞原位检测、微生物、医学病原检测鉴定等领域[8-9]。Schopf 等[10]通过琼脂糖珠表面修饰扩增引物,然后进行滚环扩增,并通过荧光探针检测,DNA检测限达到1amol;Su等[11]利用一种高灵敏的磁珠电化学发光方法,结合滚环扩增技术来分析单链核苷酸多态性,结果可以将1/100000突变型样品从野生型中区分开;Li等[12]利用纳米技术结合滚环扩增技术,通过金纳米粒子探针与酶消化的滚环扩增产物杂交,实现用肉眼比较颜色来进行检测;Cheng等[13]在双抗体免疫夹心的基础上建立了免疫滚环扩增方法,通过在引物5′ 端标记生物素,实现探针与扩增产物原位杂交检测;Yan等[14]利用了纳米金能够同时结合蛋白和核酸的特性,将用于RCA反应的DNA引物和抗体通过纳米金连接起来,形成一个更强信号的扩增反应,其灵敏度可达到5 amol/L,比传统的ELISA方法扩大5个数量级;郭艳玲等[15]用超分支滚环扩增技术对60例肺结核病人、38例非结核对照组和20例健康人的痰标本进行检测,灵敏度达740amol/L,检测菌悬液的灵敏度达200cfu/mL;在植物病原检测中,Szemes等[6]设计了11种锁式探针,采用锁式探针与微阵列相结合的方法,对11种植物病原物(真菌和线虫)进行分析,建立了特异性强、灵敏度高的检测技术体系,黄冠军[16]等据柑橘溃疡病菌(Xanthomonasaxonopodispv.citri)独有的蛋白基因序列设计特异性锁式探针进行滚环扩增,建立了柑橘溃疡病菌滚环扩增检测技术体系。本研究旨在依据番茄细菌性叶斑病菌独有的蛋白基因序列,设计特异的锁式探针及其扩增引物,建立特异、灵敏的番茄细菌性叶斑病菌超分支滚环扩增(HRCA)检测技术,为我国的口岸把关,也为番茄细菌性叶斑病菌的疫情动态监测提供新的分子检测技术。
1 材料与方法
1.1 材料
1.1.1 供试菌株
番茄溃疡病菌[Clavibactermichiganensissubsp.michiganensis(Smith)Davis et al.],由福建省农业科学院作物研究所邱思鑫提供;番茄细菌性叶斑病菌(Pseudomonassyringaepv.tomato)、番茄青枯病菌[Ralstoniasolanacearum(Smith) Yabuuchi et al.]、西瓜细菌性果斑病菌(Acidovoraxavenaesubsp.citrulliWillems et al.)、水稻细菌性条斑病菌[Xanthomonasoryzaepv.oryzicola(Fang et al.) Swing et al.]、杨桃细菌性斑点病菌[P.syringaepv.averrhoii(van Hall) Bergey]、柑橘溃疡病菌[Xanthomonascitri(Hasse) Dawson]、枯草芽胞杆菌[Bacillussubtilis(Ehrenberg) Cohn]、香蕉枯萎病菌[Fusariumoxysporumf.sp.cubense(E.F.Smith) W.C.Snyd.& H.N.Hans.]、香蕉炭疽病菌[Colletotrichummusae(Berk.& Curt.) Arx.]均由福建农林大学细菌实验室提供。
1.1.2 主要试剂和仪器
TaqDNA ligase、Exonuclease I、Exonuclease Ⅲ、BstDNA polymerase、无菌 ddH2O均购自New England Biolabs(NEB)公司。dNTPs、DNA提取试剂盒,100bp DNA ladder,分别购自Promega和Tangen公司。PCR仪(ABI公司)、核酸蛋白仪、凝胶成像系统、离心机(Eppendorf公司)。
1.2 锁式探针的设计
根据Fanelli等[17]获得的一段255 bp大小的番茄细菌性叶斑病菌特异性片段(GenBank: AJ581671.1),按照锁式探针的设计原则[6,18]采用软件Oligo和DNASTAR设计锁式探针。设计的锁式探针两端检测臂T1、T2和Pst的特异性片段碱基序列互补,锁式探针中间的一段连接区域采用小鼠基因xist(NCBI Reference Sequence: NR_001463.3)一段序列,设计好的锁式探针使用Mfold(http:∥www.bioinfo.rpi.edu/applications/mfold/)预测探针的二级结构,确保锁式探针的二级结构最小。锁式探针扩增引物根据其连接序列设计,最后,将所设计的锁式探针序列和扩增引物在NCBI数据库进行同源性比对,筛选出特异性强的锁式探针和性能稳定易于扩增的扩增引物。锁式探针、扩增引物均由上海生物工程有限公司合成,锁式探针合成时对其5′端进行磷酸化修饰。
表1番茄细菌性叶斑病菌的超分支滚环扩增所用到的锁式探针及其扩增引物
Table1PadlockprobeandprimersofHCRAforPst
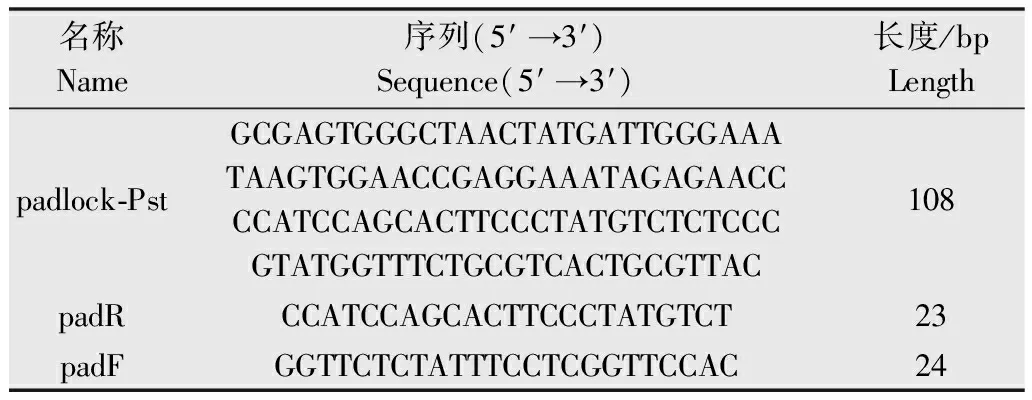
名称Name序列(5′→3′)Sequence(5′→3′)长度/bpLengthpadlock⁃PstGCGAGTGGGCTAACTATGATTGGGAAATAAGTGGAACCGAGGAAATAGAGAACCCCATCCAGCACTTCCCTATGTCTCTCCCGTATGGTTTCTGCGTCACTGCGTTAC108padRCCATCCAGCACTTCCCTATGTCT23padFGGTTCTCTATTTCCTCGGTTCCAC24
1.3 超分支滚环扩增检测技术建立与优化
1.3.1 锁式探针环化连接
将锁式探针与番茄细菌性叶斑病菌以及参照菌株的DNA模板分别进行环化连接,反应体系为10μL:10×TaqDNA连接酶缓冲液1μL,40U/μL的TaqDNA连接酶0.15 μL,400pmol/L锁式探针0.2 μL,DNA模板2 μL,无菌ddH2O补足至10μL。采用热循环法连接,反应条件为:94 ℃ 4 min;94 ℃30s,65 ℃ 5 min,15个循环;95 ℃ 15 min灭活TaqDNA连接酶。反应结束后立即将反应管冰浴5 min。锁式探针连接优化包括线性锁式探针的终浓度、DNA连接酶、连接程序选择等,本文采用的是热循环法,DNA连接酶采用的是NEB公司的TaqDNA连接酶,仅针对线性锁式探针的终浓度进行优化,设置不同浓度的线性锁式探针,按照上述方法进行连接。
1.3.2 消化反应
冰浴后的PCR反应管中加入10μL的混合液:10×核酸外切酶I缓冲液2 μL;5 U/μL的核酸外切酶I 1.5 μL;10×核酸外切酶Ⅲ缓冲液2 μL;5 U/μL的核酸外切酶Ⅲ 1μL;无菌 ddH2O补足至10μL,37 ℃反应2.5 h,彻底消化未环化的线性探针,再于95 ℃反应3h,灭活剩余的核酸外切酶。
1.3.3 超分支滚环扩增
将锁式探针连接环化消化后的产物作为模板,用扩增引物进行超分支滚环扩增,反应体系为25 μL:10×BstDNA聚合酶缓冲液2.5 μL,8U/μL的BstDNA聚合酶0.5 μL,10μmol/L 的padR 0.5 μL,10μmol/L的padF 0.5 μL,10mmol/L的 dNTPs 1μL,环化连接消化后产物2 μL,无菌 ddH2O 补至25 μL。反应条件:62.5 ℃反应1.5 h。反应结束后取7 μL于2.0%的琼脂糖凝胶上电泳,溴乙锭染色,紫外凝胶成像系统照相分析。优化时,设置不同的反应温度,温度梯度为1℃,对滚环扩增反应进行优化。
1.4 超分支滚环扩增检测的特异性
将锁式探针与番茄细菌性叶斑病菌以及参照菌株的DNA模板分别进行环化连接,按照上述步骤进行连接、消化、超分支滚环扩增,测试并比较超分支滚环扩增的特异性。
1.5 超分支滚环扩增检测的灵敏度测定
将提取的Pst的DNA,经核酸蛋白仪测定浓度,再经无菌ddH2O 10倍梯度稀释后作为模板,各取2 μL按上述步骤进行连接、消化、超分支滚环扩增,以确定锁式探针超分支滚环扩增的最低检测DNA浓度。
1.6 基于锁式探针的PCR扩增
将连接、消化后的环化锁式探针作为模板,用扩增引物进行PCR扩增,反应体系20μL:2×PCR buffer 10μL,10μmol/L的 padR 0.5 μL,10μmol/L的 padF 0.5 μL,锁式探针环化连接、消化反应后产物2 μL,无菌ddH2O 补足至20μL。反应条件:94 ℃ 4 min;94 ℃ 30s,62 ℃ 30s,72 ℃ 30s,35个循环;72 ℃ 8 min。
2 结果和分析
2.1 番茄细菌性叶斑病菌超分支滚环扩增检测技术建立
整个超分支滚环扩增反应包括线性锁式探针的连接环化,核酸外切酶消化和滚环扩增三部分。连接反应试验中优化了线性探针的终浓度;消化反应试验中分别优化了核酸外切酶I、Ⅲ的量和作用时间;滚环扩增反应中优化了反应温度。研究结果表明,线性探针终浓度为8 pmol/L时,连接效果最好(图 1),超分支滚环扩增的最适反应温度为62 ℃(图 2)。采用热循环连接法对终浓度为8 pmol/L的锁式探针进行连接,然后用10U的核酸外切酶Ⅰ和6U核酸外切酶Ⅲ消化2.5 h,可以提高连接效率,有效消除未环化的线性探针的影响。用优化后的体系进行连接、消化和滚环扩增,Pst的DNA作为靶标序列,无菌ddH2O作空白对照,获得Pst特异的HRCA产物,然后进行琼脂糖凝胶电泳,电泳结果出现了典型的阶梯状的条带分布(图3),条带的大小为108 bp+108×N(N≥0) bp。

图1 锁式探针终浓度的优化Fig.1 Optimization of the final concentration for padlock probe

图2 超分支滚环扩增的温度优化Fig.2 Optimization of the reaction temperature for HRCA

图3 番茄细菌性叶斑病菌超分支滚环扩增电泳分析Fig.3 Electrophoretic analysis of HRCA for Pst
2.2 超分支滚环扩增检测的特异性
超分支滚环扩增检测的特异性试验结果表明:在供试的10种病原菌中,只有番茄细菌性叶斑病菌能被特异性检出,其余9种病原菌均不能检出(图4a),试验使用Zaccardelli等[4]报道的PCR方法对供试的10种病原菌进行PCR扩增,只有Pst可以扩增出532 bp目标片段(图4b),与超分支滚环扩增结果相同,由此表明,建立的番茄细菌性叶斑病菌超分支滚环扩增技术对该病菌有特异性,可以用于快速检测。

图4 番茄细菌性叶斑病菌超分支滚环扩增的特异性Fig.4 The specificity of HRCA for Pst
2.3 超分支滚环扩增检测的灵敏度
超分支滚环扩增检测的灵敏度试验结果表明,HRCA检测灵敏度高,最低DNA检测浓度为500fg/μL,Pst的DNA浓度为500fg/μL时,可以检测到,但电泳条带较微弱,当浓度小于500fg/μL时,则检测不到(图 5)。用Zaccardelli 等[4]报道的PCR方法检测的灵敏度为5 pg/μL(图 5),可见HCRA方法检测灵敏度更高,比常规PCR方法的灵敏度高出一个数量级。

图5 番茄细菌性叶斑病菌超分支滚环扩增检测的灵敏度Fig.5 The sensitivity of HRCA for Pst
2.4 基于锁式探针的PCR扩增
锁式探针经过环化后进行PCR扩增,经凝胶电泳成像,得到预期的约108 bp的目标片段(图6),而供试的其他病原菌未扩增出目标片段,其特异性和HRCA的特异性是一致的(图 4,图6)。

图6 番茄细菌性叶斑病菌基于锁式探针的PCR扩增Fig.6 The padlock-dependent PCR for Pst
3 讨论
番茄细菌性叶斑病是一种危害严重的病害,其检测方法有很多种,但都有一定的缺陷,现行的血清学检测和胶体金免疫检测等常规诊断方法检测灵敏度低而容易造成假阴性,定性PCR或定量PCR检测技术均是对检测靶标DNA的扩增,增加了交叉污染的风险[19]。基于锁式探针的超分支滚环扩增(HRCA)技术仅特异性地扩增环化锁式探针,不扩增模板DNA,避免PCR扩增检测靶标DNA的富集影响,最大限度地减少交叉污染风险,具有特异性强、灵敏度高、简单易操作和高通量的特点。
锁式探针具有很高的特异性及单碱基错配识别能力,当错配位置在锁式探针的3′末端时,很容易识别而不会连接,滚环扩增后也不会产生扩增信号;但是当错配位置在5′端或3′端中部时,将不容易识别,滚环扩增后能在凝胶电泳上检测到电泳条带[6,18]。所以在设计锁式探针时,将锁式探针的3′端选择在与Pst碱基序列高度一致、而与近似种差异较大的碱基序列位置上,使得探针3′端与Pst靶标DNA完全互补,而与非靶标DNA 3′端存在错配,以保证设计的探针特异性。
线性锁式探针对滚环扩增有一定的影响,当HRCA体系中同时存在线性探针和靶标DNA时,会对滚环扩增产生背景信号,形成一定干扰,因此,把线性探针消化掉非常重要[20-21]。报道只需要用核酸外切酶Ⅰ,就能特异性地降解线性 ssDNA。但Szemes[6]经过试验证实,同时加入核酸外切酶Ⅰ与核酸外切酶Ⅲ进行消化效果更好。在本研究中,采用了核酸外切酶Ⅰ与核酸外切酶Ⅲ消化,结果与Szemes的一致。
在本研究中,进行了基于锁式探针的常规PCR试验,得到预期的108 bp的目标片段。用锁式探针常规PCR的方法,避免了锁式探针滚环扩增时未环化的线性锁式探针的非特异性影响。锁式探针最大优势就是特异性和高通量检测多种病原物,如果在同一反应体系中用锁式探针常规PCR的方法扩增番茄上的多种病原细菌,并结合反向斑点杂交技术,就可以实现番茄上多种病原细菌高通量检测,这是我们下一步的研究目标。
[1] Schneider R W,Grogan R G.Bacterial speck of tomato: sources of inoculum and establishment of a resident population[J].Phytopathology,1977,67:388-394.
[2] Wilson M,Campbell P J,Jones J B,et al.Biological control of bacterial speck of tomato under field conditions at several locations in North America[J].Phytopathology,2002,92:1284-1292.
[3] Cuppels D A,Moore R A,Morris V L.Construction and use of a nonradioactive DNA hybridization probe for detection ofPseudomonassyringaepv.tomatoon tomato plants[J].Applied and Environmental Microbiology,1990,56: 1743-1749.
[4] Zaccardelli M,Spasiano A,Bazzi C,et al.Identification and in planta detection ofPseudomonassyringaepv.tomatousing PCR amplification ofhrpZ(Pst)[J].European Journal of Plant Pathology,2005,111:85-90.
[5] Nilsson M,Malmgren H,Samiotaki M,et al.Padlock probes:Circularizingoligonucleotidesfor localized DNA detection[J].Science,1994,65(5181):2085-2088.
[6] Szemes M,Bonants P,Weerdt M D,et al.Diagnostic application of padlock probes-multiplex detection of plant pathogens using universal microarrays[J].Nucleic Acids Research,2005,33(8):e70.
[7] Lizardi P M,Huang X H,Zhu Z R,et al.Mutation detection and single-molecule counting using isothermal rolling-circle amplification[J].Nature Genetics,1998,19:225-232.
[8] 刘倩,李蓓.基于锁式探针的滚环扩增技术在植物病原检测中的应用[J].植物检疫,2009,23(3):38-41.
[9] 赵春燕,易世红,李凡.网状分枝扩增技术快速检测大肠埃希菌O 157:H7及其他产志贺样毒素大肠埃希菌[J].吉林大学学报(医学版),2006,32(1):18-22.
[10]Schopf E,Chen Yong.Attomole DNA detection assay via rolling circle amplification and single molecule detection[J].Analytical Biochemistry,2010,397:115-117.
[11]Su Qiang,Xing Da,Zhou Xiaoming.Magnetic beads based rolling circle amplification-electroch-emiluminescence assay for highly sensitive detection of point mutation[J].Biosensors and Bioelectronics,2010,25:1615-1621.
[12]Li Jishan,Deng Ting,Chu Xia,et a1.Rolling circle amplification combined with gold nanoparticle aggregates for highly sensitive identification of single-nucleotide polymorphisms[J].Analytical Chemistry,2010,82:2811-2816.
[13]Cheng Wei,Yan Feng,Ding Lin,et al.Cascade signal amplification strategy for subattomolar protein detection by rolling circle amplification and quantum dots tagging[J].Analytical Chemistry,2010,82:3337-3342.
[14]Yan Juan,Song Shiping,Li Bing,et al.An on-nanoparticle rolling circle amplification platform for ultrasensitive protein detection in biological fluids[J].Small,2010,22(6):2520-2525.
[15]郭艳玲,刘洋,姜广路,等.超分支滚环扩增技术快速检测结核分枝杆菌感染的临床应用价值[J].结核病与胸部肿瘤,2012(2):89-91.
[16]黄冠军,殷幼平,张仑,等.柑桔溃疡病菌滚环扩增检测体系的建立[J].微生物学报,2008(3):375-379.
[17] Fanelli V,Cariddi C,Finetti-Sialer M.Selective detection ofPseudomonassyringaepv.tomatousing dot blot hybridization and real-time PCR[J].Plant Pathology,2007,56:683-691.
[18]Pickering J,Bamford A,Godbole V,et al.Integration of DNA ligation and rolling circle amplification for the homogeneous,end-point detection of single nucleotide polymorphisms[J].Nucleic Acid Research,2002,30(12):e60.
[19]Wang B,Potter S J,Lin Y G,et a1.Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification[J].Clinical Microbiology,2005,43(5):2339-2344.
[20]Zhang D Y,Zhang W,Li X,et al.Detection of rare DNA targets by isothermal ramification amplification[J].Gene,2001,274(1-2):209-216.
[21]Hafner G J,Yang I C,Wolter L C,et al.Isothermal amplification and multimerization of DNA byBstDNA polymerase[J].Biotechniques,2001,30(4):852-867.
RapiddetectionofPseudomonassyringaepv.tomatousinghyperbranchedrollingcircleamplification
Wang Nianwu1,2,Wang Ting1,Shen Jianguo2,Hu Fangping1
(1.CollegeofPlantProtection,FujianAgriculturalandForestryUniversity,Fuzhou350002,China;2.FujianBureauofExit-EntryInspectionandQuarantine,Fuzhou350001,China)
A padlock probe was designed based on the sequence of the unique hypothetic protein gene in the complete genome ofPseudomonassyringaepv.tomato(Pst),according to the design principle of padlock probe and primers.Detection system of hyperbranched rolling circle amplification (HRCA) was established and optimized.The results showed that Pst could be specifically detected by this method,while other reference plant pathogens could not be detected.The detection sensitivity of HRCA was about 500fg/μL of DNA concentration,higher than that of the conventional PCR method.
Pseudomonassyringaepv.tomato; hyperbranched rolling circle amplification; padlock probe
2013-04-23
:2013-07-14
国家质检总局项目(20011K158);国家自然科学基金项目(30671463);福建省发改委项目(KY0030060)
S 436.412
:ADOI:10.3969/j.issn.0529-1542.2014.02.017
* 通信作者 E-mail: huf@fafu.edu.cn

