水葫芦胞质型谷氨酰胺合成酶EcGS1全长cDNA的克隆及序列分析
蒋丽花 傅明辉 李园枚 严国花 郑李军 陈肖丽 彭进平
(广东工业大学轻工化工学院,广州 510006)
谷氨酰胺合成酶(Glutamine synthetase,GS)是植物将无机氮转化为有机氮的第一个酶,与谷氨酸合成酶(Glutamate synthetase,GOGAT)联合作用催化氨的同化,使其转变成谷氨酰胺(Gln)和谷氨酸(Glu),Gln和Glu是高等植物体内含氮有机物的生物合成最主要的供体[1]。因此GS是植物氮代谢中的关键酶,与植物对氮素的利用[2,3],植物的产量和品质[4]、植物的抗逆性密切相关[5-7]。高等植物中存在多种GS同工酶形式。根据亚细胞定位,植物的GS分为两大类,一类是胞质型或胞液型GS(GS1),一类是质体型或叶绿体型GS(GS2)。GS1为多基因编码,目前发现在水稻中有3个[8],拟南芥有5个[9],玉米有5个[10],土豆有2个[11],杨树有3个[12]。GS2一般为单基因编码,仅在苜蓿种子的发育中发现了第二个GS2基因[13]。GS2的亚基的分子量为44-45 kD左右,而GS1的亚基的分子量小于38-40 kD。GS2亚基多肽的N端存在一个由50个左右的氨基酸组成的信号肽序列,起到指导其向质体定位的作用,C端存在一个GS2亚基特有的由16个氨基酸组成的保守区。GS1和GS2在植物体内起着不同的作用。GS1主要同化内源性蛋白质降解和氨基酸分解代谢产生的氨,并可能在氮素的转运中起作用,主要参与种子萌发时储存氮源的转运及叶片衰老时氮源的转移及再利用。GS2则同化经硝酸盐还原酶和亚硝酸盐还原酶催化产生的氨,以及同化光呼吸作用产生的氨[14]。
水葫芦(Eichhomia rassipes)学名凤眼莲,属雨久花科凤眼莲属植物,是亚热带和温带淡水水面广泛生长的一种水草。水葫芦对水体中的氮素利用效率非常高,在富营养化水体中泛滥生长,破坏水体生态平衡,但适量的水葫芦也可净化水质,用于富营养化湖泊[15,16]及河道[17]、养殖废水[18,19]、工业废水[20,21]等方面的处理。水葫芦为何能够高效利用富营养化水体中的氮素,目前所有的研究仅在生理生化的水平上[22,23],还未深入到分子水平。
本研究以水葫芦谷氨酰胺合成酶cDNA为模板,克隆水葫芦胞质型谷氨酰胺合成酶(EcGS1)基因,推测其氨基酸序列,分析其中的保守片段,预测该序列编码蛋白的亚细胞定位,比较该序列与其他植物中相关序列之间的相似性,并构建分子进化树,旨在为进一步深入研究EcGS1基因的功能,揭示水葫芦高效利用氮素的分子机理奠定基础。
1 材料与方法
1.1 材料
1.1.1 试验材料 水葫芦采自广州市安检水库。
1.1.2 主要试剂 RNAiso plus 总RNA提取试剂盒、Prime ScriptTMRT-PCR Kit、Agarose Gel DNA Purification Kit 试剂、pMD19-T vector、RACE 试剂盒均购于TaKaRa;E. coliDH5α、DNA Marker、Taq酶、dNTP均购自鼎国昌盛生物技术(北京)有限公司。其他生化试剂和常规试剂均为超纯或分析纯。
1.2 方法
1.2.1 引物设计及合成 根据已公布的其他植物GS1氨基酸保守序列,用在线软件ICODEHOP设计简并引物,结合5'-Full RACE Kit,3'-Full RACE Core Set Ver.2.0试剂盒中的克隆方法设计进行RACE PCR,所用引物由Invitrogen广州公司合成,序列信息详见表1。
1.2.2 总RNA的提取及cDNA第一链的合成 取适量幼嫩的水葫芦根,洗净,用液氮充分研磨粉末,总RNA的提取方法按照RNAiso plus说明书进行。以提取的总RNA为模板,用Prime ScriptTMRTPCR Kit试剂盒方法进行逆转录合成cDNA,产物置于-20℃保存。
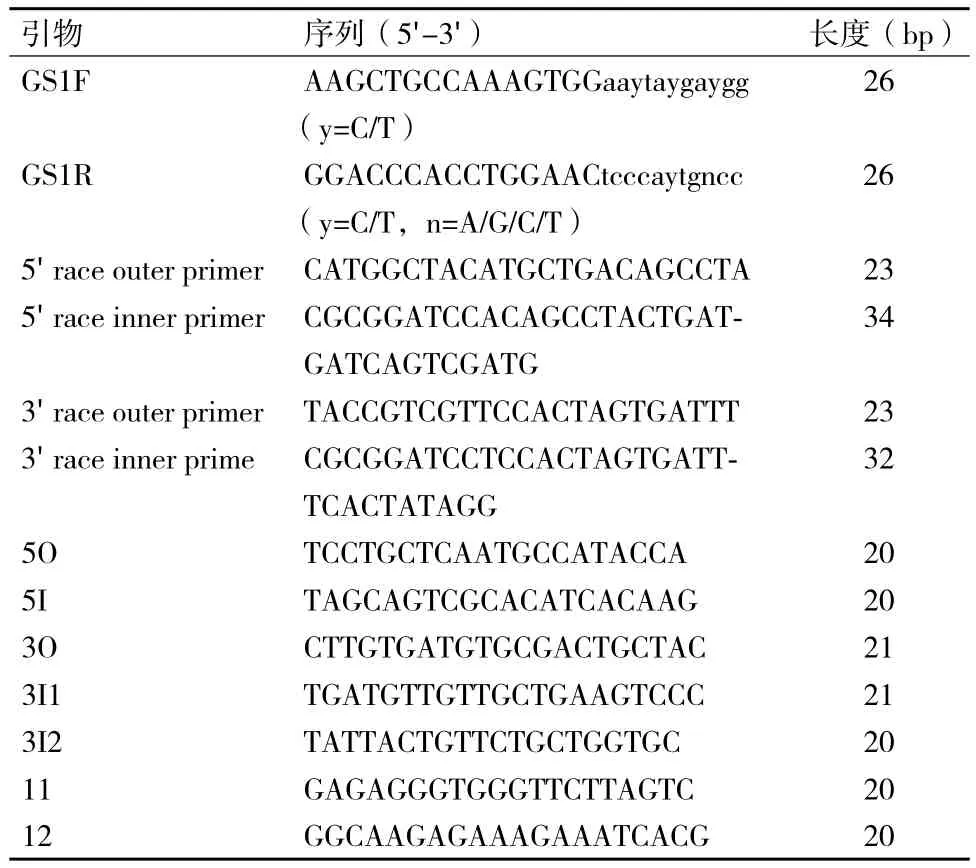
表 1 引物序列
1.2.3 EcGS1基因中间片段的扩增及克隆 以cDNA第一链为模板,采用简并引物GS1F/GS1R,进行PCR扩增。反应体系为:ddH2O 17 μL,Taq酶0.5 μL,buffer 2.5 μL,dNTP 1 μL,模板 2 μL,上下游引物各1 μL。反应程序为:94℃预变性 5 min;94℃30 s,53℃ 30 s,72℃ 1 min,30个循环;72℃延伸10 min。PCR产物电泳鉴定,回收,转入T载体,用菌落PCR筛选阳性克隆测序。
1.2.4 5' RACE扩增及克隆 引物采用Primer 5软件设计,根据EcGS1基因中间片段设计其5'端扩增的特异引物,联合5' race outer primer/5' race inner primer,进行5'端的巢式PCR,反应体系同1.2.3,反应程序为:第1轮(5O联合5' race outer primer),94℃预变性 5min;94℃ 30 s,40℃ 30 s(每循环升1℃),72℃ 1 min,20个循环;72℃延伸10 min。第2轮(5I联合5' race inner primer),94℃ 5 min;94℃ 30 s,40℃/44℃/48℃/52℃/56℃ 30 s,72℃ 1 min,30个循环;72℃延伸10 min。PCR产物电泳鉴定,回收,转入T载体,用菌落PCR筛选阳性克隆测序。
1.2.5 3' RACE扩增及克隆 根据根据EcGS1基因中间片段和5'端片段的序列设计其3'端的特异引物(3O/3I1/3I2),联合3' race outer primer/3' race inner primer 对EcGS1基因3'端进行巢式PCR,反应体系同1.2.3,第1轮(3O联合3' race outer primer),扩增程序同5' RACE的第1轮反应,第2轮(3I1联合3' race inner primer)和第3轮(3I2联合3' race inner primer)扩增程序同5' RACE的第2轮反应,PCR产物的电泳鉴定,回收,转入T载体,用菌落PCR筛选阳性克隆测序。
1.2.6 EcGS1基因全长获得 根据EcGS1基因中间片段,5'端及3'端测序结果,拼接获得全基因序列,对序列进行开放阅读框的分析,设计引物(11/12),进行PCR。扩增程序:94℃预变性5 min;94℃ 30 s,55℃ 30 s,72℃ 1 min,30个循环;72℃延伸10 min。PCR产物电泳鉴定,回收,转入T载体,用菌落PCR筛选阳性克隆测序。
1.2.7 EcGS1基因序列分析 用ProtParam工具分析EcGS1蛋白的理化性质(http://web.expasy.org/protparam/)。用PROSITE和SMART数据库对EcGS1蛋白保守结构域进行分析(http://www.expasy.Org/prosite,http://smart.Embl-heidelberg.De/)。利用TargetP程序预测EcGS1蛋白的细胞亚定位(http://www.cbs.dtu.dk/services/TargetP/)。利用NCBI中BLASTX对NCBI蛋白质全库进行相似性搜索(http://blast.ncbi.nlm.nih.gov/Blast.cgi)。利用ClustalW2进行多序列比对,用Mega4.1采用邻接法构建分子进化树。
2 结果
2.1 水葫芦总RNA的提取
用紫外分光光度仪测定提取的水葫芦总RNA,其OD260/280=2.0,OD260/230=2.5,说明所提取的总蛋白质和杂质污染较少,纯度较高。总RNA经由1%琼脂糖凝胶电泳检测(图1)。
2.2 EcGS1基因中间片段克隆及测序
PCR产物经电泳检测到一条单一条带(图 2),位于500 bp左右,进行克隆及测序,得到467 bp片段,通过BLAST分析,证实为GS相似序列。
2.3 5' RACE产物克隆及测序

图 1 水葫芦总RNA电泳图

图 2 EcGS1基因中间片段
经电泳检测5' RACE第2轮扩增产物(图 3),得到一条约400 bp片段,克隆测序后发现,去5'race inner 引物后367 bp,其84-86位为起始密码子ATG,与EcGS1基因中间片段有140 bp重叠序列。

图 3 EcGS1基因5'RACE扩增产物
2.4 3' RACE产物克隆及测序
经电泳检测3' RACE第3轮扩增产物(图 4),得到一条约900 bp条带,进行克隆测序,去3' race inner primer 后序列为883 bp,内含mRNA 3'末端polyA,与EcGS1基因中间片段有143 bp重叠序列。
2.5 EcGS1基因cDNA全长获得
将上述3条序列拼接,得到EcGS1基因的cDNA序列,共1 434 bp,提交NCBI,获得序列登录号为KF683089,采用ORF分析序列,表明其中含有1个1 071 bp完整开放阅读框序列,编码356个氨基酸残基,ATG为起始密码子,TGA为终止密码子。根据EcGS1基因的cDNA序列设计引物,用引物11/12从水葫芦基因组DNA中扩增到约1 200 bp DNA片段,将其克隆测序后得到1 183 bp片段(图5)。该测序结果与拼接结果及开放阅读框分析结果完全一致,证实了试验设计的正确性。

图 4 EcGS1基因3'RACE扩增产物

图 5 EcGS1基因全长片段
2.6 EcGS1基因编码蛋白的氨基酸序列、保守结构域及亚细胞定位
用ProtParam分析EcGS1基因所编码蛋白的氨基酸序列(图6)显示,该蛋白分子量为39.3 kD,等电点为5.52。用PROSITE数据库分析该蛋白237-253氨基酸残基为ATP结合区域(putative ATP-binding region){K-P-[LIVMFYA] -x(3,5)-[NPAT] -[GA] -[GSTAN] -[GA] -x-H-x(3)-S},11-14、185-188、251-254氨基酸残基为N-糖基化序列(N-glycosylations)N-{P}-[ST] -{P}。用SMART分析,显示EcGS1蛋白包含了一个GS beta-Grasp domain(17-97 AA)和一个GS catalytic domain(103-351 AA)。利用TargetP程序预测EcGS1蛋白序列不存在信号肽,为胞质蛋白。
2.7 EcGS1基因编码的氨基酸序列与其他植物GS1相似性比对
运用NCBI的BLASTX对蛋白质序列全库进行相似性搜索,选择其中的13条序列相似性较高的不同植物的GS1氨基酸序列,利用ClustalW2,进行多序列比对,结果(表2)显示,与其他物种的GS1氨基酸序列相似性在82%以上,其中与橡胶树的相似性最高。说明了不同植物种间GS1在氨基酸序列上保守性较高。根据水葫芦及其他植物的GS1氨基酸序列,用Mega4.1软件构建GS1氨基酸序列的分子进化树(图7)。
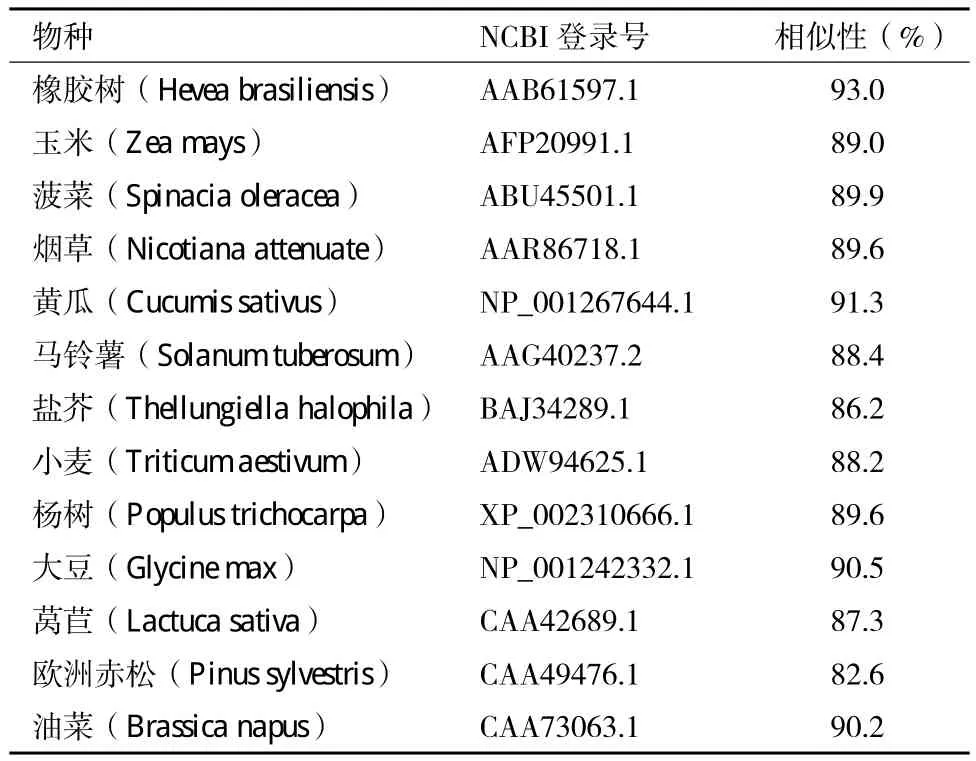
表 2 水葫芦与其他植物的GS1氨基酸序列相似性比较
3 讨论
GS1蛋白和GS2蛋白都在细胞质中合成,GS1蛋白为胞质型蛋白,GS2蛋白像多数质体型的蛋白一样,最初在细胞质中合成前体多肽,被基质内的有关酶切去N端的一段肽段后,被运输到叶绿体内[24]。本试验克隆了EcGS1基因cDNA序列,该序列编码356个氨基酸的多肽链,通过BLAST相似性分析,证实EcGS1为GS1蛋白,通过TargetP程序预测,该序列没有信号肽,为胞质型蛋白。EcGS1分子量大小为39.3 kD,与其他GS1分子量(38-40 kD)大小一致,而与GS2分子量(44-45 kD)明显不同。

图 6 EcGS1基因核苷酸序列和推导的氨基酸序列
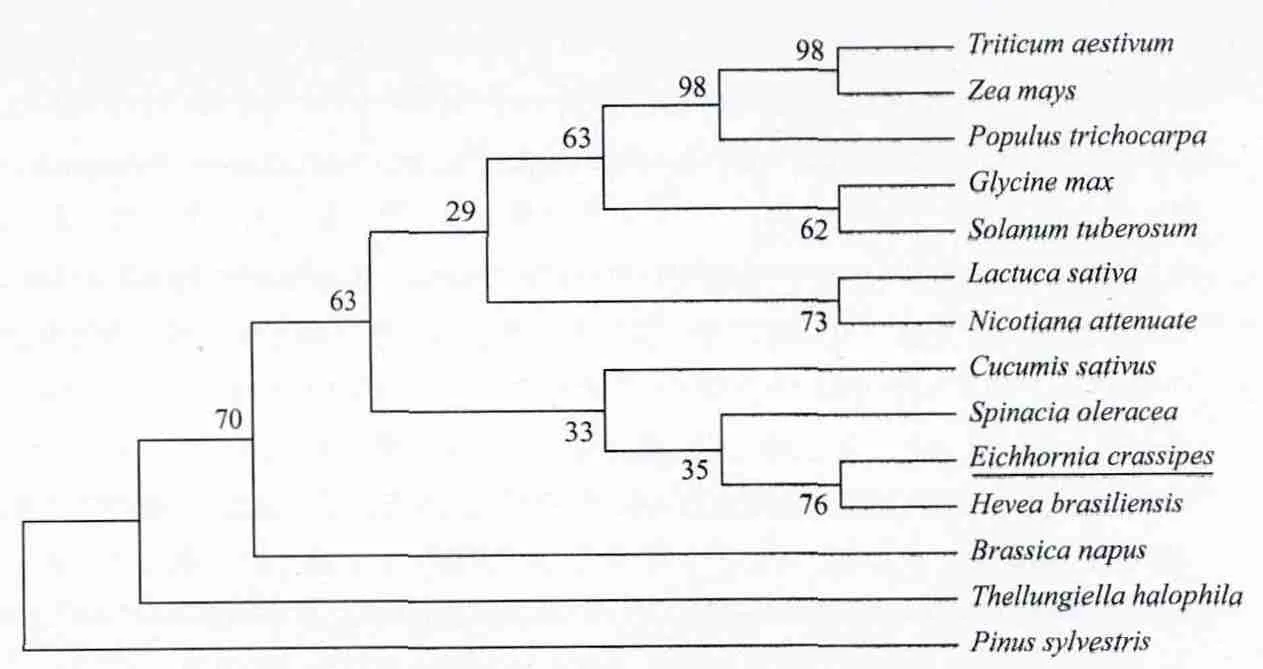
图 7 水葫芦与其他物种GS1氨基酸序列的系统进化树
EcGS1与其他13种不同类植物的GS1氨基酸序列相似性比较结果显示,EcGS1氨基酸序列与橡胶树的相似性最高,为93.0%,EcGS1氨基酸序列与欧洲赤松的相似性较高,为82.6%,说明了不同植物间的GS1在氨基酸序列上保守性较高。GS是植物利用环境中氮素的关键酶,在植物生理过程中占有重要地位,因此,在进化过程中承受了较大的选择压力,所以序列的保守性较高。通过EcGS1基因编码的氨基酸序列保守结构域的分析,发现该酶含有ATP结合序列。Llorca等[25]采用电镜和图像技术结合生化数据也证实了植物谷氨酰胺合成酶分子中存在ATP结合位点。这点是与其功能相一致的,谷氨酰胺合成酶在催化谷氨酸与NH4+合成谷氨酰胺时,需要消耗ATP[14]。生物信息学分析发现该酶还含有多个保守的N-糖基化序列,在已报道的水稻[26]、油菜[27]、茶树[28]等植物的GS1蛋白序列也存在这些保守序列。
从分子进化树图看,植物GS1的分子进化树与物种进化树并不一致。王小纯等[29]报道,GS2分子进化树与物种进化树一致,而GS1的分子进化树与物种进化树并不一致,这点与本试验结果相符合,王江和张景六[30]的分析亦得出同样结论。一个可能的原因是GS1在进化中的分歧可能早于单子叶和双子叶植物的分歧,但这点仍需要进一步数据证实。目前,与水葫芦在进化上亲缘关系较近的物种的GS1基因序列被测定的并不多,随着今后更多的GS1基因序列被测定,GS1的分子进化历程将会被更好地揭示。
有关水葫芦分子生物学的研究有助于揭示水葫芦高效利用富营养化水体中氮素的分子机理,为水葫芦的进一步开发和利用提供理论基础,能够从分子水平上揭示水葫芦这种外来入侵植物快速入侵的机理,探讨从营养代谢角度调控其生长的可能性。
4 结论
利用RACE技术克隆得到水葫芦胞质型谷氨酰胺合成酶EcGS1基因cDNA序列,序列登录号为KF683089,序列长1 534 bp,开放阅读框为1 071 bp,编码356个氨基酸的多肽链,分子量为39.3 kD,等电点为5.52。EcGS1编码的蛋白包含了ATP结合区域、N-糖基化位点、GS beta-Grasp功能区,GS催化功能区。EcGS1氨基酸序列与橡胶树的GS1相似性最高,为93.0%。
[1] 韩娜, 葛荣朝, 赵宝存, 等.植物谷氨酰胺合成酶研究进展[J] .河北师范大学学报, 2004, 28(4):407-410.
[2] Sun H, Huang QM, Su J. Highly effective expression of glutamine synthetase genes GS1 and GS2 in transgenic rice plants increases nitrogen-deficiency tolerance [J] . Journal of Plant Physiology and Molecular Biology, 2005, 31(5):492-498.
[3] Man HM, Boriel R, El-Khatib R, Kirby EG. Characterization of transgenic poplar with ectopic expression of pine cytosolic glutamine synthetase under conditions of varying nitrogen availability [J] .New Phytologist, 2005, 167(1):31-39.
[4] Fuentes SI, Allen DJ, Ortiz-Lopez A, et al. Overexpression of cytosolic glutamine synthetase increases photosynthesis and growth at low nitrogen concentrations [J] . Exp Bot, 2001, 52:1071-1081.
[5] Rana NK, Mohanpuria P, Yadav SK. Expression of tea cytosolic glutamine synthetase is tissue-specific and induced by cadmium and salt stress [J] . Biologia Plantarum, 2008, 52(2):361-364.
[6] Lee HJ, Abdula SE, Jang DW, et al. Overexpression of the glutamine synthetase gene modulates oxidative stress response in rice after exposure to cadmium stress [J] . Plant Cell Rep, 2013, 32:1521-1529.
[7] Pascual MB, Jing ZP, Kirby EG, et al. Response of transgenic poplar overexpressing cytosolic glutamine synthetase to phosphinothricin[J] . Phytochemistry, 2008, 69:382-389.
[8] Tabuchi M, Sugiyama K, Ishiyama K, et al. Severe reduction in growth rate and grain filling of rice mutants lacking OsGS1;1, a cytosolic glutamine synthetase1;1 [J] . The Plant Jounal, 2005,42:641-651.
[9] Ishiyama K, Inoue E, Tkahashi AW, et al. Kinetic properties and ammonium-dependent regulation of cytosolic isoenzymes of glutamine synthetase inArabidopsis[J] . Biol Chem, 2004, 279(16):16598-16605.
[10] Martin A, Lee J, Kichey T, et al. Two cytosolic glutamine synthetase isoforms of maize are specifically involved in the control of grain production [J] . The Plant Cell, 2006, 18:3252-3274.
[11] Teixeira J, Pereira S, Cánovas F, et al. Glutamine synthetase of potato(Solanum tuberosumL. cv.Désirée)plants:cell- and organ-specific expression and differential developmental regulation reveal specific roles in nitrogen assimilation and mobilization [J] .Experimental Botany, 2005, 56(412):663-671.
[12] Castro-Rodríguez V, García-Gutiérrez A, Canales J, et al. The glutamine synthetase gene family inPopulus[J] . BMC Plant Biology, 2011, 11:119.
[13] Seabra AR, Vieira CP, Cullimore JV, et al.Medicago truncatulacontains a second gene encoding a plastid located glutamine synthetase exclusively expressed in developing seeds [J] . BMC Plant Biology, 2010, 10:183-198.
[14] 陈胜勇, 李观康, 汪云, 等.谷氨酰胺合成酶的研究进展[J] .中国农学通报, 2010, 26(22):45-49.
[15] 濮培民.改善太湖马山水厂水源区水质的物理-生态工程实验研究[J] .湖泊科学, 1993, 5(2):171-180.
[16] 窦鸿身, 蹼培民, 张圣照, 等.太湖开阔水域凤眼莲的放养实验[J] . 植物资源与环境, 1995, 4(1):54-60.
[17] 孙文浩, 俞子文, 余叔文.城市富营养化水域的生物治理和风眼莲抑制藻类生长的机理[J] .环境科学学报, 1989, 9(2):187-195.
[18] 蒋艾青.凤眼莲对城郊污水鱼塘的净化试验[J] .淡水渔业,2003, 33(5):43-44.
[19] 袁桂良, 刘鹰.凤眼莲对集约化甲鱼养殖污水的静态净化研究[J] .农业环境保护, 2001, 20(5):322-325.
[20] Casabianca LM. Large production ofEichhornia crassipesof paper industry effluent [J] . Bioresoure Technol, 1995, 54:35-38.
[21] 袁蓉, 刘建武, 成旦红, 等.凤眼莲对多环芳烃(萘)有机废水的净化[J] .上海大学学报:自然科学版, 2004, 10(3):272-276.
[22] 贺丽虹, 沈颂东.水葫芦对水体中氮磷的清除作用[J] .淡水渔业, 2005, 35(3):7-9.
[23] 宋伟, 韩士群, 刘海琴, 等.水葫芦去除污水中氮磷的效果[J] .安徽农业科学, 2008, 36(25):1107-11079.
[24] Paula MM, Lígia ML, Isabel MS, et al. Expression of the plastidlocated glutamine synthetase ofMedicago truncatulaaccumulation of the precursor in root nodules reveals anin vivocontrol at the level of protein import into plastids [J] . Plant Physiol, 2003, 132(1):390-399.
[25] Llorca O, Betti M, González JM, et al. The three-dimensional structure of an eukaryotic glutamine synthetase:Functional implications of its oligomeric structure [J] . J Struct Biol, 2006,156:469-479.
[26] Sakamoto A, Ogawa M, Masumura T, et al. Three cDNA sequences coding for glutamine synthetase polypeptides inOryza sativaL. [J].Plant Molecular Biology, 1989, 13:611-614.
[27] Ochs G, Schock G, Trischler M, et al. Complexity and expression of the glutamine synthetase multigene family in the amphidiploid cropBrassica napus[J] . Plant Molecular Biology, 1999, 39:395-405.
[28] Rana NK, Mohanpuria P, Yadav SK. Cloning and characterization of a cytosolic glutamine synthetase fromCamellia sinensis(L.)O. Kuntzethat is upregulated by ABA, SA, and H2O2[J] . Mol Biotechnol, 2008, 39:49-56.
[29] 王小纯, 张同勋, 李高飞, 等.小麦谷氨酰胺合成酶基因克隆与其表达特性分析[J] .河南农业大学学报, 2012, 46(5):487-492.
[30] 王江, 张景六.稻属谷氨酰胺合成酶家族的系统分析[J] .植物生理学通讯, 2007, 43(2):277-282.

