成像质谱在微生物代谢产物研究中的应用
徐玉泉
中国农业科学院生物技术研究所,北京100081
微生物合成的代谢产物结构和功能多样,广泛应用于医药和农业生产领域。另一方面,这些代谢产物对于微生物自身的生长、发育以及对环境中其他相邻微生物也会产生重要的影响。在微生物群落中,微生物通过代谢产物的交换来彼此适应和彼此竞争,只有系统化全面地监测微生物产生的各种代谢产物,如聚酮、非核糖体多肽和脂肪酸等,才有可能了解微生物代谢产物的本质[1]。对于微生物代谢产物的研究通常采用活性跟踪的方法,即通过从土壤、植物、海洋或极端环境样品中分离环境微生物,在实验室条件下发酵培养微生物,最后确定从培养物中分离得到的代谢产物的结构和生物活性。该方法和微生物产生这些代谢物的环境割裂开来,无法系统化多角度地了解微生物在菌落水平上产生这些代谢物的全部信息,而且很难针对那些含量少且不稳定的微生物生物活性物质进行研究[2]。事实上,在微生物相互作用中,往往不只是单个代谢物参与化学交流。在微生物的基因组中,含有大量的基因与合成代谢产物相关,这些代谢物分子常常参与微生物之间的代谢交流,进而影响或控制微生物种内和种间代谢物的合成、表型的变化以及发育分化等生物进程[3,4]。可见,一种微生物能够产生多种活性化合物,这些化合物都有可能改变自身和相邻微生物的生理特点。针对复杂的微生物代谢研究,成像质谱技术显示出较强的应用优势。
1 成像质谱的工作流程
基质辅助激光解吸/电离成像质谱 (matrixassisted laser desorption/ionization imaging mass spectrometry,MALDI IMS)可以有效地研究不需要标记的多种生物分子,如多肽、蛋白质、聚合物和脂类在空间上的分布,一次实验就可以检测和可视化观察到多个目标分子[5,6]。其基本原理为:通过控制固定有样品的MALDI金属板在横向和纵向的移动,使MALDI激光将预先设定检测点的样品离子化,通过收集100到1 000个点的质谱数据,绘制出所测样品二维质谱分布图。

图1 成像质谱研究微生物代谢产物的工作流程[1,7]Fig.1 Workflow of microbial metabolites research by MALDI-IMS[1,7].
成像质谱技术起初被应用于医学领域,如病理学和药学研究。近年来逐渐成为研究微生物代谢产 物 的 重 要 工 具[1,7],其 主 要 的 工 作 流 程(见图1,彩图见227页图版)为:微生物在薄层琼脂培养基上培养后(1),将分析区域的微生物连同培养基一起转移到MALDI金属板上,照相后覆盖基质(2,7,8),在37℃脱水干燥,使琼脂培养基和微生物菌落固定在MALDI板上(3)。随后,样品被送入MALDI成像质谱仪中,设定微生物菌落周围需分析的由一系列微米距离激光点组成的光栅网格。通过收集和处理光栅网格中每个激光点的质谱数据,得到每个光栅网格的平均质谱。成像质谱的最后步骤是将任何相同质荷比(m/z)的离子设定为一种颜色来标明其在所检测样品上的位置(4,5,6),和分析区域的数码照片叠加后,产生可视化的生物分子在样品中的空间分布(9)。
2 成像质谱在微生物代谢产物研究中的应用
成像质谱可以直接可视化观察薄层琼脂培养基上微生物代谢产物的时空分布和不同微生物之间代谢产物的对话。具有所需样品少、不需预处理、能够同时观察多种代谢分子等优点,可以用于发现新型微生物天然产物,指导天然产物的分离,可视化研究微生物之间的化学交流,以及阐明微生物耐抗生素新机制等。
2.1 发现和指导重要代谢产物的分离
海绵和蓝藻能够合成多种次生代谢产物,是药用天然产物的重要资源[8,9]。利用成像质谱不仅能够从海洋蓝藻中观察到多种代谢产物的空间分布,而且可以从混合样品中观察到单个海绵丝状体合成的代谢产物,证明该技术可以发现合成目标代谢产物的微生物以及跟踪生物合成的进程[10]。到目前为止,成像质谱技术已经应用于包括嗜热和嗜温菌、蓝藻、海洋和陆地放线菌,以及致病细菌等不同环境的微生物,从中发现了大量已知和未知的天然产物,与分离提取、串联质谱、傅里叶变换离子回旋加速器质谱(FT-ICR-MS)以及MALDI TOF/TOF相结合,部分或完全地确定了这些代谢物的结构,如白僵菌产生的白僵菌素和白僵菌环四肽等代谢物[1];以成像质谱数据为基础,从放线菌中也发现了一些重要的多肽代谢产物,与基因组挖掘技术相结合,阐明了包括玫瑰孢链霉菌中脂糖肽类抗生素arylomycin在内的多种多肽代谢物的生物合成途径[11,12]。以上研究表明成像质谱已成为发现微生物重要活性多肽代谢物的新平台。
2.2 可视化研究微生物之间的化学对话
包括信号分子和营养物质在内的代谢物交换在所有生物系统中是一种普遍的现象。细菌通过产生各种信号分子来控制微生物种内和种间代谢物的合成、表型的变化以及发育分化等生物进程。枯草芽胞杆菌具有复杂的生命周期,使其能够在诸如土壤、伤口以及肠道各种环境中生存[13]。和这些能力相符的是,枯草芽胞杆菌10%的基因组与合成特异的分子有关[3],这些分子常常参与种内和种间的代谢交换。枯草芽胞杆菌存在细胞自食现象,即在营养缺乏情况下,有的细胞能够产生微量信号分子 SKF(sporulation killing factor)和 SDP(sporulation delaying protein),可以通过杀死不产生SKF和SDP的细胞,从死亡细胞中获取产生孢子的营养和能量来保持其物种生存[14]。由于这两个信号分子在细胞中的含量很低,传统的技术手段难以分离鉴定,无法进一步探究这两种信号分子的结构及其作用机制。借助成像质谱技术,观察到SKF和SDP信号分子并确定了它们的结构(见图2,彩图见228页图版),同时证明了在固体培养基上,SKF和SDP都能抑制枯草芽胞杆菌的生长;而在液体培养基中,只有SDP具有抑制作用[15]。
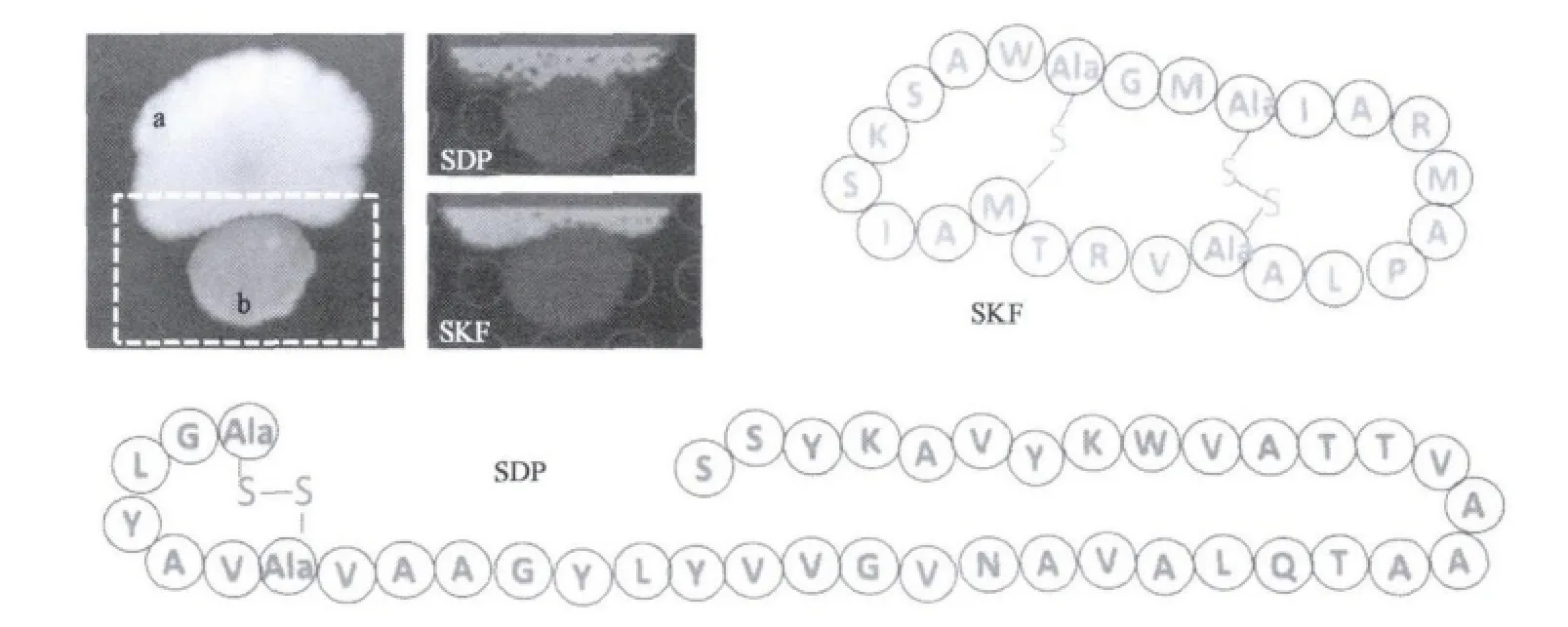
图2 枯草芽胞杆菌SKF和SDP在菌落上的分布及结构[15]Fig.2 The distribution and the structures of SKF and SDP[15].
在生态环境中,微生物往往感应自身或周围的化学物质,通过和这些化学物质的相互作用,进而影响自身表现型,微生物表现型的改变反过来又会合成新的代谢产物影响周围的环境和其他的微生物[2,16]。成像质谱可以原位地研究环境和微生物的化学交流过程,能够可视化地将微生物的表现型改变以及肉眼看不见的化学型变化与代谢物的相互作用连接在一起,动态地研究群体中微生物之间的相互作用。当枯草芽胞杆菌和天蓝色链霉菌相邻培养时,利用成像质谱观察到枯草芽胞杆菌产生的脂多肽surfactin能够抑制天蓝色链霉菌疏水多肽SapB的合成,进而导致天蓝色链霉菌不能产生气生菌丝和孢子,同时还发现surfactin对天蓝色链霉菌抗生素CDA的合成具有抑制作用[17];在海洋沉积样品的微生物群体中,面对枯草芽胞杆菌的威胁,原小单胞菌首先产生小分子多肽(m/z 2869)抑制枯草芽孢杆菌,随着枯草芽胞杆菌的靠近,可诱导原小单胞菌产生钳铁化合物promicroferrioxamine,竞争环境中的铁元素,用于自身的生存所需[18]。
2.3 阐明微生物耐抗生素新机制
很多微生物向环境中分泌天然产物以阻止竞争微生物的生长发育,相反,竞争微生物会产生或获得某种抗性来削弱或消除拮抗分子的作用[19]。利用成像质谱,通过跟踪枯草芽胞杆菌和链霉菌Mg1的代谢物交流(见图3,彩图见228页图版),发现链霉菌Mg1能够水解枯草芽胞杆菌产生的环化脂多肽surfactin(1047),使其不能抑制自身气生菌丝的产生。以此为线索,进一步发现,链霉菌Mg1的水解酶SfhA能够将枯草芽孢杆菌产生的环状surfactin水解为线性的surfactin(1065),使其失去抑制链霉菌产生气生菌丝的能力[20]。该结果说明在研究微生物相互作用中,成像质谱可以追踪天然产物修饰的过程。
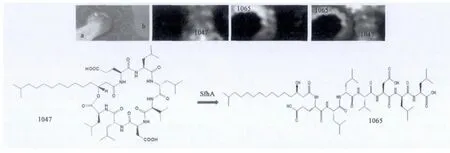
图3 链霉菌Mg1和枯草芽胞杆菌3610的成像质谱[20]Fig.1 MALDI IMS of Bacillus subtilis 3610 and Streptomyces sp.Mg1[20].
3 展望
成像质谱所需样品少,能够可视化的研究代谢产物在微生物菌落和环境中的时间和空间分布。该方法能够和微生物产生这些代谢物的环境联系在一起,可以系统化多角度地地展示微生物在菌落水平上产生这些代谢物的全部信息,发现大量已知和未知的代谢产物[1]。以成像质谱数据为指导,利用基因组挖掘等生物信息学手段,可以阐明其中一些重要代谢物的生物合成途径[11,12]。借助于成像质谱,通过观察微生物相互作用中相邻区域或生长抑制区域的代谢分子,便于发现新的生物活性物质或微生物抗性新机制[15,18,20]。成像质谱不仅用于研究微生物-微生物之间的相互作用,而且也应用于微生物-植物、微生物-昆虫相互作用的研究[21,22]。当然,化学基质的选择、检测代谢物的类型、以及培养基的成分等都会对成像质谱效果产生影响,但是随着成像质谱技术的发展以及和其它检测和成像技术的结合,如解吸电喷雾电离(desorption electrospray ionization,DESI)技术[23]、共聚焦显微技术和荧光原位杂交技术等,成像质谱在微生物代谢产物研究中将会发挥越来越重要的作用。
[1]Gonzalez D J,Xu Y, Yang Y L, et al.. Observing the invisible through imaging mass spectrometry,a window into the metabolic exchange patterns of microbes[J].J.Proteomics,2012,75(16):5069-5076.
[2]Shank E A, KolterR. New developments in microbial interspecies signaling[J].Curr.Opin.Microbiol.,2009 ,12(2):205-214.
[3]Gross H. Strategies to unravelthe function oforphan biosynthesis pathways:recent examples and future prospects[J].Appl.Microbiol.Biotechnol.,2007,75(2):267-277.
[4]Straight P D,Kolter R.Interspecies chemical communication in bacterial development[J].Annu.Rev.Microbiol.,2009,63:99-118.
[5]Seeley E H,Caprioli R M.Molecular imaging of proteins in tissues by mass spectrometry [J].Proc.Natl.Acad.Sci.USA,2008,105(47):18126-18131.
[6]Cornett D S,Reyzer M L,Chaurand P,et al..MALDI imaging mass spectrometry:molecular snapshots of biochemical systems[J].Nat.Methods,2007,4(10):828-833.
[7]Yang J Y,Phelan V V,Simkovsky R,et al..Primer on agarbased microbial imaging mass spectrometry[J].J.Bacteriol.,2012,194(22):6023-6028.
[8]Abdelmohsen U R, Bayer K, HentschelU. Diversity,abundance and natural products of marine sponge-associated actinomycetes[J].Nat.Prod.Rep.,2014,31(3):381-399.
[9]Sakai R,Swanson G T.Recent progress in neuroactive marine natural products[J].Nat.Prod.Rep.,2014,31(2):273-309.
[10]Esquenazi E,Coates C,Simmons L,et al..Visualizing the spatial distribution of secondary metabolites produced by marine cyanobacteria and sponges via MALDI-TOF imaging[J].Mol.Biosyst.,2008,4(6):562-570.
[11]Kersten R D,Yang Y L,Xu Y,et al..A mass spectrometryguided genomemining approach for natural product peptidogenomics[J].Nat.Chem.Biol.,2011,7(11):794-802.
[12]Liu W T,Kersten R D,Yang Y L,et al..Imaging mass spectrometry and genomemining via short sequence tagging identified the anti-infective agent arylomycin in Streptomyces roseosporus[J].J.Am.Chem.Soc.,2011,133(45):18010-18013.
[13]Aguilar C,Vlamakis H,Losick R,et al..Thinking about Bacillus subtilis as a multicellular organism [J].Curr.Opin.Microbiol.,2007,10(6):638-643.
[14]González-Pastor J E,Hobbs E C,Losick R.Cannibalism by sporulating bacteria[J].Science,2003,301(5632):510-513.
[15]Liu W T,Yang Y L,Xu Y,et al..Imaging mass spectrometry of intraspecies metabolic exchange revealed the cannibalistic factors of Bacillus subtilis[J].Proc.Natl.Acad.Sci.USA,2010,107(37):16286-16290.
[16]Pacheco A R,Sperandio V.Inter-kingdom signaling:chemical language between bacteria and host [J]. Curr. Opin.Microbiol.,2009,12(2):192-198.
[17]Yang Y L,Xu Y,Straight P,et al..Translating metabolic exchange with imaging mass spectrometry[J].Nat.Chem.Biol.,2009,5(12):885-887.
[18]Yang Y L,Xu Y,Kersten R D,et al..Connecting chemotypes and phenotypesofcultured marine microbial assemblages by imaging mass spectrometry[J].Angew.Chem.Int.Ed.Engl.,2011,50(26):5839-5842.
[19]Linares J F,Gustafsson I,Baquero F,et al..Antibiotics as intermicrobial signaling agents instead of weapons[J].Proc.Natl.Acad.Sci.USA,2006,103(51):19484-19489.
[20]Hoefler B C,Gorzelnik K V,Yang J Y,et al..Enzymatic resistance to the lipopeptide surfactin as identified through imaging mass spectrometry of bacterial competition [J].Proc.Natl.Acad.Sci.USA,2012,109(32):13082-13087.
[21]Nihorimbere V, CawoyH, SeyerA, et al.. Impactof rhizosphere factors on cyclic lipopeptide signature from the plant beneficial strain Bacillus amyloliquefaciens S499[J].FEMS Microbiol.Ecol.,2012,79(1):176-191.
[22]Kroiss J, Kaltenpoth M, Schneider B, et al.. Symbiotic Streptomycetes provide antibiotic combination prophylaxis for wasp offspring [J].Nat.Chem.Biol.,2010,6(4):261-263.
[23]Watrous J,Roach P,Heath B,et al..Metabolic profiling directly from the Petridish using nanospray desorption electrospray ionization imaging mass spectrometry [J].Anal.Chem.,2013,85(21):10385-10391.

