微生物降解硝基芳香族化合物(NAC)的研究进展
杨瑞红
新疆教育学院,乌鲁木齐830043
硝基芳香族化合物(nitro-aromatic compounds,NAC)是一种重要的有机化工原料,主要应用于农药、医药和染料等领域,使用量巨大,是土壤、河流和大气中的主要污染物之一[1]。这类化合物中,有些在动物皮下、腹腔和肌肉中积累可诱发癌变等;有些可分解产生NOx类的有毒烟雾;有些是氧化和光合磷酸化的强效解偶联剂[2]。据报道,肠道菌群和哺乳动物的器官中包含有非特异性的硝基还原酶,它能够催化NAC的硝基转化为更有害的亚硝基和羟氨基,对哺乳动物来讲属于强致癌物[2]。因此这类物质对健康和环境都带来潜在的危害。硝基芳香族化合物性质稳定,具有持续和多样的化学特性,不易降解,且可通过食物链富集放大来破坏生态系统。传统物理清除法不仅成本高而且有二次污染[3]。在自然界,这种外源物所致的生态压力大部分通过生物降解来解决,微生物代谢可使NAC进入生物地化循环,因此利用分子生物学手段构建质粒来改造微生物可大大促进NAC的微生物降解。
1 微生物降解NAC的过程
硝基芳香族化合物主要来源于化学工业,不是天然的营养来源,属于难于利用的、有害的环境底物。但微生物具有很强的环境适应性,能够在短期内进化出新的代谢途径或者改变已有的代谢途径来代谢这些与天然底物结构类似的人工合成底物。大多数生物自身都有硝基还原酶,能够将NAC转化为相应的胺类,但是在自然条件下,完全分解硝基芳香烃类的酶是非常少的,转化过程经过很多中间产物导致转化速度比较慢。但是只要生物异源物质在环境中持续存在,微生物群落在其中生存,就能够在短时间内驯化出可降解硝基芳香烃的不同途径。
当微生物暴露在NAC中时,它有可能经历不同的代谢过程,如:分解代谢,利用NAC作为唯一的碳/氮源和能量供应源,最后形成无机终产物如CO2和水;或者共同代谢,这是一种特异地作用于其他底物的酶的非特异性转化NAC的途径,只有在其他底物存在时NAC才可以被转化。共同代谢反应在无氧和有氧条件下均可发生,可以在外加碳源时氧化或者还原形成最终的低毒或无毒物质。
NAC在生物系统中可被还原产生二级的非生物偶联反应,形成惰性的偶氮-、氧化偶氮和聚合物。该反应由两类反应酶催化,ⅠⅡ型酶为氧气不敏感型,Ⅱ型酶为氧气敏感型[4]。反应酶可将一个或两个电子转移到硝基或亚硝基上,Ⅰ型硝酸还原酶在电子对供体和无氧下,最终产生芳香胺;Ⅱ型酶在梭菌属、大肠杆菌、植物和动物中广泛存在,可还原硝基为亚硝基[5]。生物介导的还原反应主要发生在邻位,而非对位。
1.1 无氧条件下的生物降解转化
在无氧条件下,微生物通过硝基还原酶催化NAC上的硝基接受共底物连续供给的电子对还原成亚硝基衍生物、羟胺或胺。
研究发现,在厌氧条件下,大多数聚硝基芳香烃易被降解,但只有极少数厌氧微生物能将NAC底物完全转化成CO2或甲烷,如:洛氏芽胞盐杆菌(Haloanaerobicum praevalens)可降解硝基苯为芳香胺类化合物[6];甲酸甲烷杆菌可完全转化3,4-硝基酚[7];双酶梭菌(Clostridium bifermentans)可将 2,4,6-三硝基甲苯(TNT)转化为 2,4,6-三氨基甲苯(TAT),再转化为 2,4,6-三苯甲醇[8];真氧产碱杆菌(Ralstonia eutropha)可将3-硝基酚降解为氨基酚[9];罗尔斯通菌可将4-硝基苯甲酸盐降解成3-羟基-4-对氨基苯甲酸甲酯[10];耐热醋酸棱状芽胞杆菌、Methylobacterium sp.BJ001、克雷白氏杆菌属C1、瘤胃微生物可将TNT分别还原成三甲苯醇、氨基二硝基甲苯、亚硝酸、2-羟基l-氨基-4,6-二硝基甲苯[11~14]。TNT、二硝基甲苯、3,5-二硝基苯甲酸、2,3,4-硝基酚等化合物的部分或者完全降解需要多个细菌菌团的协同参与,如:脱磷弧菌和梭菌属的纯培养可通过形成三氨基甲苯中间体而快速降解 TNT[5]。
厌氧环境下的还原率由整个分子的化学性质(环和取代基的个数和类型)来决定。随着硝基对位功能基团吸电子能力的增加,硝基还原率会相应增加。研究发现,一种颗粒厌氧污泥可以将硝基酚矿化为甲烷[15],而在氢和其他原电子供体(乙醇等)存在时,产甲烷颗粒淤泥的还原速率会上升[16]。
1.2 有氧条件下的生物修复
好氧微生物对NAC的降解主要是以单-、双-硝基芳烃作为完全矿化时的碳/氮源和能量来源。如,放射菌类可将单硝基酚羟基化,形成亚硝酸和邻-或对-二羟基苯[17]。恶臭假单胞菌2NP8菌株降解杀虫剂(对硫磷和甲基对硫磷)时,首先将其水解为4-对硝基苯酚,随后在硝基上发生羟化,此菌株还能够完全转化30个单-、双-硝基芳香烃底物[18]。目前,已分离得到多种降解NAC的好氧微生物,见表1。
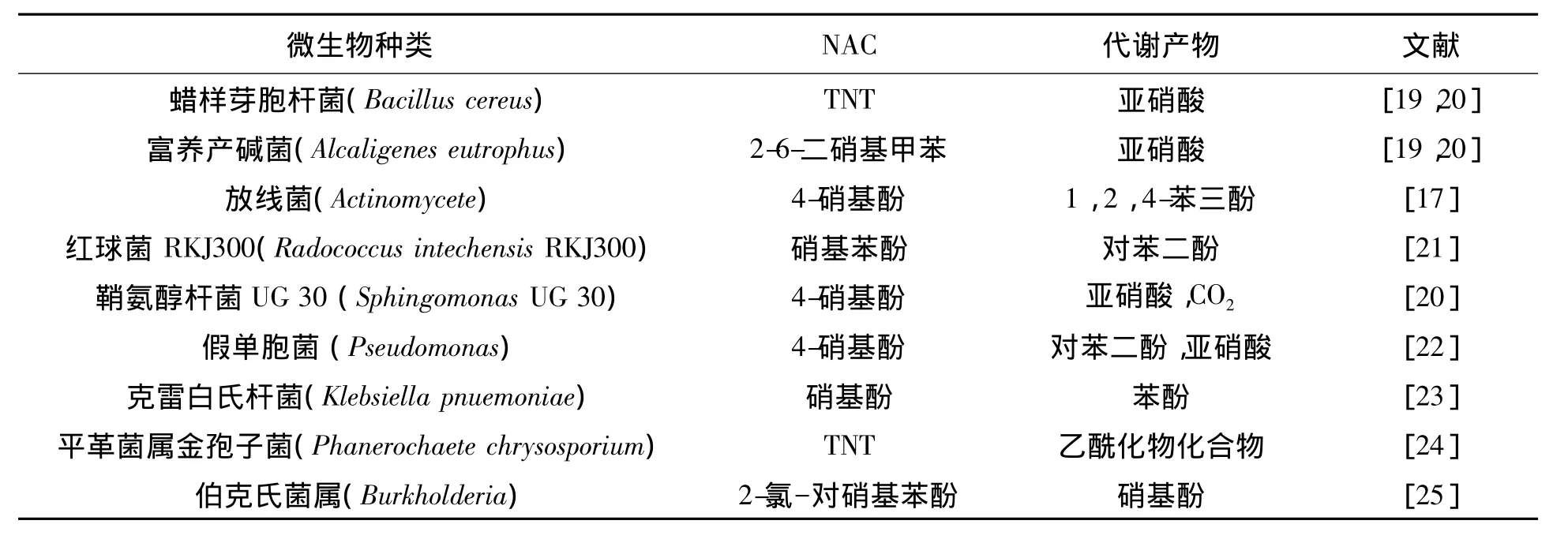
表1 好氧微生物对硝基芳烃化合物的降解Table 1 Degradation of nitro-aromatics by aerobic microorganisms.
2 生物修复方式
目前NAC的生物修复主要有原位和非原位两种。
2.1 原位生物修复
原位生物修复是在污染的原地进行,主要处于实验研究阶段,仅有少数能在实际中应用。也有研究利用植物与微生物共同在原位作用,如利用柳枝稷、矮菜豆与灭菌的根培养物或它们的混合物转化TNT[26];利用植物根际的假单胞菌进行TNT 的根际修复[20]等。
2.2 非原位生物修复
非原位生物修复是将污染物从产生地移出后进行修复。由于易操作和可控性,此类研究较多。非原位生物修复常用反应器进行处理,但成本相对高。Ziganshin等[27]将 TNT重污染的土壤与水、解脂假丝酵母AN-L15菌株和淀粉等基质混合在反应器中,最终将TNT降解,该反应中有有机酸中间产物的产生,因此必须调节pH以保证其降解率。由于反应器中承载的营养物质有限,微生物细胞存活不易,并存在重金属的抑制和细胞易洗掉等缺陷,现在又发展出固定细菌细胞技术(immobilized bacteria technology,IBT)[28],该技术用粘附/交联/截留的方法将细胞固定在不同基质上,如泥炭、琼脂糖、粘土、藻酸盐、硅藻土和k-角叉莱胶等,或自行固定在颗粒泥浆上,生物修复的加载率和修复率都较反应器法有所提高。Sreenivasulu等[29]用3%海藻胶固定芽胞杆菌成功降解了硝基酚;Peres等[30]用硅藻土固定洋葱伯克霍尔德菌(Burkholderia cepacia)PB4对4-硝基苯甲酸盐和4-苯佐卡因混合物进行了高效降解;Mulla等[31]实现了聚氨酯包裹微球菌SMN-1对土壤中二硝基甲苯的生物修复;相对于反应器中的游离细胞,固定化细胞对pH条件和pH变化更有耐受性。而且两个菌株共同固定比单独固定一类细胞效果更好,Arora等[32]在藻酸钙上共同固定硝基还原的厌氧肠道菌(Enterobacter)和矿化4-氯-2-硝基酚的好氧产碱杆菌属(Alkaligenes),最终将4-氯-2-硝基酚完全降解;在藻酸盐微珠上共同固定丛毛单胞菌属(Comamonas)JS46和JS47也成功清除了重工业废水中的对硝基苯甲酸[33]。
3 NAC微生物降解的分子生物学
利用分子生物学技术改造基因来提高微生物系统的修复性能是目前的研究热点之一。
3.1 代谢途径及酶活性的定向改造
NAC类化合物的降解过程是一个复杂的酶反应过程,提升降解速率可通过锚定限速步骤来改进,如提高限速酶的表达量或者稳定性,调节有关调控蛋白的表达量等。Cho等[34]利用 DNA shuffling连续循环得到了提高有机磷酸酯水解酶活性的变异株E.coli 22A11,产生的水解酶降解速率比野生型快25倍。Ensley等[35]通过基因工程手段增加编码基因的翻译来提升限速酶和调节蛋白的表达水平,使得改造后的大肠杆菌氧化萘的速度可以达到 5.0 g/L·h。
组成型酶高度的底物特异性使微生物不能有效代谢很多硝基-芳香烃,体外蛋白质工程可通过定向修饰来改变酶降解底物的动力学。如,改变细胞色素P450活性位点拓扑结构,使其能结合卤代脱氢酶,并影响芳烃加双氧酶的底物范围,由此构建的改造菌株能够降解目标硝基芳烃和其他有毒污染物,应用于含有毒化学物的工业废水的处理[36]。
NAC不能被有的微生物完全矿化,但经微生物转化后的终端产物可作为其他微生物的底物。因此,结合两种供体菌株的基因,构建代谢途径衔接、互补的高效工程菌株可使NAC达到有效矿化。鞘氨醇单胞菌UG30中pcp-B基因编码五氯苯酚降解酶,该基因在E.coli中异源表达,能够加速4-硝基邻苯二酚的降解并在降解p-硝基酚时形成少量亚硝酸[37]。因此,结合不同代谢途径能够创造作用更宽范围的NAC降解微生物。
3.2 质粒载体介导降解的研究
质粒可携带NAC降解相关的基因,介导微生物降解NAC,以质粒为载体构建工程菌株具有很大优势[38]。如,恶臭假单胞菌Paw中TOL质粒,编码NAC降解基因。Kadiyala等[39]构建了两种重组质粒,分别携带有阴沟肠杆菌的硝基苯还原酶基因(nfs1)和假产碱假单胞菌JS45的硝基苯硝基还原酶基因(nbzA)及羟基苯胺变旋酶A(habA)基因,这两种质粒分别在大肠杆菌JS 995和JS 996中表达,重组大肠杆菌能够快速地将硝基苯转化为对氨基苯酚。由55 kb传递性质粒介导的不动杆菌株RKJ12能以2-氯-4-对硝基甲酸作为单一碳/氮源和能源[40]。表2总结了可用于NAC降解的质粒。目前,含有转座插入lux基因的萘分解质粒的荧光假单胞菌HK44突变体是美国第一个批准野外测试的转基因生物(GMO)[41]。

表2 不同种类的硝基芳烃化合物降解质粒Table 2 Different types of nitro-aromatic compounds degradation plasmids.
3.3 基因工程菌株的降解效果
通过基因工程手段,将降解酶基因重组到目标微生物中,可解决生物修复中遇到的各种问题,在很大程度上提高降解效果,增强实际应用的可行性。Cui等[46]发现,在 E.coli中表达来源于邻单胞菌M6的甲基对硫磷水解酶基因(mpd),可将NAC水解为p-硝基酚。用Burkholderia DNT的pJS1(包含2,4-二硝基甲苯基因簇)与苜蓿中华根瘤菌(Sinorhizobium meliloti)USDA 1936构建微共生体,获得的中华根瘤菌株DHK1能够显著降低污染土壤中的2,4-二硝基甲苯含量[28]。将洋葱伯克霍尔德菌(Burkholderia cepacia)的甲苯单氧酶基因改造并导入荧光假单胞菌里,4 d即可清除小麦土壤微环境中63%的TCE[47]。尽管高效的基因工程微生物非常具有应用前景,但成功的生物修复还需要对现场条件进行评估。
4 展望
目前,已对NAC微生物降解的多种途径及其中间代谢产物的种类进行了广泛研究,但是对其分解顺序的分子基础还需更全面透彻的了解。虽然已经获得了多种降解微生物资源,但对其特性及功能酶结构、调控和分子生物学方面还需要更全面的理解。实际应用中,由于污染物种类繁多,更新变化较快,微生物在其中起到的作用也受到一定的限制,还需要开发和发现新的微生物类群,建立全面的操作性强的矿化技术。
在NAC微生物降解的研究中,重点将在以下方面展开。①降解微生物的土壤定植情况的生态位鉴定,其中基因探针的方法是展开监测的有效手段。②在硝基芳香烃污染的生态系统中引入植物修复,并与根基微生物相结合,这是一个可持续、生态友好、高效的根际修复过程。③探究超微细菌(ultramicrobacteria)种群从土层快速移除来消化有毒废物的潜力,可为原位生物修复策略中存在的问题提供了一种选择指导。
NAC微生物降解在实际中还存在生物异源物质的高极限浓度下处理酶的失活问题、在生态系统中化合物的生物利用率问题,及在比较苛刻的生化条件下降解微生物的存活问题等,只有逐步解决这些问题,微生物修复技术才可以更加有效。对于处理量的提高,优化处理参数是有效的生物修复策略所必需的。在实际应用中,仍然需要强调可持续生物修复的生态学原则。因此,探索新代谢途径、简化天然进化过程、改造出能净化广泛污染物种类的微生物是科学家不懈追求的目标。
[1]Luan F,Xie L,Li J,et al..Abiotic reduction of nitroaromatic compounds by Fe(II)associated with iron oxides and humic acid[J].Chemosphere,2013,91(7):1035-1041.
[2]Peres C M,Agathos S N.Biodegradation of nitroaromatic pollutants:from pathways to remediation [J].Biotechnol.Annu.Rev.,2000,6:197-220.
[3]Li L,Wang J,Zhou J,et al..Enhancement of nitroaromatic compounds anaerobic biotransformation using a novel immobilized redox mediator prepared by electropolymerization[J].Bioresour.Technol.,2008,99(15):6908-6916.
[4]Preuss A,Rieger P G.Anaerobic transformation of 2,4,6-trinitrotoluene and other nitroaromatic compounds[A].In:Spain J C.(ed.)Biodegradation of Nitroaromatic Compounds[M].New York:Springer-Verlag,1995,69-85.
[5]Spain J C.Biodegradation of nitroaromatic compounds[J].Annu.Rev.Microbiol.,1995,49:523-555.
[6]Oren A,Gurevich P,Henis Y.Reduction of nitrosubstituted aromatic compounds by the halophilic anaerobic eubacteria Haloanaerobicum praevalens and Sporohalobacter marismortui[J].Appl.Environ.Microbiol.,1991,57(11):3367-3370.
[7]Gorontzy T,Küver J,Blotevogel K H.Microbial transformation of nitro-aromatic compounds under anaerobic conditions[J].J.Gen.Microbiol.,1993,139:1331-1336.
[8]Lewis T A,Goszczynski S,Crawford R L,et al..Products of anaerobic 2,4,6-trinitrotoluene(TNT)transformation by Clostridium bifermetans [J]. Appl. Environ. Microbiol.,1996,62(12):4669-4674.
[9]Schenzle A,Lenke H,Fischer P,et al..Catabolism of 3-nitrophenol by Ralstonia eutropha JMP134 [J]. Appl.Environ.Microbiol.,1997,63(4):1421-1427.
[10]Peres C M,Russ R,Lenke H,et al..Biodegradation of 4-nitrobenzoate,4-aminobenzoate and theirmixtures:new strains,unusual metabolites and insight into pathway regulation[J].FEMS Microbiol.Ecol.,2001,37(2):151-159.
[11]van Aken B, et al.. Biodegradation of nitro-substituted explosives, TNT, RDX and HMX by a phytosymbioticMrthylobacterium sp.associated with poplar tissues[J].Appl.Environ.Microbiol.,2004,70:508-517.
[12]Kim H,Bennett G N,Song H.Degradation of TNT by Klebsiella sp.isolated form activated sludge[J].Biotechnol.Lett.,2002,24:2023-2028.
[13]Fleischmann T J,Walker K C,Spain J C,et al..Anaerobic transformation of 2,4,6-TNT by bovine ruminal microbes[J].Biochem.Biophys.Res.Commun.,2004,314(4):957-963.
[14]Sponza D T,Kuscu O S.p-nitrophenol removal in a sequential anaerobic migrating blanket reactor (AMBR)/aerobic completely stirred tank reactor(CSTR)system [J].Proc.Biochem.,2005,40:1679-1691.
[15]Razo-Flores E, Donlon B, Lettinga G, et al..Biotransformation and biodegradation of N-substituted aromatics in methanogenic granular sludge[J].FEMS Microbiol.Rev.,1997,20(3-4):525-538.
[16]Razo-Flores E,Lettinga G,Field J A.Biotransformation and biodegradation ofselected nitro-aromaticsunderanaerobic conditions[J].Biotechnol.Prog.,1999,15:358-365.
[17]Kulkarni M A,Chaudhari A B.Biodegradation of p-nitrophenol by P.putida[J].Bioresour.Technol.,2006,97(8):982-988.
[18]Ye J, Singh A, Ward O P, et al.. Biodegradation of nitroaromatics and other nitrogen containing xenobiotics[J].World J.Microbiol.Biotech.,2004,20:117-135.
[19]Mercimek H A, Dincer S, Guzeldag G, et al.. Aerobic biodegradation of 2,4,6-trinitrotoluene(TNT)by Bacillus cereus isolated from contaminated soil[J].Microb.Ecol.,2013,66(3):512-521.
[20]Kulkarni M,Chaudhari A.Microbial remediation of nitroaromatic compounds:an overview[J].J.Environ.Manage.,2007,85(2):496-512.
[21]Ghosh A,Khurana M,Chauhan A,et al..Degradation of 4-nitrophenol,2-chloro-4-nitrophenol,and 2,4-dinitrophenol by Rhodococcus imtechensis strain RKJ300 [J].Environ.Sci.Technol.,2010,44(3):1069-1077.
[22]Qureshi A A,Purohit H J.Isolation of bacterial consortia for degradation of p-nitrophenol from agricultural soil[J].Ann.Appl.Biol.,2002,140:159-162.
[23]Melancon E,Boopathy R.Metabolism of compounds with nitrofunctions by Klebsiella pnuemoniae isolated from a regional wetland[J].Int.Biodeterior.Biodegrad.,2004,54:269-275.
[24]Hodgson J,Rho D,Guiot S R,et al..Tween-80 enhanced TNTmineralization by Phanerochaete chrysosporium[J].Can.J.Microbiol.,2000,46:110-118.
[25]Pandey J,Heipieper H J,Chauhan A,et al..Reductive dehalogenation mediated initiation of aerobic degradation of 2-chloro-4-nitrophenol(2C4NP)by Burkholderia sp.strain SJ98[J].Appl.Microbiol.Biotechnol.,2011,92(3):597-607.
[26]Riveraa R,Medinab V F, Larsonc S L, et al.. Phytotreatment of TNT contaminated ground water[J].J.Soil Contam.,1998,7(4):511-529.
[27]Ziganshin A M,Naumova R P,Pannier A J,et al..Influence of pH on 2,4,6-trinitrotoluene degradation by Yarrowia lipolytica[J].Chemosphere,2010,79(4):426-433.
[28]Dutta S K,Hollowell G P,Hashem F M,et al..Enhanced bioremediation of soil containing 2,4-dinitrotoluene by a genetically modified Sinorhizobium meliloti[J].Soil Biol.Biochem.,2003,35(3):667-675.
[29]Sreenivasulu C,Megharaj M.Degradation of p-nitrophenol by immobilized cells of Bacillus spp.isolated from soil[J].Int.Biodeterior.Biodegrad.,2012,68:24-27.
[30]Peres C M,van Aken B,Naveau H,et al..Continuous degradation of mixtures of 4-nitrobenzoate and 4-aminobenzoate by immobilized cells of Bukholderia cepacia PB4[J].Appl.Microbiol.Biotechnol.,1999,52(3):440-445.
[31]Mulla S,Talwar M P,Bagewadi Z K,et al..Enhanced degradation of 2-nitrotoluene by immobilized cells of Micrococcus sp.strain SMN-1[J].Chemosphere,2013,90(6):1920-1924.
[32]Arora P K, Sasikala Ch, Ramana ChV. Degradation of chlorinated nitroaromatic compounds[J].Appl.Microbiol.Biotechnol.,2012,93(6):2265-2277.
[33]Field J A, Stams A J, Kato M, et al.. Enhanced biodegradation of aromatic pollutants in co-cultures of anaerobic and aerobic bacterial consortia[J].Antonie van Leeuwenhoek,1995,67(1):47-77.
[34]Cho Y G,Rhee S K,Lee S T.Influence of phenol on biodegradation ofp-nitrophenolby freely suspended and immobilized Nocardioides sp.NSP41 [J].Biodegradation,2000,11(1):21-28.
[35]Ensley B D, DeFlaun M F. Hazardous chemicals and biotechnology:past successes and future promise[A].In:Young L Y,Cerniglia C E.(Eds.)Microbial Transformation and Degradation of Toxic Organic Chemicals[M].New York:Wiley Liss Inc.,603-629.
[36]Holloway P,Knoke K L,Trevors J T,et al..Alteration of the substrate range of haloalkane dehalogenase by site-directed mutagenesis[J].Biotechnol.Bioeng.,1998,59(4):520-523.
[37]Cho C M,Mulchandani A,Chen W.Bacterial cell surface display of organophosphorus hydrolase for selective screening of improved hydrolysis of organophosphate nerve agents[J].Appl.Environ.Microbiol.,2002,68(4):2026-2030.
[38]Wagner K H,Schwarz T,Kaufmann M.Phenol degradation by an Enterobacterium:a Klebsiella strain carries a TOL like plasmid and a gene encoding a novel phenol hydroxylase[J].Can.J.Microbiol.,1999,45:162-171.
[39]Kadiyala V, Nadeau L J, Spain JC. Construction of Escherichia coli strains for conversion of nitroacetophenones to ortho-aminophenols[J].Appl.Environ.Microbiol.,2003,69(1):6520-6526.
[40]Prakash D,Kumar R,Jain R K,et al..Novel pathway for the degradation of 2-chloro-4-nitrobenzoic acid by Acinetobacter sp.strain RKJ12 [J].Appl.Environ.Microbiol.,2011,77(18):6606-6613.
[41]Ripp S,Nivens D E,Ahn Y,et al..Controlled field release of a bioluminescent genetically engineered microorganism for bioremediation process monitoring and control[J].Environ.Sci.Technol.,2000,34(5):846-853.
[42]Prakash D, Chauhan A, Jain R K. Plasmid encoded degradation of p-nitrophenol by P.cepacia[J].Biochem.Biophys.Res.Commun.,1996,224:375-381.
[43]James K D,Williams P A.ntn genes determining the early steps in the divergent catabolism of 4-nitrotoluene and toluene in Pseudomonas sp.strain TW3[J].J.Bacteriol.,1998,180(8):2043-2049.
[44]Hayatsu M,Hirano M,Tokuda S.Involvement of two plasmids in fenitrothion degradation by Burkholderia sp.strain NF100[J].Appl.Environ.Microbiol.,2000,66:1737-1740.
[45]Kulkarni M A, Chaudhari A B. Kinetic studies on biodegradation of p-nitrophenol by P.putida.[J].India J.Chem.Technol.,2006,13:60-64.
[46]Cui Z L,Li S P,Fu G P.Isolation of methyl parathion degrading strains M6 and cloning of the parathion hydrolase gene[J].Appl.Environ.Microbiol.,2001,67:4922-4925.
[47]Yee D C,Maynard J A,Wood T K.Rhizoremediation of trichloroethylene by a recombinant root-colonizing Pseudomonas fluorescens strain expressing toluene ortho-monooxygenase constitutively[J].Appl.Environ.Microbiol.,1998,64(1):112-118.

