一份水稻矮杆小粒突变体的形态特征和基因定位
曹 建, 孙学辉, 路铁刚
中国农业科学院生物技术研究所,北京100081
水稻矮杆基因在研究水稻株型发育机理,诠释植物生长发育机制中扮演着重要的角色。水稻的株高变矮是由矮杆主效基因控制,同时也受到修饰基因、调节基因以及抑制基因的共同影响。矮杆基因的作用可以导致水稻植株水平或者细胞水平的变化,如节间变短,细胞个数下降,进而植株变矮。水稻的株高发育还与植物激素有关。在已克隆的与水稻株高发育有关的基因中,与赤霉素有关的有 SD1[1]、SLR1[2]和 EUI1[3]等,与油菜素内酯(BR)有关的有 D11[4]、BRD1[5]和 D2[6]等。其中SLR1和EUI1为高杆基因,SD1为半矮秆基因,其余为矮杆基因。这些基因在赤霉素或油菜素内酯的合成或信号转导中发挥着重要作用。
水稻的籽粒大小是水稻产量的主要影响因素之一,并直接影响稻米的外观和品质。研究水稻控制水稻籽粒发育相关的基因,明确水稻籽粒生长发育的机理,对于解释水稻生殖生长的模式以及培育高产优质的水稻品种有着重要作用。目前,水稻分子生物学家已经利用突变体对小粒基因做过了大量研究,如H346/L32以及Kitaake中的小粒基因 MI1和 MI2[7~14];来源于斯里兰卡的突变体club mutant的圆短粒基因KR1[15]等。其余的小粒性状基因还控制着稻谷芒、籽粒纹理、圆籽粒以及株高等性状。伴随籽粒纹理性状出现小粒性状的基因有LGT[16],伴随圆籽粒性状出现小粒性状的基因有 RK1、RK2[17,18],伴随芒性状出现小粒性状的基因有 AN6、AN7[15],伴随矮杆性状出现小粒性状的基因有 D56、D2、D54、DK6、DWF33、DWF1 和 D38 等[6,14,19~21]。
本文以1份水稻矮杆小粒材料t129为基础,通过与水稻籼稻亚种Dualr杂交得到F1代和F2代群体,对该突变体的矮杆小粒性状进行遗传分析,以确定该突变体的遗传规律,同时利用Dular/t129杂交F2代群体定位了一个隐性矮杆小粒基因。
1 材料与方法
1.1 水稻材料
水稻矮杆小粒突变体来源于本实验室构建的T-DNA插入突变体库[22],遗传背景为粳稻品种日本晴。经过河北和海南多代自交和选择,突变体表型稳定,受种植环境影响极小,是遗传稳定的品系。本实验中,水稻野生型材料为粳稻品种日本晴(Oryza Sativa L.spp.Japonica cv.Nipponbare)和籼稻品种Dular。
1.2 表型观察
将日本晴与纯合突变体t129种植于田间,种植10行,每行10株。成熟后分别在中间两行选择20株,调查株高、千粒重、结实率。
1.3 遗传分析及基因定位的群体构建
以矮杆小粒突变体t129为母本(P1)与野生型粳稻品种日本晴(P2)进行正反交,收取F1代种子,种植后进行自交得到F2代种子。将F2代种子种植。观察杂交组合F1、F2群体中出现野生型表型和矮秆小粒表型植株的数量,对矮杆小粒突变体进行遗传分析。
利用矮杆小粒突变体t129/籼稻品种Dular(P3)的F2(P1/P3)群体作为基因定位群体。
1.4 基因组DNA提取
根据突变体t129与Dular杂交组合F2代植株表型,选取隐性表型单株10株,分成两组各5株,每组剪取等量叶片混合,构建隐性表型基因池。采用改进的CTAB法[23]提取隐性表型基因池。精细定位时用同样方法提取用于连锁分析的F2代单株DNA。
1.5 InDel分子标记分析
本实验所用InDel引物由北京擎科新业生物技术有限公司合成。利用网上所公布的粳稻亚种日本晴和籼稻亚种9311的全基因组序列进行比对,利用在籼稻粳稻之间片段大小差异设计引物,引物设计软件为DNAMAN,引物设计完成后,在日本晴和Dular两个杂交亲本之间检测多态性。然后利用多态性引物对前述Dualr/t129 F2隐性表型混池进行初定位,再用与该混池连锁的初定位标记对隐性F2代全部单株进行连锁分析,确定引物与F2代隐性表型群体的连锁关系。本研究开发的部分InDel标记见表1。
PCR 扩增体系(10 μL)为:ddH2O 6.1 μL,10× PCR Buffer 1 μL,MgCl2(25mmol/L)0.6 μL,dNTP Mix(1mmol/L)0.2 μL,引物(2 pmol,正向+反向)1 μL ,Takarar Taq(5 U/μL)0.1 μL,DNA模板(20 ng/μL)1 μL。PCR 扩增程序为:94℃预变性 5min;94℃变性 30 s,56℃退火 30 s,72℃延伸30 s,扩增 5个循环;第二步,94℃变性30 s,58℃退火30 s,72℃延伸 30 s,扩增33 个循环;最后72℃下延伸7min。反应产物在8%聚丙烯酰胺凝胶上电泳,电压200 V,时间2 h 10min。电泳完成后用银染显色,在胶片观察灯下观察结果并照相。将电泳图谱进行数值转换:与隐性亲本带型一致的记为1型带,与显性亲本带型一致的记为2型带,同时具有双亲本带型的的记为3型带。

表1 本研究开发的多态性InDel标记Table 1 Polymorphic InDel markers used in this study.
1.6 遗传作图与基因定位分析
利用Mapmaker软件对转换数据进行分析,并进行遗传作图[24],对矮杆小粒基因进行定位分析。
2 结果与分析
2.1 t129小粒突变体的表观性状的遗传分析
2.1.1 t129突变体与野生型亲本的性状差异通过田间测定,原始亲本日本晴植株平均高度为91cm,而 t129突变表型植株平均高度为51cm,突变植株高度明显下降矮化(见图1A,彩图见227页图版)。同时突变植株与原始亲本植株日本晴的粒长相比也明显缩短(见图1B),平均长度由野生表型的0.768cm缩短为0.562cm,千粒重也明显下降(见表2)。

图1 成熟期野生型t129的植株形态与籽粒粒型Fig.1 Phenotype of the wild type and t129 mutant at mature stage.
2.1.2 突变体t129的遗传分析 将纯合t129突变体与日本晴进行杂交,获得F1代杂交种子。种植F1代植株,对其植株高度和籽粒形态进行观察。如表3所示,F1代粒长均偏向于野生型亲本。如日本晴/t129的F1代粒长平均值为0.742cm,明显偏向于野生型亲本日本晴(平均粒长为0.768cm)。在日本晴/t129杂交F1代中株高性状也同样偏向于日本晴。由此可见,t129突变体矮杆小粒的性状受隐性基因控制。

表2 突变体t129日本晴的农艺性状分析Table 2 Agronomic traits of the wild type and the t129 mutant.

表3 突变体t129与日本晴及其杂种F1代株高和粒长性状统计Table 3 Statistics on plant height and grain length among parents and F1.
为了进一步明确矮秆、小粒突变表型是否由单基因控制,将F1代植株进行自交获得F2代植株群体,并对其进行遗传分析统计。如表4所示,t129突变体与日本晴的组合F2代分离群体中,野生型表型246株,突变型植株数85株,卡方值,无显著性差异,符合3∶1的孟德尔遗传理论分离比例。日本晴与t129突变体杂交组合F2代分离群体中,野生型表型植株309株,突变体表型植株107株。卡方值=3.84,同样无显著性差异,符合3∶1的孟德尔遗传理论分离比例。以上研究结果表明,矮杆小粒突变体受一对隐形单基因控制。

表4 F2代分离群体的统计分析Table 4 Statistical analysis of the phenotype of F2segregating population.
2.2 t129基因的初步定位
为了进一步克隆突变基因,将日本晴背景的纯合t129突变体与Dular杂交获得F1代,而后F1代自交获得F2代分离群体,对其突变位点进行精细定位。
用600对InDel引物对亲本Dular和日本晴进行多态性分析,有151对引物在Dular和日本晴之间有多态性。这151对引物均匀分布在水稻的12条染色体上。用此151对引物对Dular/t129 F2代群体隐性表型植株混池(5株/混池)进行初步染色体定位,发现两个DNA混池与5号染色体长臂上rm5-6标记完全连锁。进而取20株突变体表型单株和10株野生型表型单株对该标记进行验证,证明 rm5-6与 t129突变位点连锁,在rm5-6标记上下游合成InDel引物,其中有20对在亲本间具有多态性,其中多态性较好的标记是InDel3、InDel6、InDel8、InDel31、InDel43、InDel51、InDel57和InDel65,利用88个突变体表型单株将t129定位在rm5-6与InDel65之间,进一步利用426株突变表型(隐性单株)群体和新开发的分子标记最终把t129基因定位在5号染色体InDel43与InDel57之间,物理距离约430 kb(见图2),其中与InDel51共分离(见图3)。
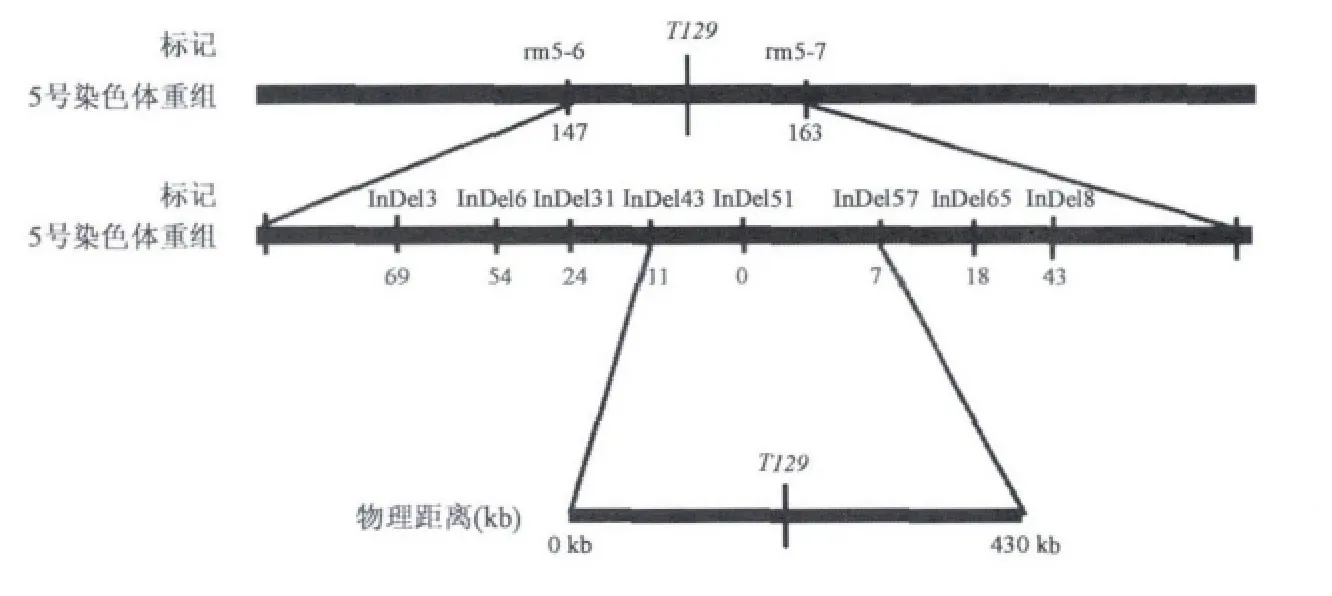
图2 t129基因在第5号染色体上的分子定位Fig.2 Molecular mapping of T129 on the chromosome 5.
3 讨论
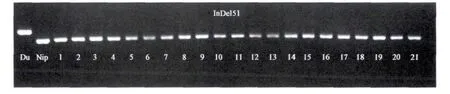
图3 t129突变位点与InDel51标记连锁分析Fig.3 Linkage analysis of the mutant locus of t129 with Indel51 marker.
水稻是研究单子叶植物的模式植物之一。水稻矮杆、半矮秆突变体在研究单子叶植物生长发育调控模式中也扮演着不可或缺的作用[25,26]。水稻矮杆、半矮秆基因在水稻植株中不仅决定了植株高矮,还在分蘖、茎秆、育性等性状中发挥作用,参与水稻植株形态建成的各种生理生化过程。目前,发现和报道的水稻矮杆、半矮秆突变体已超过了80个。研究表明大部分分离克隆的矮杆和半矮秆基因与植物的赤霉素、油菜素内酯(BR)和独脚金素内酯这三种激素的合成代谢、信号转导有关系。如前述的SD1基因编码GA20氧化酶,是赤霉素合成途径中的关键酶[27];水稻GID1基因是从一个极矮、GA不敏感突变体gid1中克隆出来的,它编码一个类似于激素敏感脂肪酶蛋白,作用于植物特有的GRAS转录因子N端的DELLA结构域,参与赤霉素信号转导[28];水稻GID2基因突变也表现出极端矮化的表型,其通过参与泛素介导的蛋白质降解负调控GA信号转导;从水稻矮杆突变体d11中克隆的CYP724B1基因在BR合成中起着关键作用[29];水稻BRD1基因发生突变可以表现出植株极端矮化,所有节间均不伸长等表型,该基因编码一个在BR合成中重要的限速酶[30];此外,调控水稻BR信号转导的主要基因如 DLT、IBH1等发生突变也会导致植株矮化[31,32]。因此,对于水稻矮杆突变体进行研究,能够进一步揭示水稻株型发育的分子机制与遗传机制。目前,在水稻理想株型育种实践中,矮杆、半矮秆突变体发挥着重要作用,对矮杆、半矮秆基因的克隆,对其参与的生物学途径的研究,能够为我们选育优良水稻品种、研究水稻生长发育的分子机理提供很大帮助。
此外,籽粒大小也是影响水稻产量的重要因素。粒长基因在遗传基础上决定着籽粒大小。从以往的报道中我们可以看出,粒长主要为数量性状[32~37]。但也有研究表明粒长受单基因控制[38]。在已克隆的与水稻粒长粒型有关的基因中,GW5和GW2与泛素介导的蛋白质降解过程有关[39,40],而 GS3 与细胞信号转导的负调控有关[41]。此外,根据前人报道,水稻籽粒的发育还受植物激素调节,如前述的在BR合成中起关键作用的 D11基因,其突变体也表现为小粒表型[4],此外,具有小粒表型的 d2、brd1突变体同样存在 BR 合成障碍[6,30]。因此,研究控制水稻籽粒发育的基因,对增进植物发育的分子机理以及细胞信号转导过程的认识大有裨益。
本研究对一矮杆小粒突变体t129进行了表型分析和初步定位。其株高约为野生型一半,籽粒粒长较野生型明显减少,是研究水稻株型发育与籽粒发育分子机理的良好材料。在分析突变体与野生型配置的正反交杂交组合后,F2群体中野生型表型与突变体表型分离比例符合3:1的遗传分离比例,由此表明t129突变体由常染色体隐性单基因控制。经过精细定位将t129定位到5号染色体长臂上,引物InDel43和InDel57之间,物理距离为430 kb,并与标记InDel51共分离。该结果为进一步研究水稻株高粒型调控基因、深入研究基因功能奠定基础。
[1]Sasaki A,Ashikari M,Matsuoka M M,et al..A mutant gibberellin-synthesis gene in rice[J].Nature,2002,416:701-702.
[2]IkedaA,Ueguchi-Tanaka M,Sonoda Y,et al..Slender rice,a constitutive gibberellin response mutant,is caused by a null mutation of the SLR1 gene,an ortholog of the height-regulating gene GAI/RGA/RHT/D8 [J].Plant Cell,2001,13(5):999-1010.
[3]Luo A,Qian Q,Yin H,et al..EUI1,encoding a putative cytochrome P450 monooxygenase, regulates internode elongation by modulating gibberellin responses in rice.[J].Plant Cell Physiol.,2006,47(2):181-191
[4]Tanabe S,Ashikari M,Fujioka S,et al..A novel cytochrome P450 is implicated in brassinosteroid biosynthesis via the characterization of a rice dwarf mutant,dwarf11,with reduced Seed Length[J].Plant Cell,2005,17(3):776-790.
[5]Hong Z,Ueguchi-Tanaka M,Shimizu-Sato S,et al..Loss-offunction of a rice brassinosteroid biosynthetic enzyme,C-6 oxidase,prevents the organized arrangement and polar elongation of cells in the leaves and stem[J].Plant J,2002,32(4):495-508.
[6]Chen Y,Wang M, Ouwerkerk PB F.. Molecular and environmental factors determining grain quality in rice[J].Food Energy Secur.,2012,1(2):111-132.
[7]Hashimoto Z,Mori N,Kawamura M,et al..Genetic diversity and phylogeny of Japanese sake-brewing rice as revealed by AFLP and nuclear and chloroplast SSR markers[J].Theor.Appl.Genet.,2004,109(8):1586-1596.
[8]Shao G,Wei X,Hu P,et al..Allelic variation for a candidate gene for GS7,responsible for grain shape in rice[J].Theor.Appl.Genet.,2012,125(6):1303-1312.
[9]Liu T M,Shao D,Xing Y Z,et al..Mapping and validation of quantitative trait loci for spikelets per panicle and 1,000-grain weight in rice(Oryza sativa L.)[J].Theor.Appl.Genet.,2010,120(5):933-942.
[10]Rabello A R, GuimarãesC M, RangelP H, et al..Identification of drought-responsive genes in roots of upland rice(Oryza sativa L.)[J].BMC Genomics,2008,485(9):1-13.
[11]Pietsch C,Sreenivasulu N,Wobus U,et al..Linkage mapping of putative regulator genes of barley grain development characterized by expression profiling [J].BMC Plant Biol.,2009,9(4):1-11.
[12]Feng B H,Yang Y,Wu J L,et al..Characterization and genetic analysis of a novel rice spotted-leaf mutant HM47 with broad-spectrum resistance to Xanthomonas oryzae pv.oryzae.[J].J.Integr.Plant Biol.,2013,55(5):473-483.
[13]Huang T Y,Wang Z,Hu Y G.Genetic analysis and primary mapping of pms4,a photoperiod-sensitive genic male sterility gene in rice(Oryza sativa)[J].Rice Sci.,2008,15(2):153-156.
[14]Yan C J,Zhou J H, Yan S, et al.. Identification and characterization of a major QTL responsible for erect panicle trait in japonica rice(Oryza sativa L.)[J].Theor.Appl.Genet.,2007,115(8):1093-1100.
[15]Xie X,Mihee Song M H,Jin F,et al..Fine mapping of a grain weight quantitative trait locus on rice chromosome 8 using near-isogenic lines derived from a cross between Oryza sativa and Oryza rufipogon [J].Theor.Appl.Genet.,2006,113(5):885-894.
[16]Furukawa T,Maekawa M,Oki T,et al..The Rc and Rd genes areinvolved in proanthocyanidin synthesis in rice pericarp[J].Plant J.,2007,49(1):91-102.
[17]Li H,Xue D,Gao Z,et al..A putative lipase gene EXTRA GLUME1 regulates both empty-glume fate and spikelet development in rice[J].Plant J,2009,57(4):593-605.
[18]Liu S,Wang F,Gao L J,et al..Genetic analysis and fine mapping of LH1 and LH2,a set of complementary genes controlling late heading in rice(Oryza sativa L.)[J].Breed Sci.,2012,62(4):310-319.
[19]Yoshimura A,Ideta O,Iwata N.NAL1 allele from a rice landrace greatly increases yield in modern indica cultivars[J].Proc.Natl.Acad.Sci.USA,2013,110(51):20431-20436.
[20]Hada H,Hidema J,Maekawa M,et al..Higher amounts of anthocyanins and UV-absorbing compounds effectively lowered CPD photorepair in purple rice(Oryza sativa L.)[J].Plant Cell Environ.,2003,26(10):1691-1701.
[21]Wang Y H,Li J Y.The plant architecture of rice(Oryza sativa)[J].Plant Mol.Biol.,2005,59(1):75-84.
[22]Wan S,Wu J,Zhang Q,et al..Activation tagging,an efficient tool for functional analysis of the rice genome[J].Plant Mol.Biol.,2009,69,69-80.
[23]Zhang W,Sun X,Yuan H,et al..The pattern of insertion/deletion polymorphism in Arabidopsis thaliana [J]. Mol.Genet.Genomics.,2008,280(4):351-361.
[24]Lim J H,Yang H J,Jung K H,et al..Quantitative trait locus mapping and candidate gene analysis for plant architecture traits using whole genome re-sequencing in rice [J].Mol.Cells,2014,37(2):149-160.
[25]Sakamoto T,Miura K, Itoh H, et al.. An overview of gibberellins metabolism enzyme genes and their related mutants in rice[J].Plant Physiol.,2004,134(4):1642-1653.
[26]Xue Y,Li J,Xu Z.Recent highlights of the China rice functional genomics program [J].Trends Genet.,2003,19(7):390-394.
[27]Sasaki A,Ashikari M,Ueguchi-Tanaka M,et al..Green revolution:a mutant gibberellin-synthesis gene in rice[J].Nature,2002,416(6882):701-702.
[28]Ueguchi-Tanaka M, AshikariM, Nakajima M, et al..GIBBERELLIN INSENSITIVE DWARF1encodes asoluble receptor for gibberellin[J].Nature,2005,437(7059):693-698.
[29]Zhang C,BaiM Y, ChongK. Brassinosteroid-mediated regulation of agronomic traits in rice[J].Plant Cell Rep.,2014,33(5):683-696.
[30]Mori M, Nomura T, Ooka H, et al.. Isolation and characterization of a rice dwarf mutant with a defect in brassinosteroid biosynthesis[J].Plant Physiol.,2002,130(3):1152-1161.
[31]Tong H, LiuL, JinY, et al.. DWARFAND LOWTILLERING acts as a direct downstream target of a GSK3/SHAGGY-like kinase to mediate brassinosteroid responses in rice[J].Plant Cell,2012,24(6):2562-2577.
[32]Zhang L Y,Bai M Y,Wu J X,et al..Antagonistic HLH/bHLH transcription factors mediate brassinosteroid regulation of cell elongation and plant development in rice and Arabidopsis[J].Plant Cell,2009,21:125-139.
[33]Li Z F,Wan J M,Xia J F,et al..Mapping quantitative trait loci underlying appearance quality of rice grains(Oryza sativa L.)[J].Acta Genet.Sin.,2003,30:251-259.
[34]Xing Y Z,Tan Y F,Xu C G,et al..Mapping quantitative trait loci for grain appearance traits of rice using a recombinant inbred line population [J].Acta Bot.Sin.,2001,43(8):840-845.
[35]Li Z K,Yu S B,Lafitte H R,et al..QTL × environment interactions in rice.I.Heading date and plant height[J].Theor.Appl.Genet.,2003,108(1):141-153.
[36]Tian F,Zhu Z F,Zhang B S,et al..Fine mapping of a quantitative trait locus for grain number per panicle from wild rice(Oryza rufipogon Griff.) [J].Theor.Appl.Genet.,2006,113(4):619-629.
[37]Yoshida S,Ik egami M,Kuz e J,et al..QTL analysis for plant and grain characters of sake brewing rice using a doubled haploid population[J].Breeding Sci,2002,52:309-317.
[38]Chang T T,Somrith B.Grain quality traits of a candidate rice variety PB-95 [J].Asian J.Plant Sci.,2003,2(6):483-484.
[39]Weng J, Gu S, Wan X, et al.. Isolation and initial characterization of GW5,a major QTL associated with rice grain width and weight[J].Cell Res.,2008,18(12):1199-1209.
[40]Song X J,Huang W,Lin H X,et al..A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J].Nat.Genet.,2007,39(5):623-630.
[41]Mao H L,Sun S Y,Zhang Q F,et al..Linking differential domain functions of the GS3 protein to natural variation of grain size in rice[J].Proc.Natl.Acad.Sci.USA,2010,107(45):19579-19584.

