赖氨酸特异性组蛋白去甲基化酶1与白血病的研究进展
郭艺迪,梁冬雪,李悦,李校堃,付学奇,胡鑫,
吉林大学 a.生命科学学院;b.白求恩医学院;吉林 长春 130012
表观遗传学指在基因的DNA序列没有发生改变的情况下,基因功能发生了可遗传的变化,并最终导致表型的变化,它通过DNA的甲基化、组蛋白修饰、染色质重塑和非编码RNA调控等4种方式来控制基因表达。
组蛋白修饰是细胞内基因表达的重要调控机制,真核细胞中,核小体是染色质的基本结构,2对组蛋白H3及H4形成四聚体,两侧各有一个组蛋白H2A、H2B紧密结合形成的二聚体,从而促成核心组蛋白八聚体。核小体是由长146 bp的DNA双螺旋结构缠绕组蛋白八聚体1.75圈形成的。在真核生物中,染色质由DNA、组蛋白、非组蛋白及少量RNA组成,其中组蛋白是真核细胞中特有的成分,富含带正电荷的精氨酸和赖氨酸,可以与带负电荷的DNA分子紧密结合。
组蛋白修饰包括乙酰化、甲基化、磷酸化和泛素化。甲基化是组蛋白修饰的重要方式,组蛋白甲基化位点多位于H3、H4的赖氨酸和精氨酸残基上。其中,精氨酸可以被单甲基化和二甲基化,而赖氨酸可以被单甲基化、二甲基化和三甲基化。组蛋白H3和H4中共有5个精氨酸残基(H3R2、H3R8、H3R17、H3R26和H4R3)可被PRMT家族组蛋白甲基化转移酶甲基化,组蛋白精氨酸甲基化既参与激活基因转录又参与抑制基因转录[1]。组蛋白共有6个赖氨酸残基(H3K4、H3K9、H3K27、H3K36、H3K79、H4K20)可被甲基化,其中H3K4、H3K36和H3K79的甲基化通常与基因转录的激活相关,而H3K9、H3K27和H4K20的甲基化通常和基因转录的抑制相关[2]。
自2000年Jenuwein等报道第一个组蛋白赖氨酸甲基化转移酶Suv39H1以来,组蛋白甲基化研究受到广泛关注[3];而对于组蛋白甲基化修饰的另一方面,去甲基化的研究始于2004年Shi等[4]对赖氨酸特异性组蛋白去甲基化酶1(lysine specific demethyl⁃ase 1,LSD1)的报道。
1 LSD1的发现
很多组蛋白修饰都是可逆的,其中以赖氨酸甲基化尤为重要,它与DNA的甲基化密切相关。但是在发现组蛋白赖氨酸甲基化后的几十年里,人们一直认为组蛋白赖氨酸甲基化是不可逆的,是一个稳定的组蛋白标记。尽管蛋白精氨酸脱亚氨酶4(pro⁃tein-arginine deiminase type-4,PADI4)可以将甲基化的精氨酸转换为瓜氨酸,但由于并非移除甲基,故PADI4并不是严格意义上的去甲基化酶[5-6]。直到2004年Shi等发现了第一个组蛋白LSD1,才明确了组蛋白的甲基化修饰是一种动态可逆的过程。LSD1又称KDM1、KIAA060、pl10b、BHC110、NPAO,是组蛋白H3-K4的特异性脱甲基酶[4],稳定存在于一些组蛋白去乙酰化酶复合物中[7-11]。
最近,有研究发现JmjC蛋白家族具有去甲基化酶活性并具有广泛的作用,能够对H3K4、H3K9、H3K36等多个位点进行去甲基化。目前研究表明,JmjC蛋白的3个亚家族JHDM1、JHDM2、JMJD2中的许多成员都具有组蛋白赖氨酸去甲基化酶活性。JmjC结构域属于金属酶中的Cupin超家族,在功能上高度保守,并且其完整性决定了JmjC蛋白的去甲基化酶活性[12]。但仅有JmjC结构也不能完成一个去甲基化催化过程,还需要有另外不同的结构域共同作用才能发挥去甲基化的催化反应,比如锌指结构域、Tudor结构域。并且,其中一些蛋白不仅具有组蛋白赖氨酸去甲基化酶活性,还能特异性结合H3K4三甲基化和H4K20三甲基化位点发挥作用[7]。
2 LSD1的结构及其催化原理
LSD1含有一个N端SWIRM结构域、一个Tower结构域和一个C端胺氧化酶结构域。很多与染色体相互作用的蛋白都含有SWIRM结构域,而Tower结构域是由从C端胺氧化酶结构域向外伸出的2个平行结构构成,因此Tower结构域把胺氧化酶结构域分成2个部分,即FAD结合结构域和催化活性中心。在三级结构中,胺氧化酶结构域的这2个部分仍然紧密结合在一起,形成一个完整的具有催化活性的结构域。这一特殊的结构对于LSD1的功能有着重要的作用。Tower结构域与CoREST的2个SANT结构域中间一个很长的连接α螺旋相互作用,使LSD1和CoREST形成紧密的复合物,进而使核小体去甲基化,Tower结构域的缺失突变会导致LSD1丧失活性(图1)。
结构同源分析显示,LSD1是核内黄素依赖的单胺氧化酶,LSD1只能使H3K4的单甲基化及双甲基化去甲基化,对三甲基化的H3K4没有作用,因为三甲基化后的N上没有多余的质子,也就没有胺氧化酶的作用位点,这一方面符合单胺氧化酶的作用机制,同时也说明存在其他类型的酶催化三甲基化H3K4的去甲基作用。研究发现,包含JmjC结构域的组蛋白去甲基化酶(JmjC domain-containin his⁃tone demethylase,JHDM)家族可以催化组蛋白三甲基(me3)的去甲基化修饰。
LSD1与底物反应时FAD从甲基化的组蛋白赖氨酸得到质子,生成FADH2,甲基化的赖氨酸失去质子生成亚胺中间物,进而FADH2被氧化生成FAD和H2O2,亚胺中间物加水后生成胺基和甲醛。LSD1催化氧化反应时胺基底物上必须有一个质子,因此只能催化单甲基化和二甲基化的赖氨酸底物去甲基化。

图1 LSD1结构域

图2 组蛋白去甲基化反应机理A:LSD1的催化反应机理;B:含有JmjC催化结构域的组蛋白去甲基化酶的催化反应机理
3 LSD1的调控
实验表明,在体外组蛋白赖氨酸去甲基化酶是有活性的,即仅仅是酶与底物的简单结合就能够充分完成去甲基化反应,这就提高了酶在活体内的调控,并且有效避免了可能的不正确的去甲基化反应发生,而其调控的方法之一正是在去甲基化酶基因表达水平上进行调控。事实上,许多去甲基化酶都能够限制胚胎发展及其在成人期间表达模式,以及对于环境刺激的应对能力。
一些去甲基化酶似乎在一个特定的生物过程中对生物体起着保护作用。例如,从裂殖酵母到小鼠等广泛的生物研究试验表明,在减数分裂中去甲基化酶LSD1在H3K4二甲基化/单甲基化中起着支持作用。哺乳动物中,LSD1在小鼠睾丸及相关组织中有较高水平的表达,而这些组织中H3K4的二甲基化表达水平相对较低[13]。果蝇的LSD1同源突变体导致性别特异性胚胎致死及存活者的不育(主要发生在雌性中),这可能是卵巢发育缺陷所致[14]。H3K4二甲基化水平在杂合子雌性的原始生殖干细胞中有较高的表达水平,而且体外实验表明果蝇的LSD1蛋白质对于H3K4二甲基化/单甲基化具有去甲基化酶活性[15],并且其在哺乳动物和果蝇之间均具有保守的酶特异性。最后,裂殖酵母LSD1同源突变体对于孢子是单倍不足的。总之,这些数据表明LSD1在减数分裂和生殖细胞中起到支持保护作用。
生化研究表明,LSD1存在于包含许多已知或假定的DNA结合转录因子的蛋白质复合体中。这些DNA结合因子包括转录起始因子TFⅡ-Ⅰ[16]、E-钙黏着蛋白启动子结合因子ZEB1/2[8]和ZNF217[17]及雄性激素受体(AR)[18],这表明DNA结合因子在某些特殊位点上对于LSD1的富集和稳定起重要作用。而有证据表明,转录因子还能调节去甲基化酶的活性以及改变它们在转录调控中的作用。最近的研究表明,LSD1也存在于包含延伸因子RNA聚合酶ELL、pTEFb、AF4和AFF4的转录延伸复合体中[19]。
有研究表明,在体促素和垂体催乳素细胞的Gh启动子中发现了2种形式的LSD1复合体[20]。在体促素细胞中,积极转录的Gh被LSD1-WDR5复合体所占据,在垂体催乳素细胞中,Gh启动子被LSD1-CoREST-CtBP辅助抑制复合物所占据,从而导致转录抑制。由于ZEB1的诱导与Gh的抑制相关,推测ZEB1直接结合到CoREST-CtBP与LSD1的复合体上,复合物的总体结构转变为抑制模型。在对AR敏感的启动子PSA的研究中也发现了相似的模型,在PSA上LSD1与KDM4C/JMJD2C相互结合约束,但是当AR结合时去甲基化的H3K9二甲基化/单甲基化被释放出来[18,21-22]。这些研究表明LSD1去甲基化酶活性可以通过DNA结合因子来调控。在这些情况下,最初的靶基因以及随后通过去甲基化酶结合在靶基因位点上的修饰可能是由于一些未知的DNA结合因子、RNA因子、局部的染色质环境或一些还不明确的机制。
4 LSD1的功能
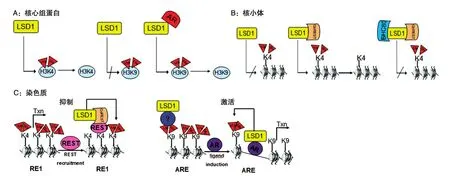
图3 LSD1协同相关蛋白质辅因子特异性调控去甲基化
LSD1作为多个转录调节复合物的组分参与转录调控,其去甲基化酶活性也受复合物中其他分子的调控。如LSD1能使组蛋白上的H3K4去甲基化,单独LSD1作用不能使H3K9去甲基化[4],然而与雄激素受体结合后LSD1也可催化H3K9去甲基化(图3A)[18]。但是不能以核小体作为底物,使核小体去甲基化,只有当LSD1与CoREST结合后才能以核小体为底物使之去甲基化[23-24],而BHC80抑制LSD1-CoREST复合物介导的核小体去甲基化作用,从而调控基因转录(图3B)[23]。在体内,LSD1存在于组蛋白去乙酰化酶复合物中,这一复合物中还包含其他蛋白如CoREST、BHC80、HDAC1/2、BRAF等,这些蛋白能调控LSD1的活性[10,24]。CoREST为LSD1发挥活性提供了一座桥梁,使LSD1能接触底物核小体组蛋白,并且还能稳定核内LSD1[23,25]。一方面组蛋白H3中赖氨酸4的去甲基化与染色质的浓缩和转录抑制相关;另一方面,组蛋白H3的赖氨酸9去甲基化与染色质开放构象和基因表达相关(图3C)。LSD1与雄激素受体结合后介导的H3K9与基因转录激活相关[18],但雄激素受体元件是如何与配体结合,通过哪些未知因子共同发挥作用的详细机制尚不清楚。
LSD1、CoREST和BHC80通常存在于同一个组蛋白去乙酰化酶复合物中。LSD1对低乙酰化组蛋白的去甲基化作用明显高于高乙酰化的组蛋白,说明转录抑制复合物中的组蛋白去乙酰酶有利于LSD1发挥去甲基酶活性。如在LSD1/CoREST复合物中,HDAC1/2催化H3K9去乙酰化,使CoREST结合于去乙酰化位点。在CoREST存在的情况下,LSD1催化H3K4me2去甲基化,而后LSD1另一个相互作用的蛋白BHC80结合于H3K4me0位点,阻止H3K4的再次甲基化作用(图4)[26]。然而,BHC80与LSD1相互作用的机制尚不清楚。
5 LSD1与白血病
在正常的造血系统生成过程中,各种转录因子通常以组合的方式对正常细胞环境中的特定靶基因发挥调节作用,造血干细胞经历了一系列的基因表达的动态调控,在促进其分化为成熟红细胞的同时也抑制了一些抑分化基因的表达。转录因子所形成的调控网络对于维持正常的造血细胞分化起到了至关重要的作用,当这种动态的调控失衡,正常的造血细胞分化过程发生紊乱,就诱发了白血病的发生。细胞分化发生异常,细胞表面压力发生改变,丧失对分化刺激因素产生反应的能力,分化障碍的未成熟红细胞不断产生,无限增殖,并大量涌入外周血液中,形成白血病。白血病的发生机制与基因层面上的调控及表观遗传学修饰的异常均具有密不可分的联系[27]。
LSD1在恶性造血系统中高表达,对于造血系统的分化具有重要的调控作用[28]。在正常的造血系统分化过程中,造血干细胞分化为早期前祖细胞进而最终分化成成熟红细胞,任何时期所产生的突变均有可能导致恶性增殖的发生发展。TAL1是早期造血干细胞特异性调控因子之一,它的异常表达会导致T细胞白血病的产生。Hu等[29]于2009年首次在TAL1复合体中分离出由LSD1、HDAC1/2、Co-REST组成的去甲基化酶复合体,经大量实验证明LSD1复合物通过表观遗传修饰作用对红细胞分化的相关基因表达发挥动态调控功能,在造血系统的生成及红细胞分化过程中具有重要意义,并在白血病的发生发展过程中充当重要角色。LSD1蛋白的过量表达可促进红细胞增殖,同时抑制红细胞分化。LSD1通过与TAL1结合调控TAL1靶基因启动子活性,改变其位点的甲基化修饰水平,从而调控机体造血系统的发生发展、红细胞的增殖分化。LSD1的表达沉默抑制红细胞分化,而过量表达LSD1蛋白同样也抑制红细胞的分化,这说明LSD1在机体造血系统中发挥重要作用,无论是未分化的红细胞还是已分化的红细胞都需要LSD1蛋白的存在,也许LSD1可通过与红细胞特异性转录因子如TAL1或Gfi1b相结合调控红细胞的分化[29-30]。
前期工作的另一方面根据大量的实验结果大胆假设了一个LSD1和TAL1的分子机制模型,如图5所示。当TAL1与LSD1、HDAC1蛋白抑制复合体相结合时,染色体结构上组蛋白发生去甲基化修饰,LSD1复合体可抑制TAL1介导的转录活性,并调控红细胞分化中特异性蛋白的表达,从而抑制红细胞的分化。但随着红细胞的分化,TAL1与LSD1的相互作用减少并发生转换,又可与其他转录激活蛋白质相互作用,如p300、PCAF、hSET1,并重新修饰组蛋白,导致染色体结构开放,提高转录水平,促进红细胞的增殖和分化。所以,LSD1以表观遗传修饰的方式调控TAL1的功能和机体造血系统。也正是TAL1-LSD1中蛋白质-蛋白质之间的相互作用,调节转录及基因表达,从而在机体造血系统及红细胞分化中发挥功能。
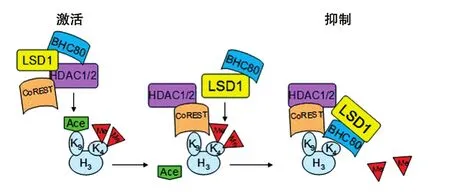
图4 LSD1介导的H3K4去甲基化调控的表观遗传机制
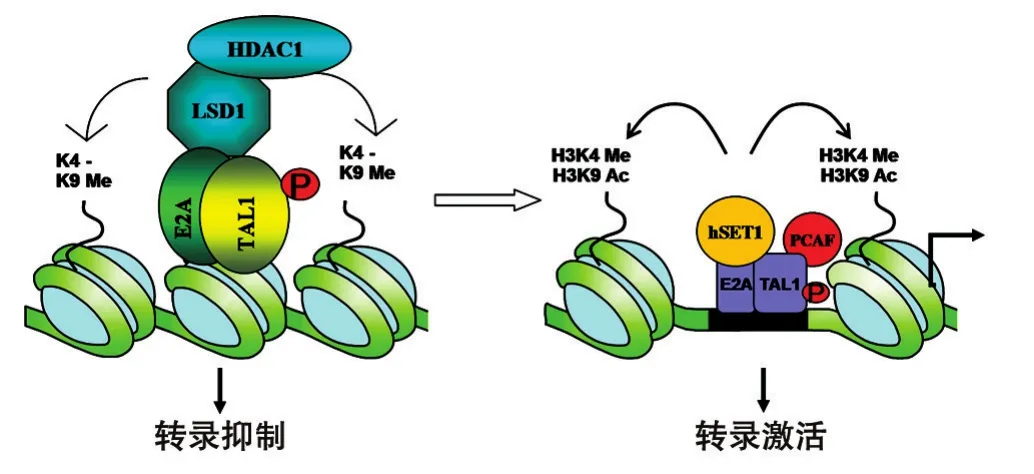
图5 LSD1调控红细胞分化及白血病发生的分子模型
Notch1信号的异常激活会导致致癌性转化并最终导致T细胞急性淋巴白血病(T-ALL)的发生,最近发现LSD1参与到了Notch1的多功能复合体中。在T-ALL中,LSD1所介导的表观遗传学修饰作用对于Notch1靶基因的激活与抑制发挥着非常重要的动态调控功能。当有Notch1的协同作用时,LSD1特异性结合于H3K9me2位点发挥去甲基化作用进而激活靶基因的表达,相反,当Notch1缺失后,这种去甲基化酶通过催化H3K4me2的去甲基化达到抑制转录的目的。LSD1的这种动态调控机制对于T-ALL的发生发展具有重要意义[31]。
LSD1在多种人类急性髓系白血病(AML)中列于具有高度表达的基因中的前5%[32]。Harris等[33]通过建立患白血病的小鼠模型,确定了LSD1在AML生成过程中所发挥的重要调控作用,建立了LSD1过表达和AML白血病之间的联系。研究发现,LSD1的敲除会使AML细胞显著丧失白血病细胞无限增殖分化的潜能,不能形成克隆,在小鼠模型中不会导致白血病的发生。由LSD1高表达介导的表观遗传修饰所导致的基因沉默,与疾病的发生发展密切相关,LSD1可能通过直接或间接的方式调控造血系统中一些重要转录因子和染色质重塑相关酶的表达,很可能通过调控一系列基因表达进而激活MLLAF9白血病相关癌基因。CHIP-Seq实验显示,LSD1沉默后,H3K4me2水平的增加是MLL-AF9启动子上可以检测到的惟一变化。LSD1抑制剂会导致被异常沉默的抑癌基因获得激活从而重新表达[34]。这些结果无一不证实了在AML白血病中,LSD1所发挥的去甲基化功能和白血病相关的致癌基因具有密不可分的关系。鉴于这些重要发现,LSD1在调控基因表达及MLL细胞分化过程中均发挥着重要作用,但LSD1在靶基因位点作用机制以及募集的蛋白复合体还有待更深一步的探究。
综上所述,LSD1在调控致癌基因的表达[33]及造血系统生成、红细胞分化过程[29]中发挥着重要作用,因而这一重要的表观遗传学调节因子可能成为白血病治疗中的重要靶标。
6 讨论
目前对于LSD1及组蛋白去甲基化酶有比较深入的研究,但仍有许多问题有待解决,今后的研究将主要关注LSD1与白血病的关系。TAL1可以结合一些辅助抑制物,如 mSin3A、HDAC及ETO-2[35-37],从而在造血过程中发挥转录抑制作用。LSD1是赖氨酸特异性的去甲基化酶,它可以特异去除单甲基或双甲基的H3K4。CoREST和HDAC1/2能够极大地增强LSD1的去甲基化酶活性[10,23-24,38]。
CoREST、HDAC1/2及其他未知蛋白能与LSD1相结合,这对于LSD1的酶活力以及转录抑制是必须的[39-40]。但这些复合物在正常或异常的环境中对于TAL1和LSD1的调控有什么不同,目前还不清楚。我们认为,在LSD1与其相关复合物之间的蛋白与蛋白的相互作用控制着它的酶活性及底物特异性,并且很有可能在TAL1/LSD1复合体中存在某些未知的成分,而正是这些成分控制着TAL1或LSD1在造血过程中的作用。敲除LSD1会造成鼠红白血病细胞MEL细胞及鼠类肝细胞中造血分化的紊乱,这表明广泛表达的LSD1可能是通过与组织特异性的转录因子相互作用控制着组织特异性的造血分化过程。因此,LSD1与造血系统特异性的抑制因子Gfi-1b相互作用,共同影响红细胞、巨核细胞及粒性白细胞的分化[30]。有研究表明[30]LSD1通过造血特异性转录因子的诱导来发挥其抑制活性,并且控制造血限制性基因的表达,例如TAL1和Gfi-1b。一系列证据表明,TAL1和Gfi-1b能够相互作用,并且可能共同存在于一条转录途径中。另外,辅助抑制物ETO-2能够影响红细胞中TAL1与Gfi-1b之间的相互作用,但目前并不确定ETO-2与TAL1、Gfi-1b及LSD1是否存在于相同的复合体中。
有报道表明,在红细胞分化过程中TAL1能够动态地与激活因子或抑制因子相互作用。我们在未分化的MEL细胞中发现TAL1与LSD1相结合并具有去甲基酶活性,但在分化后第1天这种活性就消失了,而在分化的后期复合体的活性又得到恢复[29]。对于在红细胞分化过程中TAL1/LSD1的动态相互作用,有2种可能的解释。一是,在祖细胞中,LSD1拥有限制这些细胞分化的能力;而细胞一旦分化,LSD1的功能即转变为抑制TAL1的靶基因,这些靶基因能够促进细胞分化。在这种观点的支持下,敲除LSD1后,在细胞分化过程中TAL1启动子p4.2位点上H3K4甲基化及组蛋白乙酰化水平增高。另外一种可能性是,LSD1蛋白复合体改变了其辅酶的组成,这有可能改变其酶的特异性。例如,当雄激素受体与LSD1结合,其将会通过去甲基作用抑制单甲基或双甲基的H3K9的标记改变LSD1的底物特异性,这会促进AR依赖的转录活性。因此,LSD1通过敲除某些特定的表观遗传标记进而产生一种紧凑或宽松的染色质环境,从而能够抑制或激活转录。在这种观点的支持下,LSD1应该在造血系统中具有双重功能,并且我们在Jurkat细胞中发现TAL1与LSD1形成的2种复合体。二是,LSD1与TAL1具有直接相互作用,且结合结构域为TAL1的142~185残基,TAL1的该结构域中含有一个可以被蛋白质激酶A(PKA)磷酸化的氨基酸残基Ser172,这个位点的磷酸化作用可以影响TAL的转录调控作用。有研究表明TAL1与LSD1之间的动态结合是通过PKA介导的TAL1 Ser172位点的磷酸化作用完成的,PKA信号通路可以调控红细胞分化过程和T细胞白血病生成中TAL1与LSD1的相互作用,TAL1 Ser172位点的磷酸化能够破坏TAL1与组蛋白LSD1间的结合,导致红细胞分化过程和T细胞白血病生成中某些TAL1靶基因的激活。TAL1可以被蛋白质激酶PKA在体内或体外磷酸化,尽管这一磷酸化作用不会影响TAL1的细胞内定位及其与E2A蛋白质形成二聚体的能力,但能够改变TAL1结合某些DNA的能力,从而影响其转录活性。
LSD1在造血过程中发挥怎样的生物学作用呢?LSD1通过调节激活或抑制染色质结构域来调控基因表达。它可能通过干扰正常细胞生长分化所需要的正常基因的表达模式,从而与人类疾病密切相关。TAL1的激活与许多T-ALL患者密切相关。在T细胞白血病中,TAL1抑制E2A/HEB的转录活性,并且在正常的胸腺细胞进行大量细胞分裂阶段中干扰细胞周期进程。因此,LSD1结合TAL1从而抑制其转录活性的机制可能是,LSD1通过结合TAL1而抑制E2A/HEB的活性,进而导致白血病的发生。
随着对组蛋白修饰研究的逐步深入,几种组蛋白修饰酶如组蛋白去乙酰化酶已成为肿瘤治疗的靶蛋白。已有许多研究表明,组蛋白甲基化和去甲基化间的平衡与人类包括白血病在内的多种疾病紧密相关。在前列腺癌[41]、成神经细胞瘤[42]、非小细胞性肺癌[43]、ER阴性乳腺癌[44]及膀胱癌[45]中均有LSD1的高表达,LSD1的抑制会导致这些肿瘤细胞生长障碍,相反其过表达会通过某种表观遗传修饰途径导致肿瘤的发生。近来研究发现,LSD1在白血病细胞中同样过表达[46]。以反苯环丙胺衍生物为例的多种LSD1小分子抑制剂被开发出来,其可以显著抑制白血病细胞分化,并具有增强抗白血病药物疗效的作用[47]。LSD1有望成为白血病表观遗传修饰治疗的新靶点,为白血病药物开发提供了新的方向,以期为提高患者治愈率,延长生存期提供依据。更加深入地探究LSD1与白血病发生的相关机制,具有十分重要的临床意义。
[1] Wysocka J,Allis C D,Coonrod S.Histone arginine methyla⁃tion and its dynamic regulation[J].Front Biosci,2006,11:344-355.
[2] Martin C,Zhang Y.The diverse functions of histone lysine methylation[J].Nat Rev Mol Cell Biol,2005,6(11):838-849.
[3] Rea S,Eisenhaber F,O'Carroll D,et al.Regulation of chro⁃matin structure by site-specific histone H3 methyltransferases[J].Nature,2000,406(6796):593-599.
[4] Shi Y,Lan F,Matson C,et al.Histone demethylation mediat⁃ed by the nuclear amine oxidase homolog LSD1[J].Cell,2004,119(7):941-953.
[5] Cuthbert G L,Daujat S,Snowden A W,et al.Histone deimi⁃nation antagonizesargininemethylation[J].Cell,2004,118(5):545-553.
[6] Wang Y,Wysocka J,Sayegh J,et al.Human PAD4 regulates histone arginine methylation levelsvia demethylimination[J].Science,2004,306(5694):279-283.
[7] Tong J K,Hassig C A,Schnitzler G R,et al.Chromatin deacetylation by an ATP-dependent nucleosome remodelling complex[J].Nature,1998,395(6705):917-921.
[8] Hakimi M A,Dong Y,Lane W S,et al.A candidate X-linked mental retardation gene is a component of a new fami⁃ly of histone deacetylase-containing complexes[J].J Biol Chem,2003,278(9):7234-7239.
[9] You A,Tong J K,Grozinger C M,et al.CoREST is an inte⁃gralcomponentoftheCoREST-human histonedeacetylase complex[J].Proc Natl Acad Sci USA,2001,98(4):1454-1458.
[10]Humphrey G W,Wang Y,Russanova V R,et al.Stable his⁃tone deacetylase complexes distinguished by the presence of SANT domain proteins CoREST/kiaa0071 and Mta-L1[J].J Bi⁃ol Chem,2001,276(9):6817-6824.
[11]Shi Y,Sawada J,Sui G,et al.Coordinated histone modifica⁃tions mediated by a CtBP co-repressor complex[J].Nature,2003,422(6933):735-738.
[12]Tsukada Y,Fang J,Erdjument-Bromage H,et al.Histone de⁃methylation by a family of JmjC domain-containing proteins[J].Nature,2006,439(7078):811-816.
[13]Huang Y,Fang J,Bedford M T,et al.Recognition of histone H3 lysine-4 methylation by the double tudor domain of JM⁃JD2A[J].Science,2006,312(5774):748-751.
[14]Godmann M,Auger V,Ferraroni-Aguiar V,et al.Dynamic regulation of histone H3 methylation at lysine 4 in mammali⁃an spermatogenesis[J].Biol Reprod,2007,77(5):754-764.
[15]Di Stefano L,Ji J Y,Moon N S,et al.Mutation of Drosophi⁃la Lsd1 disrupts H3-K4 methylation,resulting in tissue-spe⁃cific defects during development[J].Curr Biol,2007,17(9):808-812.
[16]Rudolph T,Yonezawa M,Lein S,et al.Heterochromatin for⁃mation in drosophila is initiated through active removal of H3K4 methylation by the LSD1 homolog SU(VAR)3-3[J].Mol Cell,2007,26(1):103-115.
[17]Cowger J J,Zhao Q,Isovic M,et al.Biochemical characteriza⁃tion of the zinc-finger protein 217 transcriptional repressor complex:identification of a ZNF217 consensus recognition se⁃quence[J].Oncogene,2007,26(23):3378-3386.
[18]Metzger E,Wissmann M,Yin N,et al.LSD1 demethylates re⁃pressive histone marks to promote androgen-receptor-depen⁃dent transcription[J].Nature,2005,437(7057):436-439.
[19]Biswas D,Milne T A,Basrur V,et al.Function of leukemo⁃genic mixed lineage leukemia 1(MLL)fusion proteins through distinctpartnerprotein complexes[J].Proc NatlAcad Sci USA,2011,108(38):15751-15756.
[20]Wang J,Scully K,Zhu X,et al.Opposing LSD1 complexes function in developmental gene activation and repression pro⁃grammes[J].Nature,2007,446(7138):882-887.
[21]Yamane K,Toumazou C,Tsukada Y,et al.JHDM2A,a JmjC-containing H3K9 demethylase,facilitates transcription activa⁃tion by androgen receptor[J].Cell,2006,125(3):483-495.
[22]Wissmann M,Yin N,Muller J M,et al.Cooperative demethyl⁃ation by JMJD2C and LSD1 promotes androgen receptor-de⁃pendent gene expression[J].Nat Cell Biol,2007,9(3):347-353.
[23]Shi Y,Matson C,Lan F,et al.Regulation of LSD1 histone demethylase activity by itsassociated factors[J].MolCell,2005,19(6):857-864.
[24]Lee M G,Wynder C,Cooch N,et al.An essential role for Corest in nucleosomal histone 3 lysine 4 demethylation[J].Na⁃ture,2005,437(7057):432-435.
[25]Lee M G,Wynder C,Bochar D A,et al.Functional interplay between histone demethylase and deacetylase enzymes[J].Mol Cell Biol,2006,26(17):6395-6402.
[26]Lan F,Nottke A C,Shi Y.Mechanisms involved in the regu⁃lation of histone lysine demethylases[J].Curr Opin Cell Biol,2008,20(3):316-325.
[27]Vu L P,Luciani L,Nimer S D,et al.Histone-modifying en⁃zymes:their role in the pathogenesis of acute leukemia and theirtherapeutic potentialInt[J].JHematol,2013,97(2):198-209.
[28]Lynch J T,Harris W J,Somervaille T C,et al.LSD1 inhibi⁃tion:a therapeutic strategy in cancer[J]?Expert Opin Ther Targets,2012,16(12):1239-1249.
[29]Hu X,Li X,Valverde K,et al.LSD1-mediated epigenetic modification is required for TAL1 function and hematopoiesis[J].Proc Natl Acad Sci USA,2009,106(25):10141-10146.
[30]Saleque S,Kim J,Rooke H M,et al.Epigenetic regulation of hematopoietic differentiation by Gfi-1 and Gfi-1b is medi⁃ated by the cofactors CoREST and LSD1[J].Mol Cell,2007,27(4):562-572.
[31]Yatim A,Benne C,Sobhian B,et al.NOTCH1 nuclear inter⁃actome revealskey regulatorsofitstranscriptionalactivity and oncogenic function[J].Mol Cell,2012,48(3):445-458.
[32]Goardon N,Marchi E,Atzberger A,et al.Coexistence of LMPP-like and GMP-like leukemia stem cells in acute my⁃eloid leukemia[J].Cancer Cell,2011,19(1):138-152.
[33]Harris W J,Huang X,Lynch J T,et al.The histone demeth⁃ylase KDM1A sustains the oncogenic potential of MLL-AF9 leukemia stem cells[J].Cancer Cell,2012,21(4):473-487.
[34]Sharma S K,Wu Y,Steinbergs N,et al.(Bis)urea and(Bis)thiourea Inhibitors of Lysine-Specific Demethylase 1 as Epi⁃genetic Modulators[J].J Med Chem,2010,53(14):5197-5212.
[35]Schuh A H,Tipping A J,Clark A J,et al.ETO-2 associates with SCL in erythroid cells and megakaryocytes and provides repressor function in erythropoiesis[J].Mol Cell Biol,2005,25(23):10235-10250.
[36]Goardon N,Lambert J A,Rodriguez P,et al.ETO2 coordi⁃nates cellularproliferation and differentiation during erythropoie⁃sis[J].EMBO J,2006,25(2):357-366.
[37]Huang S,Qiu Y,Stein R,et al.p300 functions as a tran⁃scriptional coactivator for the Tal1/SCL oncoprotein[J].Onco⁃gene,1999,18(35):4958-4967.
[38]Li Y,Deng C,Hu X,et al.Dynamic interaction between TAL1 oncoprotein and LSD1 regulates TAL1 function in hema⁃topoiesis and leukemogenesis[J].Oncogene,2012,31:5007-5018.
[39]Huang Y,Vasilatos S N,Boric L,et al.Inhibitors of histone demethylation and histone deacetylation cooperate in regulat⁃ing gene expression and inhibiting growth in human breast cancer cells[J].Breast Cancer Res Treat,2012,131(3):777-789.
[40]Charles T F,Oliver M D,Larissa L,et al.Lysine-specific de⁃methylase 1 regulates the embryonic transcriptome and CoR⁃EST stability[J].Mol Cell Biol,2010,30(20):4851-4863.
[41]Kahl P,Gullotti L,Heukamp L C,et al.Androgen receptor coactivatorslysine-specifichistonedemethylase 1 and four and a half LIM domain protein 2 predict risk of prostate can⁃cer recurrence[J].Cancer Res,2006,66(23):11341-11347.
[42]Schulte J H,Lim S,Schramm A,et al.Lysine-specific de⁃methylase 1 is strongly expressed in poorly differentiated neu⁃roblastoma:implications for therapy[J].Cancer Res,2009,69(5):2065-2071.
[43]Lv T,Yuan D,Miao X,et al.Over-expression of LSD1 pro⁃motes proliferation,migration and invasion in non-small cell lung cancer[J].PLoS One,2012,7(4):e35065.
[44]Lim S,Janzer A,Becker A,et al.Lysine-specific demethyl⁃ase 1(LSD1)is highly expressed in ER-negative breast can⁃cers and a biomarker predicting aggressive biology[J].Carcino⁃genesis,2010,31(3):512-520.
[45]Hayami S,Kelly J D,Cho H S,et al.Overexpression of LSD1 contributes to human carcinogenesis through chromatin regulation in variouscancers[J].IntJCancer,2011,128(3):574-586.
[46]Lokken A A,Zeleznik-Le N J.Breaking the LSD1/KDM1A addiction:therapeutic targeting of the epigenetic modifier in AML[J].Cancer Cell,2012,21(4):451-453.
[47]Binda C,Valente S,Romanenghi M,et al.Biochemical,struc⁃tural,and biological evaluation of tranylcypromine derivatives as inhibitors of histone demethylases LSD1 and LSD2[J].J Am Chem Soc,2010,132(19):6827-6833.

