建鲤ODC1基因型与增重的相关性分析
李红霞 李建林 唐永凯 俞菊华
(中国水产科学研究院淡水渔业研究中心, 农业部淡水渔业和种质资源利用重点实验室, 无锡 214081)
建鲤ODC1基因型与增重的相关性分析
李红霞 李建林 唐永凯 俞菊华
(中国水产科学研究院淡水渔业研究中心, 农业部淡水渔业和种质资源利用重点实验室, 无锡 214081)
构建了建鲤(Cyprinus carpio var. jian) 鸟氨酸脱羧酶 (Ornithine decarboxylase, ODC) jlODC1a基因上6个和jlODC1b基因上4个SNP位点的PCR-RFLP方法, 检测了这10个位点在12个家系约900尾建鲤选育群体中的基因型, 各位点的基因型频率存在差异, 最小等位基因频率 (MAF)为0.14—0.48。各位点不同基因型与增重相关分析结果, 其中 7个 SNPs与建鲤增重显性相关的位点, ODC1s基因上与雌鱼增重相关的SNP位点(7个)较雄鱼(4个)多。标记富集结果表明富集与建鲤增重相关的优势基因型的SNP个数越多的个体增重速度越快SNP个数越多的个体增重速度越快, 富集 4个的平均增重显著快于富集0—3的个体增重,且比0标记的快约14%, 这反映出生长为数量性状。进一步对所检测位点进行双倍型分析, 结果显示具有四个优势基因型且全部杂合优势基因型的4567 (XXXXXXACCTCTCT)组的增重最快, 比0优势基因型的增重快达 26.6%, 可以考虑用于今后的快增长建鲤的选育计划中。此外, 双倍型分析结果还表明, 不同位点之间可能存在或颉抗或协同的互作, 如1和4之间存在拮抗关系, 因此在今后的选育计划中, 在考虑标记富集的情况下还应考虑标记之间的关系。宜在选择互为协同作用优势基因型的前提下, 富集尽可能多的SNP标记。
建鲤; 鸟氨酸脱羧酶; 基因型; SNP-增重相关分析
鸟氨酸脱羧酶(Ornithine decarboxylase, ODC)为多胺合成的限速酶, 催化鸟氨酸脱羧生成腐胺, 腐胺再相继生成精脒和精胺, 腐胺、精脒和精胺统称多胺, 多胺是细胞增殖、分化的关键物质, 因此, ODC及多胺对细胞的生长、机体的生长具有重要的调节作用。研究表明, 鸟氨酸脱羧酶的活性在生长旺盛的组织内明显高于生长缓慢的细胞和组织, 可以作为细胞增殖的指标[1,2]。在养殖业中, ODC就被作为与生长密切相关的候选基因, 肌肉鸟氨酸脱羧酶活性被认为是与蛋白合成相关的生化指标。鸡(Gallus domestiaus)的鸟氨酸脱羧酶基因位于生长QTL区内, 该基因启动子上的多态性与生长和躯体框架特征显著相关[3]。在水生动物的研究中, 生长快的大西洋鲑(Salmo salar)幼鱼背轴肌肉鸟氨酸脱羧酶活性明显高于生长慢的个体[4], 能作为特定生长率的指标[5]。本实验通过测定6个个体基因序列, 在建鲤的2个ODC1基因上构建了10个SNPs位点的PCR-RFLP检测方法, 分析了10个位点在12个家系建鲤群体中的分布, 以及不同基因型与雌、雄及各自鱼种阶段、成鱼阶段增重的相关性, 筛选与生长性状相关的分子标记, 为分子辅助育种奠定基础。
1 材料与方法
1.1 材料
实验鱼为中国水产科学研究院淡水渔业研究中心宜兴养殖基地繁育的建鲤选育群体, 12家系, 繁殖后分家系于网箱养殖, 约1个月, 每家系PIT标记约90尾, 测初重等数据后混合于同一池塘养殖。养至当年底扫描标记并测定体重等数据, 减去初重计为鱼种阶段增重, 第二年继续养殖至11月份, 测定体重等数据, 减去第二次测定的体重计为成鱼阶段增重。养殖过程中存在标记丢失、鱼死亡等意外事件, 最终实验鱼约942尾。
1.2 实验方法
DNA抽提 实验鱼尾静脉采血, 蛋白酶K消化过夜, 传统酚-氯仿法提取基因组DNA[6], DNA完整性使用琼脂糖凝胶电泳检验, 浓度使用紫外分光光度计测定, A260/280一般为1.8左右。TE将DNA样品稀释成50—100 ng/μL备用。
多态位点筛选和检测 根据本实验室分离到的CcODC1s基因设计引物[7], 测定6尾建鲤ODC1s序列, 使用 Clustal W[8]软件比对, 以同一位点不同碱基出现比例大于 1/3认定为 SNPs 位点。再通过分析SNP位点附近序列特征兼顾考虑a和b特异性扩增, 设计 SNP特异扩增引物, 结果 jlODC1a与jlODC1b上分别查找到 6个和 4个可以使用PCR-RFLP方法检测的SNPs。各位点所使用的PCR引物、限制性内切酶以及不同基因型酶切片段信息见表1。在10个位点中, 有6个位于外显子上, 其中有2个为非同义突变, 均位于jlODC1a上, 分别为E6_A212G改变氨基酸M(A)→V(G), E9_G19A改变氨基酸S(G) →N(A)。PCR扩增体系为12.5 μL, 其中含模板DNA 1.0 μL, 引物0.25 μL (10 μmol/L)。反应结束取6 μL PCR产物至10 μL酶切体系, 其中含内切酶 0.2 μL, 在不同酶推荐的温度下反应3h, 然后用1.5%—3%琼脂糖凝胶电泳检测, 确定基因型。
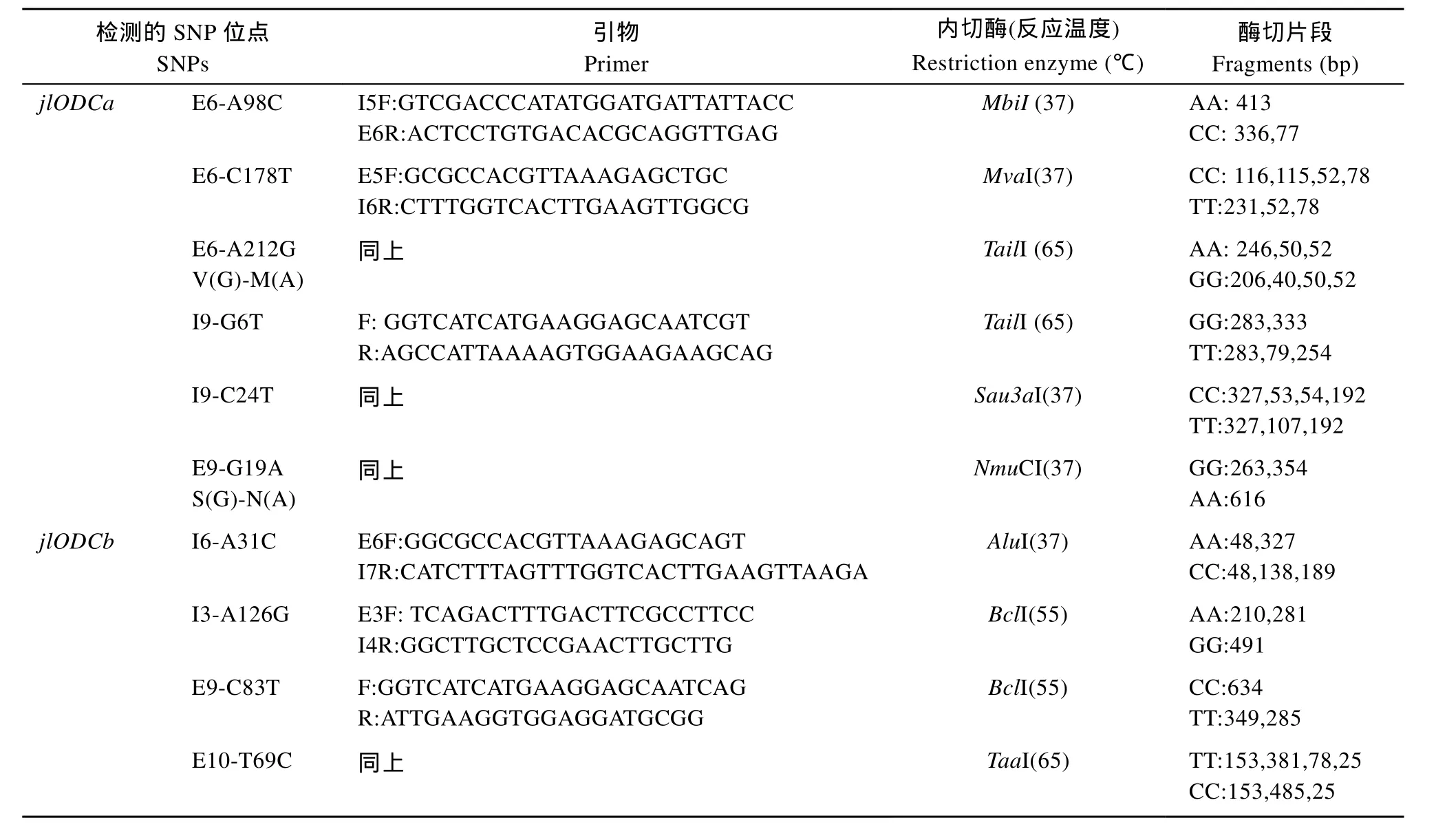
表 1 10个SNP位点的检测引物, 限制性内切酶及酶切片段Tab. 1 Primers, restriction enzymes and restriction fragments of the 10 SNP loci
SNP位点的双倍型分析 增重优势基因型a-E6-A212G(AA)记为1, a-E9-G19A(AA)记为2, b-I3-A126G (AA)记为3, a-E6-A98C (AC)记为4, a-E6-C178T(CT)记为5, b-E9-C83T(CT)记为6, b-E10-T96C(CT)记为7, 同时考虑7个位点, 所检测的建鲤可分为不含优势基因型的0组; 有1个优势基因型的4、6、7组; 有2个优势基因型的14、17、26、56、57、67组; 有3个优势基因型的146、147、167、256、456、 567组; 有4个优势基因型的1467、1567、2567、4567组以及5个优势基因型的14567组共21组个体组。其余各种优势基因型或组合的样本低于15尾的, 不列入统计范围。通过21种双倍型与增重的关联分析,筛选出可用于建鲤选育计划的基因型。
统计分析 利用SPSS软件包的GLM模型进行SNP位点不同基因型、不同双倍型与实验鱼不同阶段增重的关联分析。
2 结果
2.1 SNP位点的不同基因型检测及与增重的相关性
本文所检测的10个位点中有7个位点与增重呈显著相关, 其中, a基因外显子 6上有三个位点, A98C(AC型)、C178T(CT型)及A212G(AA型); 外显子9上一个位点, G19A (AA型); b基因外显子9上一个位点, C83T (CT型); 外显子10上一个位点, T96C (CT型); 内含子 3上一个位点 A126G (AA型)。7个位点的基因型分布存在差异, 最小等位基因频率 (MAF)为0.14—0.48(表2)。
表 2 10个SNPs位点在所检测家系中的基因型分布以及与增重的相关性Tab. 2 Genotype distribution in experiment families of 10 SNPs and correlation with weight gain (±SE)
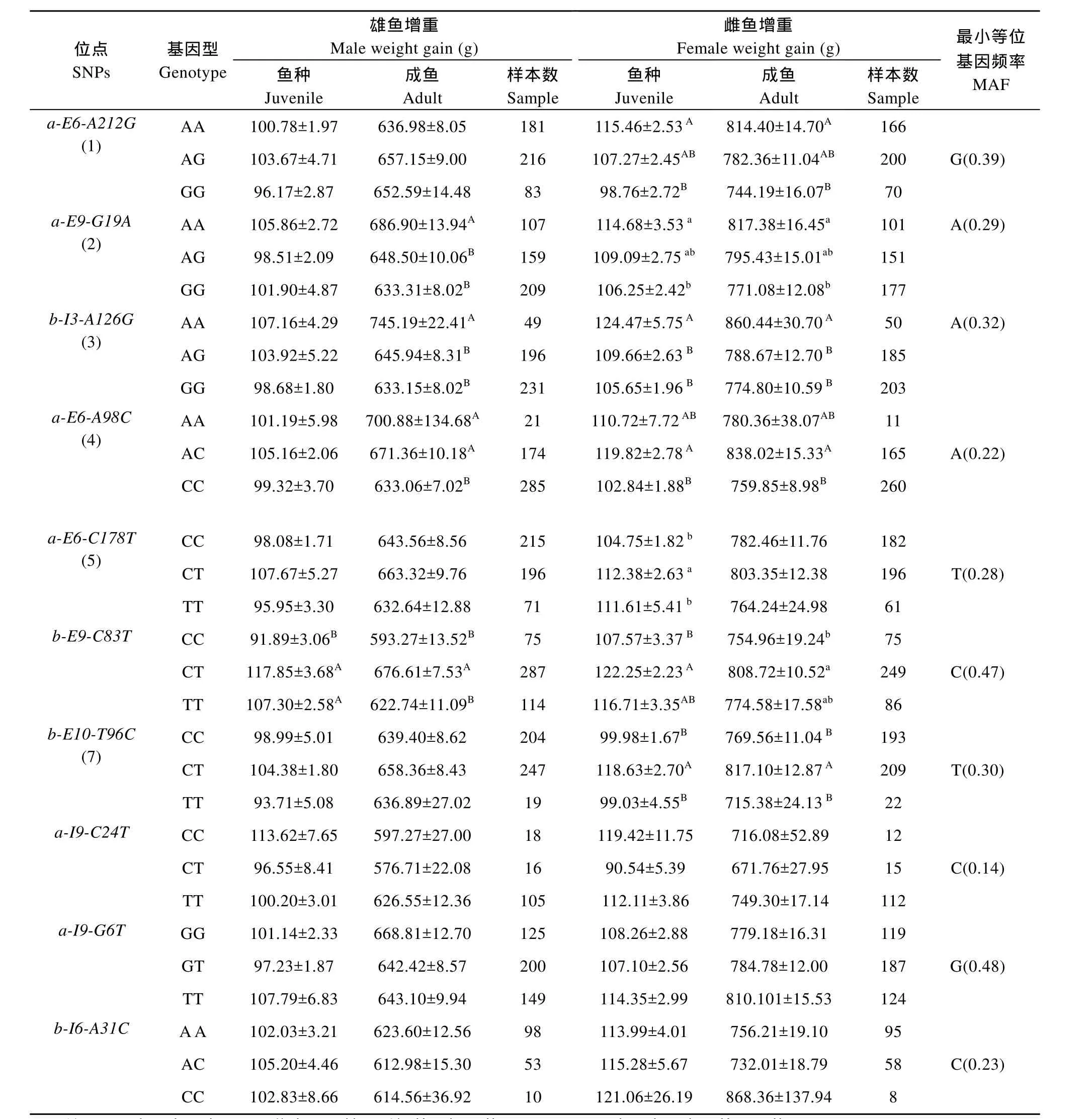
表 2 10个SNPs位点在所检测家系中的基因型分布以及与增重的相关性Tab. 2 Genotype distribution in experiment families of 10 SNPs and correlation with weight gain (±SE)
注: 不同大写字母表示同一位点不同基因型间差异极显著(P<0.01); 不同小写字母表示差异显著(P<0.05)Note: Different capital and lowercase letters indicate significant difference of different genotypes of one same SNP site at P<0.01 and P<0.05 level, respectively
雄鱼增重Male weight gain (g)位点SNPs基因型Genotype 鱼种Juvenile雌鱼增重Female weight gain (g)成鱼Adult样本数Sample鱼种Juvenile成鱼Adult样本数Sample最小等位基因频率MAF a-E6-A212G (1) AA 100.78±1.97 636.98±8.05 181 115.46±2.53A814.40±14.70A166 AG 103.67±4.71 657.15±9.00 216 107.27±2.45AB782.36±11.04AB200 G(0.39) a-E9-G19A (2) b-I3-A126G (3) a-E6-A98C (4) GG 96.17±2.87 652.59±14.48 83 98.76±2.72B744.19±16.07B70 AA 105.86±2.72 686.90±13.94A107 114.68±3.53a817.38±16.45a101 A(0.29) AG 98.51±2.09 648.50±10.06B159 109.09±2.75ab795.43±15.01ab151 GG 101.90±4.87 633.31±8.02B209 106.25±2.42b771.08±12.08b177 AA 107.16±4.29 745.19±22.41A49 124.47±5.75A860.44±30.70A50 A(0.32) AG 103.92±5.22 645.94±8.31B196 109.66±2.63B788.67±12.70B185 GG 98.68±1.80 633.15±8.02B231 105.65±1.96B774.80±10.59B203 AA 101.19±5.98 700.88±134.68A21 110.72±7.72AB780.36±38.07AB11 AC 105.16±2.06 671.36±10.18A174 119.82±2.78A838.02±15.33A165 A(0.22) CC 99.32±3.70 633.06±7.02B285 102.84±1.88B759.85±8.98B260 a-E6-C178T (5) b-E9-C83T b-E10-T96C (7) a-I9-C24T a-I9-G6T b-I6-A31C CC 98.08±1.71 643.56±8.56 215 104.75±1.82b782.46±11.76 182 CT 107.67±5.27 663.32±9.76 196 112.38±2.63a803.35±12.38 196 T(0.28) TT 95.95±3.30 632.64±12.88 71 111.61±5.41b764.24±24.98 61 CC 91.89±3.06B593.27±13.52B75 107.57±3.37B754.96±19.24b75 CT 117.85±3.68A676.61±7.53A287 122.25±2.23A808.72±10.52a249 C(0.47) TT 107.30±2.58A622.74±11.09B114 116.71±3.35AB774.58±17.58ab86 CC 98.99±5.01 639.40±8.62 204 99.98±1.67B769.56±11.04B193 CT 104.38±1.80 658.36±8.43 247 118.63±2.70A817.10±12.87A209 T(0.30) TT 93.71±5.08 636.89±27.02 19 99.03±4.55B715.38±24.13B22 CC 113.62±7.65 597.27±27.00 18 119.42±11.75 716.08±52.89 12 CT 96.55±8.41 576.71±22.08 16 90.54±5.39 671.76±27.95 15 C(0.14) TT 100.20±3.01 626.55±12.36 105 112.11±3.86 749.30±17.14 112 GG 101.14±2.33 668.81±12.70 125 108.26±2.88 779.18±16.31 119 GT 97.23±1.87 642.42±8.57 200 107.10±2.56 784.78±12.00 187 G(0.48) TT 107.79±6.83 643.10±9.94 149 114.35±2.99 810.101±15.53 124 A A 102.03±3.21 623.60±12.56 98 113.99±4.01 756.21±19.10 95 AC 105.20±4.46 612.98±15.30 53 115.28±5.67 732.01±18.79 58 C(0.23) CC 102.83±8.66 614.56±36.92 10 121.06±26.19 868.36±137.94 8
各位点的基因型分布及与增重相关性结果(表 2), b-E9-C83T 的CT型雄鱼鱼种增重极显著高于CC型 (P<0.01); a-E6-A98C (AC)、a-E9-G19A (AA)、b-I3-A126G (AA)与 b-E9-C83T (CT)的雄鱼成鱼增重与其他类型相比均达到极显著水平(P<0.01), 此次获得的 7个阳性位点均与雌鱼鱼种增重显著相关, 其中极显著相关的有 5个分别为a-E6-A98C(AC)、a-E6-A212G (AA)、b-I3-A126G (AA)、b-E9-C83T (CT)、b-E10-T96C (CT) (P<0.01); 其余 2个与雌鱼鱼种增重显著相关(P< 0.05)。与雌鱼鱼种增重极显著相关的5个位点中除了 b-E9-C83T (CT,显著水平)外, 其余4个也均与雌鱼成鱼的增重极显著相关, 另外还有a-E9-G19A (AA)位点与雌鱼成鱼增重显著相关(P<0.05)。图1为7个阳性位点的基因分型图。
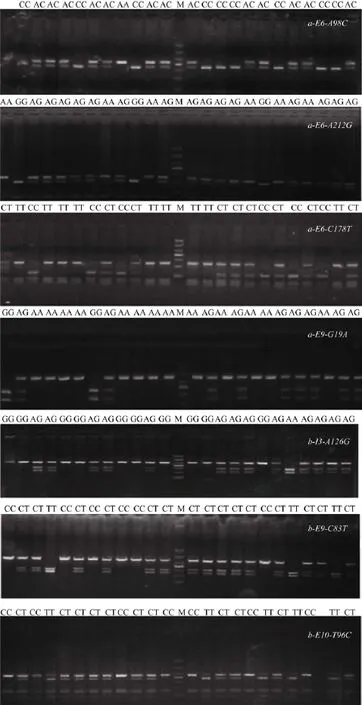
图1 7个阳性SNP 位点不同基因型的电泳结果Fig. 1 Electrophoresis patterns of different genotypes in seven SNP loci M. DNA marker (DL1000)
2.2 标记富集与双倍型分析
本次所获得的7个与增重相关标记, 4个位于jlODCa上分别为即a-E6-A98C (AC)、a-E6-A212G (AA)、a-E6-C178T(CT)、a-E9-G19A(AA); 3个位于 jlODCb上分别为 b-I3-A126G1a(AA)、b-E9-C83T (CT)、b-E10-T96C(CT)。在7个位点中, 富集个数最多的为6个(表3)。分析结果, 随着标记富集个数的增加, 建鲤增重随之增加。富集3个标记的个体增重比无标记的个体增重快13.6% (P<0.05), 4个标记个体增重极显著高于 3个标记个体增重(P<0.01)。而富集5个标记的个体增重比富集4个标记的个体增重有增高, 但差异不显著; 富集 6个标记的个体仅有12尾, 虽然平均增重极显著高于富集4个标记的个体增重,但不能列入统计范围。在所检测的建鲤群体中有 4个标记的鱼有 178尾, 约占19.1%。
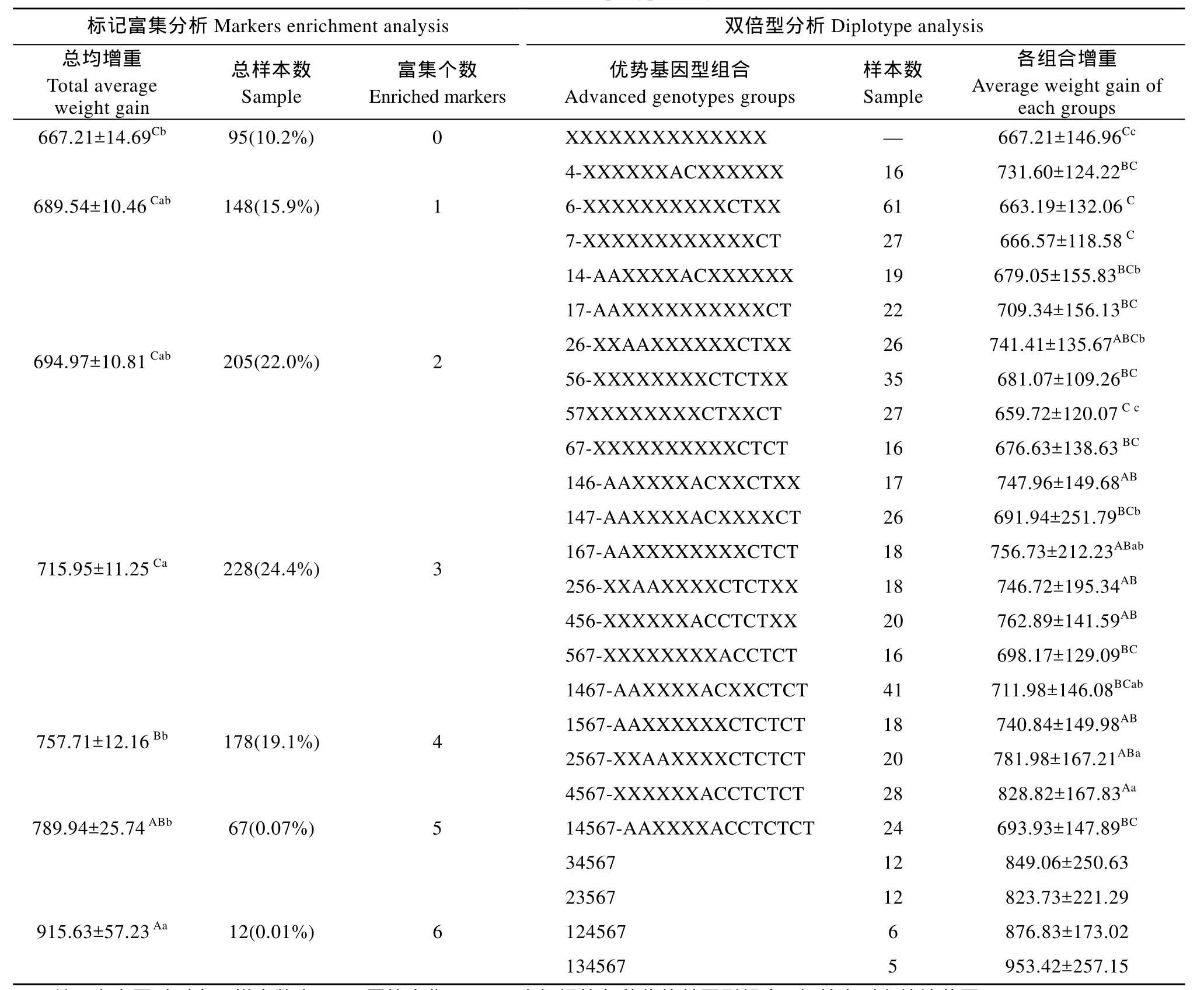
表3 标记富集及双倍型分析Tab. 3 Markers enrichment and diplotype analysis of screened SNPs
21组双倍型与增重关联分析显示, 0组与1个优势基因型各组均没有显著差异, 其中 4组增重最快达731.6 g, 较0组增重约11.7%。在有2个优势基因型的 6组中, 只有 26组增重(741.41 g)显著快于57组(659.72 g) (P<0.05); 其余差异不显著。3个优势基因型的各组差异不显著; 四个优势基因型各组中, 4567组增重为所有检测组中最高(823.82 g)极显著高于1467组(711.98 g) (P<0.01)且各组增重均大于700 g。有5个优势基因型有三组, 34567、23567和14567, 前两组样本数均为12尾, 样本大于15的仅有14567一组, 但增重仅为693.93 g, 为三组中增重最慢的。而同时有 6个优势基因型的仅有两组124567和134567, 二者虽然增重都比较快, 但由于二者样本数均少于10尾, 不能列入统计范围。
3 讨论
单核苷酸多态性 (Single nucleotide polymorphism, SNP), 主要指在基因组水平上单个核苷酸的变异所引起的DNA序列多态性, 在基因组中分布广泛。在畜禽类养殖业中, SNP位点可能与机体的各种生长或经济性状相关, 可用于现有品种改良与新品种的选育。Orexin基因5′调控区筛选到了与牛(Bos tarurs)增重相关的SNP位点[9], PNRCI基因的3′非翻译区筛选到了与菜鸭(Tsaiya ducks)繁殖性能相关的SNP位点[10]。水产养殖业中至今也有较多报道, 如斑节对虾(Penaeus monodon)[11]、罗非鱼(Oreochromis niloticus)[12]、大口黑鲈(Largemouth bass)[13]的生长相关位点。本实验室在关于SNP位点筛选方面也做了大量的研究, 如尼罗罗非鱼的 IGF-2基因[14], 吉富罗非鱼(Genetically improved farmed tilapia, GIFT)的GH[15]、GHR[16]、IGF-1[17]基因, 尤其是在建鲤的与生长相关的基因上, 如 MSTN[18]、GHR[19]、GHSR[20]、IGFBP1[21]、IGFBP3[22]、IGF-Ia[23]、FABP2[24]、FABP3[25], 筛选了多个可以应用于选育的生长相关的SNP位点。
在分离了2个建鲤jlODC1s基因并确定该基因的表达与建鲤生长相关后[7], 通过查找两基因上的SNP位点, 本实验在jlODC1a与 jlODC1b两个基因上分别构建了6和4个SNP位点的PCR-RFLP检测方法, 从实验图谱可以看出, 检测结果清晰、可靠。将 SNP位点的不同基因型与增重进行相关性分析,结果共获得了 7个与建鲤增重显性相关的位点, 其中4个位于jlODC1a基因上, 3个位于jlODC1b基因上, 与雄鱼鱼种阶段增重极显著相关的SNP位点1个 (P<0.01); 与成鱼阶段增重极显著相关的位点有4个 (P<0.01)。与雌鱼鱼种阶段增重相关的位点有7个, 分别呈显著(P<0.05)和极显著差异(P<0.01); 与雌鱼成鱼阶段增重相关的有 6个。可见在 ODC1s基因上与雌鱼增重相关的SNP位点较雄鱼多, 俞菊华等在ODC1s基因的表达研究中报道, 肌肉组织中ODC1s基因雌鱼的表达量明显高于雄鱼, 且无论雌鱼雄鱼, a基因的表达在脑、肝脏、肌肉、性腺等组织中一般都要高于 b基因的表达, 尤其是肝脏中达到显著差异水平(P<0.05)[7], 这可能也是造成本次与建鲤增重显性相关SNP位点的筛选中a基因(4个)略多于b基因(3个)的一个原因。另外, 同一位点基因型在雌、雄鱼的表现不完全一致, 如a-E6-A212G、a-E6-C178T、a-I9-C24T及b-E10-T96C四个位点均与雌鱼增重显性相关, 但与雄鱼增重并未表现出相关性; 同一位点在鱼不同生长阶段的表现也不完全一致, a-E6-A98C、a-E9-G19A及b-I3-A126G位点与雄鱼成鱼增重极显著相关, 但与雄鱼鱼种阶段增重则相关性不显著; 这些差异可能与该基因在不同性别和不同阶段的表达差异有关, 如阮瑞霞等在进行筛选吉富罗非鱼GHR上增重相关SNP时就出现类似雌雄差异的情况是与基因表达水平相一致的[16]。
标记富集结果反映了生长为数量性状, 总的来说富集标记越多的个体增重速度越快, 富集 4个标记以上的增重比0标记的快约14%。双倍型分析结果显示同时具有4个优势基因型的1467、1567、2567与4567等4组增重均大于700 g, 其中4567组增重(828.82±167.83) 明显快于其他三组, 其中极显著快于1467组(711.98±146.08) (P<0.01)。就此次研究的建鲤 ODC1s的 SNP位点, 要选择培养的是富集 4个标记, 且基因型分别为 4567 (XXXXXXACCT CTCT)基因型的个体, 其增重较 0优势基因型的个体快 26.6%, 比群体平均增重快约 13%, 可以考虑用于今后快速增长建鲤的育种计划中。本次所得 7个阳性位点中共 3个纯合型, a-E6-A212G(AA)、a-E9-G19A(AA)与 b-I3-A126G(AA), 说明纯合型对增重优势起到了一定的作用, 而在双倍型分析中 4个优势基因型的4组中, 全部为杂合型的4567组增重明显快于各包含一个纯合、三个杂合型的其他三组, 其中极显著快于1467组。就此次研究而言杂合型位点对于个体增重的贡献更多。
各位点双倍型分析结果还显示, 单独具有4(AC)的个体增重明显快于具有两个优势基因型的14、56、67、17、57、67组、三个优势基因型的147、567, 以及4个优势基因型的1467组的个体。由此, 4 (AC)为建鲤ODC1s基因上与增重相关的主效 SNP位点, 且各位点之间存在着或颉抗或协同的作用。比较 4与 14组合, 平均增重由 731.60 g降至679.05 g, 尤其4567与14567组相比, 增重由828.82 g骤降至693.93 g, 由此1与4表现为颉抗;与之相反, 比较7与17组合, 6与26组合, 二者分别由666.57与663.19增至709.34与741.41 g, 由此推测1与7、2与6表现为协同作用。本次标记富集分析与双倍型分析的结果有些不太相符, 一方面可能由于多位点一起分析后, 各小群的样本数不够大, 各位点之间是否还存在我们尚未清楚的相互关系, 而这些相互关系可能就导致标记富集数与增重之间不完全是一致的结果?本次研究最终仅得到了富集 4个标记的一种最优基因型组合, 将来是否可以筛选到富集五个甚至六个的最优基因型组合?接下来我们将扩大样本数对这个问题进一步验证, 且此次五、六个标记的各基因型组合也展现出增重明显上升的趋势, 因此继续这个思路的筛选工作具备充分的可行性, 另一方面也提示我们在今后的选育过程中, 在选育富集多SNP标记个体的同时也要注重各优势基因型的组合, 扬长避短, 在选择互为协同作用优势基因型的前提下, 富集尽可能多的SNP标记。
[1] Tsuji T, Todd R, Meyer C, et al. Reduction of ornithine decarboxylase antizyme (ODCAz) level in the 7,12-dimethylbenz (a) anthraceneinduced hamster buccal pouch carcinogenesis model [J]. Oncogene, 1998, 16: 3379—3385
[2] Favre C, Camovale C E, Monti J A, et al. Inhibition by interferon alpha-2b of rat liver regeneration: effect on ornithine decarboxylase and total protein synthesis [J]. Biochemical Pharmacology, 2001, 61(12): 1587—1593
[3] Uemoto Y, Sato S, Ohtake T, et al. Ornithine decarboxylase gene is a positional candidate gene affecting growth and carcass traits in F2intercross chickens [J]. Poultry Science, 2011, 90(1): 35—41
[4] Benfey T J, Saunders R, Knox D E, et al. Muscle ornithine decarboxylase activity as an indication of recent growth in pre-smolt Atlantic salmon, Salmo salar [J]. Aquaculture, 1994, 121(1—3): 125—135
[5] Arndt SKA, Benfey T J, Cunjak R A. A comparison of RNA concentrations and ornithine decarboxylase activity in Atlantic salmon (Salmo salar) muscle tissue, with respect to specific growth rates and diet variations [J]. Fish Physiology and Biochemistry, 1994, 13(6): 463—471
[6] Sambrook J, Fritsch E F, Maniatis T (Eds.), Huang P T, trans. Molecular Cloning-A Laboratory Manual [M]. Beijing: Science Press. 2002, 463—465
[7] Yu J H, Li H X, Tang Y K, et al. Characterization and expression analysis of ornithine decarboxylase 1 genes of Cyprinus carpio l [J]. Acta Hydrobiologica Sinica, 2013, 37(3): 566—571 [俞菊华, 李红霞, 唐永凯, 等. 鲤鱼鸟氨酸脱羧酶基因的序列特征和表达. 水生生物学报, 2013, 37(3): 566—571]
[8] Thompson J D, Higgins D G, Gibson T J. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice [J]. Nucleic Acids Research, 1994, 22: 4673—4680
[9] Zhang A, Zhang L, Zhang L, et al. Single Nucleotide polymorphism (SNP) analysis of orexin gene 5′regulatory region in Chinese indigenous cattle populations [J]. Agricultural Sciences in China, 2011, 10(8): 1273—1279
[10] Chang M T, Cheng Y S, Huang M C. A novel SNP of the PNRC1 gene and its association with reproductive traits in Tsaiya ducks [J]. Theriogenology, 2012, 78(1): 140—146
[11] Prasertlux S, Khamnamtong B, Chumtong P, et al. Expression levels of RuvBL2 during ovarian development and association between its single nucleotide polymorphism (SNP) and growth of the giant tiger shrimp Penaeus monodon [J]. Aquaculture, 2010, 308(1): S83—S90
[12] Liu F P, Bai J J, Ye X, et al. Cloning of MC4R gene and study on the association between SNPs of MC4R and growth trait in tilapia (Oreochromis niloticus) [J]. Journal of Fishery Sciences of China, 2009, 16(6): 816—823 [刘福平, 白俊杰,叶星, 等. 罗非鱼 MC4R基因克隆及与生长相关的 SNP位点. 中国水产科学, 2009, 16(6): 816—823]
[13] Yu L Y, Bai J J, Fan J J, et al. SNPs detection in largemouth bass myostatin gene and its association with growth traits [J]. Journal of Fisheries of China, 2010, 34(6): 665—671 [于凌云, 白俊杰, 樊佳佳, 等. 大口黑鲈肌肉生长抑制素基因单核苷酸多态性位点的筛选及其与生长性状关联分析.水产学报, 2010, 34(6): 665—671]
[14] Yu J H, Chen X F, Li J L, et al. Isolation of IGF2 and association of IGF2 polymorphism with growth trait in genetically improved farmed tilapias, Oreochromis niloticus L [J]. Aquaculture Research, 2010, 41(11): e743—e750
[15] Tang Y K, Yu J H, Xu P, et al. Cloning of growth hormone (GH) gene of genetically improved farmed tilapia (GIFT) and association of the single nucleotide polymorphisms (SNPs) of GH gene with the weight gain [J]. Journal of Hunan Agricultural University (Natural Sciences), 2012, 38(4): 422—425 [唐永凯, 俞菊华, 徐跑, 等. 吉富罗非鱼生长激素(GH)的分离及其SNPs与增重的相关性. 湖南农业大学学报, 2012, 38(4): 422—425]
[16] Ruan R X, Yu J H, Li H X, et al. Isolation of two growth hormone receptor genes and SNPs associated with body weight in GIFT strain tilapia Orechromis niloticus [J]. Chinese Journal of Zoology, 2011, 46(3): 37—46 [阮瑞霞,俞菊华, 李红霞, 等. 吉富罗非鱼两种生长激素受体基因的分离及与增重相关的 SNPs位点. 动物学杂志, 2011, 46(3): 37—46]
[17] Ruan R X, Yu J H, Li H X, et al. Effects of IGF-1 genotype on weight gain and body form in GIFT strain Orechromis niloticus [J]. Journal of Fishery Sciences of China, 2011, 18(3): 682—688 [阮瑞霞, 俞菊华, 李红霞, 等. 吉富罗非鱼IGF-1基因的基因型对生长和体型的影响. 中国水产科学, 2011, 18(3): 682—688]
[18] Yu J H, Li H X, Tang Y K, et al. Myostatin (MSTN) genes in Cyprinus carpio var. jian: isolation, expression, correlation between polymorphisms and body form, ADG [J]. Journal of Agricultural Biotechnology, 2010, 18(6): 1062—1072 [俞菊华, 李红霞, 唐永凯, 等. 建鲤生长抑制素基因 MSTN的分离、表达及多态性与体型、平均日增重相关性研究. 农业生物技术学报, 2010, 18(6): 1062—1072]
[19] Tao W J, Ma L J, Yu J H, et al. SNP loci associated with weight gain on growth hormone receptor genes in Cyprinus carpio var. jian [J]. Acta Hydrobiologica Sinica, 2011, 35(4): 622—629 [陶文静, 马龙俊, 俞菊华, 等. 建鲤GHR基因多态性及与增重相关的SNP位点的筛选. 水生生物学报, 2011, 35(4): 622—629]
[20] Yu J H, Li H X, Li J L, et al. Characterization of growth hormone secretagogue receptor 1(GHS-R1) genes andweight gain associated SNP loci in Cyprinus carpio var. jian [J]. Journal of Fishery Sciences of China, 2012, 19(3): 390—398 [俞菊华, 李红霞, 李建林, 等. 建鲤生长激素促泌素受体1基因的特性及其与增重相关SNP位点的筛选.中国水产科学, 2012, 19(3): 390—398]
[21] Wei K P, Yu J H, Li H X, et al. Isolation of IGFBP1 and SNP loci related to body weight gain in Cyprinus carpio var. jian [J]. Acta Agriculturae Boreali-Sinica, 2012, 27(3): 75—80 [魏可鹏, 俞菊华, 李红霞, 等. 建鲤IGFBP1基因的克隆及与增重相关的SNP位点分析. 华北农学报, 2012, 27(3): 75—80]
[22] Wei K P, Yu J H, Li H X, et al. Isolation of IGFBP3 gene and SNPs associated with body weight gain in Cyprinus carpio var. jian [J]. Chinese Journal of Zoology, 2012, 47(1): 94—102 [魏可鹏, 俞菊华, 李红霞, 等. 建鲤IGFBP3基因多态性对生长的影响. 动物学杂志, 2012, 47(1): 94—102]
[23] Li H X, Liu N N, Pu T N, et al. Isolation, SNPs identification and the correlation analysis with weight gain of IGF-Ia in Cyprinus carpio var. jian [J]. Journal of Shanghai Ocean University, 2012, 21(1): 173—179 [李红霞, 刘楠楠, 浦天宁, 等. 建鲤IGF-Ia基因的SNPs位点筛选及其与增重的相关性分析. 上海海洋大学学报, 2012, 21(1): 173—179]
[24] Xia Z L, Yu J H, Li H X, et al. Sequences analysis of jlFABP2 and the correlation between polymorphisms and body weight gain in Cyprinus carpio var. jian [J]. Hereditas, 2013, 35(5): 628—636 [夏正龙, 俞菊华, 李红霞, 等. 建鲤肠型脂肪酸结合蛋白基因的分离及其SNPs与增重的相关分析. 遗传, 2013, 35(5): 628—636]
[25] Yu J H, Li H X, Li J L, et al. Sequences analysis of jlFABP3s and the correlation between polymorphisms and body weight gain in Cyprinus carpio var. jian [J]. Journal of Fisheries of China, 2012, 36(12): 1809—1818 [俞菊华, 李红霞, 李建林, 等. 建鲤FABP3基因分离及其多态性与增重的相关分析. 水产学报, 2012, 36(12): 1809—1818]
CORRELATION ANALYSIS BETWEEN BODY WEIGHT GAIN AND ODC1 GENOTYPES IN CYPRINUS CARPIO var. JIAN
LI Hong-Xia, LI Jian-Lin, TANG Yong-Kai and YU Ju-Hua
(Key Laboratory of Freshwater Fisheries and Germplasm Resources Utilization, Ministry of Agriculture, Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214081, China)
Ornithine decarboxylase (ODC), arate-limiting enzyme in polyamine synthesis,is involved in several biological processes. PCR-RFLP was established to detect genotypes of ten SNPs (six for jlODC1a and four for jlODC1b) in about 900 individuals from 12 Cyprinus carpio var. jian families. The minimum allele frequency (MAF) was 0.14—0.48. The correlation between the genotypes of ten SNPs and body weight gain indicated that there are seven SNP loci in the female and four SNP loci in the male , and that the body weight gain is associated with the quantity of the SNPs. The individuals with four markers grew obviously faster than those with 0—3 markers, and grew 14% faster than those with 0 markers, which also reflected growth as a quantitative trait. The diplotype analysis of the screened SNPs demonstrated that individuals with four heterozygous advantage genotypes 4567 (XXXXXXACCTCTCT) were the fastest growth ones that were 26.6% faster than 0 advantage genotype individuals, which could be used in the fast-growth jian carp molecular breeding. Moreover, the diplotype analysis explored the exist of either antagonistic or synergistic interaction between different loci, suggesting that the choice of mutually synergistic advantages genotype and as much as possible SNP markers could benefit the future breeding programs.
Cyprinus carpio var. jian; Ornithine decarboxylase; Genotype; SNP-weight gain correlated analysis
Q344+.1
A
1000-3207(2014)03-0414-08
10.7541/2014.59
2013-04-16;
2014-01-27
国家科技部“863计划”项目(2011AA100401); 公益性行业(农业)科研专项(200903045); 现代农业产业技术体系建设专项资金(CARS-46); 鲤肉质相关分子标记的挖掘(2013JBFM03)资助
李红霞(1982—), 女, 山东聊城人; 硕士, 助理研究员; 主要从事水生生物技术。E-mail: lihx@ffrc.cn
俞菊华 (1966—), 女, 江苏苏州人; 博士, 研究员; 主要从事水生生物技术研究。E-mail: yujh@ffrc.cn

