凝胶条带内DNA回收测序评估DGGE可靠性
倪加加余育和
(1. 中国科学院水生生物研究所, 中国科学院水生生物多样性与保护重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
凝胶条带内DNA回收测序评估DGGE可靠性
倪加加1,2余育和1
(1. 中国科学院水生生物研究所, 中国科学院水生生物多样性与保护重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
为了评估DGGE的可靠性, 对DGGE条带中回收的DNA片段进行了测序比较分析, 并引入了DGGE可靠性指数的概念评价其可靠性。结果显示同一条DGGE条带回收的DNA来自同一属的概率为64.7%, 相同位置的DGGE条带可以被认为是同一OTU; 不同的DGGE条带回收到类似的DNA序列(16S rDNA V3区差异小于4 bp)的概率为10.5%; DGGE可靠性指数为74.8%。以上结果表明尽管DGGE技术与理论预期存在一定的差距, 但是DGGE技术基本能够反映微生物群落的多样性。
DGGE可靠性指数; DNA指纹; 变性梯度凝胶电泳; 微生物群落结构
变性梯度凝胶电泳(Denaturing gradient gel electrophoresis, DGGE)技术被广泛应用于微生物群落结构分析[1—8], 并且在PCR扩增条件、PCR引物设计、DGGE分析方法等方面得到了深入地研究和优化[9—13]。然而, 由于PCR扩增条件、变性梯度凝胶的变性范围、DGGE凝胶信息分析方法等都会对结果产生影响, 因此DGGE结果的可靠性也受到了质疑[14—16]。另外, 由于DGGE技术的理论基础是依靠DNA序列的不同造成的DNA片段变性条件不同,通过在连续的变性梯度凝胶中将 DNA片段变性来改变 DNA分子在凝胶中的迁移速率从而将序列不同的DNA片段分为不同的DGGE条带[1,14,17], 因此,在进行DGGE凝胶分析时一般都将一条DNA条带作为一个可操作分类单位(Operational taxonomic unit, OTU)来处理, 并认为位于同一变性条件的DGGE条带是相同的 OUT[1,4]; 然而, 由于共迁移等因素的影响, 在相同的DGGE条带中可能检出不同的 DNA序列, 而相同的序列也可能出现在不同的DGGE条带中[8,10,15,16]。
为了评估共迁移等因素对 DGGE结果的影响,我们对分析草鱼肠道菌群结构的DGGE凝胶条带中的DNA片段进行回收测序, 并根据所得DNA序列的相似性情况分析了同一条带中得到不同 DNA序列的概率及同一DNA序列出现在不同DGGE条带中的概率, 同时引入了 DGGE可靠性指数来评估DGGE结果的可靠性。
1 材料与方法
1.1 样本采集
实验草鱼样本于2010年12月29日采自湖北省武汉市多福科技农庄(30°08′ N, 114°23′ E), 用于分析的三尾草鱼的平均体重为(910.0 ± 72.1) g, 平均体长为(37.0 ± 0.34) cm。样本冰冻带回实验室称量体长、体重后立即在无菌条件下解剖并根据外部形态将草鱼肠道分为六段(前肠前端, FF; 前肠后端, RF; 中肠前端, FM; 中肠后端, RM; 后肠前端, FH;后肠后端, RH), 并分别从每段取约0.5 cm肠道及内含物放入2 mL离心管中–20℃保存备用。
1.2 DNA提取、PCR-DGGE分析及条带回收测序
DNA提取和PCR扩增条件参考Ni, et al.[8]。采用胶浓度为9%、变性范围为40%—70%的变形梯度凝胶对获得的PCR产物进行DGGE分析, DGGE实验条件及条带回收测序方法参考Ni, et al.[8]。
1.3 数据分析
克隆测序得到的DNA序列使用NCBI网站中的vector contamination (vecscreen) 程序去载体后使用Clustal 2.0进行序列比对, 然后使用BioEdit软件包手动去除引物序列后使用 Mothur软件中的Chimera.uchime程序分析所得序列中的嵌合体, 再使用MEGA 4软件分析非嵌合体的DNA序列的碱基差异并根据该碱基差异矩阵分析各DGGE条带中DNA序列差异情况。所得序列对应的物种信息及对应的 DNA 序列使用 Ribosomal Database Project (RDP) release 10网站中的Classifier程序和Browser程序获得。
为了分析不同DGGE条带检出相似的DNA序列的概率, 我们引入了以下计算公式:
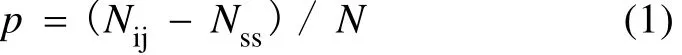
其中p为不同DGGE条带检出相似的DNA序列的概率, Nij为DNA序列碱基差异矩阵中小于定义相似性序列阈值的碱基差异值总数, Nss为同一DGGE条带中DNA序列碱基差异矩阵中小于定义相似性序列阈值的碱基差异值总数, N为DNA序列碱基差异矩阵中所有的碱基差异值总数, N = [n · (n–1)] / 2, 其中的n为用于计算碱基差异矩阵的DNA序列总数。
为了从整体上评估DGGE结果的可靠性, 我们引入了DGGE条带可靠性指数RI, 计算公式为:
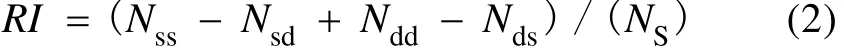
其中Nss为同一DGGE条带中相似DNA序列两两组合数, Nsd为同一DGGE条带中与其他DNA序列不相似的DNA序列两两组合数, Ndd为不同DGGE条带中检出的不相似的 DNA序列两两组合数, Nds为不同DGGE条带中检出相似的DNA序列两两组合数, NS为用于分析的 DNA序列两两组合总数。RI越大, 表征DGGE结果越可靠。
2 结果
2.1 DGGE指纹图谱和DNA测序情况
共检出77条不同的DGGE条带, 其中共有带2条, 只在一个样品中出现的条带 9条, 平均每个样品26.22±2.05条DGGE条带(图1A)。对其中的22条DGGE凝胶条带中的DNA片段进行了回收测序(图1A)。除了8号条带、19号条带和21号条带未能测得其中的DNA序列外, 其余19条带共测得50条DNA序列(18号条带只测得1条DNA序列, 11号带、12号带、13号带、17号带和20号带分别测得2条DNA序列, 其余DGGE条带分别测得3条DNA序列), 其中有2条为嵌合体序列, 分别来自于5号条带和11号条带, 后续的分析中去除了这两条嵌合体序列。
2.2 同一DGGE条带中DNA序列的相似性
在19条DGGE条带中, 除了11号和18号条带因只有一条有效的 DNA序列无法评估外, 剩余的17条DGGE条带中, 只有6条(35.3%)DGGE条带(4号、5号、9号、15号、17号和22号条带)回收的DNA 序列完整一样(0个碱基差异), 另有 5条(29.4%)DGGE条带(1号、2号、6号、12号和 20号条带)回收的DNA序列差异小于3 bp。其余的6条(35.3%)DGGE条带(3号、7号、10号、13号、14号和16号条带)中至少有两条DNA序列的差异大于30 bp。
DNA序列在 RDP数据库中比对的结果显示 1号和2号DGGE条带回收的DNA序列与Bacteroides属细菌的16S rDNA V3区序列最相似, 6号和12号DGGE条带回收的DNA序列与Cetobacterium属细菌的16S rDNA V3区序列最相似, 而20号序列则只能确定与Firmicutes门细菌的16S rDNA V3区序列最相似。为了深入分析所测DNA片段对应的细菌种间和种内16S rDNA V3区的多样性, 我们从RDP数据库中下载了 Bacteroides属和 Cetobacterium属细菌的16S rDNA序列并对其V3区进行了分析。结果显示, 12条Bacteroides fragilis的16S rDNA V3区DNA序列没有碱基差异, 10条Bacteroides thetaiotaomicron的16S rDNA V3区DNA序列存在11—13 bp差异; 而Bacteroides属的16S rDNA V3区不同种间DNA序列则存在0—18 bp差异, Cetobacterium属的16S rDNA V3区不同种间DNA序列则存在 8 bp差异; Bacteroides、Acetomicrobium 和Anaerorhabdus的 16S rDNA V3区属间差异则为28—50 bp, Cetobacterium、Clostridium、Fusobacterium、Ilyobacter、Propionigenium和Psychrilyobacter属的16S rDNA V3区属间差异则为4—14 bp。因此,同一条DGGE条带回收的DNA来自同一属的概率为64.7%。
根据DGGE条带的位置, 1号条带跟2号条带应为同一OTU, 4号条带与5号条带应为同一OTU。测序结果显示1号条带跟2号条带中的DNA序列相差0—3 bp, 都与Bacteroides属细菌的16S rDNA V3区序列最相似; 4号条带与5号条带中回收的DNA序列完全相同。这一结果显示相同位置的DGGE条带可以被认为是同一OTU。

图1 DGGE指纹图谱Fig. 1 DGGE fingerprinting
2.3 不同DGGE条带中DNA序列的相似性
我们还在不同的DGGE条带中检出序列差异小于4 bp的DNA序列, 如1号和2号条带回收的DNA序列与4号和5号条带回收的DNA序列差异为0—1 bp, 9号与20号条带回收的DNA序列差异为0—1 bp。根据公式(1)计算得到的不同DGGE条带检出相似的DNA序列的概率为10.5%。
2.4 DGGE可靠性指数
以细菌16S rDNA V3区DNA序列差异小于4 bp的序列为相似性序列标准, 使用公式(2)计算得到DGGE可靠性指数为74.8%, 表明DGGE结果能够较好地表征草鱼消化道微生物群落16S rDNA的多样性。
3 讨论
DGGE技术因其重复性高、廉价、高效而被广泛应用于微生物群落结构分析[1—8]。但由于受 PCR引物、PCR扩增条件、凝胶的变性范围等因素的影响, DGGE技术的可靠性也受到了质疑[14—16]。尽管如此, 目前为止并没有一种用于评估DGGE结果可靠性的方法被提出。我们在本文中首次引入了DGGE条带可靠性指数RI来评估DGGE结果的可靠性, 为有效评估DGGE指纹图谱的可靠性提供了技术手段。同时, 考虑到不同DGGE条带检出相似的DNA序列的概率P值会受到每条DGGE条带回收测序的 DNA序列的条数差异对结果的影响, 我们采用序列的两两组合数作为RI的自变量, 在一定程度上排除了每条DGGE条带回收测序的DNA序列的条数差异对结果的影响, 从而保证不同的研究者能够采用RI值进行横向比较。我们通过此方法对草鱼消化道微生物群落结构的DGGE结果进行的分析结果显示, 尽管同一条DGGE条带中可能回收得到不同的DNA序列, 同时类似的DNA序列也可能会在不同的DGGE条带中检出, 但DGGE结果依旧能够较好地反映环境中微生物群落结构的多样性。
由于DGGE是依靠DNA片段的变性条件将其分为不同的DGGE条带, 而不同序列的DNA片段可能有相同的变性条件, 从而导致在进行DGGE分析是在凝胶的同一变性梯度上形成相同的DGGE条带[8,14], 这将导致环境样品中的微生物多样性可能被低估; 而由于共迁移等因素的影响, 相同的 DNA片段也可能形成不同的DGGE条带[14], 这将导致环境样品中的微生物多样性可能被高估。我们的结果显示由于DGGE分辨率的影响, 环境样品中的微生物多样性被低估的概率(35.3%)高于被高估的概率(10.5%), 这也在一定程度上解释了为什么DGGE技术检出的微生物种类少于克隆文库或二代测序技术检出的微生物种类。
凝胶的变性范围被认为是影响DGGE结果的重要因素之一[10]。Gafan & Spratt[10]通过回收较宽变性范围的凝胶中的条带后使用较窄变性范围的凝胶重新分析发现, 在较宽变性范围的凝胶中被认为是相同的DGGE条带在较窄的变性范围的凝胶中则被分为不同的DGGE条带。这一结果同样暗示使用种属特异性引物进行DGGE分析得到的结果可能比使用细菌通用引物得到的结果要更可靠, 并且能够同时减小环境样品中的微生物多样性被低估和被高估的概率。
另外, 我们针对于不同属的细菌16S rDNA V3区的 DNA序列多样性进行的分析结果显示不同属的细菌其16S rDNA V3区的保守程度不同, 这可能导致在使用细菌通用引物(如本文中使用的F357-GC/R518引物)对环境微生物群落进行 DGGE分析时, 对不同门或者纲的细菌的分辨率存在差异,从而可能导致有些细菌门的多样性被低估。因此,为了能够更好地反映环境微生物群落的多样性, 我们建议在使用通用引物进行DGGE分析的同时, 应结合特定类群的特异性引物开展更进一步的 DGGE分析, 而我们引入的公式(2)则能够为横向比较不同的类群特异性引物得到的DGGE结果提供可靠性方面的评估。同时, 尽管本文中我们以草鱼消化道微生物群落为研究对象开展的DGGE分析, 我们引入的概念及分析方法完全可以拓展至其他生境微生物群落结构的DGGE结果的评估。
综上所述, 尽管与理论预期存在一定的差距, DGGE技术依旧能够较好地反映环境中微生物群落结构的多样性; 同时, DGGE条带可靠性指数RI可以有效评估DGGE结果的可靠性。
[1] Muyzer G, De Waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reactionamplified genes coding for 16S rRNA [J]. Applied and Environmental Microbiology, 1993, 59(3): 695—700
[2] Gelsomino A, Keijzer-Wolters A C, Cacco G, et al. Assessment of bacterial community structure in soil by polymerase chain reaction and denaturing gradient gel electrophoresis [J]. Journal of Microbiological Methods, 1999, 38(1): 1—15
[3] Yang C H, Crowley D E, Menge J A. 16S rDNA fingerprinting of rhizosphere bacterial communities associated with healthy and Phytophthora infected avocado roots [J]. FEMS Microbiology Ecology, 2001, 35(2): 129—136
[4] Yan Q Y, Yu Y H, Feng W S, et al. Genetic diversity of plankton community as depicted by PCR-DGGE fingerprinting and its relation to morphological composition and environmental factors in Lake Donghu [J]. Microbial Ecology, 2007, 54(2): 290—297
[5] Bell T, Ager D, Song J I, et al. Larger islands house more bacterial taxa [J]. Science, 2005, 308(5730): 1884
[6] Ni J J, Yu Y H, Hu H H. Examination of bacterial microbial community structure from Oncomelania hupensis by PCR-DGGE [J]. Sichuan Journal of Zoology, 2010, 29(5): 546—549 [倪加加, 余育和, 胡合华. 钉螺体内细菌群落结构PCR-DGGE分析方法的建立. 四川动物, 2010, 29(5): 546—549]
[7] Ni J J, Yu Y H, Feng W S, et al. Impacts of algal blooms removal by chitosan-modified soils on zooplankton community in Taihu Lake, China [J]. Journal of Environmental Sciences, 2010, 22(10): 1500—1507
[8] Ni J J, Yu Y H, Zhang T L, et al. Comparison of intestinal bacterial communities in grass carp, Ctenopharyngodon idellus, from two different habitats [J]. Chinese Journal of Oceanology and Limnology, 2012, 30(5): 757—765
[9] Boon N, De Windt W, Verstraete W, et al. Evaluation of nested PCR-DGGE (denaturing gradient gel electrophoresis) with group-specific 16S rRNA primers for the analysis of bacterial communities from different wastewater treatment plants [J]. FEMS Microbiology Ecology, 2002, 39(2): 101—112
[10] Gafan G P, Spratt D A. Denaturing gradient gel electrophoresis gel expansion (DGGEGE) – an attempt to resolve the limitations of co-migration in the DGGE of complex polymicrobial communities [J]. FEMS Microbiology Letters, 2005, 253(2): 303—307
[11] Sánchez O, Gasol J M, Massana R, et al. Comparison of different denaturing gradient gel electrophoresis primer sets for the study of marine bacterioplankton communities [J]. Applied and Environmental Microbiology, 2007, 73(18): 5962—5967
[12] Mühling M, Woolven-Allen J, Murrell J C, et al. Improved group-specific PCR primers for denaturing gradient gel electrophoresis analysis of the genetic diversity of complex microbial communities [J]. ISME Journal, 2008, 2(4): 379—392
[13] Ni J J, Yu Y H, Wu H H, et al. Effects generated by different band etraction methods in the analysis of DGGE profile [J]. Acta Hydrobiologica Sinica, 2012, 36(5): 1009—1011 [倪加加, 余育和, 吴含含, 等. 不同 DGGE谱带信息提取方法对分析结果的影响. 水生生物学报, 2012, 36(5): 1009—1011]
[14] Muyzer G, Smalla K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology [J]. Antonie van Leeuwenhoek, 1998, 73(1): 127—141
[15] Ercolini D, Hill P J, Dodd C E. Bacterial community structure and location in Stilton cheese [J]. Applied and Environmental Microbiology, 2003, 69(6): 3540—3548
[16] Ercolini D. PCR-DGGE fingerprinting: novel strategies for detection of microbes in food [J]. Journal of Microbiological Methods, 2004, 56(3): 297—314
[17] Fischer S G, Lerman L S. DNA fragments differing by single base-pair substitutions are separated in denaturing gradient gels: correspondence with melting theory [J]. Proceedings of the National Academy of Sciences of the United States of America, 1983, 80(6): 1579—1583
ASSESSING THE RELIABILITY OF DGGE BY RETRIEVING AND SEQUENCING DNA FRAGMENTS IN GEL BANDS
NI Jia-Jia1,2and YU Yu-He1
(1. Key Laboratory of Biodiversity and Conservation of Aquatic Organisms, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
To assess the reliability of DGGE, the V3 region fragments of prokaryotic 16S rDNA sequences were amplified by PCR and separated by DGGE with 40%—70% denaturing gradient in present study, and the differences of DNA sequences retrieved from DGGE bands were compared, and the concept of DGGE reliable index (RI) that was calculated by the equation RI = (Nss– Nsd+ Ndd– Nds)/Ns, was introduced. The results showed that the probability of a single band retrieving similar sequences (the sequence diversity of the V3 region of 16S rDNA is less than 4 bp) was 64.7%, and that the bands located at the same level of denaturing gradient could be considered as the same OTU. The probability that different bands retrieve similar sequences was 10.5%. The DGGE reliable index was 74.8%. Therefore, the results of DGGE still reflect the molecular diversity of environmental microbiota, although there was a disparity comparing with theoretical expectations.
DGGE reliable index; DNA fingerprinting; Denaturing gradient gel electrophoresis (DGGE); Microbial community structure
Q-331
A
1000-3207(2014)03-0454-05
10.7541/2014.64
2013-05-03;
2014-02-27
国家重点基础研究发展计划(2009CB118705); 国家自然科学基金(30970358, 31071896)资助
倪加加(1986—), 男, 山东潍坊人; 博士研究生; 主要从事微生物分子生态学研究。E-mail: nijiajia2005@126.com
余育和, E-mail: yhyu@ihb.ac.cn

