利用噬菌体展示技术淘选草鱼呼肠孤病毒的单链抗体
张 凤李周全闫利明,罗绍祥钟利桥
张晓华1袁 丽1方 勤3戴和平1
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049; 3. 中国科学院病毒研究所, 病毒学国家重点实验室, 武汉 430071)
利用噬菌体展示技术淘选草鱼呼肠孤病毒的单链抗体
张 凤1,2李周全1,2闫利明2,3罗绍祥1,2钟利桥1,2
张晓华1袁 丽1方 勤3戴和平1
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049; 3. 中国科学院病毒研究所, 病毒学国家重点实验室, 武汉 430071)
草鱼呼肠孤病毒(GCRV)是引起我国大面积草鱼幼鱼出血病暴发的主要病原, 其外衣壳蛋白 VP5和VP7在病毒入侵宿主细胞过程中起着至关重要的作用。研究以原核表达的VP7、全长VP5、VP5的N端片段及C端片段为靶蛋白, 利用已构建的噬菌体展示单链抗体文库进行淘选。经过3轮淘选后, 共获得7个针对VP7、VP5、VP5N和VP5C的单链抗体。经过验证, 识别原核表达的VP7的两个单链抗体能够成功识别天然GCRV病毒。此结果对于进一步研究GCRV与宿主细胞的相互作用机理奠定了基础。
草鱼呼肠孤病毒; VP5; VP7; 噬菌体展示; 单链抗体
草鱼呼肠孤病毒(Reovirus of grass carp, GCRV)是我国分离的第一株鱼类病毒[1,2], 隶属水生呼肠孤病毒属, 也是该属中致病性最强的一种病毒, 是引起我国大面积草鱼幼鱼出血病暴发的主要病原体,死亡率高达 80%, 给我国水产养殖业带来了极大的经济损失[3]。除了草鱼, GCRV 还能感染青鱼(Mylopharyngodon piceus)、麦穗鱼(Pseudorasbora parva)、布氏条(Hemicculter leuciclus)、稀有鲫(Gobiocypris rarus)、鲢(Hypophthalmichthys molitrix)等[4—6]。目前对于该病的防治仍以预防为主, 该病毒与宿主细胞作用的分子机理亟待揭示, 以期为草鱼出血病的防治提供新的突破。
GCRV为二十面体对称的球形颗粒, 基因组由11条(S1-S11)分段的双链RNA组成[2], 编码12种蛋白质, 包括7种结构蛋白(VP1-VP7)和5种非结构蛋白(NS1-NS5)[7]。其中, 结构蛋白VP5和VP7以200个异源六聚体的形式构成了病毒的外层衣壳蛋白, 其作用可能与病毒侵染及进入细胞的方式有关[8,9]。目前已有报道, 针对VP5和VP7的多抗血清具有中和GCRV的效应[10—12], 但是多抗血清中的多克隆抗体对病毒识别的位点多且杂, 无法特异性地研究病毒与细胞的结合及侵染过程[13]。杨倩等[14]利用传统杂交骨髓瘤方法得到了识别VP7蛋白的单克隆抗体,为建立快速检测GCRV的方法奠定了基础。但是传统的单克隆抗体制备往往价格高昂, 制备周期长,并且需要免疫小鼠, 因此限制了其大批量的生产和应用。
噬菌体展示单链抗体主要通过将工程化的抗体基因片段插入到噬菌体外壳蛋白基因中, 达到抗体与外壳蛋白融合表达并展示于噬菌体表面的目的。通过几轮对目的抗原的淘选, 目标抗体即可快速且廉价地获得[15], 此抗体在大肠杆菌中表达分泌, 产量也是无限的; 另外, 由于不需要免疫动物, 通过噬菌体展示技术获得单克隆抗体的方法更方便快捷。
本研究利用本实验室构建的噬菌体展示鼠源天然单链抗体文库, 以草鱼呼肠孤病毒外壳蛋白VP5和VP7为抗原进行淘选, 共获得了7个针对VP5和VP7的单链抗体。经过验证, 其中两个识别原核表达VP7的单链抗体能够成功识别天然GCRV病毒。这两个单链抗体将成为后续进一步研究病毒与细胞相互作用的有效工具。
1 材料与方法
1.1 质粒载体及菌种
库容量1.2×109的鼠源天然大容量单链抗体噬菌体展示文库由本实验室构建[16], 噬菌体载体pCANTAB5E及其宿主菌大肠杆菌TG1购自Pharmacia公司。采用pRSETA原核表达载体构建的GCRV VP5及VP7重组表达质粒及菌株BL21(DE3)- pLysS来自中国科学院武汉病毒研究所方勤老师实验室[17,18]。
1.2 实验材料
PCR引物为武汉擎科公司合成, 限制性内切酶XhoI和PstI购自Takara公司, PfuDNA聚合酶和T4DNA连接酶为Fermentas公司产品。HRP/Anti-M13单克隆抗体及HRP标记的鼠抗His-tag单克隆抗体购自Novagen公司, HRP标记的兔抗E tag多克隆抗体为Abcam公司产品。质粒提取试剂盒及DNA胶回收试剂盒购自北京百泰克公司。大肠杆菌培养基2YT、SOBAG和LB配方见Pharmacia和Novagen公司说明书, 大肠杆菌周质空间混合物提取缓冲液1×TES和1/5×TES缓冲液配方见Pharmacia公司说明书。氨苄青霉素(Amp)购自Amresco公司, 工作浓度为100 μg/mL; 氯霉素(Chl)购自Genview公司, 工作浓度为50 μg/mL。PBSM为含4%脱脂牛奶的PBS溶液, PBST为含0.1%Tween 20的PBS溶液。Western Blot反应底物DAB溶液的配方为: 10 mL Tris-HCl (pH 7.6, 50 mmol/L), 10 μL H2O2, 6 mg DAB。ELISA底物TMB的配方为: 2.5 mL 0.1 mol/L醋酸钠溶液 (pH 6), 250 μL TMB (溶于二甲亚砜, 60 mg/mL), 10 μL H2O2,加蒸馏水至终体积25 mL。
1.3 VP5和VP7的诱导表达
VP5和VP7的表达参照张岚岚等的方法[17,18]。简述步骤如下: 将新鲜转化的BL21(DE3)-PlysSpRSETAVP7/VP5菌株接种于LB+Amp+Chl培养基中, 37℃培养至A600=0.8, 加入IPTG至终浓度为1 mmol/L, 30℃诱导过夜(约18h)。培养产物在4 , ℃8000 g离心15min, 分离菌体和培养基上清。弃上清,将沉淀以50 g/L的浓度重悬于纯化蛋白所用的1×binding buffer中, 冰浴条件下用超声波破碎仪破碎细胞, 然后在4 , ℃ 10000 g离心10min, 分别收集沉淀和上清。
将所获沉淀和上清与诱导前菌液一起, 进行十二烷基磺酸钠—聚丙烯酰胺凝胶电泳(SDS—PAGE),之后将分离的蛋白条带转移至硝酸纤维素膜(NC膜)上, 丽春红染色并标出marker的位置。洗净后用4%PBSM (脱脂奶粉的PBS溶液, 4%为质量体积比)室温封闭NC膜1h, PBS洗三次之后, 用HRP标记的鼠抗His tag孵育1h, PBST和PBS各洗三次, DAB底物溶液中显色, 检测目的蛋白的表达情况。
参照pET载体系统使用说明书, 分别采用自然条件和尿素溶解包涵体的方式纯化VP5和VP7蛋白。尿素变性纯化的蛋白采用逐渐减低尿素浓度法使蛋白复性, 得到可溶的目的蛋白。
1.4 VP5N和VP5C的克隆表达
根据Genbank中公布的VP5基因序列(Accession No.为AF403392), 设计两对带有XhoI和PstI酶切位点的特异性引物(表1), 以pRSETAVP5质粒为DNA模版, 用PCR分别扩增VP5的N端片段(VP5N)和C端片段(VP5C)。用XhoⅠ和Pst I将VP5N和VP5C的PCR产物进行双酶切后, 与同样双酶切的pRSETA质粒载体连接, 转化到大肠杆菌BL21(DE3)–pLysS中,通过LB+Amp+Chl平板筛选, 得到阳性克隆株(称为BL21pRSETAVP5N/C)。BL21pRSETAVP5N/C的表达纯化与VP5的表达纯化步骤相同, 参见VP5的表达纯化。
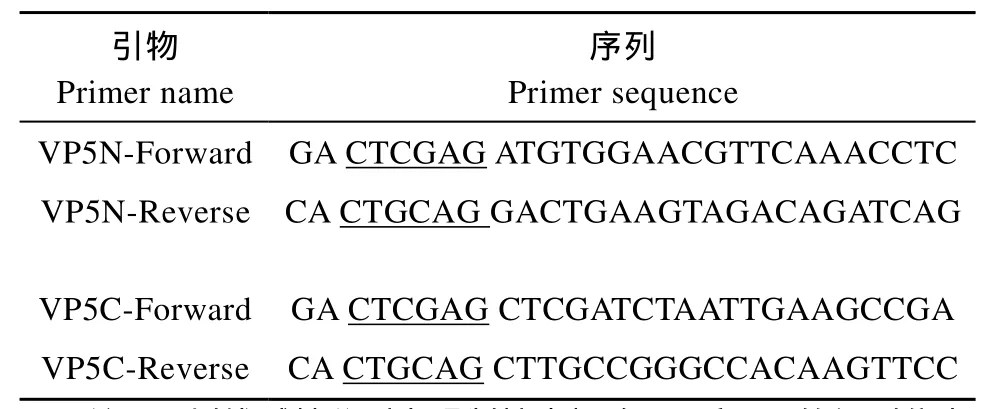
表 1 VP5N和VP5C片段PCR扩增引物Tab. 1 PCR primers for VP5N and VP5C
1.5 纯化VP7、VP5、VP5N和VP5C对小鼠天然抗体库的淘选
本实验室构建的大容量天然(Naive)噬菌体展示单链抗体文库以噬菌粒形式保存在宿主菌中, 在进行淘选之前, 需加入辅助噬菌体M13K07将其拯救成展示于PIII蛋白末端的噬菌体单链抗体(phagescFv)形式。具体方法见Barbas, et al.[19]的叙述。将VP7等抗原蛋白通过与树脂结合的方式固定, 具体操作为: 取20 μL NTA His Bind树脂(Novagen)按照说明书进行清洗、离子化和平衡之后, 分别加入10 μg用结合缓冲液稀释至200 μL的带有His标签的VP7等目的蛋白, 4℃放置过夜。为尽量减少NTA的基质效应对淘选的影响, 先将拯救好的噬菌体展示抗体库与等体积的空载树脂预保温15min, 然后将未结合的抗体库转移到VP7等抗原包被过的树脂中,于室温条件下微震荡孵育2h。接下来用蛋白纯化所用的漂洗缓冲液(含0.1%Tween-20)洗10次(3min/次),再用不含Tween-20的漂洗缓冲液洗10次(3min/次)。最后用1×洗脱缓冲液洗脱20min, 立即将洗脱下来的phage加入新鲜制备的大肠杆菌TG1(A600=0.3—0.5)中, 于37℃保温1h后涂布SOBAG平板, 30℃过夜生长。第2天用2YT-AG培养基(AG表示含100 mg/L Amp, 2%Glucose)刮下平板上的菌落, 拯救成噬菌体形式抗体后进行下一轮淘选, 如此共进行3轮淘选。
1.6 ELISA鉴定淘选阳性克隆
从最后一轮淘选的平板上随机挑取单克隆至96孔细胞培养板, 培养并拯救其形成phage-scFv, 对原核表达的VP7等靶蛋白分别进行ELISA检测, 方法如下: 将纯化的VP7等靶蛋白用PBS稀释至1000 ng/mL, 以100 μL/孔的量加入96孔板, 37℃包被1h, PBS洗三次; PBSM封闭1h后, 用PBS洗三次;加入100 μL用PBSM 1∶1稀释的含有phage-scFv的培养基上清, 37℃保温1h; PBST和PBS分别洗板三次, 加入100 μL PBSM稀释的HRP/Anti-M13单克隆抗体(1 5000), 37℃保温1h; PBST和PBS再分别洗板三次, 加入TMB底物显色, 10min后加25 μL 2 mol/L H2SO4终止反应, 酶标仪读取450nm的吸光值。
初步鉴定为阳性克隆后, 需确认scFv识别的是目的蛋白序列, 而非载体序列。方法为: 分别用目的蛋白及同载体的其他蛋白包被ELISA板, 重复上面的ELISA过程。确定为阳性克隆后, 再诱导表达为可溶性单链抗体形式进行下一步分析, 可溶性单链抗体在大肠杆菌中的表达参见张晓华等的方法[20], 简述如下: 离心获取IPTG过夜诱导后的菌体, 加入1×TES缓冲液充分重悬菌体, 再加入1/5×TES缓冲液混匀, 冰浴30min; 12000 r/min离心, 上清即为含∶有可溶性单链抗体的大肠杆菌周质空间提取物。不能够形成可溶性单链抗体的菌株, 直接取含有phage-scFv的培养基上清完成后续试验。
将最终确定为阳性克隆的单链抗体, 使用通用引物S1: 5′-GACCATGATTACGCCAAGC-3′进行基因测序(华大公司)。
1.7 ELISA和 Western Blot分析阳性单链抗体对原核表达抗原的识别
分别将原核表达纯化的VP7等抗原蛋白进行12%的SDS-PAGE电泳变性, 恒定电压140伏, 90min。将变性蛋白条带电转移到硝酸纤维素膜(NC膜)上, 恒定电流100 mA, 持续时间1h。转膜后将膜用PBSM封闭1h, PBS洗三次后, 加入用PBSM稀释的抗体, 37℃保温孵育1h; PBST和PBS分别洗膜三次, 加入PBSM稀释(1∶5000)的E tag抗体(Abcam公司), 37℃保温孵育1h, PBST和PBS分别洗膜三次,加入DAB显色液显色。
1.8 Western Blot和Dot Blot分析阳性单链抗体对真核细胞培养病毒的识别
用GCRV病毒感染草鱼头肾细胞(CIK细胞), 并获取含有病毒的细胞裂解液上清, 具体制备方法参见张岚岚等[17]。分别取细胞裂解液上清0.5、1、2、4、8 μL, 加入等体积2×loading buffer混匀, 煮沸8min后上样, 进行10% SDS-PAGE电泳分析。另外,取未经GCRV感染的CIK细胞裂解液上清8 μL进行同样操作, 作为阴性对照。后续过程与前面Western Blot检测抗体识别变性原核表达抗原相同, 区别在于, 延长PBST和PBS的洗涤时间, 以免非特异吸附影响后面的化学发光显色。Dot Blot检测步骤如下,分别取两份0.1、0.5、1、2 μL含病毒的细胞裂解液上清, 一份直接点至NC膜; 另一份加入等体积不含甘油和溴酚蓝的2 × SDS-PAGE上样缓冲液 [配方为100 mmol/L Tris-Cl (pH 6.8), 4% (m/v)SDS, 200 mmol/L DTT]混匀, 煮沸8min后, 也点至NC膜上。晾干后,用PBSM将膜封闭1h, 接着加入PBSM稀释的scFv抗体, 37℃保温1h, PBST和PBS各洗三次(10min/次),然后加入相应二抗, 37℃保温1h, PBST和PBS各洗三次(10min/次), 加入化学发光底物显色。
2 结果
2.1 VP5、VP7、VP5N和VP5C的表达与纯化
GCRV外壳蛋白VP5大小约为68 kD[21], 一方面为了易于原核表达, 另一方面, 也期望通过对截短蛋白的淘选获取识别全长的单链抗体,利用 SWISSMODEL软件(http://swissmodel.expasy.org/workspace/index.php?func= workspacemodelling&userid)对其疏水性等性质进行分析后, 将其分段克隆至pRSETA 载体, 分别命名为 VP5N 和VP5C(序列长度分别为 1110和 1134 bp,中间约有 300个碱基的重合), 与 VP5、VP7同时进行诱导表达。用1 mmol/L的IPTG诱导过夜后, 对破菌上清和沉淀进行Western Blot分析, 结果(图1)显示, 只有VP7诱导后, 在破菌上清中有较多可溶形式的目的蛋白, VP5、VP5N和VP5C均集中在包涵体沉淀中, 上清中含量很少。故 VP7在非变性条件下得到了有效纯化,而VP5、VP5N和VP5C均需在尿素变性条件下才能获得纯化蛋白, 最后通过逐渐降低尿素浓度法使其复性。图1显示纯化的VP5、VP5N和VP5C在对应的蛋白理论大小下面还有几条较弱条带, 可能是稀有密码子处终止导致的表达不完全造成的, 但这些不完全表达的片段所占含量较小, 对后续淘选过程应该不会有较大影响。

图1 VP7、VP5、 VP5N、VP5C原核表达与纯化的SDS-PAGE与Western Blot分析结果Fig. 1 SDS-PAGE and Western Blot analysis of expression and purification of four antigens (A)VP5; (B) VP5N; (C) VP7; (D) VP5C
2.2 针对原核表达VP7、VP5、VP5N和VP5C单链抗体的淘选及序列分析
以纯化的重组VP7、VP5、VP5N和VP5C为靶蛋白, 分别对本实验室构建的非免疫小鼠单链抗体库进行三轮淘选。在淘选中, 用His Bind 树脂代替传统的免疫管固定抗原, 这一做法的优越处在于,带有 His Tag的抗原蛋白通过 His Tag序列与 His Bind树脂的 Ni2+结合, 而另一端的目的蛋白就游离于溶液中, 更容易保持它原有的结构。淘选的结果是分别得到了两个针对VP7(19B7、P1A3)、VP5N(25G10、24B6)和 VP5C(26F4, 26D9)的单链抗体, 及一个针对VP5(29E12)的单链抗体。利用S1通用引物对编码单链抗体的基因序列进行测序, 翻译后的单链抗体氨基酸序列比对表明, 抗体序列差异主要集中于重链互补决定区(CDR区), 具体差异列于表2。另外,针对VP5蛋白的单链抗体29E12重链可变区(VH)缺失, 仅含有轻链可变区片段(VL), VL的CDR序列也示于表2。

表 2 scFv抗体重链可变区CDR的氨基酸序列(29E12为轻链可变区)Tab. 2 Amino acid sequence of the VH region complementary determinant regions (CDR) of isolated single-chain variable (scFv) antibodies (VL for 29E12)
对所得阳性单链抗体进行可溶性表达, 分别收集周质空间、破菌上清和破菌沉淀, 对其进行SDS-PAGE及Western Blot分析, 结果显示除了针对VP7的scFv P1A3, 针对VP5的scFv 29E12不能进行可溶性表达外, 其余阳性克隆经过IPTG诱导, 均能表达出可溶性单链抗体。对于不能形成可溶性抗体的 scFv P1A3和 29E12, 直接离心获取含有phage-scFv的培养基上清, 完成后续实验。
2.3 单链抗体对原核表达病毒蛋白特异性结合的分析
为确定所获单链抗体的特异性, 分别用 0—2 μg的VP7、VP5、VP5N和VP5C抗原包被进行ELISA检测, 每个样品设三个平行重复, 并选用同载体序列不相关的蛋白作为对照, 进行同样操作。结果如图2所示, 所有淘到的单链抗体对相应抗原进行ELISA 检测时, 在同一抗体浓度下, 随着各自抗原蛋白包被量的增加, A450都随之增加, 而对照蛋白, 则完全无此趋势,说明淘选得到的阳性单链抗体对相应抗原特异性良好。其中, scFv 19B7、P1A3对VP7, 26F4、26D9对VP5C, 25G10对VP5N的最低检测浓度均约为 50 ng; scFv 29E12对VP5的最低检测浓度稍高, 约为 100 ng; scFv 24B6对VP5N的最低检测浓度最低, 约为25 ng。同时, western结果显示(图3), 所有淘得的抗体均能够识别变性的原核表达抗原, 说明这些抗体识别的均为线性表位。另外, 识别VP5N、VP5C 的单链抗体 24B6、25G10、26F4、26D9, 对全长VP5及另一区段VP5的检测结果表明, 识别 VP5N 的 scFv 25G10也能够成功识别变性与非变性的VP5和VP5C(图2C、3B所示), 推测可能的原因是25G10的识别位点位于 VP5的中间区域, 同时VP5N的C端和VP5C的N端是同一段序列(约为100个氨基酸),故25G10可以同时识别VP5、VP5N、VP5C。但是,识别 VP5N的 scFv 24B6及识别 VP5C的 scFv 26F4、26D9均不能识别 VP5的另一区段及全长VP5(图3B、3D), 可能的解释是, 这些抗体的识别位点都不仅仅是目的蛋白序列, 而是质粒载体与目的蛋白结合处的一段序列。

图2 阳性单链抗体与对应抗原的ELISA分析结果Fig. 2 ELISA analysis of binding specificity of positive scFv antibodies to prokaryotic expressed antigens
2.4 单链抗体对真核表达病毒蛋白的识别
为确定所获单链抗体能否识别真核表达的病毒蛋白VP7和VP5, 将GCRV 感染的CIK细胞裂解液上清0.5—8 μL梯度上样进行Western Blot检测,未经GCRV感染的CIK细胞裂解液上清8 μL作为阴性对照, 原核表达VP7、VP5各4μL作为阳性对照。Western结果表明针对VP7的scFv 19B7和P1A3能够识别GCRV的变性VP7(图4A、4B所示), 而针对VP5、VP5N和VP5C的所有单链抗体均不能识别 GCRV的 VP5(结果未显示), 对此, 推测其原因,可能与真核表达的病毒VP5蛋白有着原核表达不具有的翻译后修饰有关。另外, 对于能够识别变性GCRV VP7的scFv 19B7和P1A3, 为了确认其能否识别非变性GCRV VP7, 分别取GCRV感染的CIK细胞裂解液上清及未经GCRV感染的CIK细胞裂解液上清进行 ELISA实验。但是, 背景值很高, 也没有明显阳性结果。推测原因是细胞裂解液上清, 虽经过一定的浓缩, 其蛋白种类仍然很多, 而 ELISA的包被量很有限, 目的蛋白不能有效固定。相反, NC膜的蛋白吸附量要大得多, 故尝试用Dot Blot实验确认scFv 19B7和P1A3能否识别非变性GCRV VP7。分别取变性与非变性GCRV感染CIK细胞裂解液上清0.1—2 μL梯度点膜进行Dot blot实验。0.1—2 μL变性与非变性原核表达VP7也点膜, 作为阳性对照, 0.1—2 μL未经GCRV感染的CIK细胞裂解液上清作为阴性对照。结果(图4 C3)显示, 随着样品中 GCRV浓度的增加, 化学信号也在增强,而未经GCRV感染的CIK细胞裂解液上清则无此现象, 说明scFv 19B7能够与非变性的GCRV特异性结合。根据图4 C5、C6, 在同一上样量条件下, 非变性 VP7的化学信号比变性的要强, 推测是因为, 蛋白经过变性后, 与 NC膜的结合力下降, 或者在后面长时间的PBST的漂洗中, 更容易脱落。这也解释了在GCRV感染的CIK细胞裂解液上清中, 非变性的呈现出了弱信号,而变性后的却没有信号。
而scFv P1A3的dot blot实验中则显示出很强的非特异性吸附(结果未显示), 因此无法确认其能否与非变性GCRV结合。
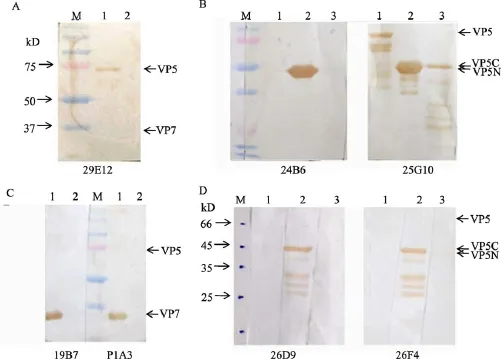
图3 单链抗体对原核表达抗原Western Blot结果Fig. 3 Western Blot assay of scFv antibodies with prokaryotic expressed antigen
3 讨论
GCRV是水生呼肠孤病毒属中致病性最强的一种病毒, 由外部的两层蛋白衣壳与内部的双链环状RNA构成, 最外层的核衣壳是由200个VP5和VP7三聚体分子组成的异源二聚体构成。根据方勤等的报道[8], VP5和VP7与哺乳动物正呼肠孤病毒(MRV)的μ1、σ3蛋白分别有20%和40%的同源性。推测其与MRV的μ1、σ3蛋白可能也有着相似的功能, 即与病毒吸附、侵染及进入细胞的方式有关[9]。
本研究利用本实验室构建的大容量天然单链抗体库对原核表达的VP5、VP7及截短的VP5N和VP5C进行淘选, 共获得了7个单链抗体。其中针对VP7、VP5N和VP5C的各有两个, 针对VP5的有一个, 它们均能够同时识别对应的变性与非变性的原核表达抗原。但是检测GCRV感染的细胞裂解液上清中的GCRV病毒时, 发现针对原核表达VP5、VP5N和VP5C淘到的单链抗体均不能识别天然GCRV中的VP5。推测其原因, 可能与真核细胞表达的病毒VP5蛋白有着原核表达不具有的翻译后修饰有关。
针对原核表达VP7淘到的两个scFv 19B7和P1A3能够识别GCRV的P7, 其中19B7可以同时识别变性与非变性的GCRV的VP7, 说明其识别位点为线性表位。P1A3可识别变性的病毒VP7(非变性的Dot Blot实验因为非特异性吸附太强, 未能得出确定结论)。在这一单克隆抗体的基础上, 接下来可以继续探究, 该抗体对病毒是否有中和效应, 同时参考哺乳动物σ3蛋白的功能区, 将GCRV的VP7分段表达, 应用该抗体揭示病毒与细胞的结合位点, 为后面深入研究提供理论基础。

图4 19B7、P1A3检测病毒VP7的Western Blot 和Dot blot结果Fig. 4 Western Blot and dot blot analysis of capsid protein VP7 in GCRV virus
本研究首次将噬菌体展示单链抗体库应用于GCRV外壳蛋白抗体的淘选中, 并成功获得了可以同时识别变性与非变性的GCRV VP7的单链抗体。相比较传统的单克隆制备方法, 它有着很多优势:淘选周期短, 获得抗体迅速; 同时因为是在大肠杆菌中表达, 抗体的产量是无限的, 并且也是廉价的;另外, 因为噬菌体展示抗体属于工程抗体, 易于后面的基因操作, 比如可通过在单链抗体基因序列后面连上某些酶基因, 直接将二者融合表达, 检测时,就可以省略二抗的步骤, 一抗孵育后即可加入该酶的反应底物, 整个过程方便快捷[22]。
另外, 构建的pRSETVP5N/C载体成功大量表达了VP5N和VP5C, 虽然针对它们淘选所获得的单链抗体不能够识别GCRV中的VP5衣壳蛋白, 但是原核表达的VP5N、VP5C对于以后研究VP5的结构与功能将会大有裨益。
总结来说, 本研究利用实验室构建的大容量天然噬菌体展示单链抗体文库成功获取了能够识别GCRV VP7的单链抗体, 此抗体将成为后面进一步研究病毒与细胞相互作用的有效工具。
[1] Chen Y X, Jiang Y L. Studies on the morphologycal and physico-chemical characterization of the hemorrhagic virus of grass carp [J]. Chinese Science Bulletin, 1983, 28: 1138—1140 [陈燕新, 江育林. 草鱼出血病病毒形态结构及其理化特性的研究. 科学通报, 1983, 28: 1138—1140]
[2] Ke L H, Fang Q, Cai Y Q. Characteristics of a novel isolate of grass carp hemorrhagicvirus [J]. Acta Hydrobiologica Sinica, 1990, 14(2): 153—159 [柯丽华, 方勤, 蔡宜权. 一株新的草鱼出血病病毒分离物特性. 水生生物学报, 1990, 14(2): 153—159]
[3] Rangel A A, Rockemann D D, Hetrick F M, et al. Identification of grass carp haemorrhage virus as a new genogroup of aquareovirus [J]. Journal of General Virology, 1999, 80: 2399—2402
[4] Ding Q Q, Yu L F, Ke L H, et al. Study on infecting other fishes with grass carp hemorrhagic virus [J]. Virologica Sinica, 1991, 6(4): 371—373 [丁清泉, 余兰芬, 柯丽华, 等.草鱼出血病病毒对其他鱼的感染性研究. 中国病毒学, 1991, 6(4): 371—373]
[5] Wang T H, Liu P L, Chen H X, et al. Preliminary studies on the susceptibility of Gobiocypris rarus to hemorrhagic virus of grass carp [J]. Acta Hydrobiologica Sinica, 1994, 18(2): 144—149 [王铁辉, 刘沛霖, 陈宏溪, 等. 稀有鲫对草鱼出血病病毒敏感性的初步研究. 水生生物学报, 1994, 18(2): 144—149]
[6] Jiang Y, Ahne W. Some properties of the etiological agent of the hemorrhagic disease of grass carp and black carp [A]. In: Ahne W, Kurstak E (Eds.), Viruses of Lower Vertebrates [C]. Berlin: Springer –Verlag, 1989, 227—240
[7] Fang Q, Shah S, Liang Y Y, et al. 3D reconstruction and capsid protein characterization of grass carp reovirus [J]. Science China Life Sciences, 2005, 48: 593—600
[8] Cheng L P, Fang Q, Shah S, et al. Subnanometer-resolution structures of the grass carp reovirus core and virion [J]. Journal of Molecular Biology, 2008, 382: 213—222
[9] Nibert M L, Schiff L A. Reoviruses and their replication [A]. In: Knipe D M, Howley P M (Eds.), Fields Virology [C]. Philadelphia, Lippincott Williams and Wilkins. 2001, 1679—1728
[10] Shao L, Sun X Y, Fang Q. Antibodies against outer-capsid proteins of grass carp reovirus expressed in E. coli are capable of neutralizing viral infectivity [J]. Virology Journal, 2011, 8: 347
[11] He Y X, Yang Q, Xu H X, et al. Prokaryotic expression and purification of grass carp reovirus capsid protein VP7 and its vaccine potential [J]. African Journal of Microbiology Research, 2011, 5(13): 1643—1648
[12] He Y X, Xu H X, Yang Q, et al. The use of an in vitro microneutralization assay to evaluate the potential of recombinant VP5 protein as an antigen for vaccinating against grass carp reovirus [J]. Virology Journal, 2011, 8: 132
[13] Reading S A, Dimmock N J. Neutralization of animal virus infectivity by antibody [J]. Archives of Virology, 2007, 152: 1047—1059
[14] 杨倩, 曹海鹏, 和永杏, 等. 一株草鱼呼肠孤病毒检测用单克隆抗体的制备及其特性. 中国免疫学杂志, 2012, 28(3): 240—241
[15] Hoogenboom H R, de Bruïne A P, Hufton S E, et al. Antibody phage display technology and its applications [J]. Immunotechnology, 1998, 4(1): 1—20
[16] Rao Y, Zhong L Q, Liao T, et al. Novel recombinant monoclonal antibodies for vitellogenin assays in cyprinid fish species [J]. Diseases of Aquatic Organisms, 2010, 93: 83—91
[17] Zhang L L, Shen J Y, Lei C F, et al. Expression of outer capsid protein VP5 of grass carp reovirus in E. coli and analysis of its immunogenicity [J]. Virologica Sinica, 2009, 24(6): 545—551
[18] Zhang L L, Shen J Y, Lei C F, et al. High Level expression of grass carp reovirus VP7 protein in prokaryotic cells [J]. Virologica Sinica, 2008, 23(1): 51—56
[19] Barbas C F III, Burton D R, Scott J K, et al. Phage Display-A Laboratory Manual [M]. New York, Cold Spring Harbor Laboratory Press. 2001, 23—35
[20] Zhang X H, Dai L F, Dai H P. Studies on expression and biochemical characteristics of single chain fragment variable A1 against white spot syndrome virus of shrimp [J]. Acta Hydrobiologica Sinica, 2006, 30(2): 141—145 [张晓华, 戴玲芬, 戴和平. 抗对虾白斑综合症病毒的单链抗体 A1 的表达和生物化学特性. 水生生物学报, 2006, 30(2): 141—145]
[21] Attoui H, Fang Q, Jaafar F M, et al. Common evolutionary origin of aquareoviruses and orthoreoviruses revealed by genome characterization of golden shiner reovirus, grass carp reovirus, striped bass reovirus and golden ide reovirus (genus Aquareovirus, family Reoviridae) [J]. Journal of General Virology, 2002, 83: 1941—1951
[22] Dai H P, Gao H, Zhao X Y, et al. Construction and characterization of a novel recombinant single-chain variable fragment antibody against white spot syndrome virus from shrimp [J]. Journal of Immunological Methods, 2003, 279: 267—275
ISOLATION OF SCFV ANTIBODIES AGAINST GCRV USING PHAGE DISPLAY TECHNOLOGY
ZHANG Feng1,2, LI Zhou-Quan1,2, YAN Li-Ming2,3, LUO Shao-Xiang1,2, ZHONG Li-Qiao1,2,
ZHANG Xiao-Hua1, YUAN Li1, FANG Qin3and DAI He-Ping1
(1. State Key Laboratory of Freshwater Ecology and Biotechnology, Institution of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China; 3. State Key Laboratory of Virology, Wuhan Institute of Virology, Chinese Academy of Sciences, Wuhan 430071, China)
Grass carp reovirus (GCRV) is the main cause of severe outbreaks of hemorrhagic disease in fingerling and yearling grass carp. The virus outer capsid proteins, VP5 and VP7, have been shown to play critical roles in the invasion of host cells. In our study, prokaryotic-expressed VP7, VP5, and the N and C terminals of VP5 protein were used as targets to select the desired antibodies from phage-displayed scFv library. After 3 rounds of panning, we selected 7 antibodies against the prokaryotic-expressed antigens -- VP7, VP5, VP5N and VP5C. Furthermore, we found that 2 antibodies against VP7 out of the total 7 were able to recognize native GCRV particles. Our study should provide an insight into the interactions between GCRV and its host cells.
Reovirus of grass carp (GCRV); VP5; VP7; Phage display; scFv antibody
Q939.4
A
1000-3207(2014)03-0430-08
10.7541/2014.61
2013-04-26;
2014-01-17
国家重点基础研究发展计划(批准号: 2009CB118704)资助
张凤(1988—), 女, 安徽人; 硕士研究生; 主要研究方向为蛋白质工程。E-mail: zhangf8810@163.com
袁丽(1979—), 女, E-mail: liyuan@ihb.ac.cn; 方勤(1961—), 女, E-mail: qfang@wh.iov.cn

