通过L-乳酸脱氢酶1的上下游DNA序列鉴定Lactobacillus sp.DMDL 9010*
刘冬梅 费永涛 王盼 陈谷 肖性龙 吴晖 唐语谦
(华南理工大学轻工与食品学院,广东广州510640)
在蔬菜加工中产量最大的品种是发酵蔬菜,尽管有2000多年悠久历史,但是工业化程度低,存在食盐含量高和亚硝酸盐超标两个突出问题.亚硝酸盐是一种潜在的致癌物,摄入过量的食盐对健康不利,因此研究降低食盐和亚硝酸盐含量的方法,对生产安全的发酵蔬菜有重要的意义[1-2].笔者课题组的前期研究表明,添加纯种Lactobacillus casei subsp. rhamnosus 719的蔬菜发酵,能很好地抑制亚硝酸盐的产生[3],也可以大幅度降低其中的食盐含量[4];还从自然发酵的蔬菜中筛选得到一株乳酸菌DMDL 9010,添加到蔬菜中进行纯种发酵很好地抑制其中亚硝酸盐的积累,同时在不添加食盐的情况下,在24h内能完成蔬菜发酵过程.
应用于菌种鉴定的分子生物学技术中,最常用的方法有16S rDNA法、重复序列聚合酶链反应(REP-PCR)指纹分析法、限制性片段长度多态性聚合酶链反应(PCR-RFLP)分析法、DNA-DNA杂交法[5-9]等.分类学家普遍将16S rDNA相似性大于97%和99%作为鉴别微生物到属和种的依据[10],然而Fox等[11]利用16S rDNA法鉴定3株16S rDNA序列相似性达到99.5%的芽孢杆菌(Bacillus subtilis),通过DNA杂交试验才确认为3株为不同的菌种;因此单独利用16S rDNA进行菌种鉴定是不准确的.PCRRFLP法可以用于种到属的鉴别[5],当16S rDNA的序列相似性达到96%以上时,PCR-RFLP法就分辨不开,即酶切图谱难以分辨[12].REP-PCR法简单,分辨力强,能鉴别16S rDNA序列相似性在99.5%以上的菌株,但是此法易被污染以及产生错误,同时还需多重对照[13].DNA-DNA杂交在细菌分类鉴定过程中起着重要的作用,已成为菌种鉴定与分类的标准,国际细菌学分类委员会于1987年规定,不同细菌的基因组DNA杂交同源性在75%及以上,同时杂交分子的热解链温度差在5℃以内时,则属于同一菌种[14],然而,这种方法的局限性在于配对杂交耗时较长,同时需要同位素的参与,还需要建立一个新的中心数据库[15].因此,本研究在16S rDNA以及生理生化特征两种传统鉴别方法的基础上,利用生物信息学的方法鉴定了一株能有效抑制蔬菜中亚硝酸盐的菌株DMDL 9010.
1 材料与方法
1.1 实验材料
1.1.1 菌种来源
乳杆菌(Lactobacillus sp.)DMDL9010菌株,由华南理工大学轻工与食品学院食品安全试验室分离筛选得到,首次被鉴定为戊糖乳杆菌(名称为Lactobacillus pentosus DMDL 9010),并于 2011年 8月19日保藏于中国微生物菌种保藏管理委员会普通微生物中心(CGMCC),保藏编号为CGMCC NO. 5172.干酪乳杆菌鼠李糖亚种(Lactobacillus casei subsp.rhamnosus 719,简称LCR 719)由华南理工大学轻工与食品学院食品安全教研室保存,保加利亚乳杆菌LB 1.83(Lactobacillus delbrueckii subsp.bulgaricus)和嗜热链球菌ST 1.204(Streptococcus thermophilus)购自广东省微生物研究所菌种保藏中心,植物乳杆菌LP 8140购自CGMCC.
1.1.2 主要试剂及仪器
MRS培养基、琼脂,购自广东环凯微生物科技有限公司;DNA提取试剂盒、TBE电泳缓冲液、DNA Marker(D-2000),购自Takara公司;20%SDS,购自广州市鼎盈贸易有限公司;95%乙醇、碘液、异丙醇,购自天津市科密欧化学试剂开发中心;亚硝酸钠,购自天津市福晨化学试剂厂;其余试剂为进口或国产的分析纯或生化试剂.主要仪器:电泳仪,北京六一仪器厂生产;快速基因扩增仪(PCR),杭州博日科技有限公司生产.
1.2 实验方法
1.2.1 亚硝酸盐的检测
参考国标GB/T5009.33—2010《食品中亚硝酸盐与硝酸盐的测定》中的盐酸萘乙二胺法测定亚硝酸盐含量,稍有修改,不经过沉淀蛋白质的步骤,直接加2mL对氨基苯磺酸溶液,混匀,静置3~5min后各加入1 mL盐酸萘乙二胺溶液(2 g/L),加水至刻度,混匀,静置15min测定反应液的吸光度.
1.2.2 降解亚硝酸盐的反应
在15 mL试管中,加入10 mL含有10.000 mg/L NaNO2已灭菌的液体MRS培养基,接入5%(体积比)的DMDL 9010的种子液,于37℃密闭状况下,静置培养24h后灭菌,取样,按照1.2.1中方法分析亚硝酸盐含量(各做3个平行).亚硝酸盐含量表示为平均值±标准偏差.
1.2.3 菌株的分子生物学鉴定
(1)菌株的16S rDNA鉴定
取过夜培养的菌液0.1~1.5 mL,12000 r/min室温离心1min,弃上清液,沉淀重悬于0.6 mL的溶菌酶溶液中,颠倒混匀5~10次后置于37℃保温40min,利用试剂盒提取菌种基因组DNA;利用其模板DNA进行PCR扩增,扩增体系见表1.PCR扩增条件为:95℃预变性5min;94℃变性30s;56℃退火30s,72℃延伸2 min,30个循环;72℃延伸5 min.电泳检测回收并将扩增得到的片段进行DNA测序,通过与NCBI数据库中的序列BLAST比对,进行16S rDNA鉴定.同时,利用MEGA 5.0软件构建系统发育树,了解该菌与已知菌的同源性以及在进化过程中的位置.

表1 菌株DMDL 9010的16S rDNA PCR反应体系1)Table 1 16S rDNA PCR reaction system of strain DMDL 9010
(2)乳酸菌的分子生物学鉴定
由于传统鉴定方法不能准确地将亲缘关系较近的乳酸菌鉴别出来,笔者分析比较植物乳杆菌与戊糖乳杆菌在NCBI中的基因组序列,找到高度保守的一段编码L-乳酸脱氢酶1的DNA序列,通过序列比对找出植物乳杆菌与戊糖乳杆菌ldhL1基因上游和下游的相似DNA序列,设计两对引物:F1,TATC-CGTACTGTGTTTCCTC;R1,ACTAGAACCAACAGCGCCGT;F2,TAGGTGGCCTTTTCGGTAGC;R2,CTCGTCTATAGCAGACGGGC.利用设计的引物进行PCR扩增,反应体系见表1.ldhL1基因上游DNA片段PCR扩增的退火温度为58℃,下游DNA片段PCR扩增的退火温度为59℃,延伸时间都是1.5min,经过1%的琼脂糖凝胶电泳检测并利用DNA回收试剂盒回收两段DNA片段,然后对两段DNA序列进行测序,并利用生物信息学方法,将两段序列利用BLAST与NCBI数据库中已知的序列进行比对,分析比对结果同时结合该菌的生理生化特征来鉴别菌株DMDL 9010.
1.3 数据分析
实验数据和绘图均采用Microsoft Excel 2003数据分析软件.
2 结果与讨论
2.1 菌株DMDL 9010降解亚硝酸盐的效果
利用5株菌DMDL 9010、LCR 719、LB 1.83、ST 1.204、LP 8140降解MRS中亚硝酸盐,经过24 h的降解后,DMDL 9010降解效果最好,能将体系中10.000mg/L亚硝酸盐降解至检测不出来,LP 8140降解效果最差,仍残留(6.489±0.052)mg/L,经LCR 719、LB 1.83、ST 1.204降解后体系中亚硝酸盐的含量分别为(0.872±0.048)、(4.798± 0.555)、(3.274±0.308)mg/L.DMDL 9010的降解效果与LCR 719、LB 1.83、ST 1.204和LP 8140相比,具有显著性(P≤0.001).可见DMDL 9010具有非常良好的亚硝酸盐降解效果,将其用于蔬菜发酵中也有非常好的效果.
2.2 菌株DMDL 9010的16S rDNA分子鉴定
用DNA提取试剂盒提取该菌的基因组DNA,以基因组DNA为模版进行PCR扩增,然后用1%的琼脂糖凝胶电泳对扩增的DNA产物进行分析,结果如图1所示.利用PCR成功获得1500 bp左右的条带,然后将16SrDNA的片段送到Takara公司进行测序,序列长为1441bp,将16SrDNA基因序列利用BLAST软件同GenBank数据库中已知的乳杆菌属内标准菌株进行多序列比对,同时绘制系统发育树,结果见图2.
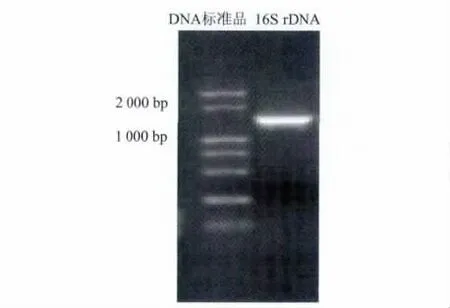
图1 PCR后DMDL 9010菌株的16S rDNA电泳图Fig.1 Gel electrophoresis of PCR-amplified 16S rDNA of strain DMDL 9010
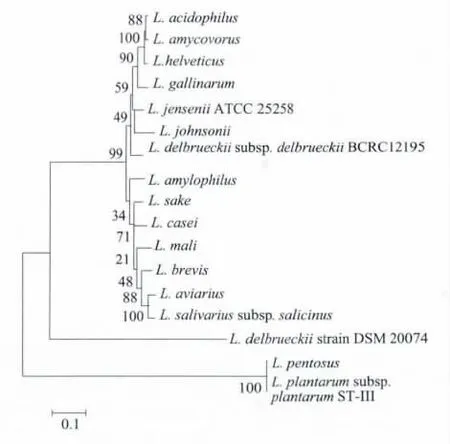
图2 基于16S rDNA基因序列建立的乳杆菌属系统发育树Fig.2 Phylogenetic tree of Lactobacilli based on 16S rDNA gene sequence
通过比对发现,菌株DMDL 9010的16S rDNA序列与植物乳杆菌和戊糖乳杆菌相似度达到99%,由此可以推断此菌株可能是植物乳杆菌或是戊糖乳杆菌.由图2可见,两种菌在进化的过程中有着较近亲缘关系.因此对16S rDNA序列分析不能提供足够的信息来鉴别它们,需要做进一步的生理生化鉴定.
2.3 菌株DMDL 9010的生理生化特征分析
对分离菌株进行的生理生化实验结果见文献[16].
经过对菌株DMDL 9010的生理生化特征进行分析,同时结合16S rDNA分子鉴定结果,发现其与《伯杰细菌鉴定手册》和《常见细菌系统鉴定手册》[17]中硬壁菌门(Firmicutes)杆菌纲(Bacilli)乳杆菌目(Lactobacillales)乳杆菌科(Lactobacillaceae)的生理生化特征一致[17].这进一步验证了16S rDNA分子生物学鉴定菌株方法的准确性及有效性.对该分离菌进行糖醇发酵试验,其中阿拉伯糖、纤维二塘、果糖、鼠李糖的发酵结果为阴性,半乳糖、葡萄糖、麦芽糖、核糖、山梨糖、蔗糖、海藻糖、木糖、棉籽糖、七叶灵、甘露糖的发酵结果为阳性.通过《伯杰细菌鉴定手册》和《常见细菌系统鉴定手册》发现植物乳杆菌和戊糖乳杆菌只有在是否能利用鼠李糖上有区别,戊糖乳杆菌标注是“d”,即80%~90%菌株阳性,植物乳杆菌是阴性.此外,有文献报道利用DNA-DNA杂交的方法成功地鉴别出植物乳杆菌与戊糖乳杆菌[18].因此,在对各种糖的利用方面,笔者得出结论:是否能利用木糖和甘油产酸可以作为鉴别两种菌的生理生化指标,3株戊糖乳杆菌中能在以鼠李糖为单一碳源生长的有1株,14株植物乳杆菌中有7株能利用鼠李糖,这与《伯杰细菌鉴定手册》存在矛盾,不能达成一致.可见,单一的生理生化指标在菌种鉴定方面有局限性.鉴于以上分析,利用新的鉴定方法来区别两种亲缘关系比较近的乳酸菌菌株显得非常必要.
2.4 利用保守L-乳酸脱氢酶1编码基因序列的分子生物学鉴定
笔者以ldhL1上下游序列为依据对菌株DMDL9010进行分子生物学鉴定相关结果,如图3-6所示.采用ldhL1上下游序列的原因及对相关结果的分析如下.
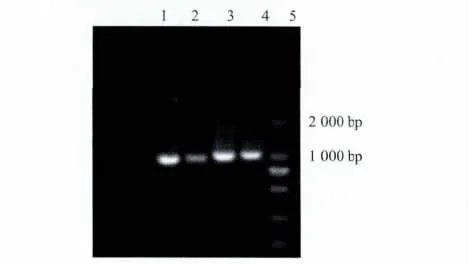
图3 PCR扩增ldhL1上下游DNA片段Fig.3 The upstream and downstream DNA product of ldhL1 amplified by PCR1,2—ldhL1下游片段;3,4—ldhL1上游片段;5—DNA标准品DL 2000

图4 菌株DMDL 9010的ldhL1上游DNA序列与NCBI中植物乳杆菌序列比对结果Fig.4 Alignment of ldhL1 upstream DNA sequence in strain DMDL 9010 with that of L.plantarum in NCBI

图5 菌株DMDL 9010的ldhL1上游序列与NCBI中戊糖乳杆菌序列比对Fig.5 Alignment of ldhL1 upstream DNA sequence in strain DMDL 9010 with that of L.pentosus in NCBI
鉴于植物乳杆菌与戊糖乳杆菌在进化上有较高相似性以及物种间的同源性,16S rDNA序列比对结果相似度为100%,同时两种菌的生理生化性质不能提供足够可靠的鉴别证据,无法有效地鉴别两种菌,但通过DNA序列分析发现两种菌具有高度保守的一段基因ldhL1(L-乳酸脱氢酶1编码基因),两种菌的ldhL1基因相似度达到90%以上,在进化过程中有较高的保守性[19].同时对NCBI数据库中已经测序的6株植物乳杆菌亚种(L.plantarum ST-III、L.plantarum WCFS1、L.plantarum 16、L.plantarum P-8、L.plantarum JDM1、L.plantarum ZJ316)与2株戊糖乳杆菌亚种(L.pentosus IG1、L.pentosus MP-10)的乳酸脱氢酶1的上游序列和下游序列差异性进行分析,发现6株L.plantarum和两株L.pentosus的上游序列由一段非编码序列和部分peptidyl-tRNA水解酶编码序列组成,对这两段序列在NCBI中DNA序列进行BLAST,发现两段序列分别在L.plantarum和L.pentosus菌种内具有较高的保守性(序列相似性99%)并且在两株菌之间差异较大(序列相似性只有78%).然后,同样地对它们的下游序列进行分析,6株植物乳杆菌的乳酸脱氢酶1序列下游有一段非编码序列和部分cell surface protein编码序列,两段序列在L.plantarum中是高度保守的(序列相似性99%),而2株戊糖乳杆菌的上游序列是一段非编码序列,BLAST发现具有种内保守性(序列相似性99%),并且两种菌的下游序列相似性较低.因此,利用两种菌共有的L-乳酸脱氢酶编码序列,即ldhL1的上游序列以及下游序列的不同作为鉴别两种亲缘关系较近乳酸菌的方法.利用引物Lps1-F、Lps1-R和Lps2-F、Lps2-R分别经过PCR扩增得到ldhL1上游序列和下游序列,然后利用1%的琼脂糖凝胶电泳检测,结果见图3,电泳条带较亮,较清晰.
将PCR扩增得到的片段进行测序,得到上游序列917 bp和下游序列843 bp.使用BLAST软件与NCBI数据库中的DNA序列进行相似性比对,将PCR扩增得到的片段进行测序,得到上游序列917bp和下游序列843bp.使用BLAST软件与NCBI数据库中的DNA序列进行相似性比对,上游序列与植物乳杆菌(L.plantarum)比对结果如图4所示.通过上游相似性比较发现,序列相似性为99%,identities 917/920,Gaps3/920(0%),Score 1679 b,即:在序列920bp中有917bp是相同的,只有3bp是不同的,得分是1679 b.同时,与L.pentosus序列的比对结果见图5,序列相似性78%,identities 719/926,Gap 32/926,Score只有534b.菌株DMDL 9010的上游序列比对结果显示,序列的位置在ldhL1的附近,序列包括部分peptidyl-tRNA水解酶编码序列以及一段非编码序列,与前面分析一致,同时由序列相似度可以初步判定菌株DMDL 9010为植物乳杆菌,而非戊糖乳杆菌.
图6是菌株DMDL 9010的下游序列与植物乳杆菌的比对结果,分析发现两株菌的序列相似性为99%,identities 840/845,Gaps4/845,Score 1530b,而与戊糖乳杆菌的序列比对发现相似性较低,在NCBI中BLAST没有戊糖乳杆菌出现.上述比对结果与之前分析一致,与植物乳杆菌相似较高的序列由cell surface protein和一段保守的非编码序列组成,同时与戊糖乳杆菌比对相似性较低.因此,可以鉴定菌株DMDL 9010是植物乳杆菌.

图6 菌株DMDL 9010的ldhL1下游序列与NCBI中植物乳杆菌序列比对ig.6 Alignment of ldhL1 downstream DNA sequence in strain DMDL 9010 with that of L.plantarum in NCBI
3 结论
从泡菜中分离到的菌株DMDL 9010具有很好的降亚硝酸盐的能力.利用传统的16S rDNA分子生物学鉴定方法和生理生化特征将分离的新菌株DMDL 9010鉴定到种的水平,但是无法在植物乳杆菌与戊糖乳杆菌之间做出准确的鉴别.笔者利用生物信息学的分析方法,找到这两种菌在进化过程中较为保守的序列,即L-乳酸脱氢酶1编码序列,以此为标记用PCR扩增其上游与下游相似度较低序列,对PCR产物进行测序以及序列比对分析,鉴定菌株DMDL 9010为植物乳杆菌.在传统菌种鉴定方法的基础上,本研究利用分子生物学以及生物信息学的方法鉴定了亲缘关系较近的植物乳杆菌与戊糖乳杆菌,为鉴别亲缘关系较近的菌株提供了一条新的思路.还需要对DMDL 9010的降解亚硝酸盐的关键酶进行研究,以阐明植物乳杆菌降解亚硝酸盐的机理.
[1] Akemi H,Kuniyasu O.Nitrate and nitrite contents in pickles of some vegetables[M].Tokyo:Nippon shokuhin kagaku Kogaku kaishi,1979:6-12.
[2] Toshirou H.The cause on the abnormal accumulation of nitrite in pickles of Chinese cabbage[M].Tokyo:Nippon shokuhin kagaku Kogaku kaishi,2001:409-415.
[3] 刘冬梅,吴晖,余以刚,等.Lactobacillus casei subsp.rhamnosus 719对泡菜中亚硝酸盐的影响[J].华南理工大学学报:自然科学版,2008,36(7):140-144. Liu Dong-mei,Wu Hui,Yu Yi-gang,et al.Effect of Lactobacillus casei subsp.rhamnosus 719 on Inhibition of Nitrites in Pickles[J].Journal of South China University of Technology:Natural Science Edition,2008,36(7):140-144.
[4] 刘冬梅,吴晖,余以刚,等.一种即食含益生菌的泡菜及其制备方法:中国,200611/23898[P].2010-02-17.
[5] Vandamme P,Pot B,Gillis M,et al.Polyphasic taxonomy,a consensus approach to bacterial systematic[J].Microbiological Reviews,1996,60(2):407-438.
[6] Chief A,Brusetti L,Borin S,et al.Genetic relationship in the‘Bacillus cereus group’by rep-PCR fingerprinting and sequencing of a Bacillus anthracis-specific rep-PCR fragment[J].Journal of Applied Microbiology,2003,94(6): 1108-1119.
[7] Dombek P E,Johnson L K,Zimmerley S T,et al.Use of
repetitive DNA sequence and the PCR to differentiate Escherichia coli isolates from human and animal sources[J].Applied and Environmental Microbiology,2000,66 (6):2572-2577.
[8] Olive D M,Bean P.Principles and applications of methods for DNA-Based typing of microbial organisms[J].Journal of Clinical Microbiology,1999,37(6):1661-1669.
[9] Socransky S S,Haffajee A D,Smith C,et al.Use of checkerboard DNA-DNA hybridization to study complex microbial ecosystems[J].Oral Microbiology and Immunology,2004,19(6):352-362.
[10] Petti C A.Detection and identification of microorganisms by gene amplification and sequencing[J].Clinical Infective Diseases,2007,44(8):1108-1114.
[11] Fox G E,Wisotzkey J D,Jurtshuk P.How close is close: 16S rDNA sequence identity may not be sufficient to guarantee species identity[J].International Journal of Systematic Bacteriology,1992,42(1):166-170.
[12] 周贤轩,杨波,陈新华,等.几种分子生物学方法在菌种鉴定中的应用[J].生物技术,2004,36(6),35-38. Zhou Xian-xuan,Yang Bo,Chen Xin-hua,et al.Application of several molecular biology method in microbe characterization[J].Biotechnology,2004,36(6):35-38.
[13] Ross T L,Merz W G,Farkosh M,et al.Comparison of an automated repetitive sequence-based PCR microbial typing system to pulsed-field gel electrophoresis for analysis of outbreaks of methicillin-resistant Staphylococcus aureus[J].Journal of Clinical Microbiology,2005,43 (11):5642-5647.
[14] Graham P H,Sadowsk M J,Keyser H H,et al.Proposed minimal standards for the description of new genera and species of root-and stem-nodulating bacteria[J].International Journal of Systematic Evolutionary Microbiology,1991,41(4):582-587.
[15] Cho J C,Tiedje J M.Bacterial species determination from DNA-DNA hybridization by using genome fragments and DNA microarrays[J].Applied and Environmental Microbiology,2001,67(8):3677-3682.
[16] 周劲松,刘冬梅,曹艳华,等.戊糖乳杆菌的鉴定及其发酵D-乳酸的研究[J].食品工业科技,2013,34 (18),174-177. Zhou Jin-song,Liu Dong-mei,Cao Yan-hua,et al.Identification of Lactobacillus pentosus and its D-lactic acid fermentation[J].Science and Technology of Food Industry,2013,34(18):174-177.
[17] 张刚.乳酸细菌[M].北京:化学工业出版社,2004: 41-47.
[18] Zanoni P,Farrow J A E,Phillips B A,et al.Lactobacillus pentosus(Fred,Petersen,and Anderson)sp.nov.,nom.rev.[J].International Journal of Systematic Bacteriology,1987,37(4):339-341.
[19] Ferain T D,Garmyn N,Bernard P,et al.Lactobacillus plantarum ldhL gene:over-expression and deletion[J]. Journal of Bacteriology,1994,176(3):596-601.
[20] Wang Y,Chen C,Ai L,et al.Complete genome sequence of the probiotic Lactobacillus plantarum ST-III[J].Journal of Bacteriology,2011,193(1):313-314.

