G/11木聚糖酶最适p H值的预测及其与氨基酸位置的关系
林源清,张光亚
(华侨大学 化工学院,福建 厦门361021)
木聚糖酶(EC3.2.1.8)是一种重要的工业用酶,可广泛应用于饲料、造纸、食品等行业.木聚糖酶的使用可大大减少造纸工业漂白过程中氯化物的用量,从而有效降低制浆造纸工业对环境的污染[1].用于造纸工业的木聚糖酶需满足耐热和耐碱条件,目前满足所需条件的酶来源于两种途径:一是从极端环境中筛选产酶菌株[2];二是通过基因工程对酶进行遗传改造[3].鉴于菌株筛选耗时长,效率低,基因工程改造越来越受研究者的关注.木聚糖酶可分为F/10和G/11家族,由于G/11家族的木聚糖酶分子较小,而且其结构更为简单,因此比较适合作为理论研究的分子模型[4].对于蛋白质的改造主要有两种策略:一是理性设计(rational design),即定点突变;二是非理性设计(irrational design),定向进化.定点突变目的明确,但需要事先了解蛋白质的结构;定向进化不需事先了解蛋白质的结构,但其筛选困难.本文利用木聚糖酶序列的信息和最适p H值,构建了氨基酸组成和最适p H值关系的模型.旨在探索影响酶最适p H值的氨基酸及其位置,为木聚糖酶的改造提供可靠的信息,以期提高研究效率.本课题组曾利用木聚糖酶的氨基酸与最适p H值关系构建BP神经网络模型,并且取得较好的预测结果[5].采用均匀设计(UD)方法,构建氨基酸组成和最适p H值关系的模型.
1 材料与方法
1.1 数据来源
G/11家族木聚糖酶的序列来源于 UniProt(http://www.uniprot.org/),木聚糖酶数据来源于文献[6].73个木聚糖酶ID号及最适p H值,如表1所示.表1中:ID为木聚糖酶在UniProt数据库中的收录号;p Hopt为文献中报道的木聚糖酶的最适p H值.
对于最适p H值在一定范围的,取其中间值.木聚糖酶的氨基酸组成分析由自行设计的软件完成.该软件不仅可以计算全段序列的氨基酸组成,还可以计算分段氨基酸组成.主成分分析由MVSP软件完成,神经网络及支持向量机由weka3.6.8软件完成.以各个木聚糖酶中全段序列及分段序列(将酶蛋白序列均分为3段,分别表示为序列的N端,C端及中间端)的20种氨基酸的组成百分比作为神经网络和支持向量机的输入,其对应的最适p H值作为结果输出.

表1 G/11木聚糖酶ID号及最适p H值Tab.1 Xylanase ID in family G/11 and the optimum p H value
1.2 均匀设计的支持向量机
在运算时,支持向量机(SVM)[7]模型和BP神经网络[8]模型都需要选择参数,以达到最佳拟合结果.因此,采用均匀设计法(UD)[9]来选择适当的运行参数.定义两个特征指标[5],即均方根误差RMSE和平均绝对误差MAE.模型预测的结果采用常用的“留一法”,即对n组数据,每次取1组作测试,其他n-1组作为训练样本,共进行n次循环,使得样本中所有数据都能进行预测.
1.3 主成分分析
主成分分析(principal components analysis,PCA)又称主分量分析,把多指标转化为少数几个综合指标,在许多领域有着有效而广泛的应用[10],是一种较为客观的综合评价方法.运用MVSP软件,可直接获得20个氨基酸变量的主成分荷载和73个个案的主成分得分.利用主成分得分与最适p H值进行拟合,拟合结果可在一定程度上综合反映氨基酸组成与最适p H值的关系.
2 结果与分析
2.1 基于均匀设计的支持向量机
利用均匀设计法,对两种不同核函数(Linear和RBF)的支持向量机运算参数进行优化,10倍交叉验证结果,如表2,3所示.表2,3中:MAE为平均绝对误差;RMSE为均方根误差.限于篇幅,仅列出最优预测结果.

表2 基于Linear核函数的支持向量机预测结果Tab.2 Result of SVM prediction based on linear kernel
由表3可知:在RBF核函数支持向量机模型中,以I端氨基酸组成作为输入,得到的预测结果最佳,即C=1,ε=0.1,γ=0.5时,其 MAE和RMSE值均最小,分别为0.84和1.17.此时,所建立的模型对木聚糖酶最适p H值预测准确率最高,故为最佳方案.

表3 基于RBF核函数的支持向量机预测结果Tab.3 Result of SVM prediction based on RBF kernel
通过比较两种核函数的预测结果,可以得知RBF核函数的整体预测结果优于Linear核函数.尽管在Linear核函数中,当惩罚值C=1,ε=0.005,其MAE为0.83,是所有预测结果中最小的.这个结果说明,在Linear核函数中运行参数取得了比较理想的结果.如果对RBF核函数进一步优化,可能会取得更好的结果.根据表3中的最优化参数C=1,ε=0.1,γ=0.5,使用支持向量机法建立最适p H值模型.通过该模型对实际测得的数据(p Hexp)进行预测,预测结果(p Hpre)如图1所示.从图1可知:该模型预测结果与实际测得结果的相关性为0.67,说明该模型可行.

图1 预测值和实测值的关系Fig.1 Relationship between experimental andpredicted transition temperature obtained
2.2 基于均匀设计的BP神经网络
为了科学地确定神经网络中连接权的初始值、最佳的隐含层神经元的个数、学习速度等参数,选择一个隐含层的神经网络,对学习速率、动态参数和隐含层结点数3个因素15水平进行均匀设计,所得的均匀设计表和训练结果,如表4所示(仅列出最优预测结果).
由表4可知:当学习速率(v)为0.06,动态参数(MP)为0.2,隐含层结点数(NHL)为8时,以I端氨基酸组成为输入的模型,对最适p H值拟合的均方根误差为1.49个p H值单位,平均绝对误差为1.09个p H值单位,具有很好的拟合效果.
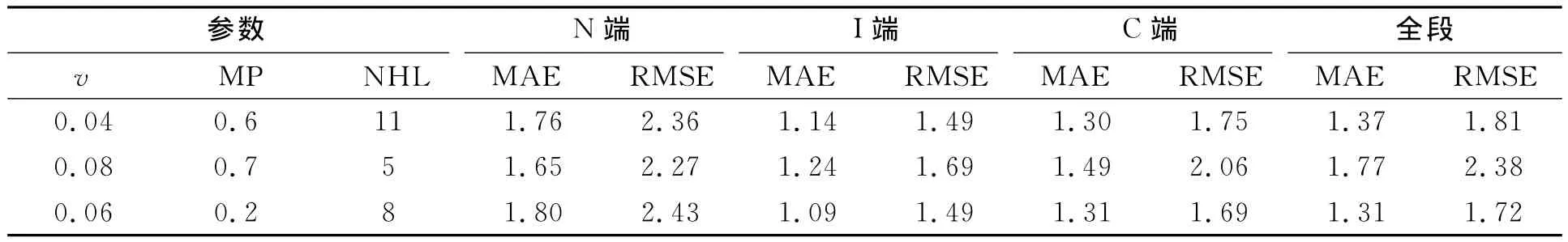
表4 BP神经网络的预测结果Tab.4 Result of BP neural network
由表2~4可知:构建的3种模型中,基于RBF核函数的支持向量机模型的整体预测结果最佳;I端的预测结果在分段预测模型中均最佳;其次是C端;最后是N端(表5).这个结果说明I端与木聚糖酶的最适p H值相关性最高.
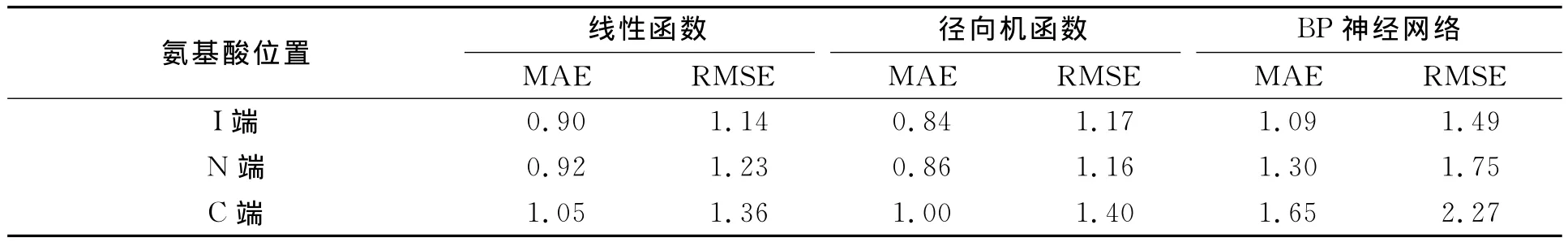
表5 3种模型3端最佳优化结果Tab.5 Optimum result of 3 segments in the three different models
2.3 氨基酸与最适p H值的相关性
原始数据运用MVSP软件做主成分分析(PCA)分析后,可得到20个氨基酸变量的主成分和73个个案主成分.根据个案得分主成分(z),做主成分与实测最适p H值的相关性图,如图2所示.

图2 主成分与最适p H值相关性图Fig.2 Relationship between principal components and optimum p H value
由图2可知:全段序列的相关性最好,R值为0.69.N端、I端、C端与最适p H值的相关性大小分别为-0.33,-0.68,0.40,其中负值表示负相关.它们与最适p H值影响的大小顺序依次为I端、C端、N端.这个结果和前文的支持向量机和BP神经网络的结果一致,验证了前文所构建模型的可靠性.值得注意的是全段序列和I端序列的R值数值相近,但是方向却相反.
由于I端序列氨基酸组成对于木聚糖酶的最适p H值影响较大.因此,仅列出I端分析结果.原始数据运用MVSP软件PCA分析后,得到20个氨基酸变量的5个主成分.
各氨基酸与5个主成分之间的关系,如表6所示.表6中:相关系数只保留一位小数,且仅列出绝对值大于0.2的氨基酸.从表6可知:第1主成分与丝氨酸(S)相关性最强,相关性高达0.8,该结果表明丝氨酸(S)是木聚糖酶的关键氨基酸;第2主成分与甘氨酸(G)、第3主成分与甘氨酸(G)、第4主成分与酪氨酸(Y)及天冬酰胺(N)的相关性显著,表明这3种氨基酸是木聚糖酶比较重要的氨基酸.Liu等[11]研究结果表明:G/11家族主成分分析的前7个主成分所代表的是该家族木聚糖酶的2级结构,分别为:卷曲、转角、折叠、转角、转角、螺旋和折叠.

表6 木聚糖酶20种氨基酸与各主成分的关系Tab.6 Relationship between 20 amino acids and principle components in xylanase
3 结束语
构建了不同的最适p H值预测模型,其中基于RBF核函数的支持向量机模型预测木聚糖酶的最适p H值的精度,比使用BP神经网络及Linear核函数的支持向量机模型更好,可做为木聚糖酶模拟的后续使用模型.采用了均匀设计的方法对构建的模型进行了参数优化,但在各因素水平的选择上仍带有一定的随意性,如果经过精心的选择,模型的预测效果还会有所改善.此外,由于木聚糖酶分子量较小、结构比较简单,只有一条多肽链,基于此酶所建立的模型对于其他具有4级结构的复杂酶类是否仍然适用仍有待探讨.
[1] 聂国兴,王俊丽,明红.木聚糖酶的应用现状与研发热点[J].工业微生物,2008,38(1):53-59.
[2] 包怡红,刘伟丰,毛爱军,等.耐碱性木聚糖酶高产菌株的筛选、产酶条件优化及其在麦草浆生物漂白中的应用[J].农业生物技术学报,2005,13(2):235-240.
[3] UMEMOTO H,YATSUNAMI R,INAMI M,et al.Improvement of alkaliphily of bacillus alkaline xylanase by introducing amino acid substitutions both on catalytic cleft and protein surface[J].Bioscience Biotechnology and Biochemistry,2009,73(4):965-967.
[4] SAPAG A,WOUTERS J,LAMBERT C,et al.The endoxylanases from family 11:Computer analysis of protein sequences reveals important structural and phylogenetic relationships[J].Journal of Biotechnology,2002,95(2):109-131.
[5] 张光亚,方柏山.木聚糖酶氨基酸组成与其最适p H 值的神经网络模型[J].生物工程学报,2005,21(4):658-661.
[6] PAES G,BERRIN J G,BEAUGRAND J.GH11 xylanases:Structure/function/properties relationships and applications[J].Biotechnology Advances,2012,30(3):564-592.
[7] VAPNIK V N.The nature of statistical learning theory[M].New York:Springer-Verlag,2000:138-167.
[8] 王轶夫,孙玉军,郭孝玉.基于BP神经网络的马尾松立木生物量模型研究[J].北京林业大学学报,2013,35(2):17-21.
[9] 方开泰.均匀设计-数论方法在试验设计的应用[J].应用数学学报,1980(4):363-372.
[10] 王志江.主成分分析法在地区企业经济效益评价中的应用[J].华侨大学学报:自然科学版,2004,25(3):322-325.
[11] LIU Liang-wei,ZHANG Jue,CHEN Bin,et al.Principle component analysis in F/10 and G/11 xylanase[J].Biochemical and Biophysical Research Communications,2004,322(1):277-280.

