ZEB2基因3′UTR区转染对人胃黏膜上皮细胞GES-1增殖、侵袭、迁移的影响*
李素丽 周 芳 张庆瑜△ 贾文亮 张安玲 韩 磊 康春生
microRNA(miRNA)是长约22 nt的短链RNA,可与miRNA反应元件(miRNA response elements,MREs)互补结合,结合后能够通过促进靶mRNA的降解和(或)抑制其翻译而负性调控基因的表达。人们对miRNA-mRNA已有足够的认识,但关于mRNA-miRNA反向作用却知之甚少。已证实MREs能够作用于miRNAs,从而调节后者的作用[1-2]。Tay等[3]证实肿瘤抑制因子PTEN假基因(PTENP1)的3′非翻译区(UTR)能与PTEN结合相同的miRNA,以miRNA依赖的方式调控细胞PTEN和P-Akt蛋白水平。miR-200a/b/c在胃癌中低表达,通过抑制E盒结合锌指蛋白(ZEB)的表达,调节肿瘤的发生发展[4-5]。因此,本研究应用人工合成ZEB2 3′UTR转染人胃黏膜上皮细胞GES-1,研究其对miR-200家族的调节作用,观察并分析其对GES-1的影响,旨在从其增殖、迁移、侵袭力方面探讨ZEB2 3′UTR导致正常胃黏膜上皮细胞恶性转化的可能性。
1 材料与方法
1.1 实验材料 ZEB2 3′UTR质粒由南京金斯瑞公司合成;QIAGEN质粒提取试剂盒购自德国QIAGEN公司,Trizol试剂及转染试剂Lipofectamine 2000购自美国Invitrogen公司,ZEB1、ZEB2单克隆抗体购自美国Abcam公司,基质金属蛋白酶(MMP)-2/9、增殖细胞核抗原(PCNA)及相应二抗均购自中杉金桥公司;dNTP mixture、反转录酶(MMLV)、RNase抑制剂购自大连Takara公司,逆转录引物及qRT-PCR引物、荧光定量miRNA PCR试剂盒、miR-200b micmics购自上海吉玛制药技术有限公司;matrigel购自美国BD公司;GES-1由天津医科大学神经肿瘤研究所馈赠。
1.2 方法
1.2.1 质粒的构建与提取 利用GenBank数据库检索ZEB2基因序列(Gen Bank,NM_001171653.1,NM_014795.3)找出其3′UTR含miR-200家族的结合序列全长,选用PGL3-Control表达载体。将序列交由南京金斯瑞公司合成,见图1。根据QIAGEN质粒中提试剂盒说明书进行质粒的提取与纯化。
1.2.2 细胞培养与分组处理 GES-1细胞用含10%胎牛血清的DMEM培养基,于含5%CO2,37℃的恒温培养箱中培养。第一部分:检测ZEB2 3′UTR转染后对ZEB1/ZEB2 mRNA、蛋白水平及miR-200a/b/c的影响;实验分为对照组、突变组和ZEB2 3′UTR组。第二部分:验证miR-200b在ZEB2 3′UTR转染后调节增殖、侵袭、迁移中的作用;实验分为对照组、ZEB2 3′UTR组、ZEB2 3′UTR+无义序列组、ZEB2 3′UTR组+miR-200b micmics组。根据Lipofectamine2000转染试剂说明书进行转染,继续处理细胞48 h后进行下一步实验。
1.2.3 qRT-PCR检测细胞miR-200a/b/c及ZEB1/ZEB2的表达变化 Trizol法提取待测细胞总RNA,按试剂盒说明书应用ZEB1、ZEB2、miR-200a/b/c特异性逆转录、PCR引物及相应组分配置反应体系。microRNA逆转录条件:16℃30 min,42℃30 min,85℃10 min。所得逆转录产物cDNA行qRTPCR检测各组miR-200a/b/c的表达水平,反应条件:95℃10 min;95℃15 s,65℃30 s,72℃30 s;共40个循环。ZEB1、ZEB2 cDNA模板合成条件:42℃1 h。PCR扩增,其特异性引物序列,见表1。扩增条件:94℃12 min,94℃30 s,58℃30 s,72℃30 s,共40个循环;72℃延伸5 min。分别以U6、GAPDH作为内参,结果判定采用2-ΔΔCT法。

Figure 1 Schematic representation of the ZEB2 3′UTR plasmid constructs图1 ZEB2 3′UTRs区质粒构建示意图

Table 1 The qRT-PCR primers sequences of ZEB1/ZEB2/GAPDH表1ZEB1、ZEB2、GAPDH qRT-PCR引物序列
1.2.4 Western blot检测细胞蛋白表达水平 用细胞裂解液RIPA裂解提取待测细胞的总蛋白。SDS-PAGE凝胶电泳分离,恒压80 V电转移至PVDF印迹膜。膜封闭液封闭1 h后,加入一抗ZEB1、ZEB2、MMP2/9、PCNA、GAPDH(1∶1 000稀释)4℃孵育过夜。用辣根过氧化物酶标记的二抗(1∶1 000稀释),室温孵育1 h,凝胶成像系统采集成像。GAPDH蛋白作为内参。以目标蛋白条带灰度值/同一样本内参条带灰度值计算目标蛋白相对表达量。
1.2.5 Transwell侵袭实验检测细胞侵袭能力 在Transwell上室聚碳酸酯滤膜(孔径8 μm)上加入预冷无血清DMEM培养基稀释的Matrigel基质胶(Matrigel∶DMEM=1∶2)100 μL,37℃30 min后待其凝固,接种100 μL无血清培养基稀释GES-1细胞(1×106个/mL),下室加入600 μL含10%胎牛血清的DMEM培养液,每组设3个复孔。37℃、5%CO2条件下培养24 h后,弃去上室液体,用棉签轻轻擦掉上层未迁移细胞,苏木精染色。DP-70荧光相差倒置显微镜(×100)采集图像,随机读取3个视野,计算穿过膜的细胞数。
1.2.6 细胞划痕实验检测细胞迁移能力 将已转染的细胞放入37℃、5%CO2培养箱中孵育。次日用200 μL微量移液枪头在6孔板内垂直及平行划痕,PBS液洗涤2次后加入无血清培养基,在倒置显微镜下观察0、48 h后划痕两侧细胞的迁移情况并拍照。距离差除以2为细胞相对迁移距离,计算细胞相对迁移率(相对迁移率=相对迁移距离/0 h时划痕边缘距划痕中线距离),重复3次并进行统计分析。
1.2.7 MTT法检测细胞增殖活性 取对数生长期细胞,常规胰酶消化制成单细胞悬液,细胞以4×103个/孔接种于96孔培养板中,每孔加入100 μL培养基,5%CO2、37℃培养箱内培养24 h后转染。取3组细胞测其吸光度(A)值,每组3个复孔,每24 h测1次。测时每孔加入20 μL(5 g/L)MTT,温育4 h后,弃去培养基,加入200 μL DMSO,振荡10 min。在酶标仪上测490 nm处各孔的A值,计算生长增殖率=(实验组A490值/对照组A490值-1)×100%。
1.3 统计学方法 应用SPSS 18.0统计软件对数据进行分析,计量资料采用均数±标准差(±s)表示,3组及以上数据间比较采用单因素方差分析,组间多重比较采用LSD-t法,以P<0.05为差异有统计学意义。
2 结果
2.1 转染ZEB2 3′UTR对GES-1细胞miR-200a/b/c及ZEB1、ZEB2 mRNA表达水平的影响 结果显示,转染ZEB2 3′UTR后,GES-1细胞中miR-200a/b/c表达水平较对照组及突变组明显下降,以miR200b下调最为显著(均P<0.01),突变组较对照组无明显改变,见图2。ZEB1及ZEB2 mRNA表达水平较对照组及突变组明显上升(均P<0.01),见图3。
2.2 Western blot检测转染ZEB2 3′UTR后ZEB1/ ZEB2蛋白表达变化 转染ZEB2 3′UTR后,GES-1细胞中ZEB1和ZEB2蛋白相对表达水平较对照组、突变组明显升高(P<0.01),见图4、表2。
2.3 转染ZEB2 3′UTR及恢复miR-200b micmics表达后对细胞侵袭、迁移能力的影响 ZEB2 3′UTR+ miR-200b micmics组miR-200b的表达水平高于其他3组,见图5。Transwell体外侵袭实验检测转染后穿过微孔的平均细胞数目,结果显示,ZEB2 3′UTR组较对照组均明显升高,而联合miR-200b micmics后,穿过小室的细胞数较ZEB2 3′UTR组明显减少(均P<0.01),见图6、表3。划痕实验结果显示,48 h后ZEB2 3′UTR组细胞运动速度明显快于对照组,迁移率显著升高,而恢复miR-200b表达后,其迁移能力有所下降(P<0.01),见图6、表3。且MMP2/9侵袭指标也出现相应变化,见图7、表4。

Figure 2 Comparison of miR-200a/b/c expression levels between three groups图2 各组细胞的miR-200a/b/c表达水平比较(n=3)
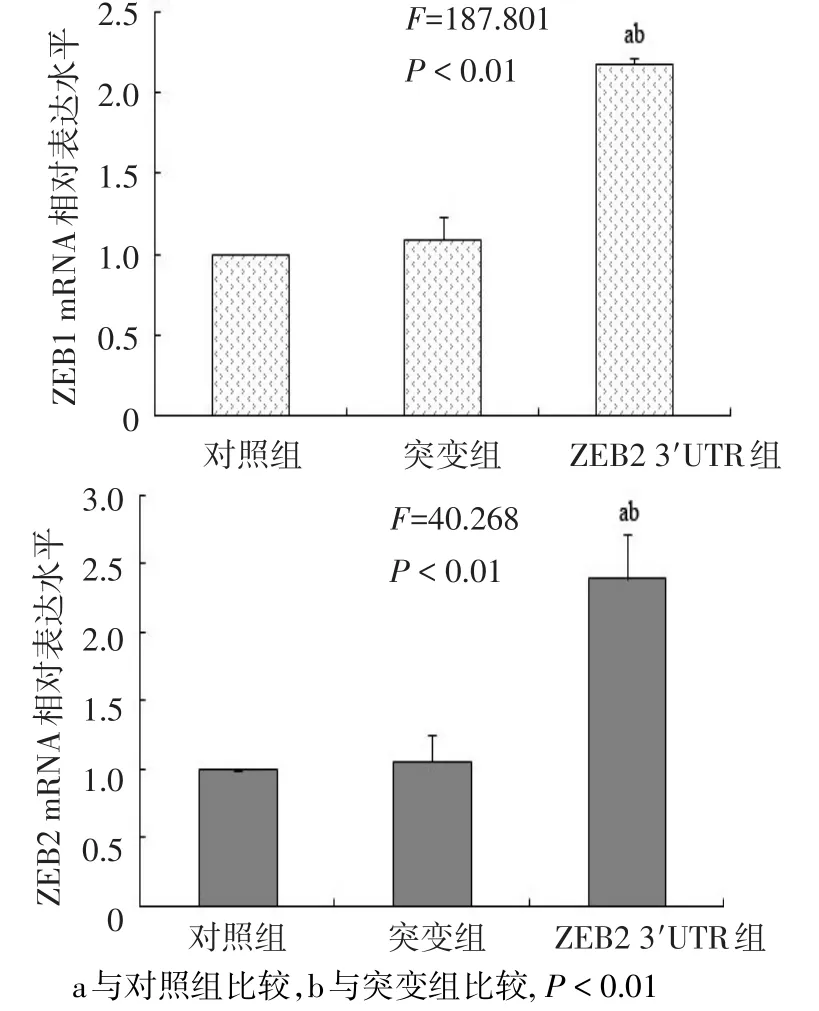
Figure 3 Comparison of ZEB1 and ZEB2 expression levels between three groups图3 各组细胞的ZEB1、ZEB2表达水平比较(n=3)

Figure 4 The protein expression levels of ZEB1/ZEB2 in three groups图4 3组细胞内ZEB1/ZEB2蛋白表达水平
Table 2 Comparison of protein expression levels of ZEB1/ZEB2 between three groups表2 各组细胞中ZEB1/ZEB2蛋白表达水平比较(n=3,±s)
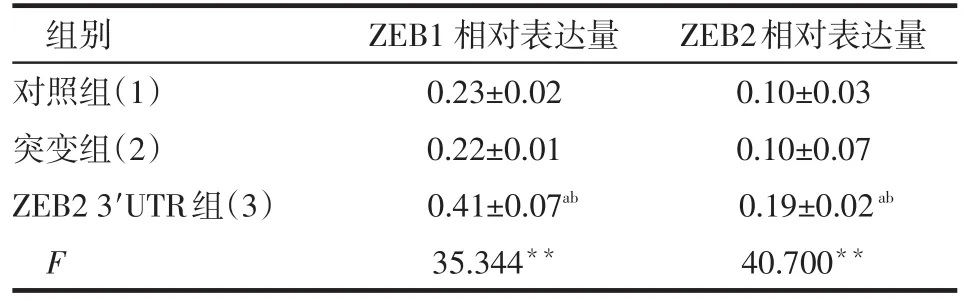
Table 2 Comparison of protein expression levels of ZEB1/ZEB2 between three groups表2 各组细胞中ZEB1/ZEB2蛋白表达水平比较(n=3,±s)
**P<0.01;a与对照组比较,b与突变组比较,P<0.05
组别对照组(1)突变组(2)ZEB2 3′UTR组(3)F ZEB1相对表达量0.23±0.02 0.22±0.01 0.41±0.07ab 35.344**ZEB2相对表达量0.10±0.03 0.10±0.07 0.19±0.02ab 40.700**

Figure 5 The expression levels of miR-200b evaluated by qRT-PCR in four groups图5 qRT-PCR检测不同处理组miR-200b的表达水平
Table 3 Comparison of the invasive capability and migrating rates after transfection between four groups表3 4组转染后细胞侵袭能力及细胞迁移率比较(n=3,±s)

Table 3 Comparison of the invasive capability and migrating rates after transfection between four groups表3 4组转染后细胞侵袭能力及细胞迁移率比较(n=3,±s)
**P<0.01;a与(1)组比较,b与(2)组比较,c与(3)组比较,P<0.01;表4同
组别对照组(1)ZEB2 3′UTR组(2)ZEB2 3′UTR+无义序列组(3)ZEB2 3′UTR+200b micmics组(4)F穿膜细胞数(个)45.67±3.21 76.33±3.78a 77.33±2.51a 47.33±3.79bc 81.353**48 h迁移率(%)24.91±2.56 52.45±2.63a 54.01±3.10a 26.33±3.02bc 94.602**
2.4 MTT检测转染后细胞增殖活性 4组之间A490值于第1天差异无统计学意义(F=0.995,P>0.05),第2~6天差异均有统计学意义(F分别为8.630、18.377、11.925、24.545、35.058,均 P<0.01);且ZEB2 3′UTR组A490值于第2~6天较对照组及ZEB2 3′UTR+miR-200b micmics组显著升高(均P<0.01);而ZEB2 3′UTR+miR-200b micmics组A490值于第1~6天和对照组比较差异均无统计学意义(均P>0.05),见图8。相应反映增殖能力指标的PCNA蛋白与生长曲线一致,出现相应变化,见图7、表4。

Figure 6 Comparison of trans-membrane cell numbers and migrating rates between four groups(×100)图6 4组细胞中穿过小室细胞数及迁移率比较(×100)

Figure 7 The protein expression levels of MMP2/9 and PCNA in four grouops图74组细胞内MMP2/9及PCNA蛋白表达水平

Figure 8 The growth curves of GES-1 in different groups图8 不同处理组GES-1细胞的生长曲线
Table 4 Comparison of protein expression levels of MMP2/9 and PCNA between four groups表4 各组细胞中MMP2、MMP9和PCNA蛋白表达水平比较(n=3,±s)

Table 4 Comparison of protein expression levels of MMP2/9 and PCNA between four groups表4 各组细胞中MMP2、MMP9和PCNA蛋白表达水平比较(n=3,±s)
组别对照组(1)ZEB2 3′UTR组(2)ZEB2 3′UTR+无义序列组(3)ZEB2 3′UTR+miR-200b micmics组(4)F MMP2 0.28±0.02 0.53±0.01a 0.55±0.05a MMP9 0.36±0.03 0.69±0.03a 0.67±0.04a PCNA 0.63±0.03 1.03±0.05a 1.01±0.06a 0.30±0.03bc 77.993**0.39±0.02bc 107.487**0.66±0.02bc 69.147**
3 讨论
3.1 mRNA-miRNA反向作用的机制 近期提出的竞争性内源RNA(competing endogenous RNA,ceR⁃NA)假说认为:mRNA、被转录的假基因和长的非编码RNA之间能够通过竞争miRNA的方式进行交互对话,这种作用依赖于“MREs”[6]。RNA之间通过竞争性结合相应的miRNA,对该miRNA的后续转录后调控作用发生有效的控制。Sumazin等[7]绘制出了恶性胶质瘤细胞中RNA相互作用网络,有超过7 000种基因编码RNA以借助miRNA的方式发生了相互作用,与癌症相关的基因起着重要的调控作用,从而参与肿瘤的发生发展过程。
3.2 3′UTR在肿瘤发生发展过程中的调节作用 PTENP1的3′UTR及PTEN ceRNA ZEB2能与PTEN结合相同的miRNA,以miRNA依赖的方式调控细胞内PTEN和P-Akt蛋白水平;其缺失可致PI3K/AKT信号激活,诱导肿瘤发生[3,8]。外源性表达“miRNA海绵”能够有效特异地抑制miRNA的功能[9]。含MREs的3′UTR具有强大的生物学活性,成为miRNA“诱饵”,螯合miRNA,从而影响它们对所表达的基因的调控。本研究中转染ZEB2 3′UTR后,ZEB1/ZEB2 mRNA及蛋白水平均升高,可能与上述机制有关。
3.3 ZEB2 3′UTR对细胞恶性转化的影响及可能机制 本研究转染ZEB2 3′UTR质粒后,miR-200a/b/c表达下调,以miR-200b降低最为明显,此变化可能与2种因素有关:一方面,特异的miRNA结合位点结构与miRNA结合后触发miRNA加尾和3′→5′修剪及亚细胞定位而影响miRNA的稳定性有关[10-11];另一方面,ZEB1/ZEB2能结合miR-200b/200a/429启动子区以调节其表达[12],ZEB2 3′UTR的导入引起miR-200家族表达降低,进而减弱其对ZEB1/ZEB2的抑制作用,升高的ZEB1/ZEB2则进一步加强了其对miR-200家族成员的表达抑制作用,形成一个正反馈调节作用。恢复miR-200b表达后,细胞恶性转化能力部分被逆转,ZEB2 3′UTR对miR-200b的调控可能是其发挥作用的主要机制之一。
ZEB2 3′UTR可以促进人胃黏膜上皮细胞的恶性转化倾向,此作用可能与转染ZEB2 3′UTR后调控miR-200家族进而影响相应的靶基因ZEB1/ZEB2的表达有关。本研究证实了非编码转录在基因规模上的影响,有望为抑制GES-1细胞恶性转化提供新的途径或方法。
[1] Seitz H.Redefining microRNA targets[J].Curr Biol,2009,19(10): 870-873.doi:10.1016/j.cub.2009.03.059.
[2] Poliseno L,Salmena L,Zhang J,et al.A coding-independent func⁃tion of gene and pseudogene mRNAs regulates tumour biology[J]. Nature,2010,465(7301):1033-1038.doi:10.1038/nature09144.
[3]Tay Y,Kats L,Salmena L,et al.Coding-independent regulation of the tumor suppressor PTEN by competing endogenous mRNAs[J]. Cell,2011,147(2):344-357.doi:10.1016/j.cell.2011.09.029.
[4]Cong N,Du P,Zhang A,et al.Downregulated microRNA-200a pro⁃motes EMT and tumor growth through the wnt/β-catenin pathway by targeting the E-cadherin repressors ZEB1/ZEB2 in gastric ade⁃nocarcinoma[J].Oncol Rep,2013,29(4):1579-1587.doi:10.3892/ or.2013.2267.
[5]申发娟,苏娟,张庆瑜,等.上调miR-200a、miR-141表达对胃腺癌细胞生长的体外研究[J].天津医药,2013,41(6):517-519.doi: 10.3969/j.issn.0253-9896.2013.06.002.
[6] Salmena L,Poliseno L,Tay Y,et al.A ceRNA hypothesis:the Ro⁃setta Stone of a hidden RNA language[J]?Cell,2011,146(3):353-358.doi:10.1016/j.cell.2011.07.014.
[7] Sumazin P,Yang X,Chiu HS,et al.An extensive microRNA-medi⁃ated network of RNA-RNA interactions regulates established onco⁃genic pathways in glioblastoma[J].Cell,2011,147(2):370-381. doi:10.1016/j.cell.2011.09.041.
[8]Karreth FA,Tay Y,Perna D,et al.In vivo identification of tumorsuppressive PTEN ceRNAs in an oncogenic BRAF-induced mouse model of melanoma[J].Cell,2011,147(2):382-395.doi:10.1016/j. cell.2011.09.032.
[9]Ebert MS,Neilson JR,Sharp PA.MicroRNA sponges:competitive inhibitors of small RNAs in mammalian cells[J].Nat Methods,2007, 4(9):721-726.
[10]Cazalla D,Yario T,Steitz JA.Down-regulation of a host microRNA by a Herpesvirus saimiri noncoding RNA[J].Science,2010,328(5985):1563-1566.doi:10.1126/science.1187197.
[11]Ameres SL,Horwich MD,Hung JH,et al.Target RNA-directed trimming and tailing of small silencing RNAs[J].Science,2010,328(5985):1534-1539.doi:10.1126/science.1187058.
[12]Bracken CP,Gregory PA,Kolesnikoff N,et al.A double-negative feedback loop between ZEB1-SIP1 and the microRNA-200 family regulates epithelial-mesenchymal transition[J].Cancer Res,2008,68(19):7846-7854.doi:10.1158/0008-5472.CAN-08-1942.

