ω-芋螺毒素的定量构效关系与虚拟筛选
丁俊杰 丁晓琴 李大禹 潘 里 陈冀胜
(北京药物化学研究所,国民核生化灾害防护国家重点实验室,北京102205)
1 引言
ω-芋螺毒素是从各种食鱼芋螺中提取的生物活性多肽,由24到31个氨基酸残基组成,对N-型或P/Q-型电压敏感的钙离子通道(VGCCs)具有很好的选择性,1-3已成为神经生物学、分子遗传学、药理学、药物化学的研究热点.ω-芋螺毒素MVIIA和GVIA是N-型VGCCs的选择性拮抗剂,具有神经保护和止痛作用,4,5已被作为先导化合物进行研究开发.6,7MVIIA的合成替代品齐考诺肽(SNX-III,Ziconotide)8疗效好、不成瘾,是第一个上市的多肽类N-型VGCC拮抗剂镇痛药物,也是唯一一个非阿片类鞘内止痛剂.9,10ω-芋螺毒素MVIIC与P/Q-型VGCCs结合,具有很强的致死性,可作为P/Q-型VGCCs的诊断探针.11ω-芋螺毒素能直接干扰神经元VGCCs的正常功能,在特异诊断试剂和新药研制开发方面具有巨大的应用前景.12-15
目前,计算机辅助药物设计已经向实用化方向迈进,16-18成为药物设计和开发的重要手段之一.但多肽类化合物由于分子量大、结构复杂、柔性较高,对其进行定量构效关系(QSAR)和计算机辅助分子设计一直是亟待解决的难题之一.19-23本文采用本课题组建立的新型多肽QSAR研究方法,24对ω-芋螺毒素多肽类似物进行了QSAR研究,分别建立N-型和P/Q-型VGCC拮抗剂的QSAR模型.在此研究的基础上,设计了VGCC拮抗剂虚拟组合多肽库.由于ω-芋螺毒素与VGCCs的作用位点及相互作用模式还未有文献报道,25因此我们采用基于配体的虚拟筛选方法,筛选出了一批高活性、高选择性的可供合成评价的多肽类似物.目前,还未见有关ω-芋螺毒素多肽QSAR和虚拟筛选的研究工作报道.
2 理论方法
2.1 QSAR模型建立
采用本课题组建立的多肽QSAR研究方法,对多肽类似物中变化的7个氨基酸残基采用描述符c(c1,c2,c3)-scales进行定量描述,按序列形成一个多肽分子的结构描述矩阵,再应用遗传偏最小二乘法(G/PLS),建立N-型和P/Q-型VGCC拮抗剂的QSAR方程.遗传算法的种群设为100,进化5000次,方程的项数设为5-20,偏最小二乘法的主成分数设为2-4,分别建立两种亚型的VGCC拮抗剂QSAR模型.经过多次平行计算,在每一个计算条件下,筛选出较优的QSAR模型.为了对7个变化的残基进行详细的QSAR研究,又将方程的项数设为22,其中包含一个常数项和21个c-scales定量描述参数,建立一个包含所有7个残基定量描述参数的QSAR模型.
2.2 虚拟组合多肽库构建
建立一个理想的虚拟组合化学集中库,应该包括尽可能多的所期望的化学结构,同时在满足一定分子多样性的前提下,尽量减小库的规模,28因此模板分子、取代位置及取代基团的化学结构的选择就成为虚拟化学库构建的关键.本文选用MVIIA和MVIIC分别作为N-型和P/Q-型VGCC拮抗剂虚拟库的模板分子,取代位点为两个模板分子氨基酸序列不同的位置,分别为6个和7个.
选取软件Cerius2提供的182个天然和非天然氨基酸结构,组成一个氨基酸残基碎片库.采用MOPAC6.0中的PM3方法,进行构象优化后,应用氨基酸结构描述符c1,c2,c3-scales对氨基酸结构进行定量描述.依据各个取代位点对构效关系的影响,分别采用相似性比较法、聚类分析方法和分子多样性选取法,确定每个取代位点的残基结构,取代基团的个数定在2-4个不等.在Cerius2的COMBICHEM I模块下,构建虚拟组合多肽库.
相似性比较法的原理是,在一定的性质空间里,根据某种距离函数计算出探针分子与其它分子间的距离,距离越小的分子与探针分子越相似.本文采用欧几里得的距离函数(Euclidean)29(见公式(1)),进行相似性比较计算,选取与探针分子结构最相似的氨基酸结构.
分子多样性选取法采用以距离为基础的最大差值(MaxMin)度量法29(见公式(2)),计算出与模板氨基酸结构差异性最大的几个氨基酸残基.

表1 训练集(Nos.1-16)和测试集(Nos.17-20)的氨基酸序列及生物活性实验值和预测值Table 1 Amino acid sequences of training set(Nos.1-16)and test set(Nos.17-20)and observed and calculated activities

其中,Dij表示探针分子与相似性分子间的距离,Xik和Xjk表示探针分子和相似性分子的第k个描述符.
2.3 虚拟组合多肽库分析
对虚拟库中的多肽分子进行构象优化,构象优化方法同2.1节.然后对多肽分子进行定量结构描述.选择了图形信息参数、结构参数(可旋转键数、氢键供体数、氢键受体数、分子量)、热力学参数(油水分配系数)、拓扑参数(分子柔性指数、Kappa分子形状指数、子图形指数、Chi连接指数、Wiener指数、Balaban指数、Zagreb指数)等共39个参数.应用主成分分析法(PCA)对数据库中所有39个参数进行主成分分析,提取前3个主要成分作为参数表征.
采用阶层式聚类分析方法(HCA)对数据库进行分析.阶层式聚类分析是对所有分子结构的参数空间进行距离运算,数值最近的进行组合,并计算出目标函数值来表征类差异,本文采用距离算数平均值方法计算目标函数值(见公式(3)).

其中,d(A,B)表示类A与类B间的最小距离;dij表示分子i和分子j间的距离;nA和nB表示类A和类B中包含的分子个数.
2.4 虚拟筛选
虚拟筛选就是计算预测化合物的一些性质,从而筛选出符合一定结构要求的化合物.虚拟筛选方法大致可以分为两类,即基于配体结构和基于靶标结构的虚拟筛选方法.18,30,31本文将采用QSAR模型预测和相似性搜索两种方法,对虚拟组合多肽库进行虚拟筛选.
QSAR模型预测采用多个QSAR方程预测平均值的方法.在建立的QSAR方程中,分别选取预测能力较好的前15个方程,组成N-型和P/Q-型VGCC拮抗剂QSAR预测方程组.对虚拟库中的多肽分子,分别应用氨基酸结构描述符c1,c2,c3-scales进行定量结构描述,然后分别应用N-型和P/Q-型QSAR方程组,对数据库中的化合物进行生物活性预测.每个化合物的最终预测生物活性采用15个方程预测结果的平均值.
相似性比较法采用欧几里得的距离函数进行计算.N-型和P/Q-型VGCC拮抗剂数据库的探针分子分别为MVIIA和MVIIC,多肽分子结构采用39个参数主成分分析的前三个主要成分进行定量描述.
3 结果与讨论
3.1 ω-芋螺毒素的QSAR研究
ω-芋螺毒素含有6个半胱氨酸,形成3对交叉的二硫键和4个loop结构,32具有一个保守的抑制剂半胱氨酸绳结结构,即由几个转角连接的三股反平行β-折叠和三个二硫键组成,此结构决定了ω-芋螺毒素的刚性骨架.33本文的训练集和测试集分子是在保留3对二硫键结构的基础上,进行几个氨基酸残基突变,因此多肽分子构象优化后,不会对分子整体构象产生较大影响,仍保持此刚性骨架结构.
ω-芋螺毒素MVIIA和MVIIC在序列上具有高度的相似性,由Discovery Studio(DS)软件的Sequence Analysis模块测得,两者的序列相似性为73.1%.但是MVIIA选择性地作用于N-型VGCCs,MVIIC主要作用于P/Q-型VGCCs,当浓度高于某一阈值时也作用于N-型.34为了设计高活性、高选择性的多肽类似物,本文分别建立N-型和P/Q-型ω-芋螺毒素的QSAR模型.应用氨基酸残基结构描述符c1,c2,c3-scales和G/PLS算法,分别产生了48个不同方程项数和主成分数的N-型和P/Q-型VGCCs的QSAR方程.为了减少单个QSAR方程造成的误差,以及尽量包含所有变化的7个残基结构对QSAR方程的影响,我们筛选出预测能力和拟合能力较好的前15个方程,组成QSAR预测方程组,用于多肽类似物的生物活性预测.QSAR方程组中的每个方程均有较好的拟合能力和预测能力,交叉验证相关系数(CV-r2)和相关系数(r2)都在0.89以上.
训练集和测试集化合物的实验活性、预测活性和预测偏差结果见表1,预测值为15个QSAR方程的预测平均值,N-型和P/Q-型VGCCs的生物活性实验值与预测值的相关系数分别为0.976和0.991,相关图见图1.可见两种亚型的QSAR方程组均有较好的预测能力.为了验证所建立的QSAR方程组的可靠性,选择生物活性数据与训练集来源一致的4个天然ω-芋螺毒素,CVIA、CVIB、CVIC和CVID作为测试集.其中,CVIA和CVID为选择性的N-型VGCC拮抗剂,CVIB和CVIC选择性不高,即作用于N-型,又作用于P/Q-型VGCCs.虽然4个测试集化合物与训练集化合物的序列相似性较低,但均具有较小的预测偏差,能够较好地反映出作用于N-型和P/Q-型VGCCs的差别.由于有两个测试集化合物的第三位残基与训练集不同,特别是CVID具有27个氨基酸残基,较其它类似物多1-2个残基,QSAR预测模型没有考虑进去,因此其预测的偏差较高一些.但是,本文构建的虚拟库,多肽分子均是针对训练集中变化的7个残基进行的结构改造,因此建立的QSAR方程组可以为数据库的虚拟筛选提供有效的筛选模型.

图1 N-型(A)和P/Q-型(B)VGCC拮抗剂训练集实验活性与计算活性相关图Fig.1 Plots of the observed and calculated activities of training set of N-type(A)and P/Q-type(B)VGCC antagonists
为了详细地研究ω-芋螺毒素中变化的氨基酸残基对VGCCs生物活性和选择性的影响,我们对包含所有7个残基定量描述参数的QSAR模型进行了细致地分析.筛选出方程长度是22条件下的最优模型,偏最小二乘法的主成分数为4,N-型和P/Q-型VGCC拮抗剂的QSAR方程的r2分别为0.953和0.983,CV-r2分别为0.901和0.961,QSAR方程的系数权重图见图2.由权重图的分析结果可见,位于loop2中第9位和loop4中第23位残基对活性的影响最大,其次是loop2中的10位、11位和loop4中的21位,loop2中的残基结构在ω-芋螺毒素选择性中起到关键作用.此结论与文献报道的实验结果35-37一致.与N-型和P/Q-型VGCCs结合的ω-芋螺毒素类似物,第9、10、11、21、23位氨基酸的c1,c2,c3-scales的方程系数正负值截然相反.氨基酸结构描述符c-scales是残基的整体描述参数,与分子立体形状、电子信息和分子构象密切相关,能定量地描述天然和非天然氨基酸二维和三维结构性质.24例如,方程系数较大的第9和23位残基,与N-型VGCCs结合的多肽类似物,c1,c2-scales的方程系数均为负值,c3-scales为正值,而与P/Q-型VGCCs结合的类似物,c1,c2-scales的方程系数均为正值,c3-scales的方程系数均为负值.化合物3对N-型VGCCs具有很高的选择性,它的9位Ser和23位Gly的c1,c2-scales描述符为较大的负值,而化合物13对P/Q-型VGCCs具有较高的结合活性,它的9位和23位均为Arg,Arg的c1,c2-scales描述符都是较大的正值.

图2 N-型和P/Q-型VGCC拮抗剂QSAR方程的c-scales系数图Fig.2 Plots of the c-scales coefficients of the QSAR equations of N-type and P/Q-type VGCC antagonists
定量构效关系研究结果可以为我们设计新型的N-型和P/Q-型VGCC拮抗剂提供重要的理论依据,并为进一步建立其虚拟组合多肽库提供有意义的构效关系信息.
3.2 虚拟组合多肽库构建
ω-芋螺毒素具有保守的刚性骨架结构,但是4个loop区里氨基酸残基变化,导致了它们作用于不同的靶标结构.38MVIIA与MVIIC分别由25和26个氨基酸残基组成,由于MVIIA与MVIIC相比,在第21位处少一个残基,因此以MVIIA骨架结构为基础,N-型VGCC拮抗剂虚拟多肽库模板分子取代基团为6个,取代基团位置在7、9、10、11、17、23位,分别命名为R1-R6,见图3(A).以MVIIC为模板分子的P/Q-型VGCC拮抗剂虚拟库取代基团为7个,取代位置在7、9、10、11、17、21、23位,命名为R1-R7,见图3(B).在保持ω-芋螺毒素骨架结构的基础上,构建虚拟多肽库.
应用氨基酸结构描述符c1,c2,c3-scales对182个氨基酸组成的碎片库,进行定量描述.据定量构效关系研究结果和文献报道,第9、23、11、21、7位氨基酸残基是决定ω-芋螺毒素选择性的关键基团,36,39-41因此采用相似性比较计算方法,选取与探针分子结构最相似的3个氨基酸残基作为取代基团.考虑到虚拟化学库的多样性,其它取代位置的残基分别采用聚类分析方法和以距离为基础的多样性选取法.第10位残基采用聚类分析的方法,把氨基酸碎片库分成三类,从每类中选取一个有代表性的氨基酸碎片.因为第10位氨基酸残基也是影响构效关系的关键残基,因此同时采用相似性比较法,选取1个与模板的残基结构最相似的分子.第17位采用分子多样性选取法,计算出182个碎片库中结构差异最大的2-3个残基作为取代基团.两个虚拟组合多肽库中各个取代位点的残基结构见图4.

图3 N-型(A)和P/Q-型(B)VGCC拮抗剂虚拟多肽库模板分子结构和取代位置Fig.3 Template molecule structures and substitution positions of N-type(A)and P/Q-type(B)VGCC antagonist virtual libraries
考虑到丙氨酸替换对ω-芋螺毒素构效关系的影响,在保留多肽分子中的二硫键不变的前提下,又建立了两个丙氨酸替换补充库,模板分子和取代位点与两个集中虚拟库相同.
分别把取代基团排列组合加载到各自的模板分子中,这样构建出容量分别为1036和1208个多肽分子的N-型和P/Q-型VGCC拮抗剂虚拟组合多肽库.
3.3 虚拟组合多肽库分析

图4 虚拟多肽库中各个取代位点的氨基酸残基结构Fig.4 Amino acid residue structures of substitution positions in virtual polypeptide libraries
为了分析虚拟组合多肽库中分子结构的差异性和多样性,对数据库进行了主成分分析和阶层式聚类分析.数据库中每个多肽分子采用39个参数进行定量结构描述,应用主成分分析法,提取出的前3个主要成分作为参数表征,两个库的前3个主要成分分别可以代表所有参数的92.59%和91.95%.主成分分析的前三个主要成分的三维空间分布见图5,由计算结果可见,数据库中的化合物都较均匀地分布于结构参数的3D空间中,说明所构建的数据库分子结构具有一定的多样性和差异性.
为了进一步对数据库中的化合物进行分析,我们对数据库中的化合物进行了阶层式聚类分析.阶层式聚类分析就是根据化合物的目标函数值,即类与类间的最小距离,把它们分成不同的层次结构,然后通过类的个数把化合物集合分成不同的类,因此它可以显示出数据库的分布情况.采用系统树图的方法显示聚类分析的结果,由N-型和P/Q-型两个虚拟库的聚类分析图可知(见图6),两个数据库分别能分成600多类和200多类,而且类与类之间的距离分布差别较大,说明数据库中的化合物具有较好的结构差异性和多样性.
3.4 虚拟筛选
分别应用N-型和P/Q-型VGCC拮抗剂QSAR方程组,对两个数据库中化合物进行生物活性预测.在对N-型数据库筛选中,预测出对N-型VGCCs的结合活性高于MVIIA的化合物47个,且它们对P/Q-型 VGCCs的结合活性(单位为 nmol∙L-1)均在5.76-6.42之间.预测出对N-型VGCCs结合活性高于9.00的化合物276个,而对P/Q-型VGCCs的结合活性都在7.5以下.在对P/Q-型虚拟库筛选中,得到对P/Q-型VGCCs的结合活性高于9.50的化合物322个,对N-型VGCCs的结合活性都在7.30以下,预测活性和选择性均好于MVIIC.由于MVIIA只作用于N-型VGCCs,而MVIIC主要作用于P/Q-型,也作用于N-型,因此MVIIC的选择性没有MVIIA强.所以我们建立的P/Q-型VGCC拮抗剂虚拟库中大部分化合物,预测出的对P/Q-型VGCCs的结合活性都高于MVIIC.通过对两个多肽数据库的活性预测分析可见,两个数据库中的多肽分子均具有较好的选择性.

图5 虚拟多肽库主成分分析的前三个主要成分三维空间分布Fig.5 Three-dimensiona space distribution of the first three principal components of PCAfor virtual polypeptide libraries

图6 N-型(A,C)和P/Q-型(B,D)VGCC拮抗剂虚拟库的聚类分析系统树图(A,B)和目标函数值分布图(C,D)Fig.6 Polts of the dendrogram(A,B)and objective function value distributoins(C,D)of HCAfor N-type(A,C)and P/Q-type(B,D)VGCC antagonists virtual libraries
对两个数据库中化合物的预测活性进行排序,各自选出活性较高的前150个化合物,组成两个新的子库,作为进一步筛选的依据.
同时,对N-型和P/Q-型两个虚拟多肽库采用相似性比较分析方法进行虚拟筛选,分别选出与探针分子MVIIA和MVIIC结构最相似的多肽类似物150个,组成两个新的子库.
把应用两种筛选方法得到的子库进行比较分析,从中筛选出共同的化合物.最终得到预测高活性和高选择性的N-型和P/Q-型VGCC拮抗剂25个.其中,筛选出的前6个N-型钙通道拮抗剂,与N-型QSAR模型的预测活性均大于9.90,与P/Q-型QSAR模型的预测活性均小于6.95.筛选出的19个P/Q-型钙通道拮抗剂,与P/Q-型QSAR模型的预测活性均大于9.65,与N-型QSAR模型的预测活性均小于7.10.可见,筛选出的这些化合物均具有较高的生物活性和选择性.表2和表3分别列出筛选出的前6个N-型和P/Q-型VGCC拮抗剂的序列和预测活性.
为了研究筛选出的多肽分子与模板分子的结构相似性和结构功能,我们对排名第一的N-型和P/Q-型多肽分子的关键残基进行了结构定量描述分析和分子表面静电势计算.图7示出MVIIA与N-型及MVIIC与P/Q-型多肽分子的6个和7个残基描述符c-scales的比对结果.结果显示,筛选出的多肽分子与模板分子的残基描述符c1,c2,c3-scales数值基本相近,特别是决定分子选择性的loop2中的第9、10、11位残基和loop4中的第23位残基,由c1,c2,c3-scales结果可以体现出N-型和P/Q-型拮抗剂选择性残基的结构性质差别,从氨基酸残基角度阐明了筛选出的分子与模板分子的结构相似性.
应用DS/Delphi模块,通过有限差分方法求解Possion-Boltzmann(PB)方程,获得模板分子和筛选分子的表面静电势分布.其中,溶液的离子强度设为0.145,蛋白质部分的介电常数设为2,溶液的介电常数设为80.网格中心设为分子中心,探针原子半径为0.14 nm.图8示出MVIIA、MVIIC与筛选排名第一的N-型、P/Q-型多肽分子的表面静电势分布情况,采用溶剂可及表面示出.蓝色代表正电区域,白色代表中性区域,红色代表负电区域.静电势的显示区间为-5.0KT/e至5.0KT/e,其中,T为温度K,K为Boltzmann常数,e是电荷单位.计算结果显示,筛选出的分子与模板分子具有较一致的静电势分布,MVIIA和MVIIC序列中不同的7个残基多数具有正的静电势.ω-芋螺毒素中具有正静电势的碱性残基,在与钙离子通道结合时起到重要作用,14,42其中第7和第23位残基较强的正静电势,是N-型拮抗剂选择性的关键.此外,筛选出的多肽分子保持了ω-芋螺毒素保守的刚性骨架结构,与模板分子具有一致的空间立体结构,例如,N-型拮抗剂的第7位残基为较大的空间体积,第23位为较小的空间体积,而P/Q-型拮抗剂与其正好相反.

表2 虚拟筛选出的前6个N-型VGCC拮抗剂预测活性及序列Table 2 Calculated activities and sequences of the top 6 N-type VGCC antagonists from virtual screening
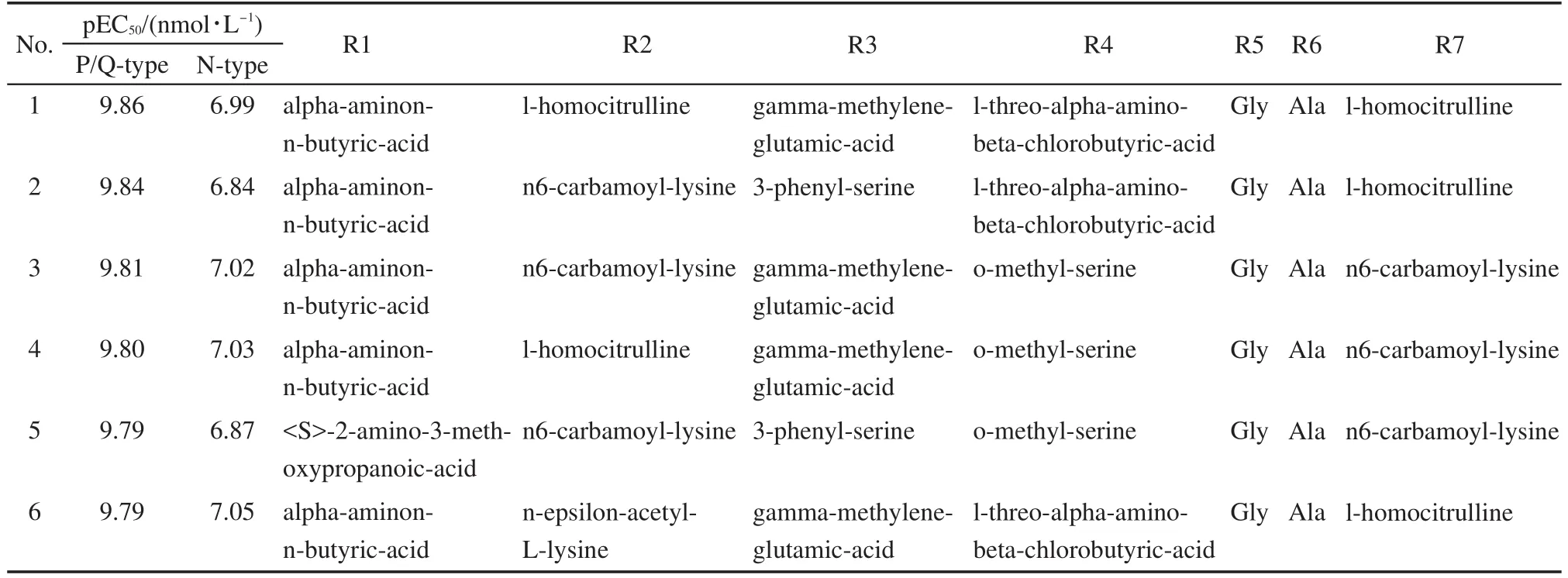
表3 虚拟筛选出的前6个P/Q-型VGCC拮抗剂预测活性及序列Table 3 Calculated activities and sequences of the top 6 P/Q-type VGCC antagonists from virtual screening

图7 MVIIA和MVIIC与筛选排名第一的N-型(A)和P/Q-型(B)拮抗剂残基描述符c-scales比较图Fig.7 Residue descriptor c-scales of MVIIAand MVIIC versus the top 1 of N-type(A)and P/Q-type(B)VGCC antagonists from virtual screening
多肽QSAR模型预测方法是基于氨基酸残基结构进行定量描述,此方法详细全面地收集氨基酸残基的二维和三维结构信息,更多地表达多肽分子的氨基酸残基结构信息.而相似性比较方法是基于整个多肽分子定量结构描述,体现多肽分子整体二维和三维结构信息,两种筛选方法各有优缺点.我们把两种筛选方法有机结合起来,相互补充,可以有效地提高筛选质量和效率.

图8 MVIIA和MVIIC与筛选排名第一的N-型和P/Q-型拮抗剂的表面静电势分布Fig.8 Electrostatic potential distributions of MVIIAand MVIIC versus the top 1 of N-type and P/Q-type VGCC antagonists from virtual screening
4 结论
对生物活性多肽ω-芋螺毒素进行QSAR研究,建立具有较好预测能力的N-型和P/Q-型VGCC拮抗剂多肽QSAR模型.设计和构建两种亚型VGCC拮抗剂的虚拟组合多肽库,并进行多重虚拟筛选,为进一步ω-芋螺毒素生物活性多肽分子设计和结构优化提供理论依据,同时为其它类型多肽分子设计提供思路和参考.目前,多肽QSAR、虚拟组合多肽库以及多肽虚拟筛选方法都在不断地发展,相信随着分子生物学、计算机科学以及多尺度模型的发展,计算机辅助分子设计技术会更加有效地指导创新多肽药物的发现和优化.
(1)Vink,S.;Alewood,P.Br.J.Pharmacol.2012,167,970.doi:10.1111/j.1476-5381.2012.02082.x
(2) Schroeder,C.I.;Lewis,R.J.Mar.Drugs2006,4,193.doi:10.3390/md403193
(3) Tetsuyuki,W.;Junichi,A.;Takeshi,M.;Takashi,M.;Seiji,I.Neurochem.Res.2003,28,705.doi:10.1023/A:1022805615926
(4) Lewis,R.J.;Dutertre,S.;Vetter,I.;Christie,M.J.Pharmacol.Rev.2012,64,259.
(5)Tetsuyuki,W.;Takashi,I.;Akinori,K.;Masayuki,X.M.;Yasuo,M.;Keiji,I.;Seiji,I.Neurochem.Res.2005,30,1045.doi:10.1007/s11064-005-7046-6
(6) Obafemi,A.;Roth,B.Pain Med.2013,14,447.doi:10.1111/pme.2013.14.issue-3
(7)Bourinet,E.;Zamponi,G.W.Curr.Top.Med.Chem.2005,5,539.doi:10.2174/1568026054367610
(8) Lynch,S.S.;Cheng,C.M.;Yee,J.L.Ann.Pharmacother.2006,40,1293.doi:10.1345/aph.1G584
(9)Alicino,I.;Giglio,M.;Manca,F.;Bruno,F.;Puntillo,F.Pain2012,153,245.doi:10.1016/j.pain.2011.10.002
(10) Sanford,M.CNS Drugs2013,27,989.doi:10.1007/s40263-013-0107-5
(11) Sasaki,T.;Kobayashi,K.;Kohno,T.;Sato,K.FEBS Lett.2000,466,125.doi:10.1016/S0014-5793(99)01772-X
(12) Wang,C.Z.;Jiang,H.;Qi,Z.W.Prog.Biochem.Biophys.2003,30,537.[王承忠,蒋 辉,戚正武.生物化学与生物物理进展,2003,30,537.]
(13) Kolosov,A.;Aurini,L.;Williams,E.D.;Cooke,I.;Goodchild,C.S.Pain Med.2011,12,923.doi:10.1111/pme.2011.12.issue-6
(14) Fedosova,A.E.;Moshkovskiic,S.A.;Kuznetsovac,K.G.;Oliverab,B.M.Biochemistry-Moscow+Series B:Biomed.Chem.2012,6,107.
(15) Kolosov,A.;Goodchild,C.S.;Cooke,I.Pain Med.2010,11,262.doi:10.1111/pme.2010.11.issue-2
(16)Zhan,D.L;Wang,S.;Han,W.W.;Liu,J.S.Acta Chim.Sin.2012,70,217.[詹冬玲,王 嵩,韩葳葳,刘景圣,化学学报,2012,70,217.]doi:10.6023/A1108313
(17) Zhang,Q.Q.;Yao,Q.Z.;Zhang,S.P.;Bi,L.M.;Zhou,Z.G.;Zhang,J.Acta Phys.-Chim.Sin.2014,30,371.[张青青,姚其正,张生平,毕乐明,周之光,张 骥.物理化学学报,2014,30,371.]doi:10.3866/PKU.WHXB201312192
(18) Hou,T.J.;Xu,X.J.Curr.Pharm.Des.2004,10,1011.doi:10.2174/1381612043452721
(19) Ding,J.J.;Ding,X.Q.;Zhao,L.F.;Chen,J.S.Prog.Chem.2005,17,130.[丁俊杰,丁晓琴,赵立峰,陈冀胜.化学进展,2005,17,130.]
(20)Audie,J.;Swanson,J.Chem.Biol.Drug.Des.2013,81,50.doi:10.1111/cbdd.2012.81.issue-1
(21) Hou,T.J.;Li,N.;Li,Y.Y.;Wang,W.J.Proteome Res.2012,11,2982.doi:10.1021/pr3000688
(22)Hou,T.J.;Li,Y.Y.;Wang,W.Bioinformatics2011,27,1814.doi:10.1093/bioinformatics/btr294
(23)Hou,T.J.;Xu,Z.;Zhang,W.;McLaughlin,W.A.;Case,D.A.;Xu,Y.;Wang,W.Mol.Cell.Proteomics2009,8,639.doi:10.1074/mcp.M800450-MCP200
(24) Ding,J.J.;Ding,X.Q.;Zhao,L.F.;Chen,J.S.Acta Pharm.Sin.2005,40,340.[丁俊杰,丁晓琴,赵立峰,陈冀胜.药学学报,2005,40,340.]
(25) Atkinson,R.A.;Kieffer,B.;Dejaegere,A.;Sirockin,F.;Lefevre,J.F.Biochemistry2000,39,3908.doi:10.1021/bi992651h
(26) Nielsen,K.J.;Adams,D.;Thomas,L.;Bond,T.;Alewood,P.F.;Craik,D.J.;Lewis,R.J.J.Mol.Biol.1999,289,1405.doi:10.1006/jmbi.1999.2817
(27) Richard,J.L.;Katherine,J.N.;David,J.C.;Marion,L.L.;Denise,A.A.;Iain,A.S.;Tudor,L.;David,J.A.;Trudy,B.;Linda,T.;Aluu,J.;Jodi-Lea,M.;Roger,D.;Peter,R.A.;Paul,F.A.J.Biol.Chem.2000,275,35335.doi:10.1074/jbc.M002252200
(28) Mason,J.S.Computer-Assisted Drug Design;Science Press:Beijing,2007;p 307.
(29)Cerius2User Guide;Molecular Simulations Inc:San Diego,2000.
(30)Drwal,M.N.;Griffith,R.Drug Discovery Today:Technologies2013,10,e395.
(31)Wilson,G.L.;Lill,M.A.Future Med.Chem.2011,3,735.doi:10.4155/fmc.11.18
(32) Ggorge,P.M.;Ramachandran,J.Annu.Rev.Pharmacol.Toxicol.1995,35,307.doi:10.1146/annurev.pa.35.040195.001515
(33) Pallaghy,P.K.;Nielsen,K.J.;Craik,D.J.;Norton,R.S.Protein Sci.1994,3,1833.doi:10.1002/pro.v3:10
(34)Adams,D.J.;Berecki,G.Biochim.Biophys.Acta2013,1828,1619.doi:10.1016/j.bbamem.2013.01.019
(35) Kazuki,S.;Cecile,R.;Nicole,M.M.;Toru,S.;Akira,O.;Atsuko,O.;Jae,I.K.;Toshiyuki,K.;Masami,T.;Michael,S.FEBS Lett.1997,414,480.doi:10.1016/S0014-5793(97)01056-9
(36)Nielsen,K.J.;Adams,D.A.;Alewood,P.F.;Lewis,R.J.;Thomas,L.;Schroeder,T.;Craik,D.J.Biochemistry1999,38,6741.doi:10.1021/bi982980u
(37) Mould,J.;Yasuda,T.;Schroeder,C.I.;Beedle,A.M.;Doering,C.J.;Zamponi,G.W.;Adams,D.J.;Lewis,R.J.J.Biol.Chem.2004,279,34705.
(38)Van Petegem,F.;Minor,D.L.Biochem.Soc.Trans.2006,34,887.doi:10.1042/BST0340887
(39) Kajuti,S.;Cecile,R.;Nicole,M.M.;Toru.S.;Atsuko,O.FEBSLett.2000,469,147.doi:10.1016/S0014-5793(00)01263-1
(40) Kazuki,S.;Cecile,R.;Nicole,M.M.;Toru,S.;Atsuko,O.;Kazushi,M.;Catherine,V.R.;Masami,T.;Michael,J.S.Biochem.Biophys.Res.Commun.2000,269,254.doi:10.1006/bbrc.2000.2284
(41) Lew,M.J.;Flinn,J.P.;Pallaghy,P.K.;Murphy,R.;Whorlow,S.L.;Wright,C.E.;Norton,R.S.;Angus,J.A.J.Biol.Chem.1997,272,12014.doi:10.1074/jbc.272.18.12014
(42) Nadasdi,L.;Yamashiro,D.;Chung,D.;Tarczy-Hornoch,K.;Adriaenssens,P.;Ramachandran,J.Biochemistry1995,34,8076.doi:10.1021/bi00025a013

