山羊CREM基因的克隆与生物信息学分析*
,,,* ,,
(1.中国热带农业科学院 热带作物品种资源研究所,海南 儋州 571737;2.山西农业大学,山西 太谷 030801)
环磷酸腺苷应答元件调节因子(CREM)作为亮氨酸拉链转录因子家族的一个成员,在精子发生过程中起着重要的作用。在精子变态过程中,染色质凝聚状态、精子细胞核的形状和大小均发生改变,过渡蛋白(TP)和精蛋白(protamine)在此过程中发挥很重要的作用,另外研究还发现过渡蛋白和精蛋白基因启动子区域均含有CRE元件,这为CREM 在精子变态过程中的作用提供了直接依据[1]。此外,CREM 还调节以下基因的表达:编码信号蛋白的基因,如蛋白激酶A 调节亚单位II,其作用为参与cAMP 信号途径[2]。许多调控精子变形的重要基因,如鱼精蛋白1和2[3]、PHGPx[4]、TP1[5]、RT7[6]、钙精蛋白[7]和ACE[8]等。其中鱼精蛋白1和2是长形精子细胞的DNA结合蛋白。另外研究还表明CREM 对肝脏的再生和增殖[9-10]、心脏功能[11-12]及下丘脑-垂体轴功能以及褪黑激素的节律产生起很重要的调节作用[13]。CREM基因表达受多重水平调节,通过多重水平调节形成不同功能活性的蛋白质,包括活化型和抑制型。
1991年,Foulkes等[14]首次通过筛选小鼠脑垂体cDNA文库,获得小鼠CREM基因 cDNA 序列,为编码341个氨基酸的蛋白质。通过氨基酸序列比对发现,与CREB存在95 %的同源性[15]。随后,人、狗、大鼠和鸡等物种的CREM基因 cDNA序列相继被克隆。但山羊CREM基因的cDNA序列、结构及功能的研究还没有报道。本研究以太行青山羊为试验动物,提取睾丸组织RNA,采用RT-PCR、5’RACE和3’RACE技术获取CREM基因cDNA序列,应用生物信息学软件分析预测氨基酸序列、功能位点、蛋白二级结构、空间结构及修饰位点,旨在为CREM翻译后表达调控机制的研究提供基础信息。
1 材料与方法
1.1 试验动物
选用5只成年太行青山羊公羊,对其实施阉割手术,采集睾丸组织,放入已处理过的冻存管后投入液氮保存。
1.2 RNA提取
依照TaKaRa RNAiso Reagent说明书操作要求提取山羊睾丸总RNA,通过Nanodrop-1000核酸蛋白测定仪检测纯度、浓度及A230/A260和A260/A280,然后琼脂糖变性电泳检测RNA的完整性,贮存于-80 ℃冰箱。
1.3 引物设计
根据GenBank中公布的人(S68271.1)、鼠(BC078899)、牛(NM_001034710)、绵羊(NM_001104935)CREM基因同源序列的ORF序列进行BLAST分析,在高度保守区域设计扩增序列特异性引物SP1,5’RACE和3’RACE引物根据TaKaRa生物公司 RACE Core Set Ver. 2.0试剂盒操作说明要求,通过Primer 5.0软件设计,引物交由上海生物工程公司合成,试验所用引物序列见表1。

表1 扩增山羊CREM基因所用引物序列Table 1 Primers used in the analysis of goat CREM gene
1.4 CREM 基因全长cDNA克隆
1.4.1 CREM基因cDNA中间片段克隆 使用SYBR® PrimeScriptTM RT-PCR reagent Kit中的反转录反应试剂进行反转录实验,构建10 μL反转录体系:1.5×PrimeScriptTM Buffer 2 μL, PrimeScriptTM RT Enzyme Mix I 0.5 μL, Oligo dT Primer(50 μM)0.5 μL, Random 6(100 μM)0.5 μL, Total RNA 0.5 μL(500 ng), RNase-Free dH2O 6 μL;反应条件:37 ℃ 15 min,85 ℃ 5 s。mRNA转录为cDNA后用作后续PCR模板,扩增引物SP1,构建15 μL体系: cDNA模板1.0 μL,10×PCR buffer 2.5 μL, 2.5 mM dNTPs 2.0 μL, Taq DNA聚合酶0.5 μL, Primer F(10 μm)0.5 μL, Primer R(10 μm)0.5 μL, ddH2O 8 μL; 反应条件:94 ℃ 预变性5 min;94 ℃ 30 s,55 ℃ 30 s, 72 ℃ 50 s, 35个循环;72 ℃延伸 8 min。反应结束后1 %的琼脂糖凝胶电泳对PCR产物进行鉴定。
1.4.2 3’RACE和5’RACE扩增 以提取的山羊睾丸组织总RNA为模板,严格按照TaKaRa生物公司RACE Core Set Ver. 2.0试剂盒说明进行3’RACE和5’RACE扩增。扩增产物经割胶回收、连接转化、定向克隆至T-Vector,测序。
1.5 序列分析
分段扩增及RACE获得的山羊CREM基因序列利用DNAStar软件中的SeqMan程序拼接,拼接产物(核酸序列)用DNAStar软件中的Edit Seq程序翻译成氨基酸序列。并通过http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html、http://smart.embl-heidejberg.de/、http://www.rcsb.org/pdb等网站预测山羊CREM二级、三级结构及功能结构域。
2 结果与分析
2.1 CREM基因 cDNA特异片段及RACE扩增结果
本试验从成年太行青山羊睾丸组织提取的RNA进行cDNA第一条链的合成,然后以山羊睾丸cDNA第一链为模板,用SP1F、SP1R引物进行扩增CREM基因cDNA特异性片段,产物经1.2 %琼脂糖凝胶电泳检测,紫外灯下成像分析,观察到一条带,为900 bp左右,与预期的目的条带大小相符,说明扩增成功。以合成的RACE cDNA为模板,通过套式PCR反应,3’RACE和5’RACE分别扩增得到约250 bp、1 000 bp的片段(图1),将所得产物进行纯化,与pMD18-T载体连接,转化到感受态细胞(E.coliJM109),筛选单克隆质粒后送上海生工测序,经GenBank BLAST结果表明,该序列属于CREM基因3’端和5’端序列。
2.2 CREM基因cDNA全长序列分析结果
2.2.1 CREM cDNA全长序列的获得 用DNASTAR软件SeqMan程序对RT-PCR和RACE产物测序结果进行拼接分析,山羊CREM cDNA 全长序列为1 183 bp(登录号:HM 802260),包含有960 bp的开放阅读框,编码319个氨基酸。该基因3’非翻译区长87 bp,3’非翻译区有真核生物典型的PolyA加尾结构(图2)。
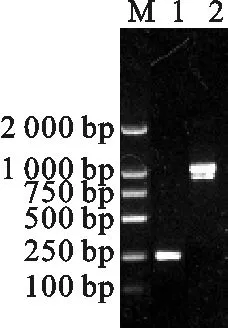
图1 3’RACE和5’RACE扩增产物M.DL 2000 Marker;1.3’RACE 扩增产物;2.5’RACE 扩增产物Fig.1 Products for 3’RACE and 5’RACE
2.2.2 功能结构域的预测 应用SMART服务器分析对结构域功能进行预测,发现在319个氨基酸序列中,73到115之间有一个“pKID”区域,259~316之间含有一个典型的“BRLZ”结构,说明属于bZIP转录因子家族。
2.2.3 跨膜区分析 用ExPASy 提供的在线TMHMM跨膜区预测软件(http://www.cbs.dtu.dk/services/TMHMM/)分析山羊CREM氨基酸序列未发现跨膜区(图4)。

图2 山羊CREM基因cDNA及其氨基酸序列Fig.2 Nucleotide sequence and predicted amino acid sequence of CREM in goat
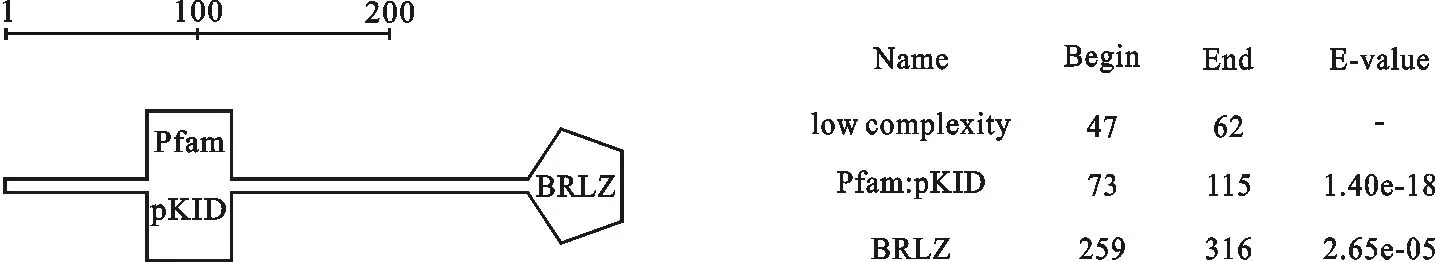
图3 山羊CREM结构功能域分析Fig.3 Function domain analysis of goat CREM
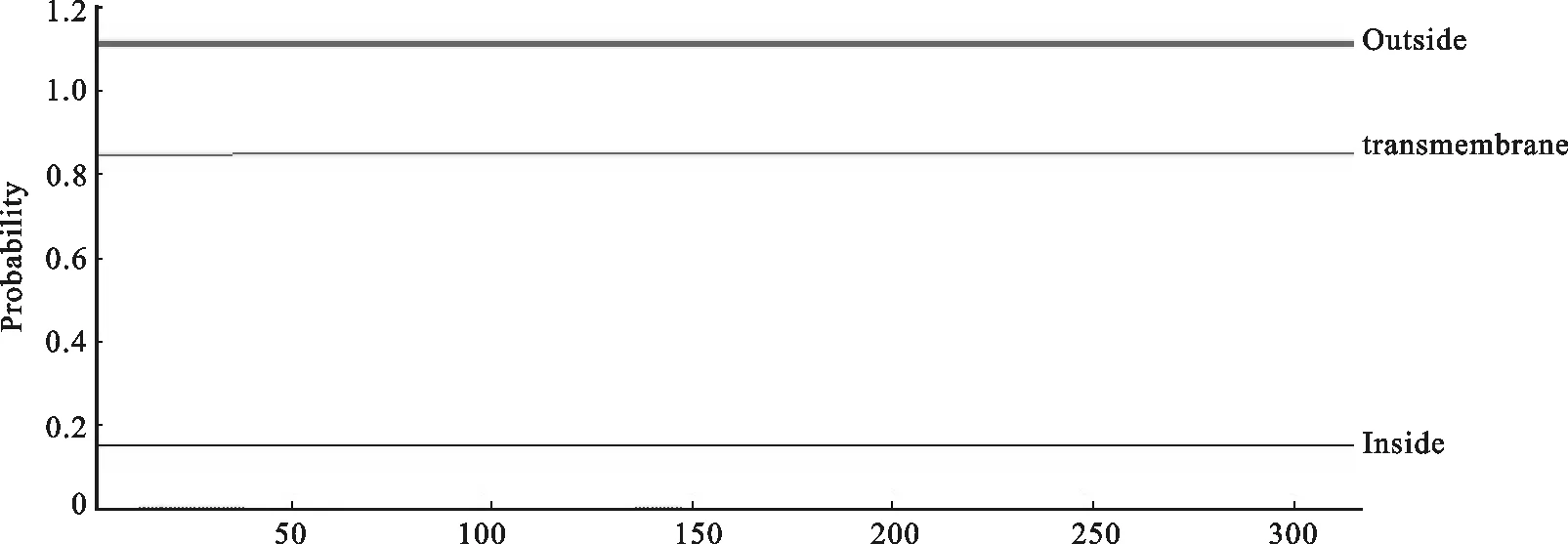
图4 TMHMM软件分析山羊CREM 跨膜区结果Fig.4 Prediction of transmembrane regions of goat CREM using TMHMM
2.2.4 二级结构的预测 通过GOR方法对二级结构预测分析结果如图5显示,山羊CREM蛋白由α-螺旋(约占37.30 %),β-折叠(约占18.81 %)和卷曲螺旋(约占43.89 %)3种模块组成,以α-螺旋和卷曲螺旋为主。
2.2.5 三级结构预测 通过SWISS-MODEL软件在线预测所推导的山羊CREM蛋白三级结构。以PDB数据库中的1 kdxB(99.9 Å)被选定为模板,待测蛋白质与模板的序列相似性达到88.889 %,建模的氨基酸由82~108,评估值为2.33172e-05,符合建模标准。预测模型见图7。由图7可见,山羊CREM蛋白三级结构包括两个核心螺旋。运用Swiss-Pdb Viewer 软件进行模型质量评价。图8为CREM预测3D模型的拉氏构象图。从结果可以看出,氨基酸在拉氏构象图中较集中的位置形成二级结构,基本判断CREM 3D模型是合理的。三级结构预测结果显示螺旋结构的全部氨基酸在第三象限的黄色区域内,说明模型可信度高。
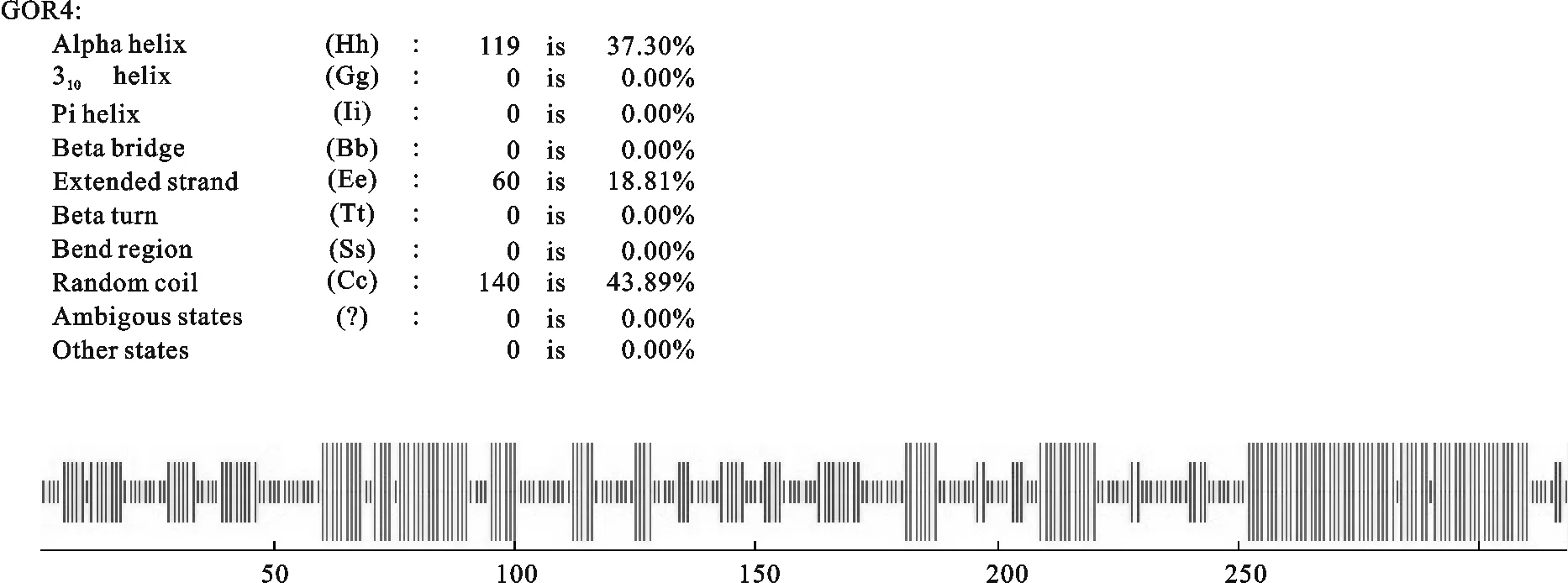
图5 山羊CREM 二级结构预测结果Hh.α-螺旋; Ee.延伸链;Cc.无规则卷曲Fig.5 Predicted secondary structure of goat CREM protein Hh. Alpha helix; Ee. Extended strand; Cc.Random coil
2.2.6 序列多重比对和分子进化树分析 将山羊CREM 基因编码区核苷酸和氨基酸序列分别与GenBank 中已登录的牛(NM_001034710.2)、绵羊(NM_001104935.1)、狗(X99115)、人(S68271.1)、褐鼠(BC078899.1)的CREM基因进行比较,结果显示核苷酸同源性分别为96.2%、89.5%、88.9%、85.0% 和90.7%,氨基酸同源性分别为99.4%、85.9%、86.8%、84.7%和96.2%。
并用基于氨基酸序列的NJ 法构建分子系统发育树(图8),结果显示山羊与牛亲缘关系最近,人次之,与小鼠、褐鼠、马、狗和绵羊的亲缘关系较为相近,与鲑鱼的亲缘关系最远。

图6 CREM蛋白的三维结构预测结果
Fig.6 Protein tertiary structure prediction of CREM
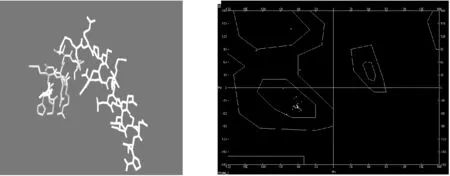
图7 CREM的三级模型拉氏构象图Fig.7 Ramachandran plot of goat CREM

图8 CREM蛋白的系统发育树分析Fig.8 Phylogenetic tree of CREM
3 讨 论
自1991年Foulkes等[14]首次获得小鼠CREM基因cDNA 序列,随后许多物种的CREM基因cDNA序列被成功克隆。本研究通过RACE技术成功克隆得到全长1 183 bp山羊CREM基因cDNA序列(登录号:HM 802260),包含有960 bp的开放阅读框,编码319个氨基酸。该基因3’非翻译区长87 bp,3’非翻译有真核生物典型的PolyA加尾结构。 3’非翻译区有一个“ATTTA”序列,与RNA的稳定性有关[15-16]。山羊CREM基因cDNA序列与牛相似,3’非翻译区仅存在一个PolyA位点,区别于小鼠等其他哺乳动物CREM基因的3’末端的两个polyA 位点[17]。对CREM氨基酸序列预测发现包含两个富含谷氨酸结构域,一个PKA磷酸化位点,以及一个碱性亮氨酸拉链结构域,均属于CREB亚族典型的功能结构域,表明本研究克隆出的CREM基因cDNA序列及氨基酸序列属于典型的bZIP转录因子家族。
通过应用SMART服务器对CREM结构域功能进行预测,发现在319个氨基酸序列中,73~115之间有一个“pKID”区域,259~316之间含有一个典型的“BRLZ”结构,pKID区域为激酶诱导结构域,是蛋白激酶对CREM蛋白进行磷酸化的部位[18],是激活CREM蛋白必不可少的区域。“BRLZ”结构为bZIP结构域,其主要作用是调控蛋白/DNA的相互作用,二聚化是蛋白结合DNA所必需的,二聚化的蛋白与CRE启动子元件结合。现已证实,许多参与精子发生过程的基因,其启动子位点均含有CREs[19]。因此,bZIP区对CREM发挥正常的生物学功能是必需的,而且也是保守的。CREM CDS序列同源性分析结果表明,与牛、绵羊、狗、人和褐鼠同源性分别为96.2 %、89.5 %、88.9 %、85.0 % 和90.7 %,氨基酸序列同源性分别为99.4 %、85.9 %、86.8 %、84.7 %和96.2 %,说明CREM在进化过程中具有较高的保守性。
蛋白质的生物功能很大程度上取决于其空间结构,致使蛋白质结构的预测成为了目前研究的热门课题。二级结构预测分析结果显示,山羊CREM蛋白有α-螺旋、β-折叠和卷曲螺旋3种模块,以α-螺旋和卷曲螺旋为主。通过同源建模,与PDB数据库中1kdxB (99.9 A)三维结构高度相似,通过Swiss-Pdb Viewer的三级结构和拉氏构象图同样也验证了CREM蛋白结构非常保守。本试验通过对山羊CREM cDNA序列成功克隆,并对其进行生物信息学分析,为后续深入探讨山羊CREM蛋白生物学功能打下基础。
4 结 论
采用RACE技术成功克隆出太行青山羊睾丸CREM基因在睾丸组织中cDNA序列,GenBank收录号为HM 802260。应用生物信息学软件分析预测了二级结构、空间结构及功能区域,通过与牛、绵羊、狗、人和褐鼠等其它物种的同源性及进化树分析,CREM具有亮氨酸拉链转录因子家族同源基因的典型序列特征和功能结构域。
参考文献:
[1] Kotaja N, De Cesare D, Macho B, et al. Abnormal sperm in mice with targeted deletion of the act (activator of cAMP-responsive element modulator in testis) gene[J].Proc Nati Acad Sci U S A,2004,101(29):10 620-10 625.
[2] Dadoune J P. Expression of mammalian spermatozoal nucleo proteins[J]. Microsc Res Tech, 2003, 61(1):56-75.
[3] Hecht N, Cavalcanti M C, Nayudu P, et al. Protamine-1 represents a sperm specific gene transcript: a study in Callithrix jacchus and Bos Taurus[J].Andrologia,2011, 43(3):167-173.
[4] Tramer F, Vetere A, Martinelli M, et al. cAMP-response element modulator-τ activates a distinct promoter element for the expression of the phospholipid hydroperoxide/sperm nucleus glutathione peroxidase gene[J].J Biochem,2004,383:179-185.
[5] Kistler M K, Sassone-Corsi P, Kistler W S. Identification of a functional cyclic adenosine 3’, 5’-monophosphate response element in the 5’-flanking region of the gene for transition protein 1 (TP1), a basic chromosomal protein of mammalian spermatids[J]. Biol Reprod, 1994, 51:1 322-1 329.
[6] Van der Hoorn F A. Identification of the testis c-mos promoter: specific activity in a seminiferous tubule-derived extract and binding of a testis-specific nuclear factor [J].Oncogene, 1992, 7(6):1 093-1 099.
[7] Sun Z, Sassone-Corsi P, Means A. Calspermin gene transcription is regulated by two cyclic AMP response elements contained in an alternative promoter in the calmodulin kinase IV gene [J].Mol Cell Biol, 1995, 15:561-571.
[8] Zhou Y, Sun Z, Means A R, et al. cAMP-response element modulator tau is a positive regulator of testis angiotensin converting enzyme transcription [J]. Proc Natl Acad Sci U S A, 1996, 93(22):12 262-12 266.
[9] Halder S, Fink M, Waterman M, et al. cAMP responsive element binding site is essential for sterol regulation of the human lanosterol 14a α-demethylase gene (CYP51)[J].Mol Endocrinol,2002,16:1 853-1 863.
[10] Acimovic J, Fink M, Pompon D, et al. CREM modulates the circadian expression of CYP51, HMGCR and cholesterogenesis in the liver[J].Biochem Biophys Res Commun,2008, 376(1):206-210.
[11] Mueller F U, Lewin G,Matus M, et al. Impaired cardiac contraction and relaxation and decreased expression of sarcoplasmic Ca2+-ATPase in mice lacking the CREM gene [J].FASEB J, 2003, 17:103-105.
[12] Isoda T, Paolocci N, Haghighi K, et al. Novel regulation of cardiac force-frequency relation by CREM (cAMP response element modulator)[J].FASEB J,2003,17:144-151.
[13] Maldonado R, Smadja C, Mazzucchelli C, et al. Altered emotional and locomotor responses in mice deficient in the transcription factor CREM[J].Proc Natl Acad Sci USA,1999,96:14 094-14 099.
[14] Foulkes N S, Laoide B M, Schlotter F, et al. Transcriptional antagonist cAMP-responsive element modulator (CREM) down-regulates c-fos cAMP-induced expression[J].Proc Natl Acad Sci U S A,1991,88(12):5 448-5 452.
[15] Shaw G, Kamen R A. Conserved AU sequence from the 3’ untranslated region of GM-CSF mRNA mediates selective mRNA degradation [J].Cell, 1986, 46:659-667.
[16] Abdel W N, Gibbs J, Mason R M. Regulation of gene expression by alternative polyadenylation and mRNA instability in hyperglycaemic mesangial cells[J]. Biochem J,1998,336:405-411.
[17] Hoeffler J P, Meyer T E, Waeber G, et al. Multiple adenosine 3’, 5’-cyclic adenosine monophosphate response element DNA-binding proteins generated by gene diversification and alternative exon splicing[J].Mol Endocrinol,1990,4:920-930.
[18] Fimia G M, De Cesare D, Sassone-Corsi P. CBP-independent activation of CREM and CREB by the LIM-only protein ACT[J].Nature,1999,398(6723):165-169.
[19] Sassone-Corsi P. CREM: a master-switch governing male germ cell differentiation and apoptosis semin[J].Cell Dev Biol,1998,9:475-482.

