HPT-mCherry融合标签在水稻转基因筛选鉴定中的应用
陈娟 ,周洁 ,严成其 ,王栩鸣 ,杨勇 ,余初浪 ,程晔 ,陈剑平
1.浙江师范大学 生化学院,浙江 金华 321000;2.浙江省农业科学院 省部共建国家重点实验室培育基地,农业部植物保护与生物技术重点实验室,浙江省植物病毒重点实验室,浙江 杭州 310021
农杆菌介导的水稻转基因技术是利用生物学手段将目的基因导入水稻基因组中的重要方法,主要用于水稻性状的改良和基因功能的研究[1-2]。自1994年Hiei等建立农杆菌介导的水稻转化体系以来[3],水稻转基因技术得到了广泛应用和发展。水稻转基因技术需要借助标记基因来筛选转化子,实验中常用的标记基因分为两类,即选择基因和报告基因[4]。目前常用选择基因对转化的材料进行较为高效的筛选,并利用其他实验进行辅助筛选。常用的选择基因包括潮霉素磷酸转移酶基因(hygromycin phosphotransferase gene,HPT)[5]和抗除草剂基因(bi⁃alaphos resistance gene,bar)[6]等。使用潮霉素筛选抗性愈伤,可能筛选得到假阳性的愈伤组织,往往需要借助PCR等方法检测进行辅助鉴定,而这些方法通常都比较耗时费力。为了提高辅助筛选的效率,目前很多研究选用报告基因进行辅助筛选。报告基因是一种编码可被检测的酶或蛋白质的基因,例如β-葡萄糖苷酸酶基因(β-glucuronidase gene,gus)和荧光蛋白基因等[7-8]。gus通过编码β-葡萄糖苷酸酶将底物水解为蓝色产物,可进行组织化学检测,但检测过程中需要破坏植物组织,限制了其在活体中的应用[9]。相比gus基因,荧光蛋白基因的检测较为简便,被广泛使用。
虽然报告基因的辅助筛选是一种比较经济有效的方式,但还是存在一些无法避免的缺陷。最主要的缺陷就是报告基因需要额外的启动子和终止子来控制其表达。启动子和终止子是控制基因表达的重要元件[4],而转基因实验中常用启动子和终止子数量有限,重复使用相同的启动子或终止子,会导致转化时发生遗传重组,存在一定的风险[4,10]。以植物转基因中常用的CaMV(花椰菜花叶病毒)35S启动子为例,该启动子可以在植物体内保持较高水平的表达,但该启动子内含有一个重组热点(TATA box),会使转基因不稳定,发生遗传重组[11]。此外,使用多个启动子和终止子会占用多个内切酶位点,使得构建转基因载体时策略设计更为复杂。在本研究中,我们尝试使用HPT-mCherry融合蛋白标签,利用HPT和mCherry筛选转基因材料。mCherry是由DsRed突变得到的单体红色荧光蛋白,在587 nm激发光下发出红色荧光,可以排除叶绿素的干扰[12-14]。利用潮霉素筛选阳性愈伤,并使用荧光显微镜观察愈伤组织和植株,操作简单,准确率较高,可以提高实验结果的可靠性。
我们构建了HPT-mCherry融合标签,用单一的融合蛋白同时实现筛选基因和报告基因的功能。实验结果显示,HPT-mCherry融合标签能有效地筛选阳性愈伤,鉴定得到阳性植株。
1 材料及方法
1.1 材料
日本晴水稻(Oryza sativa L.spp.japonica cv.Nipponbare)、根 瘤 农 杆 菌(Agrobacterium tumefa⁃ciens)EHA105均为本实验室保存;TRIzol试剂(Invit⁃rogen公司);iScriptcDNA合成试剂盒、SsoFast EvaGreen Supermix试剂盒、(Bio-Rad公司);质粒提取试剂盒(Promega公司);LightCycler 480定量PCR仪(Roche公司);NanoVue紫外分光光度计(GE公司);荧光体式显微镜(Leica公司);灯式荧光检测器(BLS公司,匈牙利)。
1.2 质粒的构建
本研究中的相关载体骨架来自pCAMBIA1300(Canberra公司,澳大利亚)。p1300URF载体(图1B)携带CaMV35S启动子驱动的HPT-mCherry融合基因。实验中以p1300UR-mC载体(图1C)作为阳性对照,携带Ubiqitin启动子(玉米泛素基因启动子)[15]驱动的mCherry基因和CaMV35S启动子驱动的HPT基因。
为了构建包含HPT-mCherry融合标签的p1300URF载体,通过序列分析,使得标记基因HPT和mCherry通过一个短的多聚甘氨酸链相连,得到融合片段。该片段包括3个小片段,片段1包含部分HPT基因和Link序列,片段2包含mCherry基因和Link序列,片段3包含载体的部分序列。首先用引物P1和P2扩增片段1、P3和P4扩增片段2、P5和P6扩增片段3,然后将上述PCR产物稀释至1/100后作为模板,用引物P1和P6通过融合PCR技术[16]合成完整的改造片段。采用凝胶回收试剂盒回收PCR产物,用RsrⅡ和SacⅡ酶切,将酶切片段连入经相同的内切酶酶切的目标载体pCAMBIA1300UR,热击转化大肠杆菌,筛选正确克隆,保菌备用。引物及序列见表1。
1.3 农杆菌介导的水稻转化
挑选饱满的种子,去壳后置于50 mL无菌离心管中,用无菌水清洗1次;加入30 mL 75%乙醇消毒1 min;弃乙醇,用无菌水清洗后,加入30 mL次氯酸钠(活性氯浓度为1.25%),搅拌后静置30 min;弃次氯酸钠,用无菌水清洗3次,转入诱导培养基[17]中,28℃培养至得到愈伤颗粒。
将含有目标载体的农杆菌悬浮于AAM培养液(D600nm=0.08~0.1)中,挑取诱导得到的水稻愈伤组织,置菌液中侵染15 min;弃上清,超净台中晾30~40 min;弃菌悬液,将愈伤组织置于共培养基[18]上,20℃暗培养3 d;用无菌水清洗愈伤组织5~6次,转移至选择培养基[18]上光照培养20 d,用Leica荧光显微镜检测带有红色荧光的愈伤组织,并将其转移至新鲜的选择培养基上进行第二轮选择,直到颗粒性的抗性愈伤组织长出;挑取抗性愈伤移入分化培养基[18]中,28℃培养至分化成苗。
1.4 转基因材料的鉴定
水稻转基因材料DNA的提取参考CTAB小量提取法[19]。用引物HPT-FL-F/HPT-FL-R进行PCR检测。PCR条件:95℃ 10 min;95℃ 30 s、58℃ 30 s、72℃ 60 s,35个循环。
1.5 转基因水稻荧光检测
用Leica荧光体式显微镜和BLS灯式荧光检测器观察转化愈伤组织及转化植株的红色荧光。

表1 实验中使用的引物序列
1.6 RNA的提取及逆转录PCR
取100 mg水稻样品置于预冷的研钵中,用液氮充分研磨,加入1 mL TRIzol试剂继续研磨至粉末,按照TRIzol试剂说明书提取水稻总RNA,最后加入20~40 μL无RNase的水溶解RNA沉淀。用iScript cDNA合成试剂盒进行逆转录,得到cDNA。
1.7 荧光定量PCR检测
用质粒提取试剂盒提取p1300URF质粒,用紫外分光光度计进行质粒浓度测量,并按下式计算质粒摩尔浓度[20]:
将质粒p1300URF进行1/10梯度稀释,分别稀释为 105、106、107、108、109、1010和 1011copies/μL 共 7个梯度的标准样品,作为模板;以mCherry-RT-F/mCherry-RT-R和HPT-RT-F/HPT-RT-R为引物,采用SsoFast EvaGreen Supermix试剂盒进行qRT-PCR(PCR 条 件 :94℃ 3 min;94℃ 20 s、58℃ 20 s、72℃ 20 s,40个循环),然后根据7个标准样品的Ct值计算模板起始浓度,并以模板起始浓度为横坐标、Ct值为纵坐标,制作标准曲线[21]。
2 结果
2.1 利用融合标签有效筛选阳性愈伤
将包含p1300URF质粒的农杆菌与粳稻品种日本晴的愈伤组织进行共培养,3 d后选择培养。实验中使用包含p1300UR-mC质粒的农杆菌进行转基因操作作为阳性对照。对选择20 d的愈伤组织进行观察,结果显示,成功转化的愈伤组织在515~588 nm激发光照射下发出红色荧光,转p1300URF愈伤组织的荧光强度较弱,但不影响mCherry作为报告基因筛选阳性愈伤组织的功能(图2A~D)。经过统计,p1300UR-mC和p1300URF分别转化了306块和301块胚性愈伤组织,通过两轮潮霉素筛选,分别获得259块和204块独立转化的表达红色荧光的抗性愈伤组织,转化效率分别为84.6%和67.8%。上述结果表明,HPT与mCherry融合后,转化效率虽然有所降低,HPT-mCherry融合标签中的HPT基因仍然可以有效筛选阳性愈伤。结合荧光观察,对照组和融合标签组都能进一步快速筛选阳性愈伤。

图1 p1300UR(A)、p1300URF(B)和p1300UR-mC(C)载体图
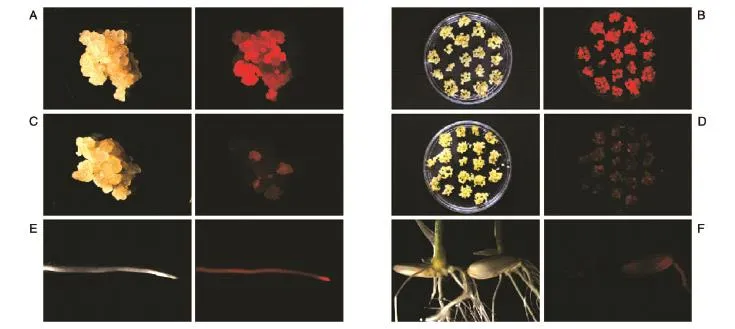
图2 转基因水稻荧光检测
2.2 利用融合标签鉴定转基因植株
对7 d苗龄的转基因水稻进行荧光检测(图2E、F),阳性对照的根、芽和茎均有较强的红色荧光,虽然受融合的HPT基因影响,转p1300URF的植株荧光强度较弱,但红色荧光仍然可以清晰地区分转化与未转化植株。为进一步验证融合标签荧光辅助筛选的有效性,我们随机选取转了14株转化植株进行PCR检测,各阳性转基因植株均扩增得到1023 bp的片段(图3),与通过荧光显微镜观察红色荧光的检测结果完全一致,说明HPT-mCherry融合标签在转基因鉴定中能够替代常用PCR检测方法,从而大量节省实验时间和相关耗材。上述结果表明,在转基因植株中,HPT与mCherry融合后相互间影响较小,可以正常用于转基因植株鉴定。
2.3 HPT和mCherry基因在mRNA水平上的表达分析
将浓度为105ng/μL(即 1.58×1013copies/μL)的质粒 p1300URF分别稀释至 105、106、107、108、109、1010和1011copies/μL作为模板,制作HPT和mCherry基因扩增效率的标准曲线,得到HPT和mCherry标准曲线的相关系数分别为0.998和0.996。提取转基因阳性愈伤和植株的总RNA进行qRT-PCR,通过绝对定量PCR分析,得到各株系中HPT和mCherry的表达量分别为107.06和107.81copies/μL。从mRNA水平证明采用融合标签后,mCherry和HPT能在转基因愈伤和植株中高效表达。参见图4。
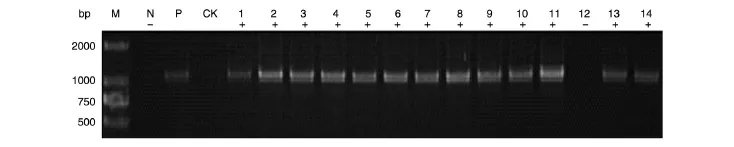
图3 转p1300URF的水稻植株HPT基因的PCR检测

图4 转基因材料的绝对定量PCR分析
3 讨论
随着水稻转基因技术的成熟,转化频率不再是T-DNA转化的主要限制因素。在水稻转基因研究中,转基因后代的鉴定和遗传分析主要利用潮霉素抗性基因的PCR或GUS酶活性进行检测,但这些方法工作量大,比较费时。mCherry是一个单体荧光蛋白,其波长较长(587/610 nm),成熟时间较短(t1/2≈0.25 h),在融合外源蛋白时受影响较少[22]。我们构建了HPT-mCherry融合标签载体,并确认了融合蛋白能有效发挥抗性基因功能,行使抗潮霉素的功能,也能有效地产生红色荧光,从而实现可视化筛选。
用HPT-mCherry融合标签进行鉴定,在避免了繁杂工作的同时保证了实验结果的可靠性。而且,该方法还避免了使用2套启动子和终止子分别控制HPT和mCherry的表达,有效减少了遗传重组的风险,同时避免了占用大量限制性内切酶位点,方便了载体的进一步使用。
[1]Nishizawa Y,Nishio Z,Nakazono K,et al.Enhanced resis⁃tance to blast(Magnaporthe grisea)in transgenic Japonica rice by constitutive expression ofrice chitinase[J].TheorAppl Genet,1999,99(3-4):383-390.
[2]唐甜甜,李浩,段永波,等.利用sgfp报告基因优化农杆菌介导的水稻遗传转化[J].生物学杂志,2012,29(1):88-91.
[3]Hiei Y,Ohta S,Komari T,et al.Efficient transformation of rice(Oryza sativa L.)mediated by Agrobacterium and se⁃quence analysis of the boundaries of the T-DNA[J].Plant J,1994,6(2):271-282.
[4]董志峰,马荣才,彭于发,等.转基因植物中外源非目的基因片段的生物安全研究进展[J].植物学报,2001,43(7):661-672.
[5]Ortiz J P A,Reggiardo M I,Ravizzini R A,et al.Hygromy⁃cin resistance as an efficient selectable marker for wheat sta⁃ble transformation[J].Plant Cell Rep,1996,15(12):877-881.
[6]Nakamura S,Mano S,Tanaka Y,et al.Gateway binary vec⁃tors with the bialaphos resistance gene,bar,as a selection marker for plant transformation[J].Biosci Biotechnol Biochem,2010,74(6):1315-1319.
[7]Vain P,Worland B,Kohli A,et al.The green fluorescent pro⁃tein(GFP)as a vital screenable marker in rice transformation[J].Theor Appl Genet,1998,96:164-169.
[8]Joersbo M,Okkels F T.A novel principle for selection of transgenic plant cells:positive selection Plant Cell Rep,1996,16(3):219-221.
[9]Miki B,McHugh S.Selectable marker genes in transgenic plants:applications,alternatives and biosafety[J].J Biotech⁃nol,2004,107(3):193-232.
[10]Cummins J,Ho M W,Ryan A.Hazardous CaMV promoter[J]?Nat Biotechnol,2000,18(4):363.
[11]Kohli A,Griffiths S,Palacios N,et al.Molecular characteriza⁃tion of transforming plasmid rearrangements in transgenic rice reveals a recombination hotspot in the CaMV 35S promoter and confirms the predominance of microhomology mediated re⁃combination[J].Plant J,1999,17(6):591-601.
[12]Baird G S,Zacharias D A,Tsien R Y.Biochemistry,mutagen⁃esis,and oligomerization of DsRed,a red fluorescent protein from coral[J].Proc Natl Acad Sci USA,2000,97(22):11984-11989.
[13]Matz M V,Fradkov A F,Labas Y A,et al.Fluorescent pro⁃teins from nonbioluminescent Anthozoa species[J].Nat Biotech⁃nol,1999,17(10):969-973.
[14]Zhou X,Carranco R,Vitha S,et al.The dark side of green fluorescent protein[J].New Phytologist,2005,168(2):313-322.
[15]Christensen A H,Quail P H.Ubiquitin promoter-based vec⁃tors for high-level expression of selectable and/or screenable marker genes in monocotyledonous plants[J].Transgenic Res,1996,5(3):213-218.
[16]Kim D,Seo H Y,Hayashi K.Construction and characteriza⁃tion of novel chimeric β-glucosidases with Cellvibrio gilvus(CG)and Thermotoga maritima(TM)byoverlappingPCR[J].Biotechnol Bioprocess Eng,2009,14:266-273.
[17]阎丽娜,李霞,吴丹.不同类型水稻材料成熟胚组织培养力的比较[J].中国农业科学,2010,43(6):1127-1135.
[18]张春雨,李宏宇,刘斌.hpt与bar基因作为水稻转基因筛选标记的比较研究[J].遗传,2012,34(12):1599-1606.
[19]Doyle J A,Doyle J L.A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J].Phytochem Bull,1987,19(1):11-15.
[20]李慧锋,李丽,张丽娟,等.PepT1基因绝对荧光定量PCR标准曲线的建立[J].山西农业大学学报,2010,30(4):332-335.
[21]李丽,赵成萍,李宏,等.质粒制备绝对定量PCR标准曲线方法的建立[J].农业生物技术学报,2011,19(6):1157-1162.
[22]Shaner N C,Campbell R E,Steinbach P A,et al.Improved monomeric red,orange and yellow fluorescent proteins derived from Discosoma sp.red fluorescent protein[J].Nat Biotechnol,2004,22(12):1567-1572.

