北部湾近海珊瑚礁区系沉积物抗菌活性放线菌类群
龚 斌,黄 蕾,张艳秋,彭春艳,熊 拯,方怀义,庞庭才,黄 鹄
(钦州学院海洋学院,广西 钦州 535000)
放线菌是最具潜在开发价值的原核生物。目前已经发现的活性代谢产物中,放线菌占一半以上[1],包括各种抗生素[2]、免疫抑制剂[3]、抗肿瘤药物[4]和多种酶类[5]等。然而经过50多年的研究,人类对陆地放线菌资源的开发已经到了极限,很难再发现新的放线菌种类和活性代谢产物[6],而海洋由于其特殊的生态环境赋予了海洋微生物独特的物种和基因特性,海洋放线菌已成为新的研究热点[7]。海洋放线菌广泛分布在各种海洋环境中,如近岸、浅滩、海洋动植物体内、海水、深海沉积物等[8]。珊瑚礁是四大海洋生态系统之一,蕴藏着丰富多样的海洋生物资源[9]。本研究的目的是从广西北部湾近海珊瑚礁区系沉积物中分离具有抗菌活性的放线菌,通过菌落形态学和孢子的形态特征,结合放线菌全基因组RAPD-PCR指纹图谱分析对放线菌进行类群划分,并通过16S rRNA基因序列Blast和进化树分析并结合生理生化特征确定放线菌的种属类群,对揭示广西北部湾海洋放线菌多样性具有较重要意义。
1 材料与方法
1.1 材料
1.1.1 试剂 PCR引物、PCR常规试剂和酶均购自TaKaRa公司;细菌基因组DNA提取试剂盒购自天根生化科技(北京)有限公司;其余试剂均购于国药集团。
1.1.2 培养基 用于放线菌分离的培养基参考了常显波等[10]的研究报道,采用培养基M1-M9对珊瑚礁区沉积物放线菌进行分离。
1.1.3 指示菌菌株 指示菌 Exiguobacterium sp.、Arthrobacter nicotianae、Vibrio parahaemolyticus、Bacillus cereus(ATCC10876)、Bacillus baekyagesm、Bacillus subtilis(ATCC6633)、E. coli(ATCC25947)均为本实验室保藏。
1.2 方法
1.2.1 放线菌的分离 5份沉积物样品来自北部湾近海珊瑚礁区,采到的样品立即放入装有海水的灭菌三角瓶中,保存于带冰袋的泡沫盒运回实验室备用。称取5 g样品,在无菌操作下加入盛有50 mL 50%无菌陈海水的三角瓶中,用涡漩振荡器充分振荡均匀,于28℃,200 r/min摇床振荡0.5 h后放置澄清,取上清采用传统的稀释平板涂布法对土壤样品进行分离,28℃培养7~30 d。
1.2.2 形态观察 根据菌落大小、形态、颜色进行初步分离筛选并纯化,所得纯培养物制成冻干牛奶管和斜面保藏于4℃备用。用淀粉琼脂培养基(5%NaCl)于28℃下进行埋片培养,分别培养7、14、21 d取出埋片,用光学显微镜 Olympus BH-2观察形态。
1.2.3 抑菌活性实验 用接种环取1环菌株孢子接种于装有5 mL发酵培养基的试管中,28℃、180 r/min培养7 d。发酵液在11000 r/min下离心15 min,上清液采用滤纸片法测定其对指示菌株的抗菌活性。细菌于适宜温度培养24 h,记录抑菌结果并测量抑菌圈直径。所有试验设3个重复。
1.2.4 放线菌的RAPD分析 采用细菌基因组DNA提取试剂盒提取放线菌总DNA,采用上海生工公司S系列随机引物,经预备实验选择扩增产物稳定、重复性好的引物,用于所有供试菌株的RAPD-PCR随机扩增多态性分析。反应体系总体积为25 μL,扩增程序:94℃变性2 min,94℃变性40 s,37℃退火1 min,72℃延伸1 min,共40个循环,最后72℃延伸6 min。扩增产物在1.4%的琼脂糖凝胶中电泳分离,用0.5%TBE电泳缓冲液,上样10 μL,3 ~4 V/cm 电泳90 min,溴化乙锭染色,用Tocan360型凝胶成像系统254 nm紫外光下检测、拍照。将有带记为“1”,无带记为“0”,然后通过MVSP软件,采用UPGMA法进行聚类分析。
1.2.5 16S rRNA基因PCR扩增和系统进化分析用引物27f(5'-AGAGTTGATCCPATGGCTCAG-3')和1541R(5'-AAGGAGGTGATCCAGCC-3')扩增放线菌16S rRNA基因序列。PCR反应条件:94℃ 变性5 min,然后94℃,30 s→50℃,40 s→72℃,1.5 min进行35个循环扩增,最后72℃延伸10 min。将PCR产物纯化后直接送上海生工生物工程技术服务有限公司,用引物27f测序。所得序列在GenBank中用Blast进行相似性分析,用ClustalX进行排列比较,用Mega 5.0软件进行系统树的构建。
2 结果与分析
2.1 放线菌的分离
从北部湾近海珊瑚礁区5个沉积物样品中成功分离到142株海洋放线菌,通过指示菌株(大肠埃希菌和枯草芽胞杆菌)筛选得到51株显示不同程度抗菌活性的菌株,这表明北部湾近海珊瑚礁沉积物是具有生物活性海洋放线菌的良好来源。51株菌经过反复筛选,得到9株具有较强抗菌活性的菌株。根据这9株菌的培养和个体形态特征,参照《伯杰氏细菌鉴定手册》初步确定其均属于链霉菌属(图1),关于菌种的详细鉴定正在进行中。

图1 部分珊瑚礁沉积物放线菌的显微镜镜检特征(400×)Fig.1 Characteristic of some Streptomyces strains under microscopic detection(400 × )

图2 9个链霉菌样品PCR-RAPD指纹多态性及UPGMA聚类分析Fig.2 RAPD-PCR and cluster analysis by UPGMA method of 9 isolated Streptomyces strains
由于链霉菌种类很多,按照菌落培养和孢子形态特征还不能区分不同的种类,本研究采用随机引物对9个链霉菌样品进行RAPD-PCR指纹多态性分析,目的是考察RAPD-PCR能否用于链霉菌的分型研究。扩增图谱中有带记为“1”,无带记为“0”,转换为数值后,进行聚类分析(图2),从聚类分析结果看,这9株链霉菌在79.8%的相似性水平上聚为2个类群:菌株5、23、44等8株菌聚在类群I,其中菌株5、23、44聚在一个亚类,菌株6、25、40、46聚在另外一个亚类;菌株42聚在类群II。
2.2 放线菌的16S rDNA序列测定和系统发育分析
对9株放线菌的16S rDNA序列分别进行了测定,结果与GenBank数据库序列进行比较。结果表明:菌株23与链霉菌 Streptomyces gardneri、Streptomyces omiyaensis、Streptomyces bungoensis 的相似性最高,达98%;菌株42与Streptomyces microflavus、Streptomyces califomicus 的相似性高达98%;菌株5和44与Streptomyces nitrosporeus的相似性达 99%。菌株 6、26、46、25、40的遗传距离较近,在系统发育树中处于同一个分枝,但16S rDNA序列上存在着一些位点的差异,菌株25、40、46聚为一类,菌株6和46与Streptomyces lateritius、Streptomyces labedae、Streptomyces chartreusis等聚为一类,序列相似性达98%。比较16S rDNA序列系统发育树和RAPD-PCR聚类分析结果可以发现,两者的聚类分析结果具有较大的相似性,说明RAPD-PCR用于链霉菌的分型研究能够较为真实地反映链霉菌不同种类的亲缘关系。

图3 链霉菌样品16S rDNA序列进化树分析Fig.3 Phylogenetic tree of 16S rDNA sequences of the isolated Streptomyces strains
2.3 放线菌的生理生化鉴定特征
部分生理生化特征鉴定结果见表1,参照《伯杰氏细菌鉴定手册》和《放线菌的分类和鉴定》,将实验结果同手册上不同种菌株进行比对,16S rRNA基因序列亲缘关系最近的放线菌种群与分离株的生理生化鉴定特征均存在差异,说明分离株为放线菌新种的可能性比较大。
2.4 放线菌的抗菌活性
采用 7种指示菌(Exiguobacterium sp.、Arthrobacter nicotianae、Vibrio parahaemolyticus、Bacillus cereus、Bacillus baekyagesm、Bacillus subtilis、E.coli)对6种放线菌的抗菌活性进行检测,结果见表2。6种放线菌均表现对2种以上的指示菌有抑菌活性,其中菌株23只能抑制革兰阴性细菌生长,菌株6、5、25、26只能抑制革兰阳性细菌生长,5和25的抑菌活性表现更强的选择性,42对革兰阴性和阳性细菌均能明显抑制,表现较为广谱的抑菌活性。这6株放线菌在抑菌活性上的差异,说明其可能分泌出多种结构功能不同的活性次生代谢产物,具有较 大的深入研究与开发的价值。

表1 链霉菌的生理生化特征鉴定Table 1 Physiological and biochemical properties of Streptomyces strains
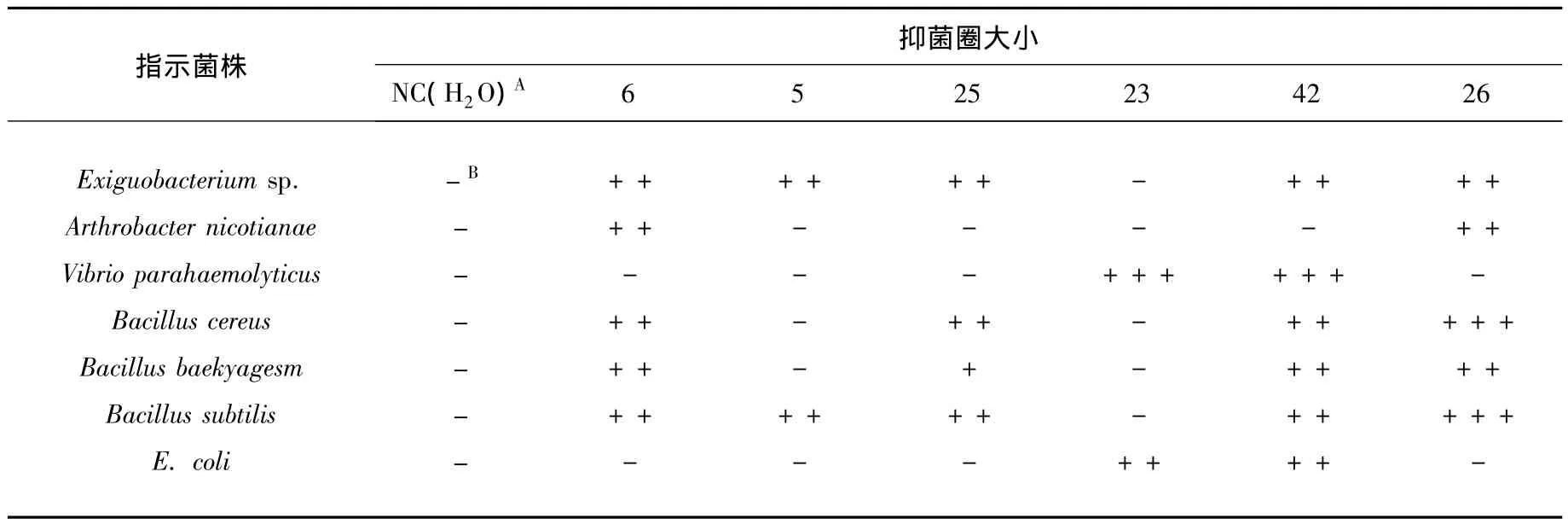
表2 放线菌次生代谢产物的抗菌活性Table 2 antibacterial activity of metabolites from Streptomyces strains
3 讨论
对来自广西北部湾近海珊瑚礁区系沉积物进行抗菌活性放线菌分离,得到9株具有较强抗菌活性的菌株。为了快速将这些放线菌进行类群划分,对9个链霉菌样品进行RAPD-PCR指纹多态性分析,通过对凝胶电泳的结果进行聚类分析,可以发现其聚类分析结果与16S rDNA序列聚类分析的结果非常接近,其中6、25、40、46在2种不同的聚类分析中均聚为一类,5、23、44聚为另一类,这说明PCR-RAPD指纹多态性分析用于链霉菌种水平的分型鉴定可以较好区分不同类群。而2种不同分析方法中菌株26表现出较大的差异,16S rDNA 序列聚类分析中26与6、25、40、46的亲缘关系较近,但在PCR-RAPD分析中亲缘关系上却表现较远,其原因可能是少数链霉菌16S rDNA序列之间的相似性很高,达98%以上,但在分类学上的地位却比较远,例如有报道表明在16S rDNA序列相似度大的链霉菌中,DNA-DNA杂交率还不到50%[11]。因此在链霉菌的分类上还需要引入一些更灵敏的方法来进行种群划分,本实验也说明在链霉菌种水平的分型研究中,PCRRAPD指纹多态性分析比16S rDNA序列聚类分析能更好区分不同种类。
由于链霉菌属内各种之间的16S rRNA基因序列相似性高,均在 98.0% ~99.9% 之间[12],所以根据16S rRNA基因序列的分析结果还不足以确定链霉菌的种类,只能判断筛选到的链霉菌菌株很可能是与其亲缘关系最近种类的一种菌株[13],为此进一步采用细菌生理生化鉴定实验来验证,结果表明得到的分离株与16S rRNA基因序列相似性最高的链霉菌在生理生化特征上均有差异,因此这些分离株可能为链霉菌新种,其分类学地位还有待进一步证明,如将该菌株与其进化上亲缘关系最近的菌株做平行生理生化鉴定实验和DNA-DNA同源杂交,才能给予其准确的分类学地位[14]。
本研究采用7种指示菌对链霉菌的抑菌活性进行测试,可以发现分离的6株菌在抑菌活性上均存在一定差异,这说明即使是亲源关系很近的分离株,由于其存在环境的区别,在活性代谢产物的合成基因上可能也存在区别,也说明近海珊瑚礁区系放线菌在活性代谢产物上存在较好的多样性,具有进一步深入研究与开发的价值。
[1]Berdy J.Bioactive microbial metabolites[J].J Antibiot(Tokyo),2005,58(1):1-26.
[2]Yu Z,Zhu H,Dang F,et al.Differential regulation of antibiotic biosynthesis by DraR-K,a novel two-component system in Streptomyces coelicolor[J].Mol Microbiol,2012,85(3):535-556.
[3]Kino T,Hatanaka H,Hashimoto M,et al.FK-506,a novel immunosuppressant isolated from a Streptomyces.I.Fermentation,isolation,and physico-chemical and biological characteristics[J].J Antibiot,1987,40(9):1249-1255.
[4]Simmons TL,Rianasolo E,Mcphail K,et al.Marine natural products as anticancer drugs[J].Mol Cancer Ther,2005,4(2):333-342.
[5]Singh R,Mo S,Florova G,et al.Streptomyces coelicolor RedP and FabH enzymes,initiating undecylprodiginine and fatty acid biosynthesis,exhibit distinct acyl-CoA and malonyl-acyl carrier protein substrate specificities[J].FEMS Microbiol Lett,2012,328(1):32-38.
[6]Lamk S.Discovery of novel metabolites from marine actinomycetes[J].Curr Opin Microbiol,2006,9(3):245-251.
[7]Chen C,Song F,Wang Q,et al.A marine-derived Streptomyces sp.MS449 produces high yield of actinomycin X2 and actinomycin D with potent anti-tuberculosis activity[J].Appl Microbiol Biotechnol,2012,95(4):919-927.
[8]田新朋,张偲,李文均.海洋放线菌研究进展[J].微生物学报,2011,51(2):161-169.
[9]赵美霞,余克服,张乔民.珊瑚礁区的生物多样性及其生态功能[J].生态学报,2006,26(1):186-193.
[10]常显波,刘文正,尹琦,等.海洋放线菌不同分离方法的比较研究[J].海洋科学,2012,36(8):35-39.
[11]李德舜,苏忠锐,孙琦,等.高温链霉菌24#的初步研究[J].微生物学报,2004,44(5):580-583.
[12]Zhu HH,Guo J,Yao Q,et al.Streptomyces caeruleatus sp.nov.,with dark blue diffusible pigment[J].INT J SYST EVOL MICR,2011,61(Pt3):507-511.
[13]Zhu HH,Guo J,Yao Q,et al.Streptomyces vietnamensis sp.nov.,a streptomycete with violet-blue diffusible pigment isolated from soil in Vietnam[J].INT J SYST EVOL MICR,2007,57(Pt8):1770-1774.
[14]Annalieas SA,Wellington MH.The taxonomy of Streptomyces and related genera[J].INT J SYST EVOL MICR,2001,51(Pt3):797-814.

