水稻耐盐QTL图谱整合
冯世超,赵宏伟,王敬国,刘化龙,赵 雪,邹德堂*
(1.东北农业大学水稻研究所,哈尔滨 150030;2.黑龙江省农垦科学院植物保护研究所,哈尔滨 150038;3.东北农业大学大豆研究所,哈尔滨 150030)
在我国现有耕地中,至少有800万hm2土地由于不当灌溉和施肥,导致土壤盐分累积而形成盐渍化[1],成为限制我国农业发展重要因素。水稻是我国主要的粮食作物之一,作为粳稻主产区的黑龙江省,盐碱地面积达40万hm2。其中,黑龙江省西部地区有盐碱土近66.7万hm2,主要分布在大庆及齐齐哈尔南部地区,其中含盐量超过1%的盐土和碱土超过10万hm2[2]。选育耐盐水稻品种以适应土地盐碱化尤为重要。
与传统育种相比,利用DNA分子标记技术结合QTL分析方法寻找与水稻耐盐性紧密连锁的分子标记,并用于分子标记辅助选育耐盐品种,是加快育种进程的有效途径,同时为抗性基因的挖掘和图位克隆奠定基础。水稻耐盐性是多基因控制的数量性状,国内外学者利用不同群体定位了许多与耐盐性相关的QTLs。龚继明等用籼稻窄叶青8号(ZYQ8)和粳稻京系17(JX17)为亲本的DH群体对水稻耐盐性进行QTL定位,以植株存活时间为指标,在1号染色体RG612和C131标记之间定位一个主效QTL-Std,以及其他7个微效QTL,贡献率10.2%~38.4%[3]。Lin等利用遗传差异较大的耐盐品种Nona Bokra和感盐品种Koshihikari所构建的F2∶3群体,定位到3个影响盐胁迫幼苗存活天数的QTL,贡献率为13.9%~18.0%,并在第7和第1染色体上检测到影响茎Na+和K+浓度的主效QTL,表型贡献率分别达48.5%、40.1%、40.6%和41.7%[4]。在以往研究中,QTL定位结果受群体数量及图谱标记密度限制,得到的QTL精度差、置信区间大,很难估计QTL的准确位置及其贡献率大小,制约QTL在育种工作中的进一步应用。Goffinet和Gerber提出的Bioercator应用程序可将前人研究获得的QTL映射到高密度参考图谱上,并通过元分析方法确定真实性QTL和缩小置信区间[5]。李雪华等对不同研究者在干旱条件下定位到的与玉米抗旱性相关的181个QTL进行整合分析,从中发掘出15个耐旱“通用QTL”[6]。吴琼等整合控制大豆生育期的98个QTLs,发现9个真实QTLs和相应的连锁标记[7]。
本研究对已经定位的水稻耐盐性QTLs进行整合,以期发现真实QTLs,为水稻耐盐性遗传机制研究、耐盐分子辅助育种及耐盐基因挖掘提供理论基础。
1 材料与方法
1.1 水稻耐盐性QTLs数据的选择、收集和整理
从www.gramene.org网站和已发表的文献收集水稻耐盐性QTLs的定位信息。收集和整理包括作图群体信息(群体大小、群体类型、群体名称和作图函数),性状信息,QTL信息(QTL名称、染色体位置、LOD值、贡献率、置信区间和加性效应)以及遗传图谱信息(标记名称及遗传距离)。如果某些QTL信息没有给出置信区间,可以根据文献[8]公式推断其95%的置信区间(见表1)。
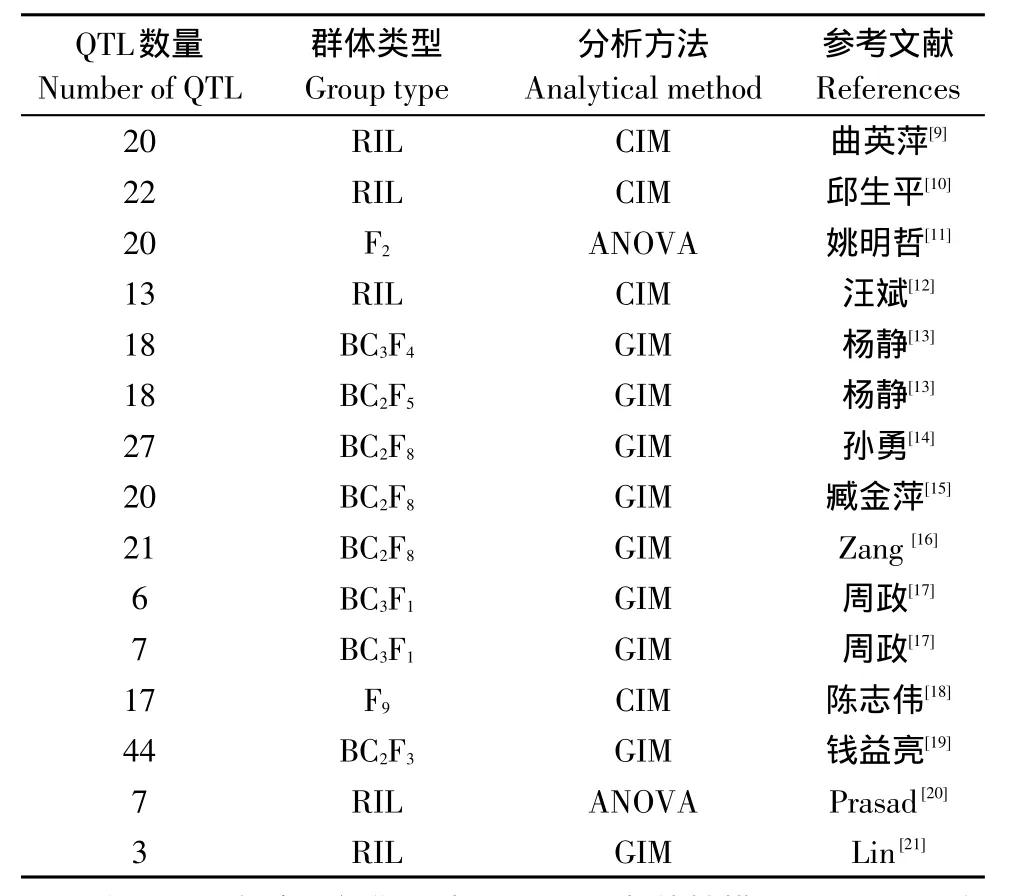
表1 已报道的水稻耐盐性QTLsTable 1 Reported rice QTLs conferring salt tolerance
式中,CI是QTL的置信区间,N是作图群体的大小,R2是QTL贡献率。公式(1)适用回交和F2群体,公式(2)适用RILs群体。

1.2 水稻耐盐性QTLs的映射
计算QTL在参考图谱上的坐标值的数学基础是齐序函数(Homothetic function)[22],计算共有标记间的距离,将原始图谱上的QTL按比例映射到参考图谱上[23],即映射(Projection)。Biomercator 2.1软件通过严格识别QTL两侧的标记名实现映射,如果左右标记其中之一不能映射,那么QTL将被舍弃。
不同QTL定位研究所用的作图群体和分子标记存在差异,构建的遗传图谱之间相同标记较少,在进行QTL映射时需选择一个高密度分子连锁图谱来作参考图谱。本研究所用参考图谱是高密度遗传连锁图谱Cornell 2001,具有较高的可信度,而且与原始图谱之间有大量共同标记,便于图谱整合及QTL映射的实现。
1.3 水稻耐盐QTLs的元分析
元分析(又称作整合分析、统合分析、荟萃分析)是将不同研究结果汇总成原始数据进行综合分析的统计方法,它可以将不同实验相近基因组区域检测到的QTL进行聚类。2000年,Goffinet和Gerber开发了元分析的算法[24]。具体过程:根据n个独立实验中QTLs,同一区域一致性QTLs由1-、2-、3-、4-和N-QTLs 5种模型模拟决定,即以最小的AIC值来确定真实性QTL。每种模型都是根据极大似然方法并通过高斯定理给出QTLs在染色体上的最大可能排列。模型中,一致性QTL位点取决于QTL最大可能分布的平均值,QTL一致性位点的方差由下面公式估计:

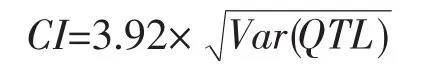
AIC值取决于模拟过程,所以它的优化使用需满足以下条件:QTL严格来源于独立实验;QTL数量10~40个;QTL在染色体上的覆盖区域不超过200 cM。若处理较大的染色体或一组较大的QTLs时,应把染色体片段分割,在每个小片段上重复进行元分析[25]。
2 结果与分析
2.1 耐盐性QTLs在水稻染色体上的分布
在BioMercator 2.1软件tools-maps projection选项下,实现水稻耐盐QTL的映射。映射结果中,有7个群体定位的119个QTLs得到映射,构建一张水稻耐盐QTL的一致性图谱。从图谱可以发现,耐盐QTL在染色体上呈不均匀分布,各条染色体上都有和耐盐相关的QTL;第1、2、3、4、5、6、8、9、11、12条染色体上分布的QTLs较多,第7和第10条染色体上分布较少;其中,第5、7、8、9、10、12条染色体上QTLs成簇分布,其他染色体上则散在分布,整合后的部分QTLs分布见图1。
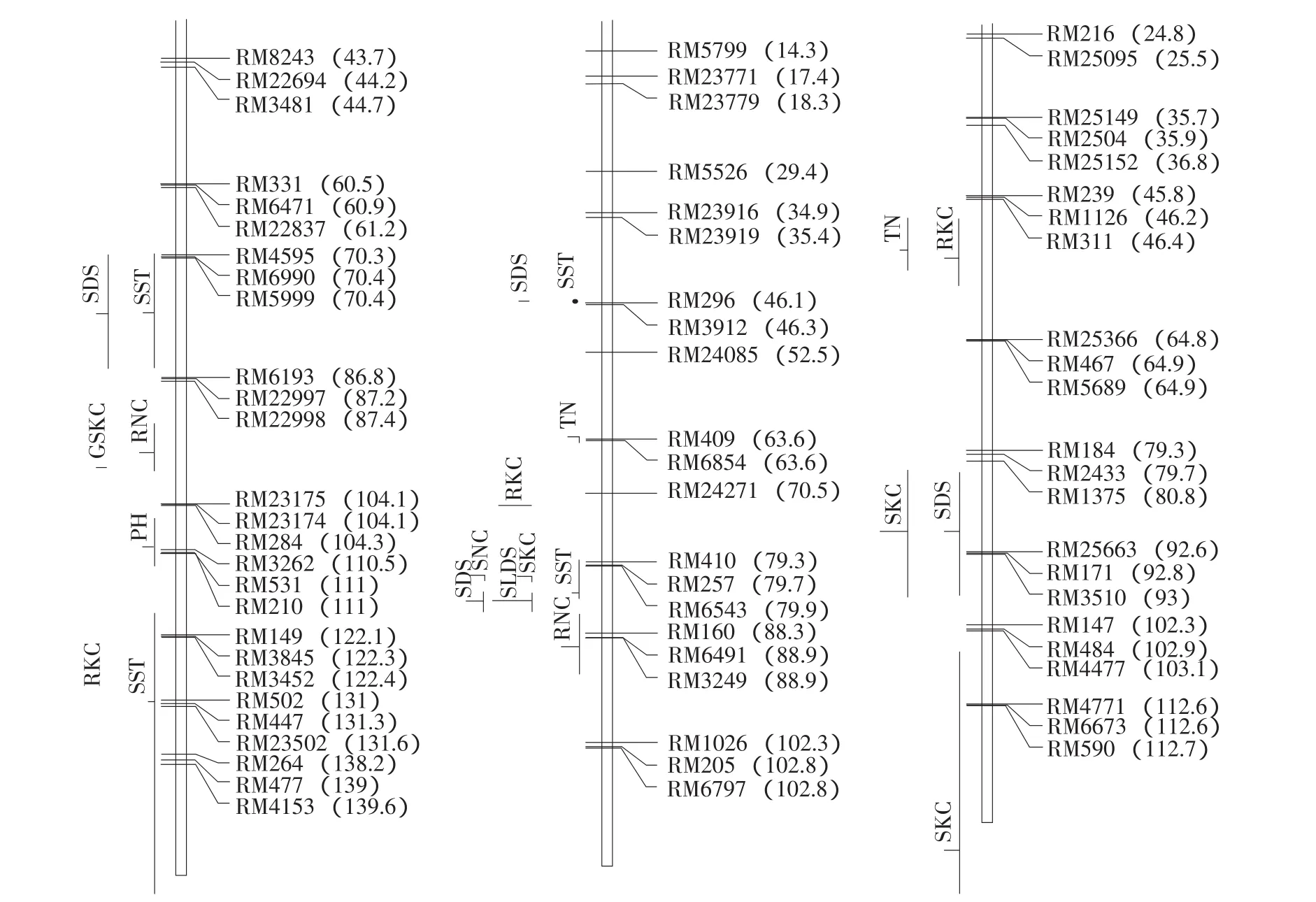
图1 部分耐盐QTLs在8、9、10号染色体上的分布Fig.1 Distribution of salt tolerance QTLs in 8,9 and 10 chromosome
2.2 耐盐性QTLs的元分析
在BioMercator 2.1软件tools-Meta-analysis选项下,进行耐盐QTL的元分析。结果以每次元分析中AIC值最小的模型为准来确定真实QTL。因为元分析只有在成簇区中产生才是有意义,所以在实际操作中忽略散在分布QTL位点产生的结果。
由表2可知,共有14个真实QTL被发现,第8和第9两条染色体上真实QTL较多。其中,在第8条染色体上,位于99.32 cM处,图距3.46 cM的QTL被发现,其标记区间为RM22998-RM23175,置信区间97.58~101.06。在第9条染色体上,位于46.1 cM处,图距2.78 cM的QTL被发现,其标记区间为RM296-RM3912,置信区间44.71~47.49。除这两个QTLs外,在第9条染色体上还有3个图距小于5 cM的真实QTL,分别位于63.93、81.89、85.58 cM处,标记区间和置信区间分别是RM409-RM 24271、RM410-RM160、RM6543-RM6491 和 63.65~64.21、81.43~82.35、84.32~86.64,第 7 条染色体有一个图距小于5 cM的真实QTL,位于144.2 cM处,标记区间和置信区间分别为RM248-RM420和144.1~144.5。
将元分析得到的真实QTL与原始QTL信息相比较表明,元分析可以明显缩小QTL的置信区间,如图2所示。

表2 水稻耐盐QTL的元分析Table 2 Meta-analysis of salt tolerance QTLs in rice
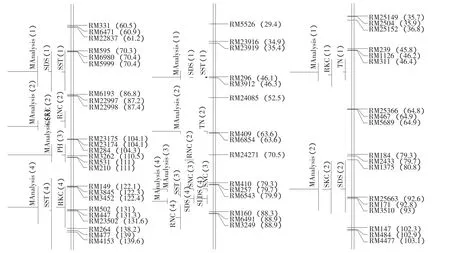
图2 8、9、10号染色体上耐盐QTL的元分析Fig.2 Meta-analysis of salt tolerance QTLs on 8,9 and 10 chromosome
位于第8染色体上110.09 cM处的真实QTL与原始QTL位置十分接近,但其图距从原来的24.5 cM缩小到8.0 cM;位于第9染色体上46.1 cM处得真实QTL图距为2.78 cM,比原始QTL图距13.60 cM缩小10.82 cM;位于第10染色体上53.72 cM处真实QTL与原始QTL位置相同,但其图距从原来的12.2 cM降到5.63 cM,其他染色体上的QTLs置信区间有不同程度的缩小。
3 讨论
3.1 图谱整合和元分析方法的应用
图谱整合把分子连锁图与经典图谱和基因组图谱进一步结合。在高密度图谱构建的基础上,通过元分析可以进一步找到真实QTL及其标记区间,因此图谱整合是发掘候选基因的新途径。尤莉等将ESTs电子定位结果与玉米耐盐性QTLs的图谱整合,经验证后发现3个重要耐盐性候选基因[26]。元分析方法在其他研究中已早有应用,Guo等对抗大豆胞囊线虫QTL进行元分析,并得到“真实QTL”及其连锁标记[27]。大多数QTL定位重要性状基因多呈数量性状遗传,受遗传背景、群体大小、基因与环境互作、统计方法等因素影响很大,单一的一次定位中难以真实评价每个QTL所能解释的遗传变异,标记的有效性较低。采用元分析方法,在整合QTL的基础上,建立数学模型,可以缩小置信区间,具有较高的可信度,提高QTL定位的准确度和有效性,可克服传统QTL分析结果的局限性,为更好诠释QTL奠定基础[7]。
通常高密度遗传图谱是精细定位QTL的前提,由于作图群体的规模过小等原因很难获得较高密度的图谱,造成已有图谱分析率较低。因此,为保证元分析的准确性,必须采用高密度的整合图谱。本研究所用的参考图谱是高密度遗传连锁图谱Cornell 2001,具有较高可信度,与原始图谱之间有大量共同标记保证QTL位点的进一步分析。
3.2 耐盐性QTL图谱整合对MAS实践的意义
随着数量遗传学和分子标记技术应用的发展,复杂数量性状的遗传行为研究成为可能。借助分子标记辅助选择技术和适当的统计方法可确定目标性状在染色体上的位置和效应。国内外研究者通过构建各种类型的群体定位许多QTL的遗传连锁图谱,将控制某一性状的QTL整合到一张相对饱和的遗传图谱上,不仅从整体把握该性状在染色体上的分布特征,而且通过元分析可发现真实QTL及其标记区间[5]。
本研究将已公开发表的水稻苗期耐冷QTL进行收集和整理,通过映射和元分析建立水稻耐盐一致性图谱,所发现的真实QTL标记区间与原区间相比都明显缩小。如第8染色体110.09 cM处发现的真实QTL与原始QTL相比其图距从原来的24.5 cM缩小到8.0 cM;位于第9染色体上46.1 cM处真实QTL比原始QTL图距缩小10.82 cM。这些真实QTL及其标记区间为水稻耐盐分子标记辅助选择奠定基础。
3.3 图谱整合的局限性
图谱整合在快速整合QTL有效缩小置信区间的同时,也有一定的局限。图谱整合需要应用大量图谱,独立实验定位的QTL数量及参考图谱上标记致密程度都影响映射的效果,所以在整合过程中存在真实QTL丢失现象,在本研究中第3、第4、第11条染色体上QTLs散布于染色体上,不能确定有无真实QTL。随着QTL定位数量增加和遗传图谱逐渐饱和,真实QTL丢失现象会得到控制[28]。
不同标记构建的遗传图谱整合,实质上就是不同标记的相互转换与结合。可考虑启用一些能对DNA结构进行分析的标记(如:SCAR、STS、EST等),通过对标记片段进行克隆和杂交实验来实现片段的转换,加强不同图谱之间的联系与整合[29]。
4 结论
本研究利用生物信息学手段,收集整理来自15个作图群体共263个与水稻耐盐性相关的QTLs信息。通过Biomercator2.1和共有标记映射,将QTLs整合到参考图谱Cornell 2001上,并通过元分析方法计算真实QTLs及其临近标记。得到以下结论,构建一张水稻耐盐QTL的一致性图谱。水稻耐盐QTL在第5、7、8、9、10、12条染色体上QTLs成簇分布,其他染色体上则散在或缺失分布。元分析共发现14个真实QTL,其中6个图距小于5 cM的真实QTLs分别位于第7、8、9条染色体上;这些一致性位点可用于耐盐水稻品种的分子辅助选育及耐盐基因的图位克隆。
[1]高继平,林鸿宣.水稻耐盐机理研究的重要进展——耐盐数最性状基因SKC1的研究[J].生命科学,2005,17(6):563-565.
[2]刘功,李锐,王连敏,等.浅谈黑龙江省盐碱地利用[J].黑龙江农业科学,2007(2):108-109.
[3]龚继明,何平,钱前,等.水稻耐盐性QTL的定位[J].科学通报,1998(17):1847-1850.
[4]刘涛,王敬国,刘化龙,等.水稻苗期耐冷QTL的图谱整合[J].作物杂志,2010(3):15-19.
[5]Lin H X,Zhu M Z,Yano M,et al.QTLs for Na+and K+uptake of the shoots and roots controlling rice salt tolerance[J].Theoretical and Applied Genetics,2004,108(2):253-260.
[6]Li X H,Li X H,Hao Z F,et al.Consensus map of the QTL relevant to drought tolerance of maize under drought conditions[J].Sci Agric Sin,2005,38(5):882-890.
[7]吴琼,齐照明,刘春燕,等.基于元分析的大豆生育期QTL的整合[J].作物学报,2009,35(8):1418-1424.
[8]Andaya V C,Mackill D J.Mapping of QTLs associated with cold tolerance during the vegetative stage in rice[J].Journal of Experimental Botany,2003,54(392):2579-2585.
[9]曲英萍.水稻耐盐碱性QTLs分析[D].北京:中国农业科学院,2007.
[10]邱生平.水稻耐盐性的遗传分析及耐盐相关基因的克隆[D].南京:南京农业大学,2005.
[11]姚明哲.太湖流域粳稻地方品种韭菜青耐盐性的遗传分析[D].南京:南京农业大学,2002:69.
[12]汪斌,兰涛,吴为人.盐胁迫下水稻苗期Na+含量的QTL定位[J].中国水稻科学,2007,21(6):585-590.
[13]杨静.利用双向导入系剖析水稻耐盐QTL定位的遗传背景效应[D].哈尔滨:东北农业大学,2009.
[14]孙勇.水稻耐盐QTL表达的遗传背景与环境效应研究[D].北京:中国农业科学院,2007.
[15]藏金萍.水稻抗旱,耐盐QTL表达的遗传背景效应及抗旱,耐盐的遗传重叠研究[D].北京:中国农业科学院,2008.
[16]Zang J,Wang Y,Yang J,et al.Dissection of genetic overlap of salt tolerance QTLs at the seedling and tillering stages using backcross introgression lines in rice[J].Science in China.Series C,Life Sciences/Chinese Academy of Sciences,2008,51(7):583-591.
[17]周政.不同抗逆性选择对水稻产量相关性状的影响以及高产,抗旱和耐盐QTL的定位[M].北京:中国农业科学院,2010.
[18]陈志伟.利用一个籼粳交重组自交系群体定位水稻苗期耐盐性QTLs[D].南京:南京农业大学,2007.
[19]钱益亮,王辉,陈满元,等.利用BC2F3产量选择导入系定位水稻耐盐QTL[J].分子植物育种,2009,7(2):224-232.
[20]Prasad S R,Bagali S P.Molecular mapping of quantitative trait loci associated with seedling tolerance to salt stress in rice(Oryza sativa L.)[J].Curr Sci,2000,78:162-164.
[21]Lin H X,Zhu M Z,Yano M,et al.QTLs for Na+and K+up take of the shoots and roots controlling rice salt tolerance[J].Tag Theoretical and Applied Genetics Theoretische and Angewandte Genetik,2004,108(2):253-260.
[22]Yamanaka N,Ninomiya S,Hoshi M.et al.An informative linkage map of soybean reveals QTLs for flowering tfrne[J].Leaflet Mophobgy and Regions of Segregation Distortion DNA Res,2001(8):61-72.
[23]Wang Y,Yao J,Zhang Z F,et al.Comparative analysis of QTL integrated mapping and statistical analysis of QTLs affecting plant height in maize[J].Chin Sci Bull,2006,51(15):1776-1786.
[24]Goffinet B,Gerber S.Quantitative trait loci:A meta-analysis[J].Genetics,2000,155(1):463-473.
[25]Arcade A,Labourdette A,Chardon F,et al.Biomercator version 2.1[M].UMR:Génétique végétale,2004.
[26]尤莉,邱法展,张祖新,等.整合玉米基因表达与遗传分析资料发掘耐渍性候选基因[J].河北农业大学学报,2007,30(3):9-13.
[27]Guo B,Sleper D A,Lu P,et al.QTLs Associated with resistance to soybean cyst nematode in soybean:Meta-analysis of QTL location[J].Crop Sci,2006,46:595-602.
[28]张耿,祝丽英,陈景堂.玉米产量相关性状QTL通用图谱的构建及功能候选基因的开发[J].华北农学报,2008,23(6):20-27.
[29]姜振.家蚕RAPS-SSR-AFLP三种分子标记综合连锁图谱的构建及图谱整合[D].苏州:苏州大学,2010.

