绵羊肺腺瘤内、外源性env基因比较分析
孔汉金,张克山,刘永杰,尚佑军,吴 斌,刘湘涛
(1.中国农业科学院兰州兽医研究所 家畜疫病病原生物学国家重点实验室 国家口蹄疫参考实验室 兰州730046;2.华中农业大学 农业微生物学国家重点实验室,武汉 430070)
绵羊肺腺瘤(ovine pulmonary adenomatosis,OPA)是由外源性绵羊肺腺瘤反转录病毒(exogenous Jaagsiekte sheep retrovirus, exJSRV)引起的成年绵羊慢性、进行性、接触传染性肺脏疾病[1]。除了大洋洲外,本病已在全球各地发生[2]。该病潜伏期长,病羊临床主要表现为咳嗽,呼吸困难,大量浆液性鼻漏,病理学表现为肺泡和支气管上皮出现肿瘤性增生等典型特征[3]。JSRV感染绵羊后,在自身编码的逆转录酶作用下形成了前病毒及其两端的长末端重复序列(LTR), 并通过整合酶整合到宿主细胞基因组中暂时或长期存在[4]。前病毒DNA编码区内有典型反转录病毒相互重叠的gag,pol,pro,env基因结构[5]。
几乎所有真核生物中都存在有与水平传播的致病性外源性逆转录病毒相对应的内源性反转录病毒(Endogenous ret-rovirus,ERVs),ERVs是远古反转录病毒感染宿主种系后并可垂直传播的宿主基因组中的基因片段[6,7]。关于ERVs 的功能目前仍不完全清楚,在正常绵羊和山羊基因组内发现含有15~20 拷贝的与exJSRV 序列密切相关的内源性绵羊肺腺瘤病毒(endogenous Jaagsiekte sheepretrovirus,enJSRV), 在每个特定物种的个体中都存在[8]。enJSRV基因组结构包括两端非编码区(5′, 3′LTR)及gag, pol, pro, env等 4 个编码基因。JSRVenv基因编码病毒囊膜前体蛋白经水解后产生表面蛋白(SU)和跨膜蛋白(TM)[9]。Exenv与Ⅱ型肺泡上皮细胞Hyal2受体结合,激活PI3K/Akt和MAPK信号通路,促进细胞内特异性转录因子,导致细胞转化的发生和病毒大量增殖从而诱发OPA肿瘤形成[10]。研究表明外源性env基因是JSRV诱导Ⅱ型肺泡上皮细胞转化的必需基因[11],但所有内源性env基因不能诱导细胞转化[12]。Exenv与外源性Enenv有着相同的受体Hyal2,可通过竞争性与受体结合阻止exJSRV感染。而且研究证实Enenv在孕体发展和胎盘形态发生过程中扮演重要生理角色[13,14]。
本文克隆并测定了Enenv序列,进行了Enenv和Exenv核苷酸、推导的氨基酸、基因结构及其蛋白B细胞表位比较分析,为进一步深入探究env功能提供了依据。
1 材料与方法
1.1 材料山东某羊场健康绵羊肺脏。ExTaqDNA 聚合 酶、dNTP、IPTG、X-gal、DNA Marker DL2000,pMD18-T 购自宝生物工程有限公司;Tissue/blood DNA提取试剂盒购自QIAGEN公司。质粒小提试剂盒、凝胶回收试剂盒购自ENZA公司。
1.2 绵羊基因组DNA的提取取绵羊肺脏样品约100 mg 置于研钵中,加入液氮研成粉末,参照组织基因组DNA提取试剂盒(QIAGEN)说明书的要求和步骤,提取总DNA。经紫外分光光度计检测浓度和纯度,-80℃保存。
1.3 引物设计 基因扩增参照GenBank公布的内源性绵羊肺腺瘤病毒env(AF153615)基因序列,应用primer 5.0软件设计如下引物。上引物:TTCAGCAGCCCAGCGATTT;下引物:AGGG AGCTTAGGTACTTGTCC,均由上海生物工程有限公司合成。以1.2提取的DNA为模板,加入上游引物和下游引物进行PCR反应。反应体系:模板5 uL,premix ExTaq25 uL, 上 下 引 物 各 1 uL,ddH2O 补充至 50 uL。反应条件:94℃预变性 5 min;94℃变性 1 min,55℃退火 45 s,72℃延伸 3 min,35个循环;最后72℃延伸10 min,4℃保存。扩增结束后,取反应液5~10 uL,1%的琼脂糖凝胶电泳检测。参照PCR 产物纯化试剂盒(ENZA)说明,将PCR 产物切胶回收纯化,与pMD18-T 载体连接,连接产物转化到大肠杆菌DH5α感受态细胞,涂布含氨苄青霉素、X-gal和IPTG的选择平板上培养12~16 h, 然后进行蓝白斑筛选,挑取白色单菌落接种含LB的细菌瓶,摇床培养6 h后进行菌液PCR鉴定。扩增条件和反应参数与上述PCR反应相同。
1.4 序列测定和遗传进化分析将上述PCR鉴定阳性的菌株送上海美吉测序公司测序,应用DNAStar软件通过Cluster W方法对测得的基因序列和GenBank公布的参考序列分别进行核苷酸和氨基酸水平的同源性分析和构建遗传进化树。
1.5 Enenv和Exenv蛋白结构与功能比较分析
1.5.1 Enenv和Exenv蛋白质一级结构分析 以外源性绵羊肺腺瘤病毒美国代表株JSRV21(AF105220)env基因作为Exenv用于本次研究的比较对象,运用在线Expasy工具ProtParam分析Enenv和Exenv所编码的蛋白分子理化特性。利用DNAStar元件的Kyte_Doolittle方法预测两者编码蛋白的亲水性和疏水性,利用TMHMM2.0和Expasy中的SOSUI和SignalIP分析工具预测两者编码蛋白的跨膜区以及信号肽。
1.5.2 Enenv和Exenv蛋白质二级结构分析 利用DNAStar 分析软件对Enenv和Exenv进行蛋白质二级结构预测并作比较分析。利用Emini软件对氨基酸序列的带电量, 表面暴露区进行分析。综合考虑蛋白质亲水性,表面可能性,抗原性指数等参数,确定两基因编码蛋白的B细胞抗原优势表位。
1.5.3 Enenv和Exenv蛋白质结构域与功能分析 含有保守氨基酸序列的基序组成结构域,是蛋白质序列功能,结构和进化的单元。利用简单模块构架搜索工具(SMART)和Interpro数据库预测和比较分析Enenv和Exenv所编码蛋白的结构功能域。
1.5.4 Enenv和Exenv蛋白质三维结构分析 将Enenv和Exenv基因推导的氨基酸序列提交Swiss-Model,使用RasMol分析软件得到两者三维结构模型并进行比较分析。
2 结果
2.1 序列测定及系统进化分析PCR扩增得到大小约2000 bp的基因片段,与预期结果一致(图略),序列测定结果表明Enenv基因大小为1836 bp,编码611个氨基酸,GC含量为39.22%。将该基因序列提交GeneBank,获得序列号为(JX843793),命名为enJSRV-SD。从Genbank上获得外源性env参考序列美国代表株JSRV21,分析发现Exenv基因全长1848 bp,编码615个氨基酸,GC含量41.18%。Enenv和Exenv的核苷酸同源性为88.1%,差异主要集中在1200~1795区段,位于env基因的TM区,多为单个碱基的改变,且发现Enenv和Exenv相比在1756~1770区段缺失了一段序列。Enenv和Exenv推导的氨基酸序列同源性为92.0%,两者主要差异位于478~599区段,Exenv氨基酸序列TM区发现有YRNM序列,而Enenv在相同位置为XKNM,在其他区段也未发现致病exJSRV特有的YXXM基序。
将本次测得的内源性env序列与Genbank公布的其他内源性和外源性env序列进行比较分析。本次测定的Enenv与国内外发布的enJSRVenv基因推导的氨基酸水平同源性为97.2%~99.2%;与内源性绵羊肺腺瘤病毒南非代表株enJS56A1(AFl53615)的氨基酸同源性最高为99.2%;与exJSRVenv的同源性较低,推导的氨基酸水平同源性为91.5%~92.0%。且与同属山羊动物鼻肿瘤病毒env基因(AY197548)推导的氨基酸同源性为89.4%(图1)。通过遗传进化树可以发现本次克隆的Enenv基因与AFl53615参考毒株亲源关系最近,处于同一分支(图2)。
2.2 Enenv和Exenv蛋白结构与功能比较分析
2.2.1 Enenv和Exenv蛋白质一级结构分析 蛋白一级结构分析发现Enenv和Exenv两者在蛋白分子量,理论等电点,正负电氨基酸个数,脂肪系数和半衰期以及总平均疏水指数等理化性质上具有相似的特性。但两者的不稳定系数有所不同,根据Protparam定义,不稳定系数分值小于40的蛋白稳定,表明内源性env蛋白不稳定(V:40.68),而Exenv稳定(V:36.66)。用Kyte-Doolittle法进行亲水性分析发现Enenv和Exenv具有较强的亲水性,由图3可看出两者亲水区主要都集中在为1~57、97~108、142~151、169~184、228~243、427~438、453~494、592~612等8个区段。使用SOSUI元件分析发现Enenv和Exenv蛋白都含有两个跨膜区,且在相同位置处(381~403、548~570)(图 4)。使用big-PI Predictor元件预测两蛋白都未发现GPI位点,使用Signal IP分析表明两者也都无信号肽。
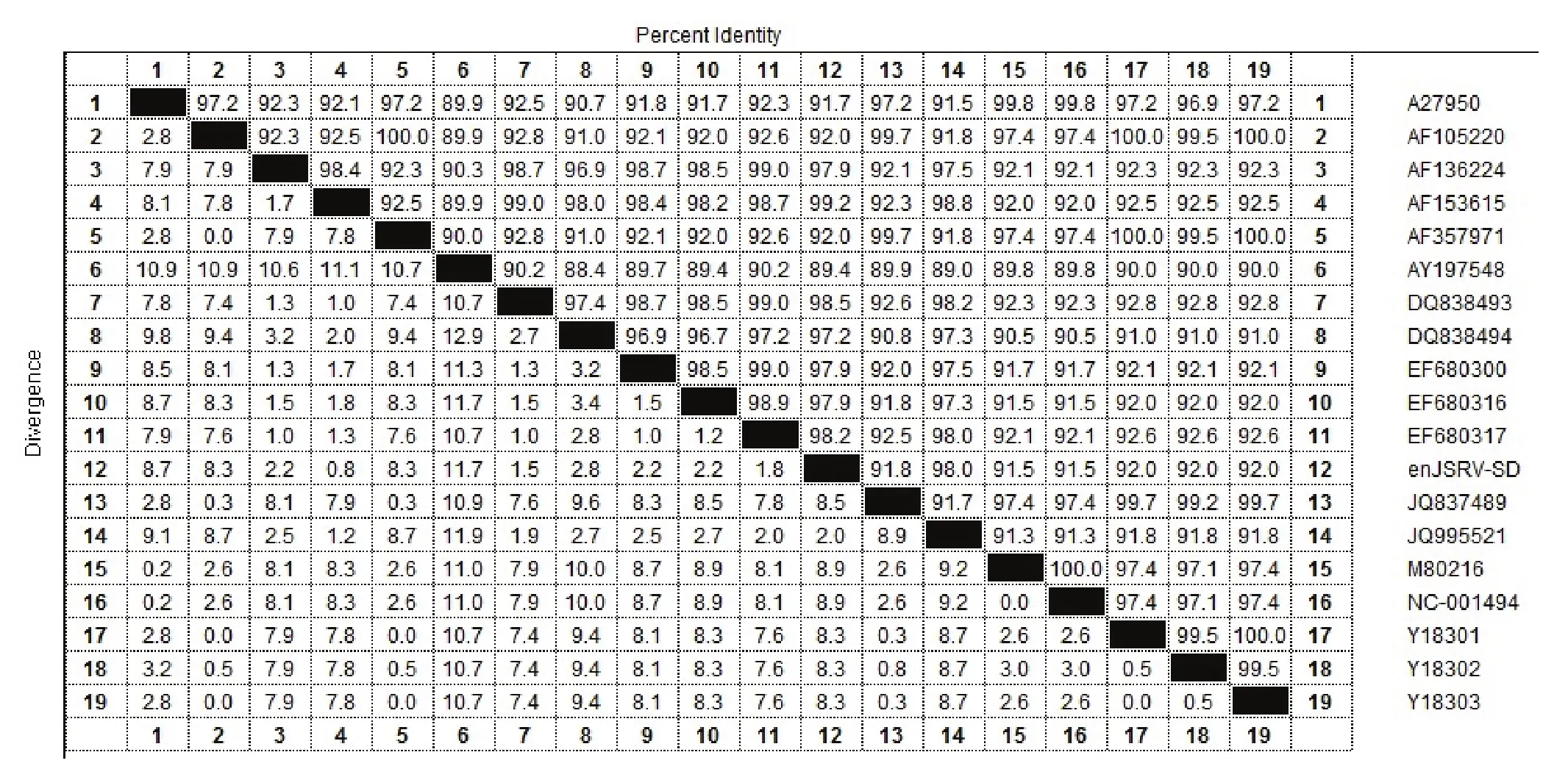
图1 enJSRV 和 exJSRV 各代表株间氨基酸同源性分析Fig.1 The identity of enJSRV and exJSRV amino acid sequences of different strains
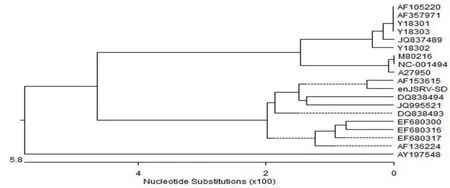
图2 enJSRV 和 exJSRV 各代表株间氨基酸水平系统进化树Fig.2 The Phylogenetic tree for different strains of enJSRV and exJSRV amino acid sequences
2.2.2 Enenv和Exenv蛋白质二级结构分析 Enenv推导的蛋白在第350~369区段,第505~517区段和第573~606区段为α螺旋区域;β折叠区域有43个区段,不规则地分布于整个Enenv蛋白;有28个转角区域和26个无规则卷曲区域。Exenv和Enenv二级结构基本相似,不同的是Exenv在第540~560区段只有1个α螺旋区域,而Enenv有2个,但enJSRV-SD在第25位置处多出1个α螺旋区域;Enenv在第520~600区段的β折叠区域分布明显要比Exenv密集,且Exenv在第610处多1个转角区域(图5)。
2.2.3 Enenv和Exenv蛋白质结构域与功能分析 结果表明Enenv和Exenv蛋白结构域极其相似,都含有1个GP41结构域,该结构域在Enenv蛋白中位于第399-608区段,而在Exenv蛋白中位于399~597。Enenv蛋白都含有2个跨膜结构域,且分布在第381-403、548-570区段,经SMART分析发现由于该区段有其他结构域覆盖而未显现出来。Enenv和Exenv蛋白质二级结构的抗原表位分析结果见图6。综合α-螺旋和β-折叠结构少、富含β-转角和无规则卷曲区域,选择抗原性指数(antigenicity)大于0、亲水性(hydrophilicity)指数大于0、氨基酸位于蛋白分子表面可能性大于1并且邻近柔性结构等参数,Enenv蛋白的B细胞表位可能位于30-35、171-175、178-181、228-233、237-243、477-479和608-610氨基酸区域内或附近。Exenv蛋白的B细胞表位与Enenv蛋白基本相同,可能位于16-20、30-35、178-181、228-233、477-479和 608-610氨基酸区域内或附近。
2.2.4 Enenv和Exenv蛋白质三维结构分析 将内外源性env基因推导的氨基酸序列提交Swiss-Model, 使用PyMOL显示工具得到其三维结构模型(图7)。两者预测的三维模型结构基本相似,从图中可明显看到Exenv蛋白比Enenv蛋白在肽链末端多出1个α螺旋。
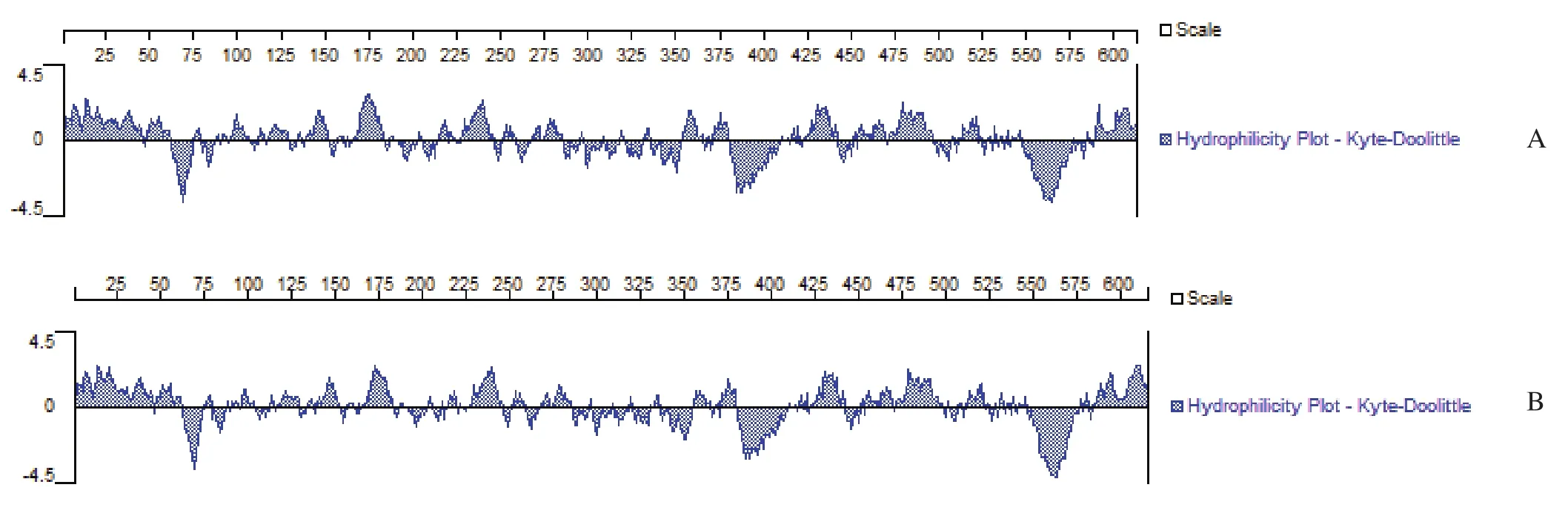
图3 Enenv(A)和Exenv(B)蛋白亲水性(Kyte-Doolittle法)Fig.3 The hydrophilicity plot of Enenv(A) protein and Exenv(B) protein

图4 内源性env(A)和外源性env(B)编码蛋白的跨膜区Fig.4 The transmembrane domain of Enenv(A) protein and Exenv(B) protein
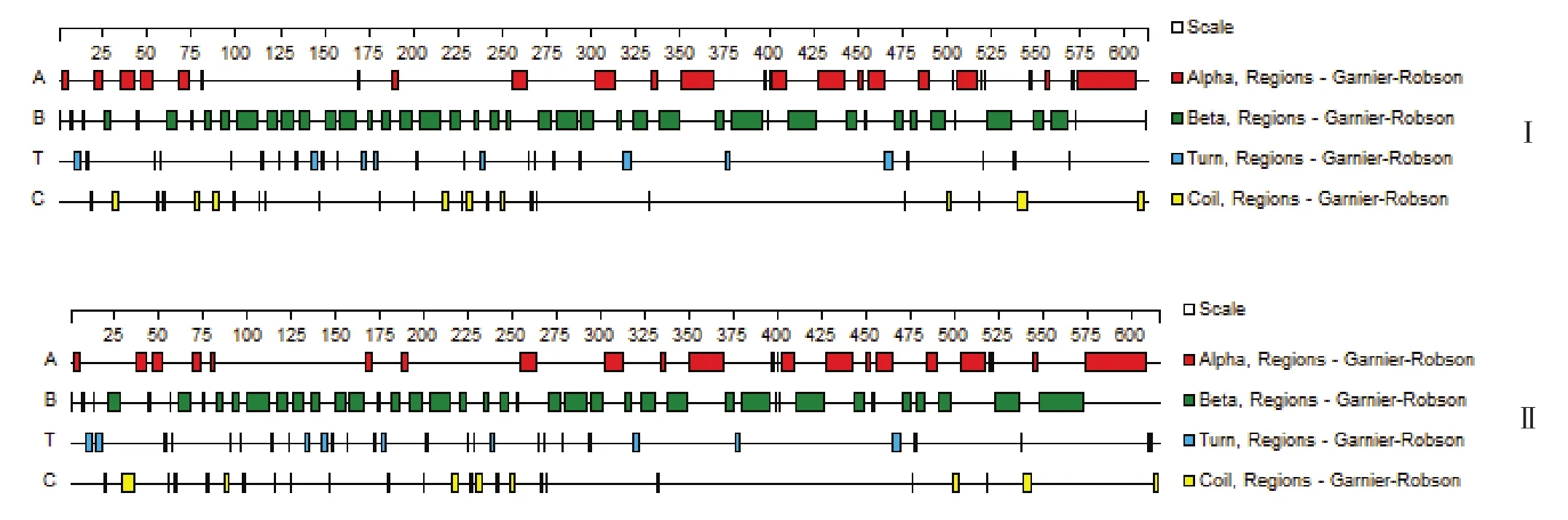
图5 内源性env(Ⅰ)和外源性env(Ⅱ)蛋白二级结构预测结果(Gamier-Robson 方法)Fig.5 Secondary structure of Enenv(Ⅰ )and Exenv(Ⅱ )protein predicted by the method of Gamier-Robson
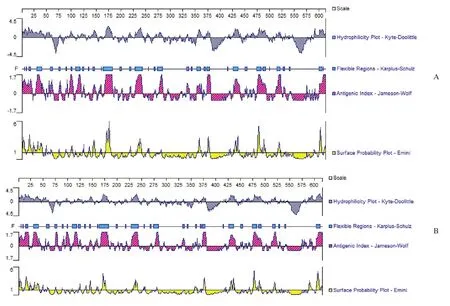
图6 内源性env(A)和外源性env(B) 蛋白的亲水性、抗原指数及其表面可能性分析Fig.6 Analysis of hydrophilicity plot, antigenic index and surface probability of the Enenv(A) protein and Exenv(B) protein

图7 内源性env(A)和外源性env(B)蛋白三维结构预测图(带状结构)Fig.7 The predicted 3-D structure for env protein of Enenv and Exenv generated by PyMOL(Ribbons model)
3 讨论
本研究以山东某羊场健康羊肺脏组织提取的基因组DNA为模板扩增enJSRV的env基因,测序后预测其二三级蛋白结构和抗原表位并与外源性绵羊肺腺瘤病毒美国代表株JSRV21 env作比较分析。序列分析发现Enenv和Exenv在核苷酸水平和结构基因编码蛋白的特征上具有一定的差异。对内外源性env基因核苷酸序列比较发现两者的突变位置主要发生在env基因的TM区,这与文献报道的exJSRV与enJSRV之间的核苷酸序列变异集中在LTR 的U3 和Env基因的TM区相一致[9],且符合反转录病毒变异的一般规律。
遗传进化树分析显示enJSRV-SD与古老型enJS56A1(AF153615)处在同一分支,具有较近的亲缘关系,可见至今我国山东绵羊染色体上保留的enJSRV-SD是古老型的,遗传相对稳定。还发现参与序列比较的内外源性绵羊肺腺瘤病毒分别处于两个不同的大的进化分支上,这也在一定程度上可以作为区别内外源性绵羊肺腺瘤鉴定诊断方法之一[5]。在所有的外源性肺腺瘤病毒env蛋白的胞质尾部包含高度保守的YXXM基序,酪氨酸(Y)磷酸化后作为PI3K/p85调节亚基的结合位点,外源性肺腺瘤病毒可通过env蛋白的胞质尾与其相应受体结合诱导靶细胞信号转导失调,导致细胞增生癌变。而无转化特性的内源性绵羊肺腺瘤病毒的env蛋白无此基序[15]。本次研究扩增的Enenv基因推导的氨基酸序列为HKNM支持了这一结论。
目前预测蛋白质疏水性最为广泛的方法是Kyte-Doolittle法。内外源性env基因编码的蛋白质二级结构总平均疏水指数(GRAVY )分别为-0.044和-0.041,表明内外源性env蛋白都为亲水性。跨膜区预测发现内外源性env蛋白都含有两个跨膜区,并且分布位置大致相同,env基因编码前体蛋白经水解后产生TM和SU,而TM实质是env蛋白的跨膜结构域。
对内外源性env蛋白的二级结构预测发现两者也极为相似,这说明两者可能具有某些相似的功能。结构域功能比较分析发现两者都含有GP41蛋白结构域,该结构域常常存在于各种逆转录病毒的囊膜蛋白上,比如HIV和SIV的囊膜蛋白的GP41亚基,其作用是调控病毒入侵阶段的膜融合[16],其蛋白衍生物可以阻止HIV-1进入人类CD4+细胞和其他细胞系,目前该蛋白已成为研发抵抗HIV和相似逆转录病毒的基因治疗研究的热点[17]。
蛋白质二级结构分析与表位分布有较大关系,螺旋区段及折叠区段因其结构规则,有氢键维持结构,不易形变,主要起稳定结构作用,而转角区域和无规则卷曲区域多位于球蛋白分子表面,在此改变多肽链方法的阻力较小,故易于形变,有利于与抗体结合,从而成为抗原表位的可能性较大。综合考虑这几种因素,预测发现内外源性env蛋白的B细胞抗原表位分布位置也大致相同。Env蛋白作为exJSRV的囊膜成分,存在潜在的黏多糖结合位点。然而对于JSRV感染,机体缺乏针对病毒蛋白的特异性细胞和体液免疫应答[2],这可能是由于绵羊在胸腺T淋巴细胞发育期间表达enJSRV env蛋白能与Hyal2结合从而造成了机体对JSRV的免疫耐受[18]。
内外源性env蛋白具有相似的三维结构表明内外源性env蛋白可能具有相同的受体,事实上两者都可与受体Hyal2结合启动机体内细胞不同的信号途径。enJSRV不仅参与绵羊形成针对exJSRV感染的免疫耐受,而且还与孕体发展有关。反刍动物的妊娠识别信号胎盘干扰素的分泌与enJSRV env蛋白和Hyal2结合有关,怀孕12 d时,enJSRV env转录和表达抑制会造成胚胎植入前发育期孕体的不正常扩展,由固体形式变成丝状,从而不能附着子宫壁上[19]。并且Enenv蛋白与其受体Hyal2共表达能通过调节细胞融合促使巨细胞合胞体斑块形成,而巨细胞合胞体斑块又是胎盘形成的关键[20,21]。目前env蛋白与Hyal2在空间结构作用的具体机制仍不清楚。Enenv的胞质尾区没有酪氨酸残基序列YXXM以及其他部位的序列差异,这可能导致Enenv三维结构与Exenv有着微小的差异,这种差异使得Enenv蛋白不能结合PI3K和激活Akt下游信号通路,最终导致Enenv蛋白无致瘤活性。本次研究通过对绵羊肺腺瘤内、外源性env基因的比较分析,为内外源性Env蛋白功能研究提供了线索,进而为绵羊肺腺瘤病毒的致病机制研究提供了理论依据。
[1]Griffiths D J, Martineau H M, Cousens C,et al.Pathology and Pathogenesis of Ovine Pulmonary Adenocarcinoma[J].J Comp Pathol 2010, 142(4): 260-283.
[2]Sharp J M, DeMartini J C.Natural history of JSRV in sheep[J].Curr Top Microbiol, 2003, 275: 55-79.
[3]刘淑英, 马学恩.绵羊肺腺瘤病研究进展[J].动物医学进展, 2003, (1): 19-22.
[4]Holland M J, Palmarini M, Garcia-Goti M,et al.Jaagsiekte retrovirus is widely distributed both in T and B lymphocytes and in mononuclear phagocytes of sheep with naturally and experimentally acquired pulmonary adenomatosis[J].J Virol, 1999, 73(5): 4004-4008.
[5]Johnson C, Fan H.Jaagsiekte Sheep Retrovirus and Lung Cancer[J].Cancer Associated Viruses, 2012: 755-791.
[6]Boeke J, Stoye J.Retrotransposons, endogenous retroviruses, and the evolution of retroelements[M].Retroviruses Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY 1997: 343-435.
[7]Lee Y N, Bieniasz P D.Reconstitution of an infectious human endogenous retrovirus[J].Plos Pathog, 2007, 3(1):119-130.
[8]周艳喜, 苏佳, 李靖, 等.内源性绵羊肺腺瘤病毒全基因组序列测定与序列分析[J].中国兽医学报, 2012, (7):962-966.
[9]York D, Vigne R, Verwoerd D,et al.Nucleotide sequence of the jaagsiekte retrovirus, an exogenous and endogenous type D and B retrovirus of sheep and goats[J].J Virol,1992, 66(8): 4930-4939.
[10]Zavala G, Pretto C, Chow YHJ,et al.Relevance of Akt phosphorylation in cell transformation induced by Jaagsiekte sheep retrovirus[J].Virology, 2003, 312(1):95-105.
[11]Maeda N, Palmarini M, Murgia C,et al.Direct transformation of rodent fibroblasts by jaagsiekte sheep retrovirus DNA[J].Proc Natl Acad Sci, 2001, 98(8):4449-4454.
[12]Arnaud F, Caporale M, Varela M,et al.A paradigm for virus-host coevolution: Sequential counter-adaptations between endogenous and exogenous retroviruses[J].Plos Pathog, 2007, 3(11): 1716-1729.
[13]Palmarini M, Gray C A, Carpenter K,et al.Expression of endogenous betaretroviruses in the ovine uterus:Effects of neonatal age, estrous cycle, pregnancy, and progesterone[J].J Virol, 2001, 75(23):11319-11327.
[14]Dunlap K A, Palmarini M, Adelson D L,et al.Sheep endogenous betaretroviruses (enJSRVs) and the hyaluronidase 2 (HYAL2) receptor in the ovine uterus and conceptus[J].Biol Reprod, 2005, 73(2): 271-279.
[15]Liu S L, Miller A D.Oncogenic transformation by the jaagsiekte sheep retrovirus envelope protein[J].Oncogene,2007, 26(6):789-801.
[16]He Y X, Cheng J W, Li J J,et al.Identification of a critical motif for the human immunodeficiency virus type 1 (HIV-1) gp41 core structure: Implications for designing novel anti-HIV fusion inhibitors[J].J Virol, 2008, 82(13):6349-6358.
[17]Zahn R C, Hermann F G, Kim E Y,et al.Efficient entry inhibition of human and nonhuman primate immunodeficiency virus by cell surface-expressed gp41-derived peptides[J].Gene Ther, 2008, 15(17): 1210-1222.
[18]Spencer T E, Mura M, Gray C A,et al.Receptor usage and fetal expression of ovine endogenous betaretroviruses:Implications for coevolution of endogenous and exogenous retroviruses[J].J Virol, 2003, 77(1): 749-753.
[19]Bazer F W, Burghardt R C, Johnson G A,et al.Interferons and progesterone for establishment and maintenance of pregnancy: interactions among novel cell signaling pathways[J].Reprod Biol, 2008, 8(3): 179-211.
[20]Dunlap K A, Palmarini M, Spencer T E.Ovine endogenous betaretroviruses (enJSRVs) and placental morphogenesis[J].Placenta, 2006, 27: S135-S140.
[21]Dunlap KA, Palmarini M, Varela M,et al.Endogenous retroviruses regulate periimplantation placental growth and differentiation[J].Proc Natl Acad Sci USA, 2006,103(39): 14390-14395.

