SVA反转录转座子的研究进展
谈丹丹,洪道俊,吴裕臣
(南昌大学第一附属医院神经内科,江西南昌 330006)
反转录转座子约占人类基因组的34%,引起的人类疾病约0.27%,其中占主导的为长散置元(long interspersed element-1,L1)、Alu 序列、SVA(SINEVNRT-Alu)(short interspersed element of retroviral origin,SINE-R;variable numble of tandem repeats,VNTR;Alu-like)[1]。以往对L1和Alu序列研究较多,而近年来,已认识SVA的基本结构,SVA在人类基因组中的重要作用逐渐受到重视。目前发现SVA与人类基因组的进化及多种疾病的发生密切相关。
1 SVA基本结构
SVA为复合序列,主要由SINE-R、VNTR、Alulike组成,故称SVA[2]。SVA的基本结构分为5部分[3],从5'端开始依次是 CCCTCT六聚体重复元件、Alu-like、富含 G-C 的 VNTR、SINE-R、加尾信号与多聚A尾序列。另外,SVA两侧翼均有靶位点重复(target-site duplication,TSD)(图1)。

图1 SVA基本结构Fig 1 The basic structure of the SVA
Alu-like由两个反义Alu片段及两者中间的单拷贝序列而组成。VNTR由36~42 bp或49~51 bp的序列拷贝而成[4],可能衍生于恒河猴的SVA2序列,对SVA的启动至关重要,可能有助于增强RNA的稳定性。SINE-R由包膜(envelope,env)基因及一个起源于古老的内源性反转录病毒的长末端重复序列组成[5]。CCCTCT六聚体重复元件及Alu-like缺失会导致SVA反转录转座启动率明显减少,故推测CCCTCT六聚体重复元件、Alu-like与SVA反转录转座启动密切相关。
2 SVA与人类基因组
2.1 SVA在人类基因组的分布
SVA是一种非长末端重复序列、非自主性反转录转座子,典型长度平均约2.0 kb,但SVA插入时长度700~4 000 bp,在人类基因组中,约2 700拷贝,占0.2%,目前已鉴定出2 762种[6]。
2.2 SVA亚家族与人类基因组的进化
反转录转座子是引起基因组不稳定的重要因素,反转录转座子插入引起基因组改变及物种变异,不断丰富和促进了生物的进化。SVA是最年轻的仍活跃的原始人类特异的非长末端重复序列,约起源于2500万年前[7]。目前共发现7种 SVA亚家族[8-9],分别为 SVA-A、SVA-B、SVA-C、SVA-D、SVAE、SVA-F、SVA-F1,其中 SVA-A、SVA-B、SVA-C、SVA-D起源于人类-猩猩分化之前,SVA-E、SVA-F、SVA-F1则只出现在人类时期。
SVA中VNTR区域富含G-C,与基因组的稳定性密切相关。随着进化,SVA的G-C富集区密度明显下降[8],导致SVA呈不断活跃趋势。人类基因组中约40%的SVA具有多态性,SVA-E和SVA-F插入多态性频率分别为37.5%和27.6%。
活跃的SVA可导致产生转录变异体[10],使人类基因组不断的改变与进化。
SVA DNA甲基化也可能是物种差异及进化的重要影响因素,在人类精子中SVA DNA呈明显的低甲基化,而在黑猩猩精子中则为甲基化。研究显示,人类精细胞中35%的同源SVA甲基化水平低于50%,而黑猩猩精细胞中仅有6%的同源SVA甲基化水平低于50%[11]。
3 SVA插入与疾病
3.1 SVA反转录转座
SVA通过DNA-RNA-DNA的途径实现反转录转座,新生儿的SVA反转录转座率约为1/916,次于Alu 序列(1/21)及 L1(1/212)[6]。由于本身不能编码蛋白质,SVA必须借助于L1编码的胞内酶才能实现反转录转座。在L1介导下,SVA DNA转录生产mRNA后再反转录生成cDNA,最后cDNA插入并整合到DNA上,从而导致基因组发生改变。
SVA基因组插入呈现出L1介导反转录转座和靶引物反转录的典型特征:1)插入发生在L1的核酸内切酶识别位点5'-TTTT/AA-3';2)SVA插入的两侧翼有4~20 bp靶位点重复序列;3)不同长度的多聚A尾;4)5'端截断;5)内部重排或倒置;6)3'端转导[12]。
基因组中SVA插入的检测方法有多种:Souther杂交、Norther杂交、PCR、反转录PCR等,其中,PCR最常用。利用PCR时,根据人类BLAT或NCBI的BLAST软件,可针对 SVA两侧翼 TSD、SINE-R区域、Alu-like区域或具体被检测基因(如TAF1基因)等多种特异序列来设计引物,然后扩增目的基因,并利用自动测序仪或2%琼脂糖凝胶电泳分析扩增产物,检测SVA插入。例如,对福山型肌营养不良的患者进行基因诊断,可使用一种三引物PCR及2%琼脂糖凝胶电泳的方法,快速检测FKTN基因中SVA 插入突变[13]。
3.2 SVA插入与疾病
现已证实SVA反转录转座子是人类致病的重要因素。SVA反转录转座主要发生于未分化或低分化细胞,与遗传性疾病及肿瘤等密切相关。目前已发现由SVA插入引起的人类疾病有8种(表1)[6,14]。
3.3 SVA致病的相关机制
SVA介导的插入突变、选择性剪接、外显子重组及差异性甲基化等[12],可导致DNA结构、转录及翻译等发生改变,从而致病。SVA介导的选择性剪接可分为外显子捕获、外显子选择等,继而引起外显子重组,使基因转录及表达异常。
外显子捕获使基因结构及表达的蛋白质发生改变,从而导致多种疾病,如福山型肌营养不良(Fuku-yama-type congenital muscular dystrophy,FCMD)、中性脂质贮积病伴肌病(neutral lipid storage disease associated with subclinical myopathy,NLSDM)、常染色体隐性遗传性高胆固醇血症(autosomal recessive hypercholesterolemia,ARH)等。例如,FCMD 是一种最常见的常染色体隐性遗传性先天性肌营养不良,该病在日本人群中的发病率为1/90,由于fukutin蛋白缺乏导致α-抗肌萎缩相关糖蛋白低糖基化,从而导致发病。编码fukutin蛋白的FKTN基因包括10个外显子,87%的FKTN基因突变来源于同一祖先建立者的第10个外显子下游3'端非编码区一个约3 kb的SVA插入[13]。研究发现,SVA插入后激活了第10个外显子内一个新的供体衔接点,通过RNA剪接,新的供体衔接点与SVA内一纳体衔接点连接,形成了新的外显子即第11个外显子[15],这个过程称为外显子捕获。SVA插入介导的选择性剪接事件,导致FKTN基因编码的fukutin蛋白缺乏及功能障碍,从而致病。最新研究表明,反义寡核苷酸靶向结合剪接点的治疗方法可有效恢复fukutin蛋白,解救SVA插入导致的福山型肌营养不良[16]。
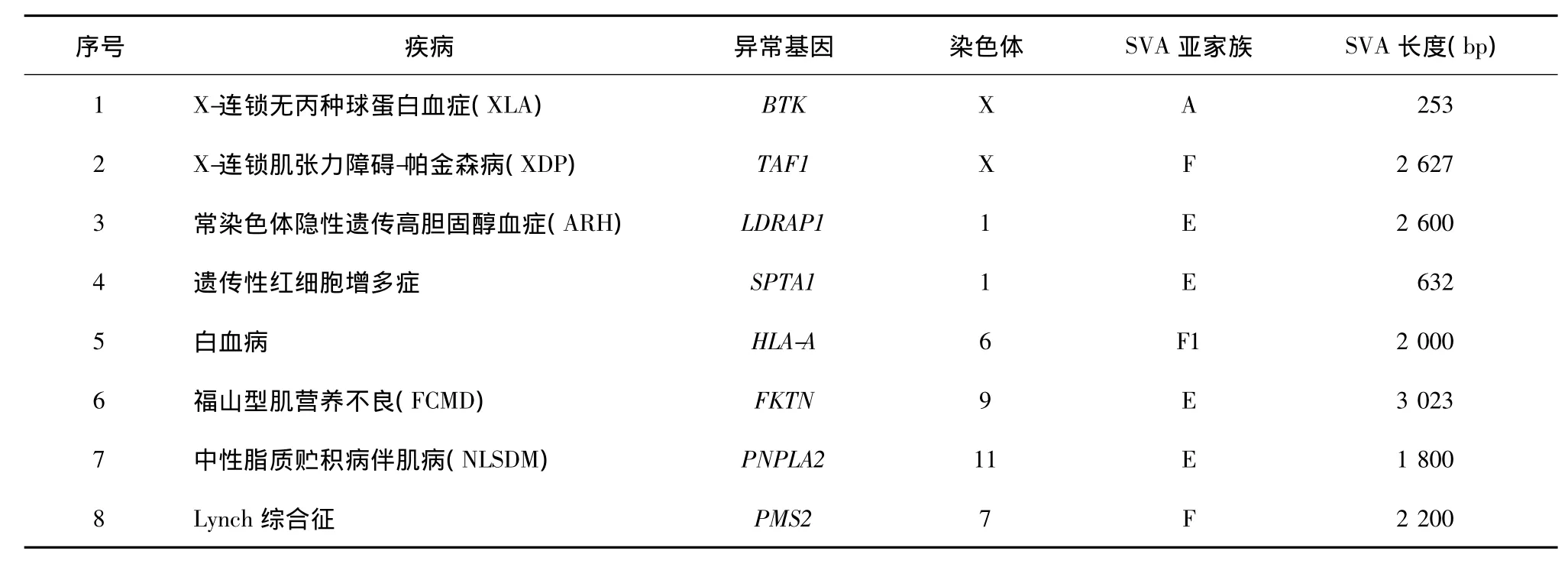
表1 SVA插入与疾病Table 1 The SVA insertions and diseases
SVA介导的外显子选择事件也可导致疾病的发生,例如 X-连锁无丙种球蛋白血症(X-linked agammaglobulinemia,XLA)。SVA插入中断了 BTK基因的第9个外显子5'端剪接点,造成外显子跳跃性识别,这种外显子选择引起的外显子重排可导致正常编码的蛋白质缺失,继而发病。另外,在BTK基因第9号外显子末端之前12 bp处即SVA插入位点也发现了Alu序列插入,表明该位点容易发生插入突变[17]。
目前认为,DNA甲基化程度可影响基因的表达。研究发现,SVA插入到X染色体的TAF1基因第32个内含子中,并伴SVA DNA甲基化[18],从而导致TAF1 mRNA表达减少,使尾状核中神经元特异性蛋白TAF1蛋白表达减少,继而干扰许多神经元基因的转录(如多巴胺D2受体)[19],可引起X-连锁肌张力障碍-帕金森病(X-linked dystonia-parkinsonism,XDP)。
SVA介导的插入突变可直接导致的基因结构改变,是其最简单致病机制。例如,SVA插入到SPTA1基因的第5个外显子中,继而产生了一个5'端截短且相对于前体倒置的结构,导致该基因编码的α收缩蛋白结构异常,引起遗传性红细胞增多症[4]。但是,目前还有一些由SVA插入而导致的疾病(如白血病、Lynch综合征)具体发病机制尚不清楚。研究发现,HLA-A基因整体缺失引起的白血病伴随着基因下游SVA插入[20],可能由于SVA插入后灭活HLA-A基因而致病。再如,研究发现,一个2.2 kb 5'端截短的SVA插入到PMS2基因第7个外显子中,导致错配修复基因PMS2突变,成为Lynch综合征一个新的致病因素[18]。
4 展望
SVA反转录转座子导致的疾病陆续被发现,近几年有关SVA介导的基因突变及功能异常的报道也逐渐增多,SVA正逐渐引起人们的关注。越来越多的证据表明,最年轻的活跃的反转录转座子SVA既对人类基因组的进化起着重要影响,也促进了人类疾病的发生。进一步认识SVA对研究人类基因组进化、人类疾病的发病机制及其诊断与治疗等有着重要意义。
[1]Konkel MK,Batzer MA.A mobile threat to genome stability:The impact of non-LTR retrotransposons upon the human genome[J].Semin Cancer Biol,2011,20:211 -221.
[2]Shen L,Wu LC,Sanlioglu S,et al.Structure and genetics of the partislly duplicated gene RP located immediately upstream of the complement C4A and the C4B genes in the HLA class Ⅲ region.Molecular cloning,exon-intron structure,composite retroposon,and breakpoint of gene duplication[J].J Biol Chem,1994,269:8466 -8476.
[3]Hancks DC,Goodier JL,Mandal PK,et al.Retrotransposition of marked SVA elements by human L1s in cultured cells[J].Hum Mol Genet,2011,20:3386 -3400.
[4]Ostertag EM,Goodier JL,Zhang Y,et al.SVA elements are nonautonomous retrotransposons that cause disease in humans[J].Am J Hum Genet,2003,73:1444 - 1451.
[5]Faulkner GJ.Retrotransposons:mobile and mutagenic from conception to death[J].FEBS Lett,2011,585:1589 -1594.
[6] Hancks DC,Kazazian HH Jr.Active human retrotransposons:variation and disease[J].Curr Opin Genet Dev,2012,22:191 -203.
[7]Cordaux R,Batzer MA.The impact of retrotransposons on human genome evolution[J].Nat Rev Genet,2009,10:691-703.
[8]Wang H,Xing J,Grover D,et al.SVA elements:a hominidspecific retroposon family[J].J Mol Biol,2005,354:994 -1007.
[9]Hancks DC,Ewing AD,Chen JE,et al.Exon-trapping mediated by the human retrotransposon SVA[J].Genome Res,2009,19:1983 -1991.
[10] Kim DS,Hahn Y.Identification of human-specific transcript variants induced by DNA insertions in the human genome[J].Bininformatics,2011,27:14 -21.
[11]Molaro A,Hodges E,Fang F,et al.Sperm methylation profiles reveal features of epigenetic inheritance and evolution in primates[J].Cell,2011,146:1029 - 1041.
[12]Raiz J,Damert A,Chira S,et al.The non-autonomous retrotransposon SVA is trans-mobilized by the human LINE-1 protein machinery[J].Nucleic Acids Res,2012,40:1666-1683.
[13]Watanabe M,Kobayashi K,Jin F,et al.Founder SVA retrotransposal insertion in Fukuyama-type congenital muscular dystrophy and its origin in Japanese and Northeast Asian populations[J].Am J Med Genet A,2005,138:344 -348.
[14]van der Klift HM,Tops CM,Hes FJ,et al.Insertion of an SVA element,a non-autonomous retrotransposon,in PMS2 intron 7 as a novel cause of Lynch syndrome[J].Hum Mutat,2012,33:1051 -1055.
[15]Nakamori M,Thornton C.Muscular dystrophy:A hidden ancestral legacy trumped[J].Nature,2011,478:46 -47.
[16]Taniguchi-Ikeda M,Kobayashi K,Kanagawa M,et al.Pathogenic exon-trapping by SVA retrotransposon and rescue in Fukuyama muscular dystrophy[J].Nature,2011,478:127-131.
[17]Conley ME,Partain JD,Norland SM,et al.Two independent retrotransposon insertions at the same site within the coding region of BTK[J].Hum Mutat,2005,25:324 -325.
[18]Makino S,Kaji R,Ando S,et al.Reduced neuron-specific expression of the TAF1 gene is associated with X-linked dystonia-parkinsonism[J].Am J Hum Genet,2007,80:393-406.
[19]Pasco PM,Ison CV,Muňoz EL,et al.Understanding XDP through imaging,pathology,and genetics[J].Int J Neurosci,2011,121:12 -17.
[20]Takasu M,Hayashi R,Maruya E,et al.Deletion of entire HLA-A gene accompanied by an insertion of a retrotransposon[J].Tissue Antigens,2007,70:144 -150.

