1株中度嗜盐菌质粒基因组文库的构建及2个膜蛋白基因的克隆和序列分析
朱文华,董冠群,滕长财,侯 娟,吕 彦,孙业盈*
(1.滨州医学院 药学院,山东 烟台 264003;2.烟台大学 生命科学学院,山东 烟台 264005)
中度嗜盐菌(modrately halophilic bacteria)近年来正受到越来越多的重视,与此相关的中度嗜盐菌的分离,鉴定,基因组学研究以及耐盐机理的研究也日益广泛和深入。中度嗜盐菌是生活在极端高盐环境中的微生物,具有较强的耐盐能力,能在含有0% ~32%NaCl的环境中生长,最适盐度为3% ~15%[1-2]。中度嗜盐菌广泛分布在不同的高盐环境中如盐湖、海洋、盐碱地和盐渍发酵食品等[3-6]。嗜盐细菌主要通过2种机制来适应高渗透压的外界环境:一种是通过在细胞内积累高浓度的盐离子如KCl和NaCl等,来使细胞内的渗透压升高,从而平衡外界环境的高渗透压,如Halobacteriaceae和Salinibacter类嗜盐菌都是通过此机制来实现耐盐的;另一种是通过在细胞内合成或者积累高浓度的相容性溶质,来升高细胞内的渗透压,抗衡细胞外的高渗透压对细胞的影响[7]。虽然,目前研究人员已经发现了部分嗜盐菌的耐盐策略,但是对其如何通过怎样的基因表达调控网络来实现这一过程还不清楚。因此对嗜盐菌进行深入的基因组学,转录组学和蛋白组学的研究,发掘克隆相关的基因,对它们进行功能研究就显得尤其重要。先前的研究中从蜢子虾酱中分离纯化鉴定了1株中度嗜盐菌,通过测定其16S rDNA序列,并通过系统发育分析将该菌初步鉴定为枝芽胞杆菌属(Virgibacillus)中的盐脱氮枝芽胞杆菌(Virgibacillus halodenitrificans)[8]。本研究进一步通过构建该中度嗜盐菌的质粒基因组文库,经测序,比对分析克隆了2个膜蛋白基因,并对它们进行了生物信息学的相关分析。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒 嗜盐菌提取于天然发酵的蜢子虾酱,购买于山东省烟台市烟台大学农贸市场。质粒pUC19,具有氨苄青霉素抗性。
1.1.2 试剂 HindⅢ-HF内切酶、T4DNA连接酶购自 NEB公司;Taq酶、CIAP(Alkaline Phosphatase)购自TaKaRa公司。
1.2 方法
1.2.1 细菌DNA的提取,酶切与酶切片段纯化用BioTeKe Corporation细菌基因组提取试剂盒提取嗜盐菌MKY20的总DNA,然后用限制性内切酶HindⅢ-HF处理,建立50 μL的反应体系,酶切2 μg细菌基因组DNA;将酶切片段经琼脂糖凝胶电泳后,用TaKaRa MiniBEST Agarose Gel DNA Exraction Kit回收片段大小为1.5~9 kb的DNA片段。
1.2.2 载体制备 提取适量的pUC19质粒以限制性内切酶HindⅢ-HF完全酶切后,试剂盒回收,然后用CIAP进行去磷酸化处理,抑制质粒的自身环化,降低假阳性克隆的比率。
1.2.3 连接与转化 通过T4DNA连接酶连接经脱磷酸处理的pUC19质粒载体与酶切DNA片段。将得到的连接产物加入到100 μL大肠埃希菌感受态细胞中,置冰浴30 min;冰浴后放入42℃的恒温水浴锅中热击90 s,热击处理后迅速加入0.7 mL LB培养液,放置在37℃的恒温摇床中震荡培养1 h,然后取0.1 mL转化菌液涂布固体LB平板于37℃过夜培养。
1.2.4 载体插入片段大小的鉴定 随机挑取转化培养的单克隆,以其 DNA为模板,利用M13(47)和M13(48)引物进行菌落PCR,然后把PCR扩增后产物进行琼脂糖凝胶电泳,和DNA Maker比对确定片段大小。
1.2.5 测序与比对分析 随机选取一定数量的插入片段的克隆,送上海美吉生物科技公司测序。经生物学软件分析,筛选具有完整的开发阅读框(ORF)的克隆。用DNASTAR软件推导ORF编码的氨基酸序列,提交到GenBank进行BLAST比对,对ORF所编码的蛋白质的同源性进行分析。将部分ORF编码的氨基酸序列和其它物种的该蛋白的氨基酸序列用ClustalW比对。跨膜区的预测通过在线程序 http://www.cbs.dtu.dk/services/TMHMM/进行。
2 结果与分析
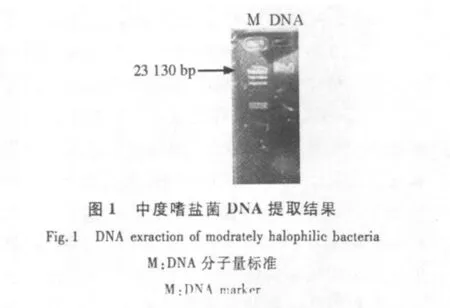
2.1 中度嗜盐菌基因组DNA的提取,酶切和片段富集
提取中度嗜盐菌MKY20总DNA,经琼脂糖凝胶电泳进行检测,结果如图1,DNA片段大小在23.1 kb左右。用限制酶 HindⅢ-HF酶切总DNA,酶切产物经琼脂糖凝胶电泳结果如图2,第一泳道所示,片段大小不一,弥散分布。切胶回收1.5~9 kb之间的片段,经电泳检测,如图2第二泳道所示,结果表明大片段明显集中,去除掉了小于1.5 kb的片段。
2.2 质粒基因组文库的构建及阳性克隆的检测
酶切后回收大小在1.5~9 kb的片段,然后与PUC19质粒载体(经HindⅢ-HF酶切并进行去磷酸化处理)连接,连接产物转化大肠埃希菌DH5a,构建中度嗜盐菌MKY20的质粒基因组文库。随机挑选转化单克隆菌落,用载体引物M13(47)和M13(48)进行PCR扩增,鉴定插入片段大小,经凝胶电泳检测,结果如图3所示,插入片段大小都在2~4 kb,重组质粒占总转化质粒的90%以上,证明了基因组质粒文库的可靠性。由此估算该基因组文库的容量大约为5×103个克隆,以平均每个克隆插入3 kb计算,该文库可覆盖约15 Mb的遗传信息。
2.3 NhaC基因的克隆及序列分析
从中度嗜盐菌MKY20的质粒基因组文库中某个随机测序的克隆中获得了一完整的ORF,经过BLAST比对分析表明该ORF所编码的蛋白质与Na+H+逆向转运蛋白(Na+H+antiporter,NhaC)相似,该蛋白存在于单细胞的原核生物到多细胞的真核生物的细胞质膜上,起到调节细胞内pH值和Na+浓度的功能[9-11]。进一步的生物信息学分析表明中度嗜盐菌MKY20 NhaC基因全长ORF为1446 bp,编码481个氨基酸的蛋白质,推测的分子量为50.5 ku,共10个跨膜区域(图4),GenBank登陆号为JX849200。



2.4 MscS基因的克隆及进化分析
在质粒基因组文库中随机测序的克隆中获得了另外一个具有完整的ORF的克隆,经BLAST比对分析表明该ORF所编码的蛋白质分别与一离子通道蛋白(small conductance mechanosensitive channel,MscS)具有较高的同源性。该MscS基因的全长ORF为822 bp,编码273个氨基酸的蛋白质,推测分子量为 30.0 ku,GenBank登陆号为JX849202。采用ClustalW软件,对该菌的MscS蛋白的氨基酸序列与GenBank中其他4种菌的MscS蛋白相应氨基酸序列进行了比对分析(图5),结果表明中度嗜盐菌MKY20的MscS蛋白与其他4种菌的MscS蛋白有较高的同源性,并且都具有3个跨膜区,其中第3个跨膜区的氨基酸序列最保守。

图5 不同菌属MscS蛋白氨基酸序列的比对分析Fig.5 Alignment analysis of MscS amino acid sequence from different species
3 讨论
本研究的实验对象是从蜢子虾酱中分离出来的1株中度嗜盐菌,经16S rDNA测序比对以及生理生化的鉴定,确定为盐脱氮枝芽胞杆菌(Virgibacillus halodenitrificans)。目前,还没有该菌种基因组学方面的研究报道。细菌的基因组大小一般在4 M左右,因此本研究在构建盐脱氮枝芽胞杆菌基因组文库时选择了能够承载几个kb的质粒载体。由于小片段包含的遗传信息比较小,并且小片段相比大片段插入质粒的概率要大的多,因此为了使每个克隆尽可能的包含更多的遗传信息,在构建文库之前,对酶切的基因组片段进行了筛选,只选择了大于1.5 kb的酶切片段进行文库构建,文库鉴定结果也表明所构建的基因组文库的克隆中插入片段均大于1.5 kb,这样就保证了每个克隆的遗传信息的承载量,便于从克隆中筛选基因全长。
Na+H+逆向转运蛋白(antiporter)是存在于质膜上的一类蛋白家族,在包括微生物、植物、动物和人中都存在,它们在维持细胞内部pH、Na+浓度等方面起着重要的调节作用[9]。在植物和微生物的研究中均已表明Na+/H+逆向转运蛋白是生物耐盐的关键因子,能够维持高盐胁迫下生物体的正常生长代谢[10-11]。本研究中从盐脱氮枝芽胞杆菌的质粒基因组文库中筛选到Na+H+逆向转运蛋白,经比对分析确定为NhaC,鉴于NhaC在其他微生物中已被证明具有耐盐功能[12],因此本研究所克隆的盐脱氮枝芽胞杆菌的NhaC基因可作为该菌的一个候选耐盐基因。机械敏感性离子通道(Mechanosensitive(MS)ion channels)存在于从单细胞的微生物到多细胞的动植物的质膜中,可以察觉细胞外的机械性的胁迫如重力,接触等,还可以对细胞内环境如渗透压力和膜损伤起反应[13]。大肠埃希菌是目前研究机械敏感性离子通道的最好模型,该蛋白可以分为2类,一类是MscL(Mechanosensitive channel of Large conductance),另一类是MscS(Mechanosensitive channel of Small conductance)[13]。研究表明MS通道在细菌渗透胁迫中具有重要的保护作用[14],因此,推测MS通道在高盐环境中生存的微生物自我保护也是必不可少的。在盐脱氮枝芽胞杆菌的质粒基因组文库中筛选到了一MscS基因,故该基因也可作为该菌的一个耐盐候选基因。盐脱氮枝芽胞杆菌质粒基因组文库的构建以及2个膜蛋白基因的克隆,将为进一步研究盐脱氮枝芽胞杆菌的耐盐机制,及其在盐渍食品中的作用提供了基础。
[1]Vreeland RH.Mechanisms of halotolerance on microorganisms[J].Crit Rev Microbiol,1987,14(4):311-356.
[2]任培根,周培谨.中度嗜盐菌的研究进展[J].微生物学报,2003,43(3):427-431.
[3]Swan BK,Ehrhardt CJ,Reifel KM,et al.Archaeal and bacterial communities respond differently to environmental gradients in anoxic sediments of a California hypersaline lake,the Salton Sea[J].Appl Environ Microbiol,2010,76(3):757-768.
[4]Sorokin DY,Tourova TP,Galinski EA,et al.Extremely halophilic denitrifying bacteria from hypersaline inland lakes,Halovibrio denitrificans sp.nov.and Halospina denitrificans gen.nov.,sp.nov.,and evidence that the genus name Halovibrio Fendich 1989 with the type species Halovibrio variabilis should be associated with DSM 3050[J].Int J Syst Evol Microbiol,2006,56(2):379-388.
[5]Xu XW,Wu YH,Zhao Z,et al.Halomonas saccharevitans sp.nov.,Halomonas arcis sp.nov.and Halomonas subterranea sp.nov.,halophilic bacteria isolated from hypersaline environments of China[J].Int J Syst Evol Microbiol,2007,57(7):1619-1624.
[6]Hezayen FF,Younis MAM,Hagaggi NSA,et al.Oceanobacillus aswanensis strain FS10 sp.nov.,an extremely halotolerant bacterium isolated from salted fish sauce in Aswan City,Egypt[J].Global J Mol Sci,2010,5(1):1-6.
[7]Ma Y,Galinski EA,Grant WD,et al.Halophiles 2010:life in saline environments[J].Appl Environ Microbiol,2010,76(21):6971-6981.
[8]孙业盈,单长民,武玉永.蜢子虾酱中度嗜盐菌的分离、鉴定及特性研究[J].中国酿造,2011,236(11):94-97.
[9]Mager T,Rimon A,Padan E,et al.Transport mechanism and pH regulation of the Na+/H+antiporter NhaA from Escherichia coli:an electrophysiological study[J].J Biol Chem,2011,286(26):23570-23581.
[10]刘广发,曾活水,陈启伟,等.假单胞菌Na+/H+逆向转运蛋白基因nhaA的克隆与鉴定[J].遗传学报,2005,32(3):309-314.
[11]李静,刘明,孙晶,等.Na+(K+)/H+转运蛋白NHX基因的研究进展[J].大豆科学,2011,30(6):1035-1039.
[12]Liew CW,Illias RM,Mahadi NM,et al.Expression of the Na+/H+antiportergene(g1-nhaC)of alkaliphilic Bacillus sp.G1 in Escherichia coli[J].FEMS Microbiol Leet,2007,276(1):114-122.
[13]Sachs,F.Stretch-activated ion channels:what are they?[J].Physiology(Bethesda),2010,25(1):50-56.
[14]Hoffmann T,Boiangiu C,Moses S,et al.Responses of Bacillus subtilis to hypotonic challenges:physiological contributions of mechanosensitive channels to cellular survival[J].Appl Environ Microbiol,2008,74(8):2454-2460.

