HRM-非标记探针法检测IL-15基因SNPs方法的建立*
王 玥,郑启城,姚秀云,邓 兵,宪 莹,于 洁
(重庆医科大学附属儿童医院血液肿瘤科/儿童发育疾病研究省部共建教育部重点实验室/儿科学重庆市重点实验室/重庆市儿童发育重大疾病诊治与预防国际科技合作基地 400014)
HRM-非标记探针法检测IL-15基因SNPs方法的建立*
王 玥,郑启城,姚秀云,邓 兵,宪 莹,于 洁△
(重庆医科大学附属儿童医院血液肿瘤科/儿童发育疾病研究省部共建教育部重点实验室/儿科学重庆市重点实验室/重庆市儿童发育重大疾病诊治与预防国际科技合作基地 400014)
目的建立用于检测白细胞介素-15(IL-15)基因单核苷酸多态性(SNP)位点的高分辨率熔解曲线(HRM)-非标记探针的方法。方法采用HRM-非标记探针法及小片段扩增法对80名健康儿童IL-15基因4个SNPs位点进行基因分型,采用基因测序法进行验证并同时与小片段扩增法分型结果进行比较。结果HRM-非标记探针法与基因测序分型结果一致,准确率为100%;未加入温度内标的小片段扩增法不能区分野生纯合子和突变纯和子。结论HRM-非标记探针法是对已知SNP位点突变研究的一种廉价、简便、准确的基因分型技术,适合于对已知的SNP位点或基因突变进行分型和检测。
白细胞介素15;多态性,单核苷酸;高分辨率熔解曲线分析;非标记探针法
高分辨率熔解曲线(high resolution melting,HRM)分析技术是一种新的DNA突变检测技术,其原理是通过掺入饱和荧光染料实现对DNA双链熔解过程的监控,再利用Tm值的差异和熔解曲线的形状来区别突变[1]。HRM属于全闭管式操作,前期的PCR和后期的熔解曲线分析均在一个管内完成,不需要对PCR产物进行分离和处理。与单链构象分析、高效液相色谱、变性梯度凝胶电泳、毛细管凝胶电泳等方法相比,HRM减少了交叉污染的概率并且节省了后期操作的成本和时间。此外,由于HRM不对PCR产物进行酶切等操作,不影响产物完整性,因此分析后的样品能够继续用于后续试验,如凝胶电泳、测序等,利用HRM技术进行基因分型的常用方法有两种:小片段扩增法和非标记探针法。目前,国内较多报道是采用小片段扩增法进行基因突变的筛查和基因分型,其弱点是对野生纯合子和突变纯合子以及对G/C突变及A/T突变的区分能力较差,需要额外引入温度内标或已知基因分型的样品进行区分[2]。而使用非标记探针法直接针对SNPs位点进行设计,能够较好地区分野生纯合子和突变纯合子,尤其是G/C突变和A/T突变,故较适用于已知SNPs的基因分型,但目前在国内应用HRM-非标记探针法进行基因分型的研究报道较少。
白血病是儿童最常见的恶性肿瘤,年发病率为3~5/10万,占儿童恶性肿瘤的35%以上,其中又以急性淋巴细胞白血病(acute lymphoblastic leukemia,ALL)为主,占70%~80%;该病发病机制未完全阐明,并且在现行化疗方案下仍有部分患儿无法获得临床缓解或者发生复发[3-4]。2003年,本院对193例ALL患儿治疗情况进行总结,发现仍有9%无法达到临床缓解;随访22例进入维持治疗期的患儿,其中4例复发[5]。因此,为进一步提高ALL患儿的治愈率,应继续寻找与ALL治疗不缓解以及复发相关的危险因素。最近,一项基于全基因组范围的SNPs筛查研究发现,位于IL-15 3′端非翻译区(3′-UTR)的5个SNPs位点(rs17007695、rs35964658、rs10519613、rs10519612和rs17015014)与儿童ALL治疗后微小残留病阳性密切相关[6-7]。为进一步证实上述位点与中国白血病患儿发病及预后的相关性,本研究选取IL-15SNPs位点为研究对象,拟对IL-15SNPs位点进行基因分型,而其中关键的技术的是IL-15SNPs的基因型检测。鉴于HRM技术的优点,以及待分析的IL-15SNPs位点中包含C/G突变,因此本文选取了基于Light Scanner96HRM检测仪的HRM-非标记探针法,并应用该方法首先对健康儿童的IL-15 3′-UTR单个核苷酸多态性位点进行基因分型,现将结果报道如下。
1 资料与方法
1.1 一般资料 所有80例样本来源于既往本院进行儿童流行病学调查时所冻存的健康体检儿童外周血样本。采用TIA Namp Genomic DNA试剂盒(北京天根生化科技有限公司)提取基因组DNA。
1.2 HRM 检测
1.2.1 引物及探针的设计和合成 在NCBI的SNP数据库中输入SNP识别号,获取SNP位点前后250bp片段(参考序列 NC_000004.11,IL-15范 围:142,557,749-142,655,140)。利 用 Primer-Blast网 站 (http://www.ncbi.nlm.nih.gov/tools/primer-blast)设计高特异性引物,并将产物长度限制在100~300bp。采用Oligo6软件对引物进行验证,剔除含有会影响扩增效率的引物二聚体、发夹结构的引物对。设计非标记探针与野生型互补,其3′末端采用 C-3spacer法封闭。rs17007695上游:5′-TGA GGC TAC GTC AGA GCT AGG CAT-3′,下游:5′-CCA CCT CGA GCC TGG TAC AAC A-3′,探针:5′-TAC CAT TGG CTT TCT TTG AAA ATC ACA CAT-3′(250bp);rs35964658 上 游:5′-CCC TTA GCC CCC AGC AAT GAG C-3′,下游:5′-TGG AGA AGG TGT GGC TTA CCC C-3′,探 针:5′-TCA AAT GAC CAC ACT TTA ATT TTC CAG C-3′(297bp);rs10519613上游:5′-GCA AAG AAT GTG AGG AAC TGG AG-3′,下游:5′-TGC CTT CAT TTC TAA GAG TTC ATC TG-3′,探针:5′-ACT CGG CAT TTC AAA TGT GCT GTC A-3(254bp);rs17015014上游:5′-ACA CTG GGG CTG AAG CAC ATC T-3′,下 游:5′-AGC CAA CAC CCT CAT CCC TTT GC-3′,探针:5′-GAA GCA AGT TTG CTG TAA AGA TGC TA-3′(194bp)。引物和探针均由上海生生物工程有限公司合成。
1.2.2 不对称PCR扩增 使用 Mastercycler梯度聚合酶链反应(PCR)仪(德国Eppendorf公司)进行PCR扩增。PCR反应用2×Hotstrat Taq PCR Master Mix试剂(北京天根生化科技有限公司)进行扩增,荧光染料用高分辨率的饱和LC Green荧光染料试剂(美国Idaho公司)。反应体系包括:DNA模板1 μL(1ng/μL),PCR Master Mix(2× )5μL,上 游 引 物 (1 μmol)、下游引物(5μmol)各1μL,探针(2.5μmol)1μL,LC Green荧光染料(10×)染料1μL。PCR扩增条件为:95℃ 预变性120s;然后95℃30s,63℃30s退火,72℃30s延伸,共55个循环以促进引物的消耗,确保单链的大量合成;95℃60s,15℃停止,促进异源双链核酸分子形成。
1.2.3 HRM分析进行基因分型 采用Light Scanner96HRM检测仪(美国Idaho公司)对PCR产物进行基因分型。检测温度范围为60~85℃,每升高0.1℃采集1次荧光亮度数据。获得原始熔解曲线图后,Light Scanner96分析软件即自动对背景信号和孔间荧光信号进行校正。选定探针峰熔解曲线范围后,软件生成标准化后的熔解曲线图,并行自动基因分型。
2 结 果
Light Scanner96HRM检测仪一次扫描可对96个样本进行熔解曲线分析,每次分析用时约8min。60~85℃范围内共产生两个熔解曲线峰,低温区的为探针峰,高温区的为产物峰。通过将分析范围限制在探针峰区域并经行转换生成衍生图后,纯合子的熔解曲线为单峰、杂合子为双峰。由于所有的非标记探针设计与野生型互补,故野生型的Tm值高于突变型,Tm差值为3~6℃。根据探针峰的数目、峰型以及Tm值,采用Light Scanner软件对样本进行自动分型,见图1。笔者采用非标记探针法、小片段扩增法及测序法对这80份健康汉族儿童标本进行了以上4个位点的测定。非标记探针法分型结果与测序结果一致,准确率为100%(表1);另一方面,由于未加入高低温内标,小片段扩增法仅能够区分杂合型和纯合型,但对进一步区分野生纯合型和突变纯合型的能力较差。rs17015014G>C位点48个样本的两种分型方法结果,见图2。
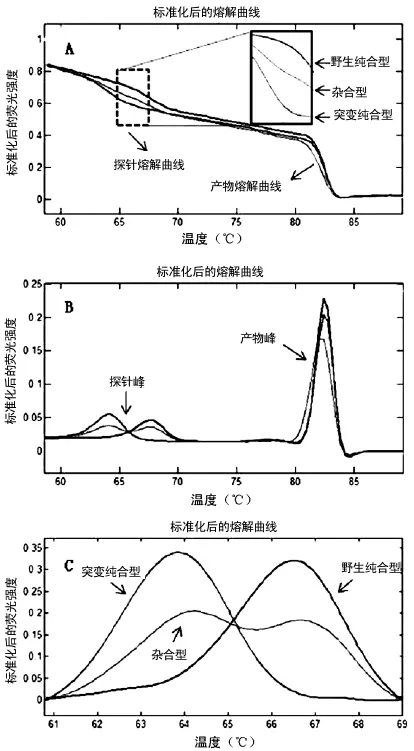
图1 HRM-非标记探针法的基因分型
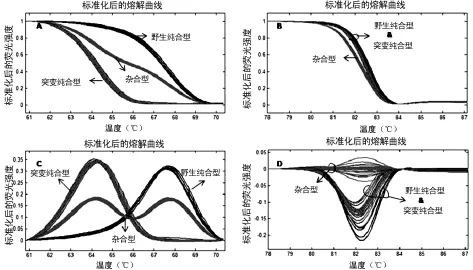
图2 非标记探针法与小片段扩增法分型比较

表1 80例样本基因分型结果
2 讨 论
HRM分析是近年来兴起的SNPs筛查和基因分型的技术,并且因其高通量、高准确性以及廉价的特点而受到了越来越多的关注和应用[8-9]。常用的HRM分析法有小片段扩增法和非标记探针法。小片段扩增法多被用于测序前的突变筛查,以减少测序的工作量。其缺点是在用于G/C突变(Ⅲ类突变)或A/T突变(Ⅳ类突变)的基因分型时,由于Tm值相差太小,往往不容易将野生纯合子和突变纯合子分开。在本实验中也进行了小片段扩增法分析,但未能够得到准确的分型结果,考虑与所设计的扩增产物片段较大(194~297bp)以及未加入高低温内标作孔间温度校正有关。此外,高通量HRM检测仪(96/384孔)由于存在孔间温度差异问题,需要在每个反应孔内都加入一对同样经过3′端封闭的高低温内标进行温度校正,因此在费用方面并不比非标记探针法更低廉。
非标记探针法基本原理同小片段扩增法,都是通过比较Tm值差异进行基因分型。但与小片段扩增法比较产物的熔解曲线不同,非标记探针法比较的是探针与产物的熔解曲线。由于加入的探针长度较短(20~40bp),增加了的Tm值差距(>3℃),因此非标记探针法在区分G/C突变(Ⅲ类突变)和A/T突变(Ⅳ类突变)时更具有优势。本实验涉及的4个SNP位点中即包含一个G/C突变。结果显示非标记探针法对该位点的区分清楚、分型准确,以测序法为标准其准确率可达100%。Liew等[10]对小片段扩增法和非标记探针法进行了对比,发现在扩增片段小于100bp时,小片段扩增法与非标记扩增法的分型结果一致,准确率都能够达到100%;但当扩增片段大于200bp时,由于小片段扩增法中野生/突变纯合子间的Tm值差异减小,从而导致了分型准确性下降。另外,由于有研究提到了可以采用低分辨率的荧光定量PCR仪对非标记探针进行基因分型,因此,笔者也试图利用Bio-Rad CFX96荧光定量PCR仪对PCR产物进行熔解曲线分析,但结果未见熔解曲线中有明显的探针峰,并且利用产物峰亦不能分型,考虑可能与机器激发光源和LCGreen染料不匹配和不能将数据导出到Light Scanner软件进行分析有关。
非标记探针法中,探针设计是实验成功的关键。经验表明,22bp以上的探针长度能够提供足够强的探针峰荧光强度;探针Tm值最好在55~70℃,以避免与引物二聚体Tm范围重叠(通常在75~80℃)及影响产物的扩增。尽管通过以上方法已能够对大部分SNPs或基因突变进行分型检测,但更复杂的基因分型,比如基因突变合并单核苷酸多态性、相邻位点的多个基因突变等,仍然给非标记探针法带来了挑战。由于受到同一扩增片段中的相邻的突变位点或SNP位点的干扰,野生型和突变型的探针峰熔解曲线可能会很相似,因此无法顺利进行区分。Poulson等[11]以隔离-探针 PCR(iolated-probe PCR,IP-PCR)实验平台为基础,通过设计两个Tm值差距较大的非标记探针成功地对同一扩增片段中相隔46bp的两个SNP位点同时进行基因分型,节约了实验成本;Margraf等[12]则是通过采用“遮蔽技术”,即将非标记探针上的一个或多个突变/SNPs位点进行遮蔽(错配法、位点删除法或通用碱基法),排除了邻近单核苷酸多态性位点或突变位点带来的干扰,清楚的识别出了存在突变的样本。这些方法的提出使非标记探针法具有了对较复杂的突变/SNPs位点进行分型的能力,拓宽了非标记探针法的应用范围。
综上所述,非标记探针法在区别Ⅲ、Ⅳ类突变时较小片段扩增法更具有优势、实验成本更低廉,更适合于对已知的SNPs位点或基因突变进行分型和检测。
[1]Wittwer CT,Reed GH,Gundry CN,et al.High-resolution genotyping by amplicon melting analysis using LC Green[J].Clin Chem,2003,49(6):853-860.
[2]张建佚,冯洁,杨泽,等.高分辨率熔解曲线小扩增子法结合混样法在线粒体13928G>C突变分析中的应用[J].中国医药生物技术,2009,4(6):445-448.
[3]张义琼,朱易萍,廖清奎.儿童急性淋巴细胞白血病治疗和预后进展[J].中国实用儿科杂志,2006,21(10):786-789
[4]Möricke A,Reiter A,Zimmermann M,et al.Risk-adjusted therapy of acute lymphoblastic leukemia can decrease treatment burden and improve survival:treatment results of 2169unselected pediatric and adolescent patients enrolled in the trial ALL-BFM 95[J].Blood,2008,111(9):4477-4489.
[5]于洁,徐酉华,宪莹,等.儿童急性淋巴细胞白血病3年临床总结[J].重庆医学,2003,3(10):1326-1329.
[6]Yang JJ,Cheng C,Yang W,et al.Genome-wide interrogation of germline genetic variation associated with treatment response in childhood acute lymphoblastic leukemia[J].JAMA,2009,301(4):393-403.
[7]Lin D,Liu C,Liu H,et al.The role of interleukin-15polymorphisms in adult acute lymphoblastic leukemia[J].PLoS One,2010,5(10):e13626.
[8]Vossen RH,Aten E,Roos A,et al.High-resolution melting analysis(HRMA):more than just sequence variant screening[J].Hum Mutat,2009,30(6):860-866.
[9]Montgomery JL,Sanford LN,Wittwer CT.High-resolution DNA melting analysis in clinical research and diagnostics[J].Expert Rev Mol Diagn,2010,10(2):219-240.
[10]Liew M,Seipp M,Durtschi J,et al.Closed-tube SNP genotyping without labeled probes/a comparison between unlabeled probe and amplicon melting[J].Am J Clin Pathol,2007,127(3):341-348.
[11]Poulson MD,Wittwer CT.Closed-tube genotyping of apolipoprotein E by isolated-probe PCR with multiple unlabeled probes and high-resolution DNA melting analysis.[J].Biotechniques,2007,43(1):87-91.
[12]Margraf RL,Mao R,Highsmith WE,et al.RET proto-oncogene genotyping using unlabeled probes,the masking technique,and amplicon high-resolution melting analysis[J].J Mol Diagn,2007,9(2):184-196.
To establish the high resolution melting analysis with unlabeled probes for genotyping IL-15 gene SNPs*
Wang Yue,Zheng Qicheng,Yao Xiuyun,Deng Bing,Xian Ying,Yu Jie△
(Department of Hematology-Oncology,the Affiliated Children's Hospital of Chongqing Medical University/the Key Laboratory of Child Development Disease Research of Education Ministry/the Key Laboratory of Paediatrics of Chongqing/Chongqing International Science and Technology Cooperation Base for Major Child Disease Development and Prevention,Chongqing400014,China)
ObjectiveWe were going to establish the high resolution melting analysis with unlabeled probes for genotyping IL-15gene SNP.MethodsGenotypes of the IL-15gene SNPs were determined in 80unrelated Han healthy children using High resolution melting curve analysis with unlabeled probes,small amplification method,genotype results were corroborated by gene sequencing.ResultsThe genotype result from unlabeled probes method and gene sequencing were consistent,the accuracy rate was 100%;small amplification method without temperature inter calibration could not differentiate the wild homozygous and mutant homozygous.ConclusionHigh resolution melting curve analysis with unlabeled probes is a cheap,convenient,fast and accurate genotype technology for known SNP sites.
interleukin-15;polymorphisms,single nucleotide;high resolution melting;unlabeled probes
10.3969/j.issn.1671-8348.2012.34.001
A
1671-8348(2012)34-3577-03
国家“十一五”科技支撑计划资助项目(2007BAI04B03);重庆市卫生局医学科研项目基金资助项目(2011-2-208)。 △
,Tel:13906005672;E-mail:yujie167@yahoo.com。
2012-07-02
2012-09-18)

