ScYLV和SrMV的PCR引物优化设计
王洪星 ,张雨良 ,罗志文 ,杨文君 ,刘志昕
(1.中国热带农业科学院热带生物技术研究所/农业部热带作物生物技术重点开放实验室,海口 571101;2.海南大学环境与植物保护学院,儋州571737)
聚合酶链式反应(PCR)是体外扩增DNA的一种技术,其能够在短时间内根据极微量的模板序列扩增出大量特异性DNA片断,扩增过程类似于核裂变。Mullis博士在1983年发明了该技术,PCR技术是现代分子生物学中最有价值的技术之一(David Clark,2005),随着科学技术的发展其又衍生出多种类型的相关技术,包括差异显示PCR、实时定量PCR、巢式PCR、多重PCR和不对称PCR等(萨姆布鲁克,2002)。
PCR方法的广泛应用及其相关技术的顺利进行均离不开PCR引物的合理设计。可以说,PCR引物的合理设计是任何PCR反应能否进行及其产物的特异性、产率高低的关键。目前许多计算机软件如Primer Premier 3.0,Primer Premier 5.0,Oligo 6.0等均可用于引物的设计。但由于软件自身存在的局限性,其设计的引物并无法满足实验者不同需要。目前最常用的是计算机辅助设计,即实验者根据PCR引物设计的原则并结合自己的经验人工设计引物,然后选择合适的软件分析引物的合理性。
1 材料和方法
1.1 实验材料及软件
实验用甘蔗样品采自海南两院甘蔗种植基地。 Trizol(Invitrongen),SYBR Green supermix(TaKaRa)、RTPCR试剂盒PrimeScript TM One-Step Ver.2.0购自宝生物工程(大连)有限公司,PCR产物回收试剂盒(上海生物工程有限公司),反转录酶、Taq酶及其它生物试剂均购自北京全式金生物科技有限公司,其它药品为国产分析纯。Primer Premier 5.0、Oligo6.0、DNAMAN等软件于http://www.bio-soft.net下载。
1.2 甘蔗总RNA提取和RT-PCR
从待检测甘蔗植株上取幼嫩组织,以TRIzol法(余爱丽,2004)得到较理想的RNA样品。提取的总RNA溶解于100μL dd H2O中,取4μL RNA溶液稀释200倍,采用UV-760紫外分光光度计测量260nm、280nm的OD值,计算OD260/280的比值并用1.2%的普通琼脂糖凝胶电泳在约5V/cm的电压下,快速电泳检测并照相。
以所提取的总RNA为模板,利用宝生物工程 (大连)有限公司SuperScript III反转录体系进行,PCR反应按全式金公司TaqDNA聚合酶操作说明进行,并优化反应程序。
1.3 PCR引物的设计与目的序列测序
1.3.1 普通PCR引物设计与目的序列测序 根据甘蔗黄叶病毒 (ScYLV)genebank相关基因系列 (登录号AF369923.1;AF369924.1;AF369925.1;AF369926.1;AF369927.1;AF369928.1;AF369929.1;AF141385.1;AF141385.1)、 高粱花叶病毒 (SrMV) 相关基因序列 (登录 号:NC_004035.1;DQ530434.1;U57358.2;AY648298.1)通过DNAMAN软件比对,找出保守序列,综合考虑引物设计的各项原则,运用DNAMAN、Primer Premier 5.0和Oligo6.0进行基因同源性分析和引物设计,使引物序列G+C含量介于45%~60%,引物长度介于18~25bp,目的片段介于400~1000 bp(见表1)。
对PCR的各引物浓度及反应的退火温度进行优化,筛选出不同病毒PCR反应体系的最佳反应模式。PCR产物经1.0%琼脂糖凝胶电泳;切胶后用回收目的片段,克隆于pMD18-T载体,送上海生物工程公司测序。
1.3.2 多重PCR引物设计扩增 利用上述结果,结合考虑多重PCR引物设计的各项原则,运用Primer Premier 5.0和Oligo6.0进行引物验证,以挑选出一对特异引物(表1)。
1.3.3 实时定量PCR引物设计 综合考虑多重PCR引物设计的各项原则, 运用 Primer Premier 5.0、Oligo6.0和DNAMAN进行基因同源性分析和引物设计。对PCR的各引物浓度及反应的退火温度进行优化,筛选出PCR反应体系的最佳反应模式(表2)。切胶回收,测序;连接pMD18-T载体、转化、提取质粒,稀释10倍,利用SYBR Green I荧光染料法做标准曲线。

表2 用于实时定量PCR扩增的引物特征
1.4 带有酶切位点的引物设计与目的序列测序
根据ScYLV相关基因 (登录号AY23679.1、GU190159.1、HM640267、AF157029.1、AF369927.1、GU190159.1、AF157029.1、AM072750.1、AY236971.1、GU570006.1),综合考虑酶切位点引物设计的各项原则,运用Primer Premier 5.0、Oligo6.0和DNAMAN进行基因同源性分析和引物设计,设计出克隆Coat Protein(CP)基因和P0蛋白基因的引物(表3)。
对PCR的各引物浓度及反应的退火温度进行优化,筛选出不同基因片段PCR反应体系的最佳反应模式。切胶回收、双酶切,连接pMD18-T载体、转化、提取质粒,双酶切质粒,凝胶电泳,回收目的片段,再送检测序。

表3 用于扩增ScYLV相关序列的带酶切位点的引物特征
2 结果与分析
2.1 样本总RNA电泳结果
用TRIzol法分别从感病植株叶片中提取RNA,琼脂糖凝胶电泳显示各条带清晰,无明显的拖尾现象,RNA具有较好的完整性(图1)。
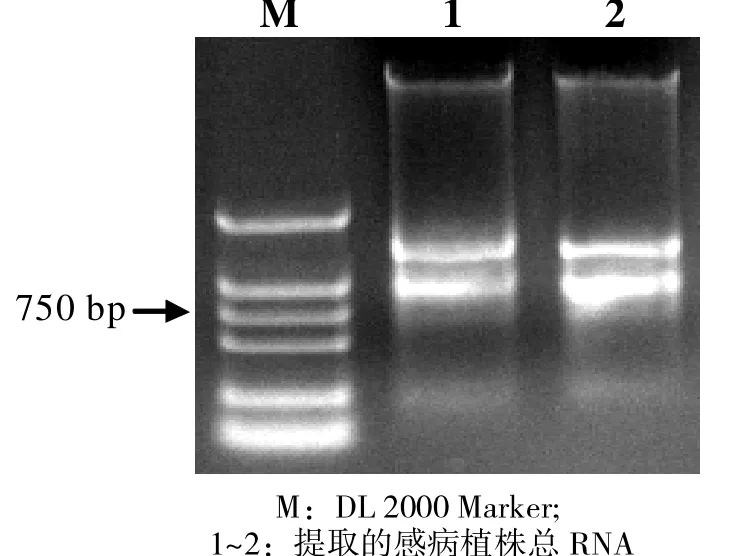
图1 Trizol法提取甘蔗病叶总RNA 1%琼脂糖凝胶电泳
2.2 PCR引物效果检测
2.2.1 普通PCR引物效果检测 所设计PCR引物扩增效率高、特异性好、电泳结果呈单一亮带,且各引物无特异性条带,引物设计成功。ScYLV和SrMV均见特异性反应条带,经测序,1~5的条带大小依次为:833、470、591、400、346 bp,与预期产物大小相同(图 2)。
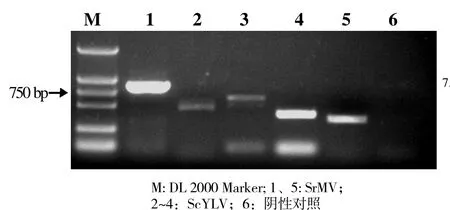
图2 引物特异性试验
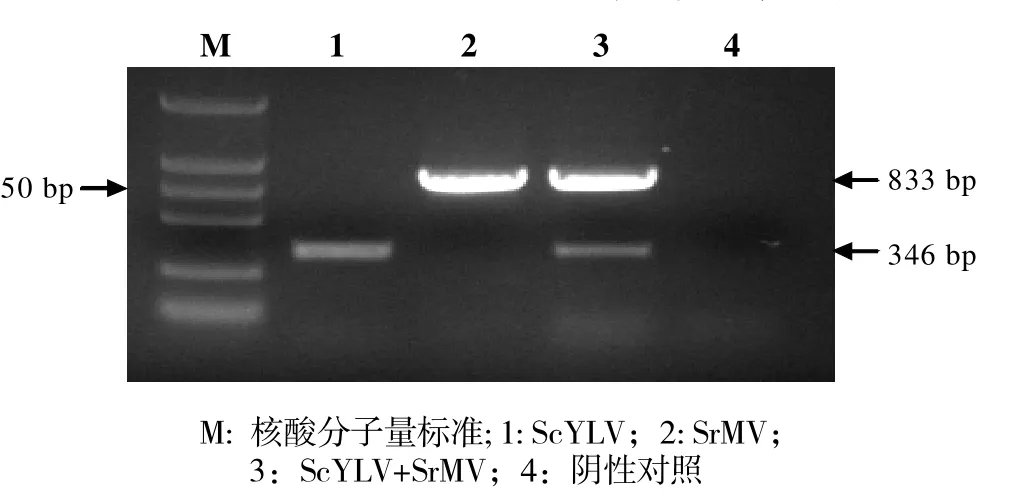
图3 多重RT-PCR特异性试验
2.2.2 多重PCR引物设计效果检测 特异性试验ScYLV和SrMV均见特异性反应条带,而且引物之间与模板之间也未产生明显的相互干扰,引物设计成功。经测序,序列大小分别为346和833bp,与预计产物大小相同(图3)。
2.2.3 实时定量PCR引物设计效果检测 试验以制备的ScYLV标准品 (浓度分别为 108、107、106、105、104、103、102、101)采用10倍浓度的梯度稀释。通过梯度稀释样本的设置,用扩增曲线达到域值时的反应循环数(Ct值)做回归曲线,结果如图4:可以得到较理想的标准曲线,线性关系良好 (R2=0.988),扩增效率为99.8%,引物设计成功,可以定量检测ScYLV。
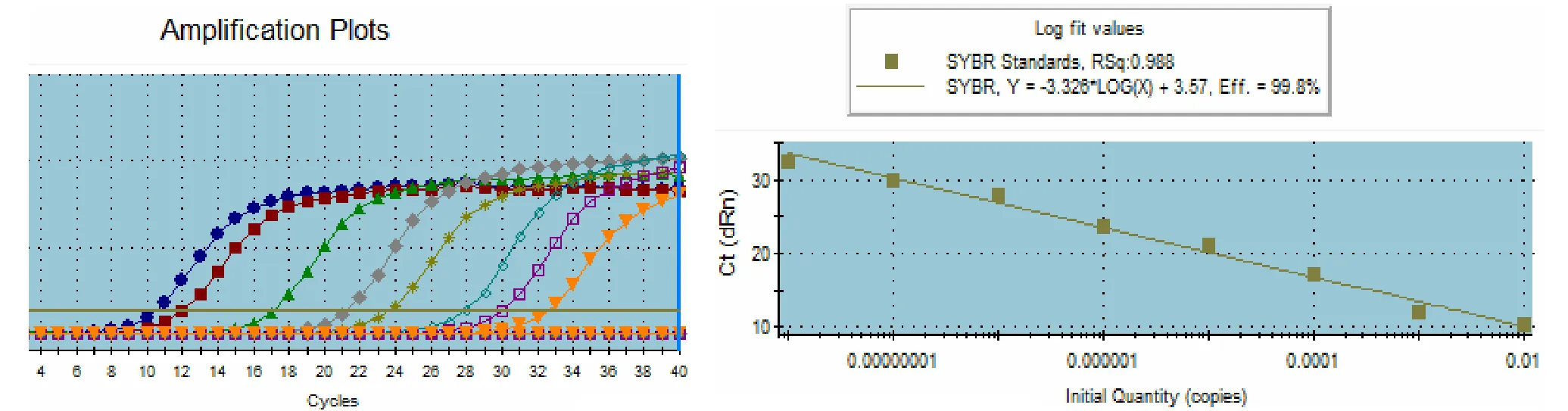
图4 ScYLV荧光定量标准曲线

图5 PMD-19重组质粒的酶切图谱
2.3 带有酶切位点的引物设计效果检测
PCR产物经双酶切后的电泳结果 (图5)表明,所设计引物扩增效率高、特异性好,可用于后续试验。ScYLV CP、P0可以顺利克隆表达,经测序,条带大小分别为591 bp和771 bp,与预期产物大小相同,均成功连有酶切位点,经蛋白分析后,CP基因在表达载体中表达出来的融合蛋白大约43kD;P0基因在表达载体中表达出来的融合蛋白大约51 kD。
3 讨论
PCR引物设计的目的是为了找到一对合适的寡核苷酸片段,使其能有效地扩增模板DNA序列。因此,引物的优劣直接关系到PCR的特异性成功与否。设计PCR引物,首先要解决引物评估问题,即回答什么是合适的引物,以及这些引物的相互干扰是否在可接受的范围内。PCR引物设计问题中要考虑的生物参数分为两部分:首先是先确定单个引物的约束参数,据此可以区分引物的优劣;其次是根据不同PCR的要求确定引物间的约束参数,据此来评价引物之间的干扰是否在可接受的范围内。
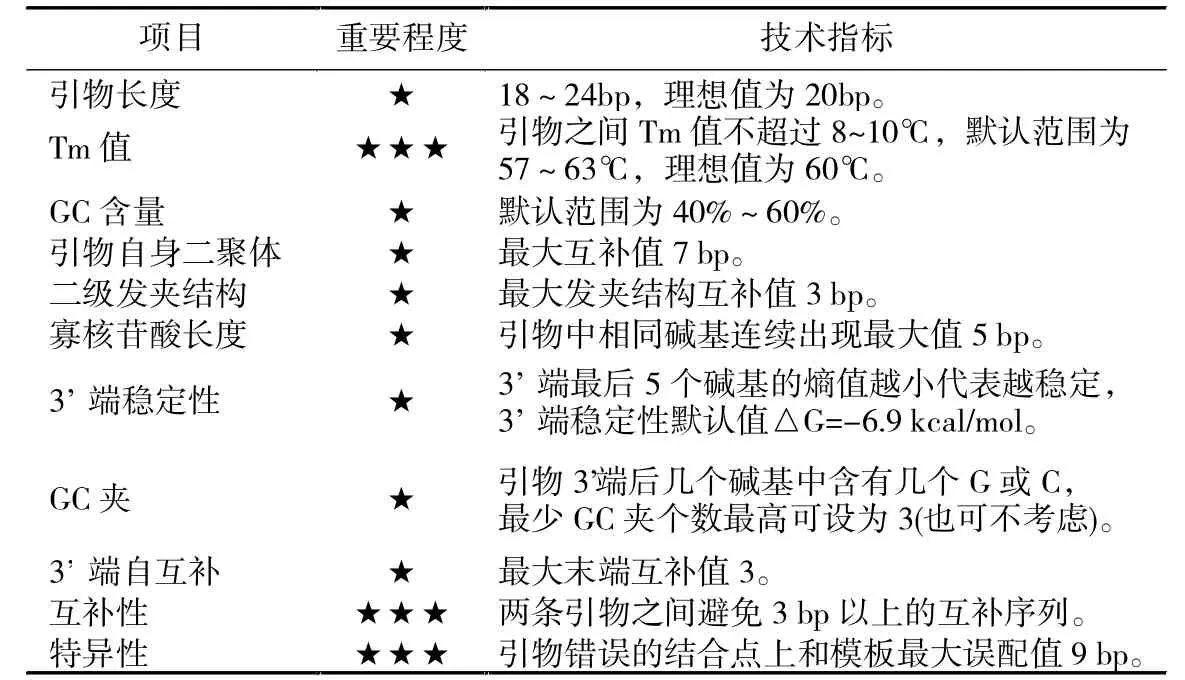
表4 单条引物约束参数
3.1 普通PCR引物的约束参数
根据对文献的总结,对单个引物考虑了如下 11个约束条件 (表 4): 引物长度(Abdelsalam.K.,2003 )、Tm 值(SantaLucia.J.Jr.,1998)、引物 GC 含量(Dieffenbach.C.W.等,1993)、引物自身二聚体、二级发夹结构、寡核苷酸长度、3'端稳定性(Steven.Rozen,2007)、GC 夹(F.John.Burpo,2001)、3'端自互补、互补性、特异性(Lin.W.M.,1993)。
3.2 多重PCR的引物约束参数
在多重PCR反应中,引物的设计至关重要,是影响多重PCR反应成败的关键。在多重PCR反应中,单条引物的长度最好介于18~24 bp,引物太长,容易导致引物之间相互缠绕,严重影响多重PCR反应结果。同时,为了避免引物的非特异性扩增,引物必须高度特异。此外,要充分考虑到不同引物对之间互补的碱基序列,特别是引物间引物二聚体的形成,这将严重影响到引物的有效性。所有的引物对最好有相近的退火温度,引物的碱基构成最好是4种碱基的比例相当,G+C的含量最好介于40%~60%。如果可能的话,引物的序列以1~2个GC开头或结尾,避免3'端以A结尾。将引物设计好后,设计的引物序列应该与数据库中的核苷酸序列进行比对。如果扩增位点非常相似,存在着交叉反应,那么在引物的3'末端至少有1~2个特异性的碱基用于目的基因的特异性扩增。在多重PCR反应中,引物的浓度影响着多重PCR反应的结果。试验结果证明,太高的引物浓度或者太低的引物浓度在多重PCR的反应中都应该避免(Henegariu.O.,1997)。太高的引物浓度或许抑制了多重的反应结果,同时太低的引物浓度很可能是无效的。
根据(Peter.M.V,2004)对多个引物之间,以及引物对,考虑了如下7个约束条件(表5):引物长度差、退火温度差、引物间3'端互补、产物长度、产物间长度差、引物间互补、特异性。

表5 用于多重PCR引物约束参数
3.3 实时定量PCR引物约束参数
对于实时定量PCR有两种类型:(1)染料法。一般用SYBRGreenⅠ染料较为常见,其引物设计参数根据(Moravec.T,2003),引物设计的基本原则是提高扩增的效率和检测特异性。一般遵循以下原则(表6)。(2)荧光探针检测法。要同时考虑引物和探针的质量。通常先满足普通PCR引物的设计要求,然后寻找合适的探针位置,再进行探针的设计,然后再将引物和探针之间进行比对分析(Kalender,2008)。用于实时定量PCR探针技术指标如表6。

表6 用于SYBR GreenⅠ染料法和探针法实时定量PCR引物约束参数

表7 带有酶切位点的引物约束参数
3.4 带有酶切位点的引物约束参数
对于带有酶切位点的引物设计,考虑了6个约束条件(表7):酶切位点、目的序列、酶切位点的位置、酶切效率、引物长度、特异性。一般都是做基因克隆表达,除基本满足一般的引物要求外还有更高的要求。根据实验需要,确定需要扩增的DNA序列,并知道其CDS区序列(编码结构基因区,即从起始密码子区至终止密码子区)可登陆NCBI网站(http://www.ncbi.nlm.nih.gov/)查询。
3.5 影响引物设计成功率的因素
引物设计的优劣,是影响实验结果的重要因素。有些引物反应性能差,可以通过反应条件优化得到改善,但大多数情况改善的效果并不明显,因此,预先设计反应性能好的引物非常重要。针对引发PCR实验失败的三大主要因素(Andreson.R.,2008):(1)引物和模板结合位点数目;(2)引物 GC 含量;(3)预期 PCR 扩增产物。引物设计的关键点主要有三:(1)特异性高:DNA模板上不存在与引物的错配部位或错配效率较低;(2)对引物解链温度Tm值的精确估算是确定PCR反应退火温度的前提条件,决定着实验的效率和精确性(Chavali.S,2005)。因此Tm值和GC含量在适当范围;引物利用效率高:引物内部和上、下游引物之间无互补序列;(3)引物的长度、扩增片段长度等参数也要加以考虑。
引物设计主要借助于生物软件,如:Primer3(Rozen.S.,2000),Primerpremier (Singh.V.K,1998),0ligo(Rychlik.W,2007)等,但其重点在单引物的设计优化上,当中也有个别引物设计软件考虑到了引物之间设计优化,基于不同实验要求,引物设计中,由于实验的复杂性,几乎没有某一个引物设计软件能完美地完全满足要求。因此利用引物设计软件中不同技术指标参数。再加上人为的分析和选择因素不但可以大大提高引物设计的成功率,而且可以不必花费太多时间和精力。经实验总结可以归纳一下,主要有以下三点:第一,模板的因素。尽量避免选择一些困难模板,其中有一些模板本身的GC含量偏高或偏低,导致引物的GC含量不能控制在合适的范围。另外还有一些模板基因家族很大或者与其DNA序列相近的基因很多,导致引物的PCR扩增不特异。这些最终都造成PCR扩增的失败;第二,引物的筛选,运用Primer Premier 5.0设计了一组引物后,经过初次筛选得到几对适合要求的引物,还要在初次筛选的基础上进一步用Oligo6.0验证,然后筛选出能够进行特异高效PCR扩增的引物。
选出理论上较合适的引物以后,可以有以下两个步骤,保证引物设计的成功率:一是要将得到的一系列引物分别在Genebank中进行回检。也就是把每条引物在比对工具http://www.ncbi.nlm.nih.gov/blast/的blast中进行同源性检索,弃掉与基因组其它部分同源性比较高的引物,也就是有可能形成错配的引物。一般连续10bp以上的同源有可能形成比较稳定的错配,特别是引物的3'端应避免连续5~6bp的同源。二是如果以mRNA为模板设计引物时还应注意:首先利用生物信息学的知识大致判断外显子与内含子的剪接位点 (例如http://CCR-081.mit.edu/GENESCAN.html的GENES-CAN工具或者GeneParser软件),然后弃掉正好位于剪接位点的引物,实验证明,按照上述原则设计合成的引物和探针,得到了高扩增效率和稳定的检测结果。
引物设计只是为PCR反应提供一种可能性,实验中由于影响PCR实验的因素较多,有时需要不断摸索条件,引物设计软件只是一种工具,只能为PCR实验设计提供比较可靠的引物,加速最佳条件的摸索,最终达到成功实现PCR实验的目的。
[1]Andreson.R.,Mols.T.,Remm.M.Predicting failure rate of PCR in large genomes[J].Nucleic Acids Res,2008,36(11):66-67.
[2]Abdelsalam.K.Bioinformatic tools and guideline for PCR primer design[J].African Journal of Biotechnology, 2003,2(5):91-95.
[3]Chavali.S.,Mahajan.A.,Tabassum.R.,Bharadwaj.D., Oligonucleotide properties determination and primer designing:a critical examination of predictions.[J].Bioinformatics,2005,21(20):3918-3925.
[4]Dieffenbach.C.W.,Lowe.T.M.,Dveksler.G..S.General concepts for PCR primer design[J].PCR Methods and Appl.,1993,3(3):30-37.
[5]David Clark.Molecular Biology:Understanding the Genetic Revolution[M].U.S.A:ELSEVIER Inc.,2005:52-73,634-661.
[6]F.John.Burpo.A critical review of PCR primer design algorithm and cross-hybridization case study[Z].USA:Stanford University,2001:1-11.
[7]Henegariu.O.,Heerema.N.A.,Dlouhy.S.R.,et al.Multiplex PCR:critical Parameters and Step-by-Step Protocol[J].Biotechniques,1997,23(3):504-511.
[8]萨姆布鲁克J,拉塞尔D W(黄培堂等译).分子克隆实验指南(第3版)[M].北京:科学出版社,2002:597-701.
[9]Kalendar R.L.D.,Schulman A.H.FastPCR Software for PCR Primer and Probe Design and Repeat Search[J].Genes,Genomes and Genomics,2009,3(1):1-14.
[10]林万明.PCR技术操作与应用指南[M].北京:人民军医出版社,1993:25-40.
[11]Moravec.T.,Cerovska.N.,Boonham.N.The detection of recombinant,tuber necrosing isolates of Potato virus Y(PVY(NTN))using a three-primer PCR based in the coat protein gene[J].J.Virol.Methods,2003,109(1):63-68.
[12]Peter.M..V.,John.M.Butler.AutoDimer:a screening tool for primer-dimer and hairpin structures[J].BioTechniques,2004,37(2):226-231.
[13]Rozen.S.,Skaletsky.H.Primer3ontheWWWforgeneralusersandforbiologistprogrammers[J].MethodsMol.Biol.,2000,132:365-386.
[14]Rychlik.W.OLIGO7 primer analysis software[J].Methods Mol.Bio.,2007,402:35-60.
[15]SantaLucia.J.,Jr.A unified view of polymer,dumbbell,and oligonucleotide DNA nearest-neighbor thermodynamics[J].Proc Natl Acad Sci USA,1998,95(4):1460-1465.
[16]Steven.Rozen.,Helen.Skaletsk.Primer3 Input Help[Z].USA:Whitehead Institute for Biomedical Research,MIT,2007.
[17]Singh.V.K.,Mangalam A.K.,Dwivedi.S.,Naik.S.Primer premier program for design of degenerate primers from a protein sequence[J].Biotechniques,1998,24(2):318-319.
[18]余爱丽.斑茅抗逆性评价及其BADH基因的克隆表达[D].福建农林大学,2004:27-28.

