华东部分地区猪群中猪萨佩罗病毒分子流行病学调查
兰道亮,吉文汇,王长松,孙 焕,陈莫林,崔 立,童光志,华修国*
(1.上海交通大学农业与生物学院 上海市兽医生物技术重点实验室,上海200240;2.中国农业科学院上海兽医研究所,上海200241)
猪萨佩罗病毒(Porcine sapelovirus,PSV)是微球形,无囊膜包裹的一类RNA病毒,属小RNA病毒科,由于最初从腹泻猪的肠道中分离得到,所以先前统属于肠道病毒属中的猪肠道病毒Ⅱ群,称为猪肠道病毒8型(PEV8)[1]。近年来,由于对肠道病毒基因组及进化关系的深入研究,该病毒已单独划分为萨佩罗病毒属[2]。PSV可引起猪脑脊髓灰质炎、肺炎、繁殖障碍、心包炎、心肌炎、皮肤损伤、腹泻等多系统综合征,同时也可出现无明显特征的亚临床症状,但无论其以何种形式暴发,都会给养猪业带来威胁。该病最早于20世纪60年代在英国发现[3],随后在北美、日本、澳大利亚等世界范围内的其他国家相继报道[4-8],而且在饲养猪群中感染率很高[9-10]。
近年来,随着规模化养猪业的发展及畜产品流通渠道的增多,我国猪群中肠道、呼吸道、生殖器官疾病以及脑脊髓炎等多系统综合征频繁发生,长期以来猪圆环病毒2型(PCV-2)、猪繁殖与呼吸综合征病毒(PRRSV)、猪细小病毒(PPV)、猪捷申病毒(PTV)被认为是引起该类疾病的主要病原体[11],但PSV这个经典肠道病毒却一直没有引起相应的重视。目前为止,我国还未见对PSV的系统性研究报道。但鉴于PSV也可引起类似的临床症状且可混合感染,使我们不得不考虑PSV这一感染因素。目前,猪病的发生越来越复杂,在控制重点传统病的同时,对于一些我国还没有开展研究或研究力度不够的传染病进行深入的研究也十分必要。因此,在我国开展PSV的研究工作,了解PSV流行情况,建立快速的病原学诊断方法,具有重要的意义。本研究主要通过设计的PSV鉴定引物,应用RT-PCR方法对华东地区猪群中PSV进行调查,通过序列比对分析其遗传进化关系,为了解该病毒在华东地区的分布情况提供基础数据。
1 材料与方法
1.1 材料
1.1.1 样本采集 2009年10月至2010年12月,从上海、江苏、安徽等地的15个大中型养猪场(200头~1 000头母猪)共采集1月龄~5月龄临床健康猪粪便样本960份,其中从上海市崇明、浦东、闵行、南汇、金山区的6个猪场采集350份,从江苏省宿迁市、盐城市、镇江市、苏州市的4个猪场采集310份,从安徽省宿州市、阜阳市、滁州市、黄山市的5个猪场采集300份(表1),采集的新鲜猪粪便样品立即置-80℃冰箱冻存。
1.1.2 主要试剂及仪器 Trizol为Invitrogen公司产品;Prime Script 1st Strand cDNA Synthesis Kit pMD-18T载体为宝生物工程(大连)有限公司产品;PCR Mix、AxyPrep DNA凝胶回收试剂盒为上海前尘生物技术有限责任公司产品;DH5αE.coli为天根生化公司产品。PCR仪PX2为美国Thermo Hybaid公司产品;核酸电泳仪Powerpac universalTM为美国Bio-Rad公司产品。

表1 采样地区及样品数量Table 1 The areas and numbers of specimen
1.2 方法
1.2.1 样本的处理及总RNA的提取 取在-80℃保存的样品,冰上溶化。用PBS液将样品按照1∶10的比例稀释,涡旋振动5min。在4℃条件下以12 000r/min离心10min,取上清。处理后的粪便上清供下游提取遗传物质。然后取100μL粪便上清,利用Trizol法提取总RNA,具体操作方法按照Invitrogen产品说明书提取步骤进行。
1.2.2 RT-PCR检测 参考Palmquist J M等[12]设计的PSV 5′保守区的通用检测引物,扩增目的片段为 163bp。上游 引 物 PSV1:5′-GTGGCGACAGGGTACAGAAGAG-3′;下游引物 PSV-2:5′-GGCCAGCCGCGACCCTGTCAG-3′。以提取的样本RNA为模板,按照Prime Script 1st Strand cDNA Synthesis Kit的说明书进行反转录,合成cDNA。反应参数为:42℃30min,85℃5s。然后取10μL反转录产物进行PCR反应,反应体系为:模板10μL,PCR mix 25μL;上游引物1μL;下游引物1μL;ddH2O为13μL。反应条件为:94℃3min;94℃30s,56℃40s,72℃40s,35个循环;72℃5 min,4℃结束反应。
1.2.3 PSV RdRp基因及VP1基因序列扩增 参考GenBank中已有PSV基因序列,自行设计扩增RdRp及VP1基因全长引物。RdRp基因扩增引物:上游引物 RdRp-F:5′-GCAGCCCTGTTGAAGAGAGACT-3′;下游引物RdRp-R:5′-ATA TGTTCACGCCGCCTAAAA-3′;VP1基因扩增引物:上游 引 物 VP1-F:5′-ATTGCCTAYACACCACCTGG-3′;下 游 引 物 VP1-R:5′-GCAGGTCTTCTCCCACAAAC-3′;参照1.2.2所述方法,对 PSV RdRp基因及VP1基因全序列进行RT-PCR扩增。
1.2.4 序列的克隆及测序 扩增后的PCR产物利用10g/L和50g/L凝胶电泳进行检测。特异性目的条带经割胶、纯化回收后连接pMD-18T载体,随后进行T/A克隆,挑取阳性重组质粒,送上海生工生物工程技术服务有限公司测序。
1.2.5 系统进化分析 本试验所得序列及其参考序列均整理成Fasta格式,通过ClustaXv1.8比对后保存生成的文件采用Mega 4.0系统进化分析软件构建系统进化树。本试验中使用的小RNA科不同种属病毒名称及参考株序列号如下:
AiV,aichivirus(FJ890523);ASV,avian sapelovirus(NC-006553);BEV,bovine enterovirus(AF123432);EMCV,encephalomyocarditis virus(NC-001479);PEMCV,porcine encephalomyocarditis virus(DQ835184);ERAV,equine rhinitis A virus(NC-003982);ERBV,equine rhinitis B virus(NC-003077);FMDV,foot-and-mouth disease virus (GU384683); HAV,hepatitis A virus(EU526088); HEV, human enterovirus(AB678778,HM185056,JN542510,AB601882);HPeV,human parechovirus(AB668033);HRV,human rhinovirus(JN990704,JN614997);LjV,ljungan virus(NC-003976);PEV,porcineenterovirus(Y14459,AF363455);PKV,porcine kobuvirus (GU298977);PSV,porcine sapelovirus(NC-003987,AY392543,AY392556,AY392544,AY392538);PTV,porcine teschovirus (NC-003985);SHAV,simian hepatitis A virus(SHVAGM27);SV,simian sapelovirus(EU789367)。
2 结果
2.1 PSV检测结果
960份临床粪便总样本,经RT-PCR检测,共有165份样本为PSV阳性样本,总体阳性率为17.2%。各地区PSV阳性率如表2所示,其中安徽地区的PSV阳性感染率要明显高于上海和江苏地区(P<0.05)。进一步分析不同年龄段猪的PSV阳性率,我们发现10周龄~20周龄的猪PSV阳性率最高,达到25.6%,大于20周龄的猪PSV阳性率最低为11.6%(表3),这表明10周龄~20周龄的猪的PSV感染率要显著高于其他周龄的猪(P<0.05)。

表2 华东地区15个猪场PSV的流行情况Table 2 Prevalence of PSV in 15farms located in East China

表3 不同年龄段猪的粪便样本中PSV检测结果Table 3 The prevalence of PSV in swine feces of different ages
2.2 系统进化分析
2.2.1 RdRp及VP1基因序列分析 从上海、江苏、安徽三省市的阳性样本中,各随机选取8个样本,扩增其RdRp基因及VP1基因全序列,并测序。然后,对这选取的24株PSV中国华东株分别进行RdRp基因及VP1基因的同源性比对。结果表明,这24株PSV中国华东株的RdRp基因间的同源性为87.0%~99.9%,同源性较高,而VP1基因间的同源性为72.4%~99.4%。
2.2.2 以RdRp基因为基础的系统进化分析 利用ClustaX v1.8,将24株PSV中国华东株的RdRp基因序列与小RNA病毒科其他代表毒株的RdRp基因序列进行核苷酸比对,然后使用Mega 4.0系统进化分析软件构建系统进化树(图1),本研究分离的毒株与GenBank中已有的PSV V13株聚为一类,且与SV-2和ASV同属于萨佩罗病毒属,而与小RNA病毒科的其他成员相距较远。说明本研究分离的毒株为PSV,符合ICTV的分类条件。
2.2.3 以VP1基因为基础的系统进化分析 将24株PSV中国华东株的VP1基因序列与微RNA病毒科其他代表毒株的VP1基因序列进行核苷酸比对,然后构建系统进化树(图2),上海、江苏和安徽三省市的毒株交错分布在一起,不存在明显的地区差异,但大体可分为3个亚群,其中7株与Gen-Bank中已有的PSV V13、16-S-X、26-T-XII株聚为一个亚群,3株与GenBank中已有的PSV Sek1562/98、Po 5116株聚为一个亚群,而剩下的又聚为一个亚群。
3 讨论
PSV很早便于英国发现,随后在北美、日本、澳大利亚等世界范围内的其它国家相继有报道,而且在正常饲养猪群中感染率超过20%[9]。PSV可引起猪肠道、呼吸道、生殖器官疾病以及脑脊髓炎等多系统综合征,同时也可出现无明显特征的亚临床症状,所以在疾病检测和防控方面容易被忽略。目前,我国尚未对PSV开展深入研究,而且PSV在我国的流行情况还不十分清楚。
本研究通过RT-PCR方法对中国华东地区猪群中PSV的感染率进行了分子流行病学调查,从上海、江苏、安徽三省市的13个地区收集的960份临床健康猪粪便样本中,共检测到PSV阳性样本165份,总体阳性率17.2%,并且在所检测的13个地区均检测到该病毒,这表明PSV在华东地区普遍存在。在所检测的3个省市中,安徽省猪群平均感染率最高为22.3%,上海市猪群平均感染率最低为13.1%,在所检测的13个地区中,情况类似,安徽省的阜阳地区猪群感染率最高为25.3%,上海市的崇明地区猪群感染率最低为10.0%,而江苏省地区猪群的感染率介于二者之间。造成不同地区的感染率差异的原因可能与地区间的养殖状况以及饲养管理方式有关。进一步分析不同年龄段猪的PSV阳性率,我们发现10周龄~20周龄的猪PSV阳性率最高,达到25.6%,表明在此年龄段的仔猪最易感,这与PEV的感染状况类似[12-13]。我国仔猪的哺乳期在一般为4周~9周,由于哺乳期仔猪受到母源抗体保护,对PSV具有一定的抵抗力,而相关抗体在断奶3周后降至最低[14-15],所以易于受到PSV感染。这也提示加强断奶仔猪,尤其是10周龄~20周龄仔猪的饲养管理,对于防控PSV有重要意义。总体来说,我国华东地区猪群中PSV的感染流行率,与国外报道的情况基本相同。但临床健康猪群中20%以上的感染率不能不视其为危害养猪业的重要潜在威胁。
随后从上海、江苏、安徽三省市的阳性样本中,随机选取24个样本,扩增其RdRp基因及VP1基因全序列,进行相关系统进化分析。以RdRp基因为基础构建进化树,分析其与小RNA病毒科其他种属之间的关系,结果表明本研究分离的毒株与已有的PSV株归为一类,且与SV-2和ASV同属于萨佩罗病毒属,而与小RNA病毒科的其它成员相距较远。由于RdRp基因在小RNA病毒科各种属成员中具有较高的保守性,成为区分小RNA病毒科中不同种属成员的有力依据。同时,从RdRp基因进化树中也可发现萨佩罗病毒属与肠道病毒属各归为一类,存在一定的遗传距离,这与国际病毒分类委员会将PSV从原先的PEV肠道病毒属中单独划分出来的分类相符。进一步以PSV的外层衣壳蛋白基因VP1为基础构建进化树,分析24株PSV中国华东株间的进化关系。由于外层衣壳蛋白VP1在小RNA病毒科各种属间具有多变性及良好的抗原性,所以被认为是划分小RNA病毒科各种属间不同亚型的有力依据[9,16]。VP1基因进化树分析表明上海、江苏和安徽三省市的毒株交错分布在一起,不存在明显的地区差异,但大体可分为3个亚群,而且已有的国外PSV均包括在这3亚群中。目前,PSV只有1个血清型,但PSV是否存在不同的基因亚型尚不清楚。Tseng C H等[2]证实PSV存在3种类型的抗原变异株,与本研究的进化分析结果相似,这提示PSV可能至少存在3种不同的抗原基因亚型。但关于PSV的亚型分型国际上尚无定论,还需进一步深入研究加以证实。
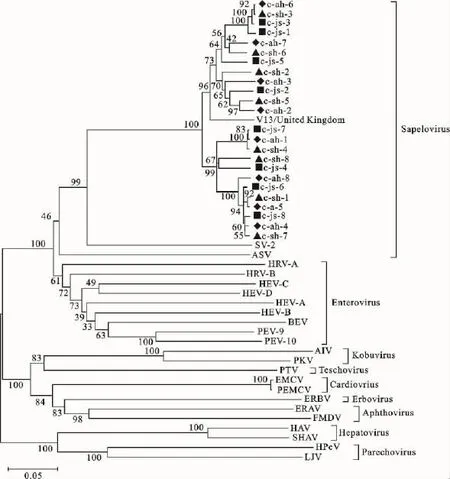
图1 基于RdRp基因序列构建的系统进化树Fig.1 The phylogenetic tree constructed based on the RdRp gene sequence

图2 基于VP1基因序列构建的系统进化树Fig.2 The phylogenetic tree constructed based on the VP1gene sequence
[1] Kaku Y,Sarai A,Murakami Y.Genetic reclassification of porcine enteroviruses[J].J Gen Virol,2001,82(Pt 2):417-424.
[2] Tseng C H,Tsai H J.Sequence analysis of a duck picornavirus isolate indicates that it together with porcine enterovirus type 8and simian picornavirus type 2should be assigned to a new picornavirus genus[J].Virus Res,2007,129(2):104-114.
[3] Lamout P H,Betts A O.Studies on enteroviruses of the pig.IV.The isolation in tissue culture of a possible enteric cytopathogenic swine orphan(ECSO)virus(V13)from the faeces of pig[J].Res Vet Sci,1960(1):152-59.
[4] L'Ecuyer C,Greig A S.Serological and biological studies on porcine enteroviruses isolated in Canada[J].Can Vet J,1966,7(7):148-154.
[5] Dunne H W,Kradel D C,Clark C D,et al.Porcine enteroviruses:a serologic comparison of thirty-eight Pennsylvania isolates with other reported North American strains,Teschen,Talfan,and T80serums-aprogress report[J].Am J Vet Res,1967,28(123):557-568.
[6] Morimoto T,Watanabe M.Serological identification of porcine enteroviruses isolated in Japan [J].Natl Inst Anim Health Q(Tokyo),1964,4:177-182.
[7] Dunne H W,Wang J T,Ammerman E H.Classification of North American porcine enteroviruses:a comparison with European and Japanese strains[J].Infect Immun,1971,4(5):619-631.
[8] Forman A J,Pass D A,Connaughton I D.The characterisation and pathogenicity of porcine enteroviruses isolated in Victoria[J].Aust Vet J,1982,58(4):136-142.
[9] Sozzi E,Barbieri I,Lavazza A,et al.Molecular characterization and phylogenetic analysis of VP1of porcine enteric picornaviruses isolates in Italy[J].Transbound Emerg Dis,2010,57(6):434-442.
[10] Buitrago D,Cano-Gomez C,Aguero M,et al.A survey of porcine picornaviruses and adenoviruses in fecal samples in Spain[J].J Vet Diagn Invest,2010,22(5):763-766.
[11] Yi J,Liu C.Molecular characterization of porcine circovirus 2isolated from diseased pigs co-infected with porcine reproductive and respiratory syndrome virus [J].Virol J,2010(7):286.
[12] Palmquist J M,Munir S,Taku A,et al.Detection of porcine teschovirus and enterovirus type II by reverse transcriptionpolymerase chain reaction[J].J Vet Diagn Invest,2002,14(6):476-480.
[13] Zell R,Krumbholz A,Henke A,et al.Detection of porcine enteroviruses by nRT-PCR:differentiation of CPE groups IIII with specific primer sets[J].J Virol Meth,2000,88(2):205-218.
[14] McCormick B M,Driesen S J,Connaughton I D,et al.Prevalence of enteroviral and parvoviral antibodies in pig sera[J].Res Vet Sci,1986,41(3):397-401.
[15] Cropper M,Dunne H W,Leman A D,et al.Prevalence of antibodies to porcine enteroviruses and porcine parvovirus in body fluids of fetal pigs from small vs large litters[J].J Am Vet Med Assoc,1976,168(3):233-235.
[16] Huang J,Gentry R F,Zarkower A.Experimental infection of pregnant sows with porcine enteroviruses[J].Am J Vet Res,1980,41(4):469-473.
[17] Krumbholz A,Dauber M,Henke A,et al.Sequencing of porcine enterovirus groups II and III reveals unique features of both virus groups[J].J Virol,2002,76(11):5813-5821.

