河南汉族人群6q24-25.1区域STR基因座的遗传多态性
张建勋,乐晓萍,杨 洋,朱运良
郑州大学基础医学院法医学教研室郑州450001
(2010-11-04收稿 责任编辑 李沛寰)
微卫星DNA又叫短串联重复序列(short tandem repeat,STR),作为第二代遗传标记,在上世纪80年代末期之后广泛应用于疾病基因的定位克隆,其在肿瘤细胞染色体不稳定性的研究、人类及其基因组的进化和法医学等方面也具有不可替代的作用[1-2]。许多肿瘤细胞在6号染色体长臂上发生杂合性缺失(loss of heterozygosity,LOH)。6q21、6q23-24、6q24-25、6q27 等是几个 LOH 的热点区域[3-6]。为了进一步精细定位这些缺失区域,需要更多的相距更近的STR基因座。作者对河南汉族人群6q24-25.1区域共11 Mb范围内STR基因座的多态性进行研究,为其进一步在法医学和复杂疾病基因的定位等方面的应用提供依据。
1 对象与方法
1.1 研究对象 120例河南汉族无关个体的血液样本采集于2008年5月,来自郑州市中心血站。其中男65例,女55例。
1.2 微卫星的查找和引物的设计 从GenBank上下载6q24-25.1区域的DNA,序列从139 000 000~150 000 000,用Tandem Repeats Finder在线软件在这段DNA序列上共查找到了14个STR基因座,平均间距为700 kb,重复单位为四核苷酸,这14个STR基因座的基本参数见表1。再用Primer3软件设计这些STR基因座的PCR引物,使扩增产物大小在300 bp以内,引物序列见表2。引物由上海生工生物工程技术服务公司合成。
1.3 PCR扩增 取血样,采用酚-氯仿法提取DNA,-20℃保存。扩增体系20μL,内含DNA模板1 ng,上下游引物各 1.5 μL(5 μmol/L),dNTP 0.4μL(10 mmol/L),Taq聚合酶 1 U,10 ×Buffer(含 Mg2+1.5 mmol/L)2 μL,ddH2O 12.6 μL。扩增条件:95℃预变性2 min;94℃ 35 s,62℃ 45 s,72℃ 40 s,每一循环退火温度降低0.5℃,共12个循环;然后94 ℃ 35 s,55 ℃ 45 s,72 ℃ 40 s,20个循环;最后72℃ 5 min,12℃保存。采用6 g/L非变性聚丙烯酰胺凝胶电泳,用银染法显示电泳条带。然后检测等位基因片段大小,用阿拉伯数字按从小到大的顺序分别表示大小不同的等位基因。

表1 14个STR位点的基本参数

表2 14个STR基因座的引物序列
1.4 统计学处理 采用PowerStatsV12.xls(美国Promega公司)对14个STR基因座进行分析,可得到如下常用参数:多态信息含量(polymorphic information content,PIC),杂合度(heterozygosity,H),个体识别率(discrimination power,DP),匹配概率(match probability,MP),非父排除率(excluding probability of paternity,PE),父权指数(paternity index,PI)。
2 结果
2.1 等位基因及其基因频率 120个无关个体6号染色体的14 个 STR 基因座分别检出5、6、2、3、6、6、7、4、7、4、4、5、4、4 个等位基因,各等位基因频率见表3。
2.2 14个STR基因座的群体遗传学参数 14个STR基因座的群体遗传学参数见表4。结果显示:D6SZ2、D6SZ6、D6SZ7、D6SZ8、D6SZ9 和 D6SZ12 这6个STR基因座的H、PIC和DP较高。
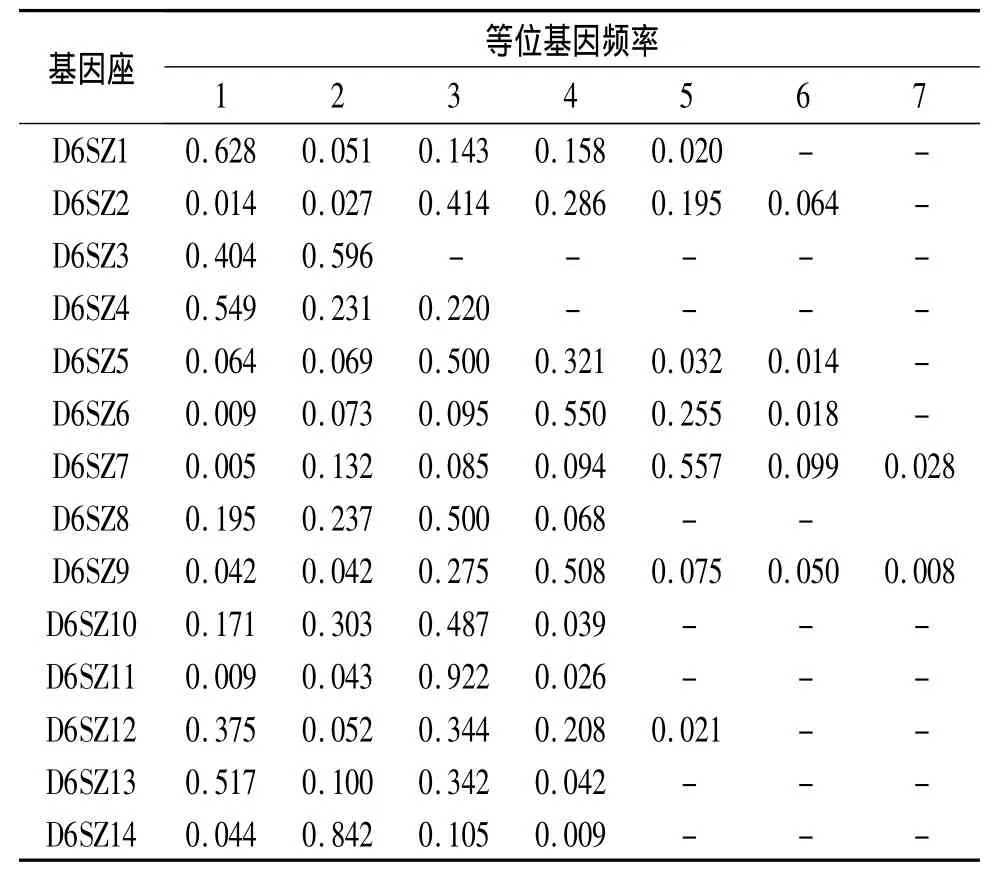
表3 14个STR基因座等位基因频率(n=120)
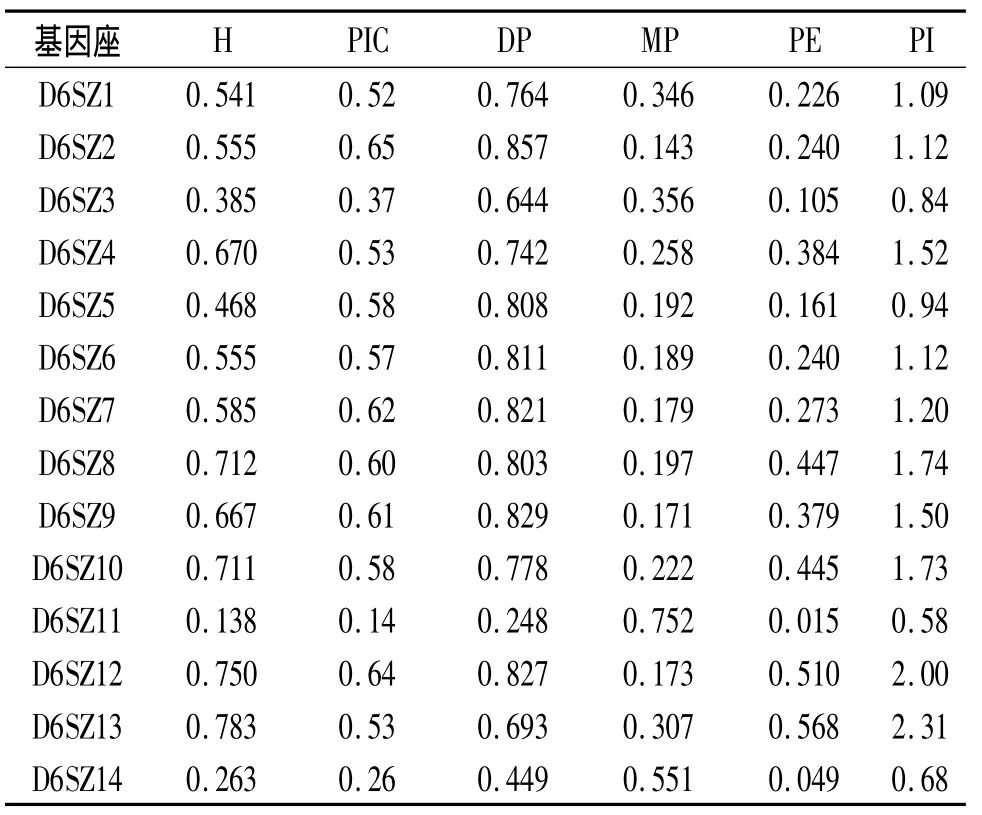
表4 14个STR基因座的群体遗传学参数(n=120)
3 讨论
人类肿瘤的发生是一个多步骤多基因异常积累的过程,包括原癌基因的激活和抑癌基因的失活。抑癌基因失活的经典机制是Kundosn的二次打击学说,可以用LOH来发现和定位新的候选抑癌基因。6q24-25区域是多种肿瘤 LOH 的热点区域[3,7]。该研究在人类基因组6q24-25.1区域中共查找到了14个STR基因座,通过对河南汉族120例健康无关个体群体调查,得到了河南汉族人群这14个基因座的群体遗传学数据。其中 D6SZ2、D6SZ6、D6SZ7、D6SZ8、D6SZ9和D6SZ12这6个STR基因座的PIC为0.57 ~0.65(>0.50),DP 为 0.803 ~0.857(>0.80),说明这6个STR基因座是一组良好的遗传标记,适用于法医学个体识别和亲子鉴定。为将这些遗传标记更好的用于法医学实践,还需进一步评估这些STR基因座在6号染色体是否存在连锁不平衡,以便发现目的基因进行基因诊断[8]。
作者挑选四核苷酸重复的微卫星。与二核苷酸及三核苷酸重复微卫星相比,四核苷酸重复微卫星PCR扩增时不容易产生影子带。微卫星的平均距离为700 kb。再加上数据库中已公布的此区域的微卫星,就可以得到更为精细的微卫星图,从而为此区域复杂疾病基因的定位候选克隆提供更为精细的物理图。
对缺失区域的精细分析有助于了解缺失的机制及其与肿瘤发生的关系。hZAC基因是一个定位于6q24.2区域的新的候选抑癌基因[7]。有一个频繁发生的区域被限定在hZAC基因周围的D6S292-D6S310-D6S311之间,此区域已在乳癌中被鉴定[9]。根据人类基因组图谱可知hZAC定位于6q24区域的D6S308和D6S978之间,物理位置为144 261 437~144 385 735 bp,恰好在该报道的D6SZ7和D6SZ8之间。作者希望用新查找到的这些高密度的多态性的微卫星标记将此区域定位于更精确的范围,为进一步精确分析断裂点打下基础。
[1] Xiong M,Jin L.Comparison of the power and accuracy of biallelic and microsatellite markers in population-based genemapping methods[J].Am J Hum Genet,1999,64(2):629
[2] Payseur BA,Place M,Weber JL.Linkage disequilibrium between STRPSand SNPSacross the human genome[J].Am J Hum Genet,2008,82(5):1039
[3] Lemeta S,Salmenkivi K,Pylkkanen L,et al.Frequent loss of heterozygosity at 6q in pheochromocytoma[J].Hum Pathol,2006,37(6):749
[4] Bignone PA,Lee KY,Liu Y,et al.RPS6KA2,a putative tumor suppressor gene at 6q27 in sporadic epithelial ovarian cancer[J].Oncogene,2007,26(5):683
[5]康睿,曹励之,俞燕,等.中国儿童急性淋巴细胞性白血病染色体6q16.3-21区域候选肿瘤抑制基因的定位与鉴定[J].生物化学与生物物理研究所研究进展,2006,33(1):65
[6] Valleley EM,Cordery SF,Carr IM,et al.Loss of ZAC/PLAGL1 in diffuse large B-cell lymphoma is independent of promoter hypermethylation[J].Genes Chromosomes Cancer,2010,49(5):480
[7] Cvetkovic D,Pisarcik D,Lee C,et al.Altered expression and loss of heterozygosity of the LOT1 gene in ovarian cancer[J].Gynecol Oncol,2004,95(3):449
[8]府伟灵.基因诊断治疗与预测医学[J].第三军医大学学报,2009,31(1):24
[9] Abodollahi A,Pisarcik D,Roberts D,et al.LOT1(PLAGL1/ZAC1),the candidate tumor suppressor gene at chromosome 6q24-25,is epigenetically regulated in cancer[J].JBiol Chem,2003,278(8):6041

