乳酸菌蛋白代谢研究进展
白凤翎,张柏林,赵宏飞
(1.渤海大学生物与食品科学学院,辽宁 锦州 121000;2.北京林业大学生物科学与技术学院,北京 100083)
乳酸菌蛋白代谢研究进展
白凤翎1,张柏林2,赵宏飞2
(1.渤海大学生物与食品科学学院,辽宁 锦州 121000;2.北京林业大学生物科学与技术学院,北京 100083)
由于乳酸菌缺乏各种生物合成途径,不能合成生长必需的一些氨基酸、维生素等物质,营养要求也十分苛刻。乳酸菌拥有胞外蛋白酶,水解乳中酪蛋白形成寡肽和氨基酸,通过寡肽转运系统Opp、二肽和三肽转运系统DtpP和DtpT将水解产物运输进入细胞,胞内的各种肽酶裂解形成游离氨基酸供细胞合成代谢,满足乳酸菌的生长需要。乳酸菌蛋白代谢研究对提高乳酸菌的生长水平、建立高密度培养体系以及促进奶酪等乳制品发酵过程中风味物质及其风味前体物质的形成研究具有重要意义。
乳酸菌;蛋白质;代谢
乳酸菌是一类革兰氏阳性球状、球杆状和杆状细菌,能够在厌氧或兼性厌氧条件下发酵己糖产生乳酸,形成与人类生活密切相关的发酵产品。由于乳酸菌属于分类地位差异性很大的细菌类群,对乳酸菌生理代谢进行研究开展比较缓慢。深入了解乳酸菌这一特殊类群细菌的生物学特征,对提高乳酸菌代谢水平、开发乳酸菌发酵产品具有非常重要的理论和实际应用价值。
自然环境中乳酸菌能够很好地利用栖息地中的物质生长,与乳中乳酸菌能够很好地适应利用乳糖作为碳源和能源一样,乳酸菌也很好地应用酪蛋白作为氮源。乳酸菌不能同化无机氮源,因此,它们必须降解蛋白质和多肽来满足细胞合成代谢对氨基酸的需要。1974年Mckay等[1]首先证明了乳酸菌必须依靠自身蛋白水解系统降解乳中酪蛋白才能满足其生长的需要,他们研究发现,乳酸乳球菌蛋白酶基因质粒的治愈菌株不能在乳中进行高密度生长,如果在乳中添加水解酪蛋白,则质粒治愈菌株就会同正常菌株一样生长。
乳中乳酸菌已经进化具有降解大分子酪蛋白为小分子片段的高效系统,能够满足在乳中生长对氨基酸的需要。该蛋白水解系统包括三组主要成分:第一组为将大分子酪蛋白水解成多肽的胞外蛋白酶;第二组是将多肽转运进入细胞的多个转运体系中的一个系统;第三组包含将转运进入细胞多肽进一步水解形成自由氨基酸的多种肽酶(图1),这些氨基酸最终进行代谢或合成蛋白质。本文对乳酸菌蛋白代谢的研究进展进行综述,对深入了解掌握乳酸菌的蛋白代谢机制,提高乳酸菌的生长水平和建立高密度培养体系具有借鉴作用。同时,乳酸菌蛋白代谢研究也可为奶酪等乳制品发酵过程中风味物质及其风味前体物质的形成方面提供理论基础。
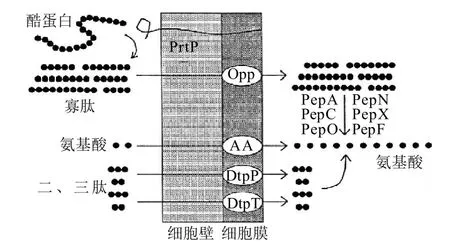
图1 乳球菌的酪蛋白水解系统[1]Fig.1 Proteolytic system in Lactococci
1 蛋白酶系统
虽然人们认为乳酸菌可能具有各种各样的蛋白水解系统,但是被广泛研究的且大多数乳酸菌都拥有的是乳酸乳球菌的蛋白水解系统。对于乳酸乳球菌和其他乳中生长的乳酸菌而言,酪蛋白是氨基酸态氮的主要来源,然而乳中非蛋白氮和自由氨基酸是有限的(<300mg/L),随着乳酸菌生长很快就被消耗掉。乳酸乳球菌属于复合氨基酸自养型微生物,需要尽可能多的氨基酸,因此酪蛋白水解对其生长是十分必要的。乳酸乳球菌拥有一个大的、没有活性的蛋白酶原表达的与细胞膜结合的丝氨酸蛋白酶(PrtP)(>200kD)。如果对蛋白质跨过细胞膜有直接作用的前导序列被除去的话,则剩余蛋白就被锚定在细胞壁上,然而,该蛋白酶原只有被成熟蛋白(PrtM)加工后才具有活性。PrtM可能是将PrtP自动降解成小片段,导致酶对蛋白区的水解,形成成熟的分子质量为180~190kD 的PrtP。
不同的乳酸乳球菌(Lactococcus lactis)菌株中拥有相类似的蛋白酶,然而它们的氨基酸序列具有小的差异。乳酸乳球菌中特殊的酪蛋白底物和PrtP水解产物不尽相同。例如,PⅢ型A群蛋白酶水解αs1-酪蛋白、β-酪蛋白和κ-酪蛋白,而PⅠ型E群蛋白酶具有较强的水解β-酪蛋白活性,对αs1-酪蛋白和κ-酪蛋白水解能力较弱。进一步而言,乳酸乳球菌中PrtP和PrtM系统的功能性差异较小。乳酸菌在乳中迅速生长二者都是必需的,当细胞在低含量肽培养基中生长时,基因PrtP和PrtM被诱导表达,而在肽丰富的培养基中则被抑制[2]。
来自于PrtP对β-酪蛋白的水解作用有超过100多种酪蛋白水解产物,这些产物大多数是4到10个氨基酸残基大片段的大分子寡肽(4~30 氨基酸),没有形成游离氨基酸、二肽和三肽[2]。由PrtP形成首要的也是最丰富的寡肽是由β-酪蛋白的C端产生的,似乎最初的水解作用引起酪蛋白打开,导致其他的裂解片段暴露出来[3]。
2 氨基酸和多肽转运系统
虽然有学者认为在多肽被转运前胞外蛋白酶一定参与降解多肽的过程,但到现在为止,由PrtP形成的多肽水解体系还没有发现,至少也没发现多肽酶。相反,乳酸乳球菌和其他乳酸菌拥有各种各样的氨基酸和多肽转运系统,能够转运大小、极性和结构不同的底物。这些转运系统有些是具有高特异性的,而有些的特异性相对较差,同时在转运过程中也消耗能量。
如前所述,乳中氨基酸的浓度太低不能支持乳酸菌的生长需要。乳酸乳球菌至少有10种氨基酸转运子,大多数转运子具有结构相似性,其底物具有特异性。如果培养基中含有足够的游离氨基酸,这些转运系统能够使足够的氨基酸进入细胞内,支持细胞生长。然而,这暗示了转运子的首要功能是从细胞质中分泌或流出过多的氨基酸来维持适当的细胞内氨基酸池的比例[4]。如果多肽是氨基酸的必需、首要氮源的话,那么一些由细胞内多肽产生的氨基酸可能积累比同化吸收快些,这些游离氨基酸能够通过氨基酸转运子以相反或流出的方向扩散到细胞外降低它们的浓度差。如果氨基酸的流出是伴随偶合离子的话(如质子扩散),则获得质子动力(proton motive force,PMF)的增加值。氨基酸的扩散提供能量甚至有可能驱动多肽的吸收[4]。
与氨基酸转运子具有鲜明对比的是多肽转运系统,它们能够很好地满足乳酸菌在乳中生长的需要。已经确证有三组多肽转运系统,包括转运二肽和三肽的两组DtpP和DtpT和转运寡肽的寡肽转运系统Opp。DtpT是一个含有463个氨基酸组成的依赖PMF转运大单体,与水溶性多肽具有亲和力。已经被获得去掉dtpT基因的突变菌株不能表达DtpT和转运一些多肽。在化学限定培养基上dtpT基因缺陷菌株生长很差。然而,这些突变菌株在乳中的生长不受影响,表明DtpT 在乳中不是必需的。乳酸菌的另一个转运二肽和三肽的DtpP转运系统是一种ATP依赖性转运系统,对水溶性多肽具有高度亲和性,对乳球菌在乳中生长也不是必需的[5]。
乳酸菌中第3种也是最重要的是寡肽转运系统Opp。由于二肽和三肽都不是由酪蛋白释放的,因此对乳酸菌在乳中生长DtpP和DtpT都不是必需的,乳酸乳球菌依赖寡肽和Opp来满足所有氨基酸的需要。实验证明不能表达编码Opp系统的基因突变菌株不能转运多肽,也不能在乳中生长[6-7]。尽管最初还不知道寡肽是由Opp转运的,乳酸乳球菌乳酸亚种中Opp系统的很多结构和遗传学特征已很好地被研究清楚[8]。Opp复合体属于转运系统中的ABC(ATP结合盒)家族,它含有5个亚基:包括两个转运膜蛋白OppB和OppC,两个ATP结合蛋白OppD和OppF和1个膜连接底物结合蛋白OppA。编码Opp的5个基因形成oppDFBCA的操纵子,一个编码肽酶PepO的基因紧接着也定位OppA 的下端,和Opp操纵子有共同转录区[7]。
虽然Opp系统可以转运多种多样的寡肽,在PrtP从β-酪蛋白释放出100多种多肽中,只有10~14种多肽作为Opp的底物。所有这些寡肽含有4~11个氨基酸残基[3],进一步分析它们含有高水平的缬氨酸、脯氨酸和谷氨酸,中等水平的丙氨酸、亮氨酸、异亮氨酸、赖氨酸和丝氨酸。十分重要的是这些寡肽提供除了组氨酸以外的所有必需氨基酸供乳球菌在乳中生长需要。
3 肽 酶
蛋白质代谢的第三步即最后一步是Opp积聚的多肽水解成片段。来自乳酸乳杆菌和乳酸乳球菌20多种不同的肽酶的生化和遗传学特征已经研究清楚(表1)。内肽酶(裂解中间多肽键)和外肽酶(裂解末端肽键)广泛分布,后者中氨基肽酶已经报道,然而C端肽酶还没有发现。一般情况下,需要内肽酶、氨基肽酶、二肽酶和三肽酶的共同协调作用才能完全利用由Opp系统转运进入细胞积聚的多肽。尽管有相当多的讨论,基于遗传学和生理学的见证(信号肽的缺乏和锚定序列以及免疫标定实验等)更容易接受的是这些肽酶是细胞内酶。乳酸菌肽酶的底物大小、特性和其他一些特征已经引起人们的浓厚兴趣,这不仅是因为肽酶生理学的重要性,而且在奶酪制造和成熟等方面肽酶发挥着非常重要的作用。
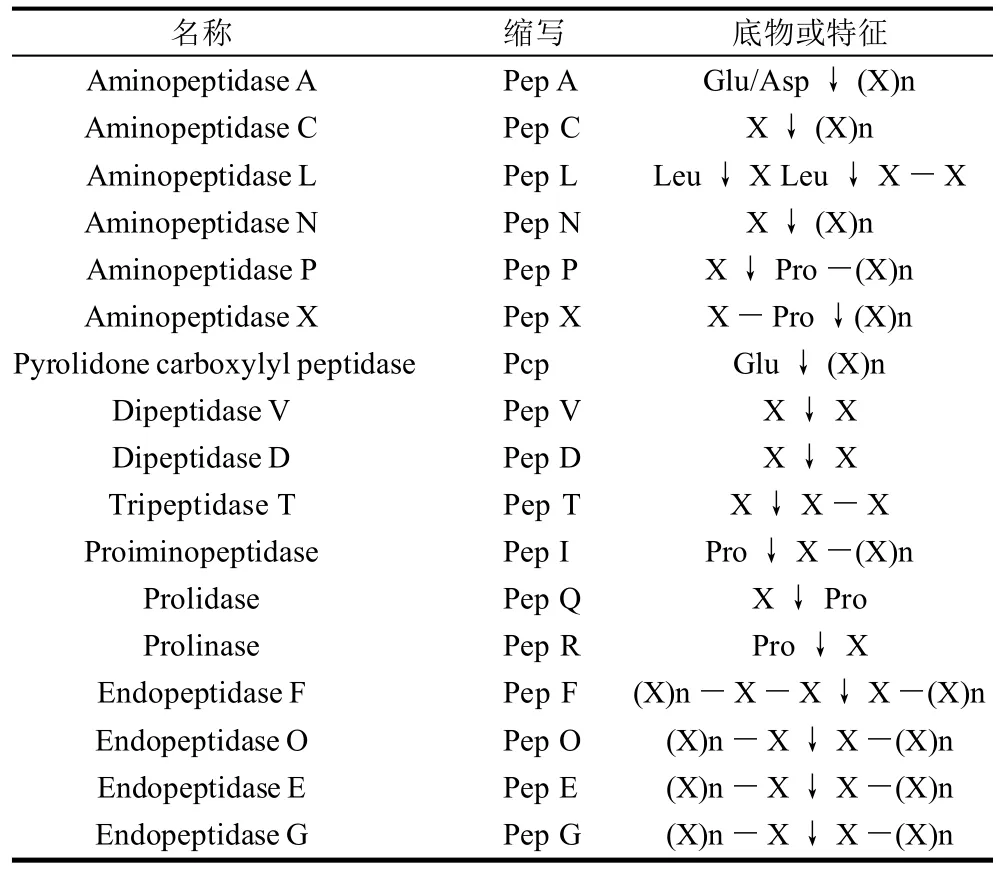
表1 乳酸菌的肽酶Table 1 Previously reported LAB-derived peptidases and their characteristics
3.1 内肽酶
几种内肽酶包括乳酸乳球菌中PepF、PepO[9]和瑞士乳杆菌中的PepE、PepG、PepO[10]已经被报道。大多数内肽酶是含有典型锌结合序列的金属酶,可以对各种大小的多肽进行水解。更有趣的是一些内肽酶如PepF在pH7.5~9.0范围内具有最佳活性,在奶酪成熟的pH值范围(<6.0)活性很低。因此,这些肽酶在细胞生理学和奶酪成熟中的作用很小。此外,乳中多肽酶突变菌株不受影响[9,11]。
3.2 二肽酶和三肽酶
来自培养基中积累或细胞内寡肽水解的二肽和三肽被二肽酶和三肽酶进一步水解成游离氨基酸。几种二、三肽酶已经被纯化,基因已被克隆[11]。尽管这些酶有各种各样自身的生物化学和物理学特性,但它们都是基于底物的特性而发挥作用。一些二肽酶和三肽酶具有非常重要的功能。几种二肽酶属于脯氨酰氨基酸二肽酶或氨酰基脯氨酸二肽酶,水解N端或C端具有脯氨酸的肽。例如,乳酸乳球菌中的PepQ和瑞士乳杆菌中的PepR分别裂解X ↓ Pro和Pro ↓ X 的二肽[12-13]。另一些水解含脯氨酸二肽和三肽的肽酶也被报道[14]。乳酸乳杆菌中三肽酶水解三肽PepT具有特征性[15]。
3.3 氨基肽酶
氨基肽酶广泛存在于乳酸菌中,它是水解N端肽键释放N端氨基酸的酶[16]。以酶的特征对氨基肽酶进行分类,一般的或特异性较差的氨基肽酶PepN和PepC水解2~12个氨基酸的寡肽,一般很少有水解含脯氨酸二肽的活性,这些酶很好地存在于乳酸乳杆菌和乳酸乳球菌中。
因为β-酪蛋白富含脯氨酸,很多PrtP寡肽产物含有脯氨酸。如前所述,含脯氨酸多肽通常对一般的肽酶是特异性较差的底物,相反特异性较强的如PepA、PepX、PepP、PepR和PepI等能够水解这些含脯氨酸多肽。像其他肽酶一样,氨基肽酶因底物的大小和特性不同而多种多样。例如,乳酸乳球菌中PepP的底物是具有X-Pro-Pro-(X)n序列4~10个氨基酸的寡肽[17]。相反,乳酸乳球菌中PepX水解相似的寡肽之外,还能水解二肽和一些不含脯氨酸的多肽[18]。一般的和特异性强的氨基肽酶对奶酪生产都特别重要,因为许多多肽不进行降解的话就具有苦味。
4 结 语
由于乳酸菌缺乏必要的生物代谢途径,不能合成生长所必需的氨基酸和维生素等生长因子,乳酸菌必须从培养基中获得这些生物活性物质才能满足其快速生长需要。乳酸菌依靠胞外蛋白酶水解乳中酪蛋白形成寡肽和氨基酸,这些水解产物通过寡肽转运系统Opp、二肽和三肽转运系统DtpP和DtpT运输进入细胞,最后由细胞内的各种肽酶裂解形成游离氨基酸供细胞合成代谢。由此可以表明乳酸菌在自然生长环境中已经形成了一整套完善的蛋白代谢系统,这是生物与环境相互作用的结果。
近年来对乳酸菌蛋白代谢机制的进一步研究主要集中在分子生物学调控方面[19-20],通过深入研究可从基因水平掌握乳酸菌的蛋白代谢机制。从而利用代谢调控手段提高乳酸菌的生长数量和代谢水平,推动乳酸菌研究的迅速发展,获得高水平的发酵产品。
[1] MCKAY L L, BALDWIN K A. Simultaneous loss of proteinase and lactose-utilizing enzyme activities in Streptococcus lactis and reversal by transduction[J]. Appl Microbiol, 1974, 28(3): 342-346.
[2] JUILLARD V, LAAN H, KUNJI E R S, et al. The extracellular P-type proteinase of Lactococcus lactis hydrolyzes-casein into more than one hundred different olipeptides[J]. J Bacteriol, 1995, 177(12): 3472-3478.
[3] KUNJI E R S, FANG G, JERONIMUS-STRATINGH C M, et al. Reconstruction of the proteolytic pathway for useβ-casein by Lactococcus lactis[J]. Mol Microbiol, 1998, 27(6): 1107-1118.
[4] KUNJI E R S, MIERAU I, HAGTING A, et al. The proteolytic systems of lactic acid bacteria[J]. Antonievan Leeuwenhoek, 1996, 70(2): 187-221.
[5] FOUCAUD C, KUNJI E R S, HAGTING A, et al. Specificity of peptide transport systems in Lactococcus lactis: evidence for a third system which transports hydrophobic di-and tripeptides[J]. J Bacteriol, 1995,177(80): 4652-4657.
[6] KUNJI E R S, HAGTING A, de VRIED C J, et al. Transport of βcasein-derived peptides by the oligopeptide transport system is a crucial step in the proteolytic pathway of Lactococcus lactis[J]. J Biol Chem,1995, 270(4): 1569-1574.
[7] TYNKKYNEN S, BUIST G, KUNJI E, et al. Genetic and biochemical characterization of the oligopeptide transport system of Lactococcus lactis[J]. J Bacteriol, 1993, 175(12): 7523-7532.
[8] DETMERS F J M, KUNJI E R S, LANFERMEIJER F C, et al.Kinetics and specificity of peptide uptake by the oligopeptide transport system of Lactococcus lactis[J]. Biochemistry, 1998, 37(47): 16671-16679.
[9] MONNET V, NARDI M, CHOPIN A, et al. Biochemical and genetic characterization of PepF, an oligopeptidase from Lactococcus lactis[J]. J Biol Chem,1994, 269(51): 32070-32076.
[10] CHRISTENSEN J E, DUDLEY E G, PEDERSON J A, et al. Peptidases and amino acid catabolism in lactic acid bacteria[J]. Antonievan Leeuwenhoek, 1999, 76(1): 217-246.
[11] MIERAU I, TAN P S T, HAANDRIKMAN A J, et al. Cloning and sequencing of the gene for a lactococcal endopeptidase, an enzyme with sequence similarity to mammalian enkephalinase[J]. J Bacteriol, 1993,175(7): 2087-2096.
[12] BOOTHE M, JENNINGS P V, FHAOLAIN I N, et al. Prolidase activity of Lactococcus lactis subsp. cremoris AM2:partialpurification and characterization[J]. J Dairy Res, 1990, 57(2): 245-254.
[13] VARMANEN P, STEELE J L, PALVA A. Characterization of a prolinase gene and its product, and an adjacent ABC transporter gene from Lactobacillus helveticus[J]. Microbiology, 1996, 142(4): 809-816.
[14] BAANKRIES R, EXTERKATE F A.Characterisation of a peptidase from Lactococcus lactis subsp.cremoris HP that hydrolyzes di-and tripeptides containing proline or hydrophobic residues as the amino terminal amino acid[J]. System Appl Microbiol, 1991, 14(2): 317-323.
[15] SAVIJOKI K, PALVA A. Purification and molecular characterization of a tripeptidase (PepT) from Lactobacillus helveticus[J]. Appl Environ Microbiol, 2000, 66(2):794-800.
[16] MIERAU I, KUNJI E R S, VENEMA G, et al. Casein and peptide degradation in lactic acid bacteria[J]. Biotechnol Genet Eng Rev, 1997,14(4): 279-301.
[17] MARS I, MONNETT V. An amino peptidase P from Lactococcus lactis with original pecificity[J]. Biochim Biophys Acta, 1995, 1243(3): 209-215.
[18] MAYO B, KOK J, VENEMA K, et al. Molecular cloning and sequence analysis of the X-propyl dipeptidyl aminopeptidase gene from Lactococcus lactis subsp. cremoris[J]. Appl Environ Microbiol, 1991, 57(1): 38-44.
[19] MAGNANI D, BARRE O, GERBER S D, et al. Characterization of the CopR regulon of Lactococcus lactis IL1403[J]. J Bacteriol, 2008, 190(2): 536-545.
[20] LARSEN R, van HIJUM S, MARTINUSSEN J, et al. Transcriptome analysis of the Lactococcus lactis ArgR and AhrC regulons[J]. Appl Environ Microbiol, 2008, 74(15): 4768-4771.
Progress in Protein Metabolism of Lactic Acid Bacteria
BAI Feng-ling1,ZHANG Bo-lin2,ZHAO Hong-fei2
(1. College of Biotechnology and Food Science, Bohai University, Jinzhou 121000, China;2. College of Biological Science and Biotechnology, Beijing Forestry University, Beijing 100083, China)
Due to the lack of various biosynthetic pathways, lactic acid bacteria (LAB) are nutritionally fastidious and unable to synthesize some essential amino acids and vitamins. LAB possess extracellular proteinase and this enzyme hydrolyzes dairy casein into oligopeptides and amino acids, which can be transported into cells by the oligopeptide transport system (Opp) and the di-tripeptide transport systems (DtpT and DtpP), followed by the hydrolysis of intracellular oligopeptides by endogenous peptidases into free amino acids supporting the synthesis and metabolism of LAB cells during their growth. Hence, studies of protein metabolism in lactic acid bacteria have great significance for improving LAB growth, establishing high-density culture systems and promoting the formation of flavor compounds and their precursors during the fermentation of cheese and other dairy products.
lactic acid bacteria;protein;metabolism
Q936
A
1002-6630(2010)19-0381-04
2010-06-28
国家“863”计划项目(2006AA10Z344;2008AA10Z335);辽宁省教育厅重点实验室项目(2008S005)
白凤翎(1964—),男,教授,博士,研究方向为食品微生物学与食品生物技术。E-mail:baifling@yahoo.com.cn
——细胞生长因子

