圆齿野鸦椿CLE多肽的系统鉴定及功能分析





摘 要 采用生物信息学的方法,鉴定和分析了圆齿野鸦椿(Euscaphis konishii Hayata)CLE基因家族组成及其表达调控作用机制。结果:从圆齿野鸦椿中共鉴定出15个CLE基因,分布在圆齿野鸦椿的6条染色体上,其编码的前体肽大小介于94~920 aa,蛋白质相对分子质量介于10 705.66~101 725.48 KD,理论等电点介于5.84~12.01。多序列比对和偏好性分析表明,圆齿野鸦椿CLE与拟南芥[Arabidopsis thaliana (L.) Heynh.]CLE的同源多肽较为一致,但是部分圆齿野鸦椿CLE在第9位氨基酸上表现出了对丝氨酸的偏好性。系统进化分析表明,圆齿野鸦椿CLE基因成员分为6大亚家族,分别为Group1(0个)、Group2(0个)、Group3(CLE1、CLE2)、Group4(CLE8、CLE9、CLE10、CLE13、CLE14、CLE15)、Group5(CLE11、CLE12)、Group6(CLE3、CLE4、CLE5、CLE6、CLE7)亚组。功能分析发现,CLE3、CLE4在根部维管束组织中表达,影响根的初生木质部发育,CLE9、CLE10、CLE8、CLE13、CLE14、CLE15在根部韧皮部中表达,可以通过间接或直接相互作用,引导根的初生韧皮部细胞从分裂向分化转化。
关键词 圆齿野鸦椿(Euscaphis konishii Hayata);CLE多肽;系统进化;功能分析
中图分类号:Q789 文献标志码:A DOI:10.19415/j.cnki.1673-890x.2025.01.001
CLE(CLAVATA3/Embryo surrounding region-related)多肽是一种非常重要的植物生长调节剂,它在植物生长发育过程中,与细胞间的信号传递有关[1],在分生组织细胞分裂和分化的维持中发挥着非常关键的信号转导调节作用[2]。圆齿野鸦椿(Euscaphis konishii Hayata)属于省沽油科(Staphylea)野鸦椿属(Euscaphis),为落叶小乔木或灌木。圆齿野鸦椿除了具有观赏价值外,还是功效极为出色的药材,其根起到清热解毒、祛湿的作用[3]。关于CLE多肽研究发现,铁皮石斛(Dendrobium officinale Kimura et Migo)中存在17个CLE基因[4],杨树(Populus L.)中具有50个CLE基因[5],番茄(Lycopersicon esculentum Mill.)中含有15个CLE基因[6]。吴云飞等研究发现CLE多肽也调控维管组织分化与增殖的平衡,CLE40能促进干细胞的分化,对维持根顶端分生组织干细胞的动态平衡起着重要作用[7]。TDIF(Tracheary element differentiation inhibitory factor)是一种由百日草(Zinnia elegans Jacq.)和其他植物产生的小分子肽类信号物质,在植物导管形成过程中具有重要作用。李晓旭研究发现百日草TDIF氨基酸序列与拟南芥CLE41/CLE44序列完全相同,TDIF/CLE41/CLE44类信号物质是调控植物导管分化和组织发生的关键分子[8]。有研究表明,在拟南芥中,过表达CLE41/CLE44会导致原形成层细胞消耗,并造成导管断裂,因此TDIF/CLE41/CLE44的作用是可以促进原形成层增殖,并对原形成层分化为导管进行抑制[9-11]。李天笑等提出PgCLE9、PgCLE14、PgCLE16等基因对人参不定根分支数有抑制作用,而PgCLE5通过促进不定根的分支来增加不定根的分支数[12]。同时,在拟南芥的初生根系统中,这些成员都能够显著抑制主根和侧根的生长。目前关于圆齿野鸦椿的分子研究集中在果色形成机制上[13],对圆齿野鸦椿根生长发育的分子研究较少。本研究通过对圆齿野鸦椿CLE多肽功能分析及鉴定,进一步挖掘圆齿野鸦椿中的功能多肽及其信号转导在根生长发育中的功能,为圆齿野鸦椿初生根调控提供一定的理论支撑。
1 "材料与方法
1.1 "圆齿野鸦椿CLE基因的查找
在获得圆齿野鸦椿基因组数据的基础上[14],以模式植物拟南芥[15]作为参考,鉴定圆齿野鸦椿CLE多肽基因。在TAIR10数据库(https://www.Arabidopsis.org/)中以“CLAVATA3”作关键词搜索[16],获取拟南芥的CLE多肽蛋白序列,利用ATCLE的peptide sequence,进行BLASTP检索,E值≤10-10的基因作为候选基因。同以上方法,再用候选基因的peptide sequence进行二次查找。
1.2 "圆齿野鸦椿CLE基序图谱的绘制
把圆齿野鸦椿CLE多肽序列和拟南芥CLE多肽序列合成一个文档,以fasta格式,打开方式选择MEGA7.0软件,点击“Align selected with MUSCLE”,再点击“Align Protein”,进行多序列比对,根据已经报道的拟南芥CLE多肽,找到对应的圆齿野鸦椿保守序列,利用在线软件WebLogo(http://weblogo.berkeley.edu/logo.cgi)绘制CLE基序图谱,进行偏好性分析。
1.3 "多序列比对与系统进化树构建
利用MEGA 7.0软件对获得的CLE蛋白序列进行多重序列比对。以多序列比对结果为基础,最大似然算法构建系统进化树,Bootstrap重复测试(ReplicATions)设置为1 000[17],输出方式为Newick格式,打开FigTree v1.4.4软件美化进化树,对圆齿野鸦椿CLE多肽进行亚家族分类。
1.4 "圆齿野鸦椿CLE基因表达热图谱的构建
根据本课题组测得的圆齿野鸦椿枝条、果实、叶片、花瓣、花瓣+萼片、雄蕊和雌蕊转录组数据,获得CLE基因表达量的FPKM(Fragments per kilobase of transcript per million fragments mapped)值,打开TBtools-Ⅱ(Toolbox for Biologists)v1.108软件[18],选择Gra-phis中的HeatMap,粘贴EXCEL中整理好的基因表达量数值,点击Start,得到表达量热图,利用Show Contronl Dialog调整色阶梯度,使得热图具有最好的分辨率。最后以jpg格式输出热图。
2 "结果与分析
2.1 "圆齿野鸦椿CLE多肽的鉴定与理化性质
利用在线预测软件SignalP 4.1Server(http://www.cbs.dtu.dk/services/SignalP/)[19],对圆齿野鸦椿CLE基因信号肽位置进行预测,获得15个圆齿野鸦椿CLE基因,比对基因号,将其命名为Ej-CLE+阿拉伯数字(见表1)。
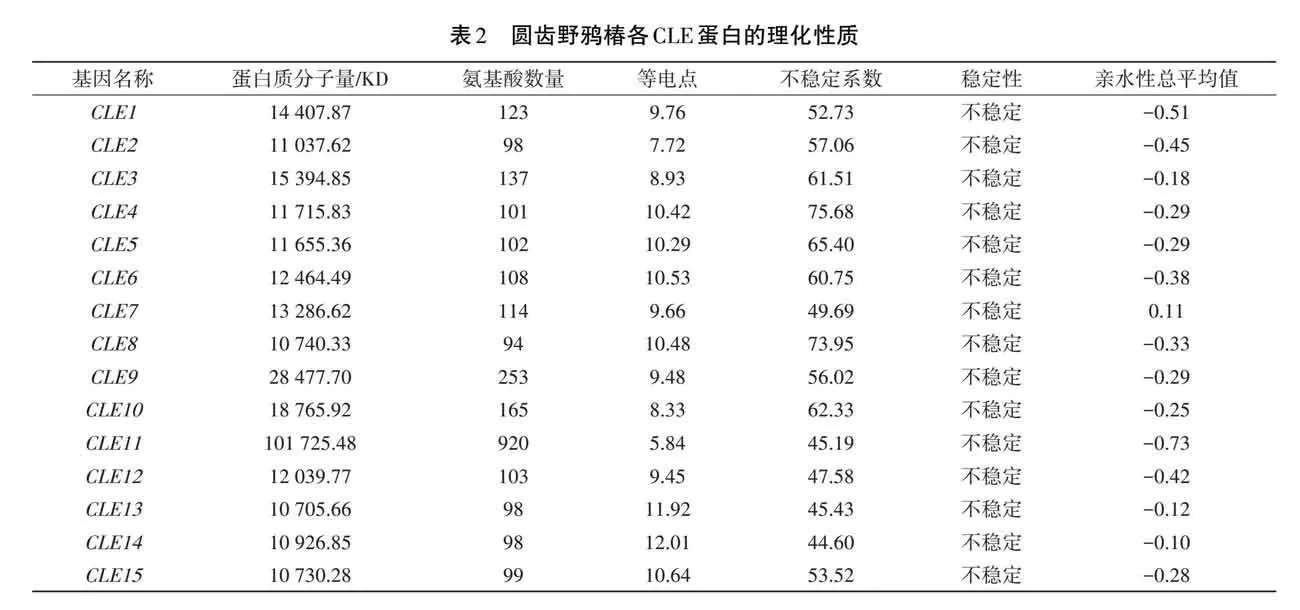
通过对圆齿野鸦椿CLE多肽基因家族的理化性质分析(见表2),CLE基因家族编码的长度介于94~920 aa;蛋白质相对分子质量介于10 705.66~101 725.48 KD;理论等电点介于5.84~12.01,偏酸性的只有EjCLE11,为5.84,其他14位成员均为碱性,平均为9.7,表明该基因家族多肽很大程度上偏碱性;不稳定系数介于44.6~75.68,亲水性总平均值大多为负数,表明圆齿野鸦椿的CLE蛋白大多为不稳定的亲水蛋白[20],仅CLE7编码的蛋白为不稳定的疏水蛋白,其他的都为不稳定的亲水蛋白。
2.2 "圆齿野鸦椿CLE多肽基序
根据拟南芥和圆齿野鸦椿CLE基因编码蛋白序列的多序列比对结果,圆齿野鸦椿CLE多肽的基序由12个氨基酸组成。其中,拟南芥CLE13与圆齿野鸦椿CLE3具有相同的基序,拟南芥CLE10和CLE9与圆齿野鸦椿CLE4具有相同的基序,圆齿野鸦椿CLE13与拟南芥CLE25具有相同的基序。利用MEGA7.0对圆齿野鸦椿CLE多肽与拟南芥CLE多肽的同源性进行了比较,发现它们与拟南芥CLE多肽的同源性较高(见图1)。
利用Motif在线预测软件MEME Suit v5.5.2(https://meme-suite.org/meme/tools/meme)对圆齿野鸦椿和拟南芥的CLE基因进行预测分析,并结合webLogo在线软件(http://weblogo.berkeley.edu/logo.cgi)绘制CLE基序图谱(如图2)。通过圆齿野鸦椿15个CLE基序图谱发现,每一个CLE基序都由12个氨基酸组成,不同CLE成员间氨基酸序列相似度较高。圆齿野鸦椿作为CLE蛋白家族的重要代表之一,在其CLE基序图谱中也显示出这些成员的一些保守和变异的特征。15个圆齿野鸦椿CLE基序的保守位点包括:第1位的精氨酸(Arg),第4、7、9位的脯氨酸(Pro),第6位的甘氨酸(Gly)及第11位的组氨酸(His)等。这些氨基酸残基在CLE蛋白成员中起着核心性质或者重要结构性质的作用[21]。结果发现,圆齿野鸦椿CLE多肽与拟南芥CLE多肽同源程度较高(见图2、图3),基序序列的偏好性类似。拟南芥CLE基序的保守位点包括:第1位的精氨酸(Arg),第4、7、9位的脯氨酸(Pro),第6位的甘氨酸(Gly),以及第11位的组氨酸(His)。
2.3 "圆齿野鸦椿CLE基因的染色体定位
利用TBtools-Ⅱ(Toolbox for Biologists) v1.108软件的Gene LocATion Visualize from GTF/GEE功能,输入圆齿野鸦椿的注释文件,根据位置信息将15个CLE基因定位到圆齿野鸦椿染色体上(图4,见封三),有15条CLE基因分布在6条染色体上,其中分布在4号染色体上的有6条,分别为CLE4、CLE5、CLE8、CLE10、CLE13、CLE15;2号染色体上分布有CLE2、CLE3、CLE7;7号染色体上分布有CLE7、CLE11;5号染色体上分布有CLE1、CLE6;8号染色体上分布有CLE12;3号染色体上分布有CLE14;1号、6号染色体上无CLE基因分布。
2.4 "圆齿野鸦椿CLE基因结构
采用TBtools-Ⅱ(Toolbox for Biologists) v1.108软件对15个圆齿野鸦椿CLE基因进行基因结构的可视化分析,发现近一半的基因有内含子,其中5个CLE基因有1个内含子,2个CLE基因有2个内含子,仅有1个CLE基因有多个内含子(图5-A,见封三)。
采用MEME(https://meme-suite.org/meme/)在线软件分析15个圆齿野鸦椿CLE蛋白全长序列特征,发现有5个保守基序(图5-B,见封三),只有CLE5、CLE13、CLE14和CLE15包含Motif5,CLE1和CLE2包含Motif4,且只有CLE11不包含Motif2,所有基因都包含Motif1。Motif1包含了CLE多肽核心功能序列,位于CLE多肽蛋白序列的C端。
2.5 "圆齿野鸦椿CLE系统进化树的构建
根据以上方法利用MEGA7.0软件对拟南芥CLE蛋白序列和圆齿野鸦椿CLE蛋白序列构建系统进化树,根据拟南芥的CLE家族分类关系可以将这些成员分为6个亚组,分别用Group1、Group2、Group3、Group4、Group5、Group6表示(图6,见封三),此外,15个圆齿野鸦椿CLE基因系统进化树的分组结果与该结果也比较一致。其中Group1和Group2没有圆齿野鸦椿CLE基因家族成员,仅有拟南芥CLE基因聚类在一起;Group4中基因家族成员最多,有CLE8、CLE9、CLE10、CLE13、CLE14、CLE15共6组基因;其次为Group6,有CLE3、CLE4、CLE5、CLE6、CLE7共5组基因;Group3和Group5基因家族成员都为2个,Group3有CLE1和CLE2,Group5有CLE11和CLE12。在这6个亚组中,Group3、Group4和Group5距离较近,Group1、Group2和Group6距离较远。
2.6 "圆齿野鸦椿CLE基因家族成员表达模式
通过上述方法筛选出15个圆齿野鸦椿CLE基因表达量的FPKM,利用TBtools-Ⅱ(Toolbox for Biologists) v1.108软件绘制热图谱(图7,见封三)。
果实中圆齿野鸦椿CLE基因的表达量普遍较低,其中CLE1、CLE2、CLE3、CLE4、CLE5、CLE6、CLE7、CLE11、CLE13、CLE14的表达量最低,表明这些CLE基因在果实中的调控作用较小,但在果实Ⅰ中圆齿野鸦椿CLE9、CLE10、CLE12表达量较高,果实Ⅱ中圆齿野鸦椿CLE8、CLE15表达量较高。在枝条中圆齿野鸦椿CLE2、CLE6、CLE7、CLE13、CLE14的表达量较果实高很多,表明这些基因主要参与圆齿野鸦椿枝条调控。圆齿野鸦椿在叶片上CLE基因表达量普遍较低,仅有CLE4有较高的表达量,表明圆齿野鸦椿CLE基因在叶片中的调控作用较小。在花瓣和萼片中,圆齿野鸦椿CLE基因表达量普遍较低,表明圆齿野鸦椿CLE基因在花萼和萼片中的调控作用较小。在花瓣Ⅱ中,圆齿野鸦椿CLE1、CLE10、CLE14表达量较高,花瓣Ⅰ中圆齿野鸦椿CLE4、CLE5表达量较高,其余表达量均较低,表明圆齿野鸦椿CLE1、CLE4、CLE5、CLE10、CLE14在花瓣中的调控作用较其他CLE基因大。在雄蕊中仅CLE3表达量较高,雌蕊中仅CLE11表达量较高,表明圆齿野鸦椿CLE3和CLE11在雄蕊和雌蕊中的调控作用较大。
结合圆齿野鸦椿CLE基因亚家族分类可知,Group3和Group6的CLE基因在果实中的调控作用较小,Group4和Group5的CLE基因在果实Ⅰ和果实Ⅱ中的表达量较高,Group3、Group4和Group6的CLE基因在枝条、花瓣中的调控作用较大,表明Group3和Group6、Group4和Group5的关系较近,Group3和Group6的CLE基因共同调控圆齿野鸦椿花瓣、枝条的生长发育,Group4和Group5的CLE基因共同调控圆齿野鸦椿果实的生长发育。
3 "讨论
拟南芥[Arabidopsis thaliana (L.) Heynh.]的基因结构较简单,在当今的植物发育学研究中,基于模式植物的生命机理研究越来越多。近年来,文献数据库PubMed(http://www.ncbi.nlm.nih.gov/pubmed)推动了植物分子生物学方面的研究[22]。对于目前已经报道的关于CLE基因多肽的研究,拟南芥CLE多肽系统鉴定及功能研究比较透彻,其蛋白是一类小分子多肽,参与调控植物生长发育过程中的细胞分化、干细胞维持和根系统发育等关键生理过程。通过对圆齿野鸦椿基因组数据的分析得知,圆齿野鸦椿基因组中存在多个可能编码CLE蛋白的基因,这些基因在圆齿野鸦椿不同组织中的表达水平也存在差异,表明它们可能有不同的生物学功能。
拟南芥CLE1、CLE3、CLE4、CLE7与植物固氮相关,同时影响植物侧根发生,其在植物的次生根中特异表达[23]。因此推测圆齿野鸦椿CLE11和CLE12可能执行相似的功能,影响侧根生长。CLE41、CLE44、CLE45等基因编码的拟南芥CLE蛋白在拟南芥干细胞的维持和分化中发挥了重要作用,调节根尖中的细胞分裂、分化及芽梢生长方向等生理过程,CLE3和CLE29基因编码的蛋白则参与了茎尖分生和根伸长等生理过程调控。因此,可以合理推测圆齿野鸦椿CLE蛋白也可能参与植物的干细胞维持和分化、根系和茎的生长发育等关键生理过程。
一些研究表明,在茎尖和根尖中,拟南芥CLE多肽激活了WUSCHEL(WUS)和CLAVATA3(CLV3)基因对应的信号通路,这些基因编码激素受体蛋白和转录因子,参与调节茎尖和根尖中植物干细胞的自我更新和身份维持[24-26]。有研究表明,ATCLE40/CLE40信号途径和CLV2/CRN信号途径在拟南芥根发育中发挥着重要的调节作用,对了解植物根部组织的形成和调控机制具有重要意义[27-28],因此推测与ATCLE40聚类在一起的圆齿野鸦椿CLE6也有相似的功能,维持根顶端分生组织干细胞的动态平衡。
维管系统又称为维管组织系统(Vascular tissue system),包括植物体内所有的维管组织,在植物的生长发育过程中起到物质运输[29]、机械支持和信号传导等重要作用[30]。在拟南芥中,CLE45多肽在韧皮部中表达,外源施加ATCLE45多肽能够抑制根初生韧皮部的分化,并抑制临近半胞细胞和筛管分子前体细胞的发育,最终起到抑制根初生韧皮部分化的作用[31]。CLE9/10分泌肽也可以通过与BAM受体结合来抑制形成层细胞向木质部的分化[25]。而CLE25多肽则通过与CLERK-CLV2受体复合物结合来促进维管系统中形成层细胞向韧皮部的分化[32]。最新的研究发现,CLE25及其对应受体BAM,能参与长途信号传导以应对缺水胁迫,产生从根到芽的信号,诱导ABA合成并导致气孔关闭[33]。因此推测与ATCLE45聚类在一起的CLE8、CLE9和CLE10也有类似的功能,抑制根初生韧皮部的分化;CLE3、CLE4也与拟南芥CLE9/10有类似功能;CLE13、CLE14、CLE15和ATCLE25聚类在一起,推测CLE13、CLE14、CLE15可能执行相似的功能。
本研究使用生物信息学的方法,首次系统分析圆齿野鸦椿CLE多肽基因家族,发现圆齿野鸦椿有15个CLE基因家族成员,这些基因与CLE多肽功能序列高度保守,每个基因都包含Motif1,大部分CLE都有内含子。系统进化分析表明,CLE3、CLE4在根部维管束组织中表达,影响根初生木质部的发育,CLE8、CLE9、CLE10、CLE13、CLE14、CLE15在根部韧皮部中表达,影响根初生韧皮部细胞的分化。本文为深入研究CLE多肽在圆齿野鸦椿生长中的作用奠定了研究基础,有助于更好地理解植物生长发育的分子机制。
参考文献:
[1] 吴锦斌,宋银,乔睿,等.CLE多肽参与植物分生组织维持与分化平衡的信号转导研究[J].生命科学,2013,25(4):421-426.
[2] 王彩丽.植物多肽类激素调控分生组织活性的分子机制研究[D].天津:天津大学,2019.
[3] 王俞岑.圆齿野鸦椿研究进展[J].现代园艺,2019(3):35-36.
[4] 诸燕,丁兰,陈忆乾,等.铁皮石斛CLE基因家族鉴定与功能分析[J/OL].浙江农业学报:1-9[2024-07-10].
[5] 刘艺森.杨树CLE信号分子对植物分生组织活性的调控机制分析[D].天津:天津大学,2018.
[6] 张宇.模式植物CLE多肽基因家族的生物信息学分析与鉴定[D].天津:天津大学,2017.
[7] 吴云飞,储黄伟,周志刚,等. A类CLE多肽调节拟南芥根尖韧皮部的发育[J].植物生理学报,2014,50(10):1515-1522.
[8] 李晓旭.拟南芥CLE多肽激素信号通路相关基因的鉴定及功能研究[D].北京:中国农业科学院,2016.
[9] FISHER K, TURNER S. PXY, a receptor-like kinase essential for maintaining polarity during plant vascular-tissue development[J]. Current Biology, 2007, 17(12):1061-1066.
[10] Zhang L, Shi X, Zhang Y, et al. CLE9 peptide-induced stomatal closure is mediated by abscisic acid, hydrogen peroxide, and nitric oxide in Arabidopsis thaliana[J]. Plant Cell Environ, 2019, 42(3): 1033-1044.
[11] 王卓毅,孙挺,徐沛.药食同源作物赤小豆(Vigna angularis) CLE基因家族鉴定与表达调控分析[J/OL].分子植物育种:1-13(2022-10-17)[2023-05-11].
[12] 李天笑,李琛,孙彻,等.人参CLE多肽的系统鉴定及其功能分析[J].分子植物育种,2022,20(22):7391-7399.
[13] YUAN X Y, SUN W H, ZOU X X, et al. Sequencing of Euscaphis konishii endocarp transcriptome points to molecular mechanisms of endocarp coloration[J]. International Journal of Molecular Sciences, 2018, 19(10): 3209.
[14] SUN W H, LI Z, XIANG S, et al. The Euscaphis japonica genome and the evolution of malvids[J]. The Plant Journal: for Cell and Molecular Biology, 2021, 108(5): 1382-1399.
[15] 徐平丽,张传坤,孙万刚,等.模式植物拟南芥基因组研究进展[J].山东农业科学,2006(6):100-102.
[16] 张亚楠.园艺植物心皮(室)数基因CLAVATA3生物信息学分析及遗传评价[D].晋中:山西农业大学,2019.
[17] 潘玉欣,王巍杰,胡金山.拟南芥和水稻UXS基因家族生物信息学分析[J].河南农业科学,2011,40(11):38-42.
[18] 陈程杰,夏瑞. TBtools——大数据时代下的国产生物软件[J].科学观察,2022,17(6):33-35.
[19] 荣誉磊,周志林,赵冬兰,等.甘薯、番茄、拟南芥中SPL转录因子的生物信息学分析[J].江苏农业科学,2021,49(20):74-83.
[20] 张宁,温银元,王金荣,等.苦参8-异戊烯基转移酶基因蛋白家族生物信息学分析[J].山西农业科学,2018,46(5):692-695,772.
[21] 张宇.模式植物CLE多肽基因家族的生物信息学分析与鉴定[D].天津:天津大学,2015.
[22] 岳靖. TDIF多肽激素在杨树生长发育中的调控机制研究[D].天津:天津大学,2020.
[23] 白婧平. TDIF多肽与生长素在拟南芥根发育中的相互作用机制研究[D].天津:天津大学,2020.
[24] ITO Y, NAKANOMYO I, MOTOSE H, et al. Dodeca-CLE peptides as suppressors of plant stem cell differentiation[J]. Science, 2006, 311(5788): 842-845.
[25] KINOSHITA A, NAKAMURA Y, SASAKI E, et al. Gain-of-function phenotypea of chemically synthetic CLAVATA3/ESR-related(CLE) peptides in Arabidopsis thaliana and Oryz sativa[J]. Plant Cell Physiol, 2007, 48(12):1821-1825.
[26] FIERS M, GOLEMIEC E, XU J, et al, The 14-amino acid CLV3, CLE19, and CLE40 petides trigger consumption of the root meristem in Arabidopsis through a CLAVATA2-dependent pathway[J]. Plant Cell, 2005, 17(9): 2542-2553.
[27] STAHL Y, WINK R H, INGRAM G C, et al. A signaling module controlling the stem cell niche in Arabidopsis root meristem[J]. Current Biology, 2009, 19(11): 909-914.
[28] STAHL Y, SIMON R. Is the Arabidopsis root niche protected by sequestration of the CLE40 signal by its putative receptor ACR4[J]. Plant Signaling amp; Behavior, 2009, 4(7): 634-635.
[29] 李欣.杨树类TDIF多肽基因家族对维管组织的发育调控[D].天津:天津大学,2018.
[30] 袁娜,李阳,杨郁文,等.棉花CLE多肽家族的全基因组鉴定与生物信息学分析[J].棉花学报,2019,31(4):263-281.
[31] 韩惠宾. SpCLE多肽调控拟南芥维管组织发育及其杨树CLE基因家族分析[D].西安:陕西师范大学,2015.
[32] REN S C, SONG X F, CHEN W Q, et al. CLE25 peptide regulates phloem initiation in Arabidopsis through a CLERK-CLV2 receptor complex[J]. Journal of Integrative Plant Biology, 2019, 61(10): 1043-1061.
[33] 张哲.植物CLE家族基因的鉴定和聚类[D].武汉:华中农业大学,2018.
(责任编辑:易 "婧)

