结合肺先验与协同深监督的肿瘤自动分割




DOI:10.3969/j.issn.10001565.2024.04.010
摘要:计算机断层扫描图像中复杂肺肿瘤(CLT)的自动分割面临2个挑战:1) 肿瘤与邻近组织之间的类间不区分;2)肿瘤内的类内不一致性.为了解决这2个问题,提出将肺肿瘤与肺之间关系的语义上下文先验纳入分割模型中,以便于模型学习到语义上下文特征,并从宏观角度重新思考CLT的分割.利用信息熵对肺形状的解剖先验进行建模.在三分类的U-Net网络中嵌入提出的新型注意模块,从而通过特定领域的知识来指导训练过程.另外,设计了一个可以获得肿瘤边界结构图以及保持肿瘤内部特征一致性的边界增强辅助网络.在此基础上,开发了一个协同深度监督网络框架(CLT-ASegNet),该框架利用混合多尺度语义特征融合进一步提高了模型的判别能力和收敛速度.CLT-ASegNet在CLTCTI分割数据集和Lung16数据集上进行了评估.实验结果表明,所提出的CLT-ASegNet可以有效分割肺肿瘤.
关键词:注意力机制; 复杂肺肿瘤分割; 语义上下文先验; 协同深度监督
中图分类号:TP391.4文献标志码:A文章编号:10001565(2024)04041410
Automatic segmentation of tumors combining lung
prior and synergetic deep supervision
WANG Bing1,2, JU Mengyi2, YANG Ying3, ZHANG Xin4, ZHAI Junhai1,2
(1. Hebei Key Laboratory of Machine Learning and Computational Intelligence, Baoding 071002, China;
2. College of Mathematics and Information Science,Hebei University, Baoding 071002, China;
3. Radiology Department, Hebei University Affiliated Hospital, Baoding 071000, China;
4. College of Electronic Information Engineering, Hebei University, Baoding 071002, China)
Abstract: There are two challenges in automatic segmentation of complex lung tumors (CLT) on computed tomography (CT) images: 1) The class indistinction between tumors and adjacent tissues; 2) Intra-class inconsistencies within tumors. In order to solve these two challenges, the semantic context prior of the relationship between lung tumor and lung is proposed to be incorporated into the segmentation model, so that the model can learn the semantic context features, and the segmentation of CLT can be reconsidered from a macro perspective. The anatomical prior of lung shape is modeled using information entropy. The proposed novel attention module is embedded in the three-classified U-Net network, so as to guide the training process through domain-specific knowledge. In addition, a boundary enhancement auxiliary
收稿日期:20231206;修回日期:20240319
基金项目:河北省自然科学基金资助项目(F2021201020);河北省自然科学基金青年科学基金资助项目(F2024502006)
第一作者: 王兵(1967—),女,河北大学教授,主要研究方向为机器学习、计算机视觉等.E-mail:wangbing@hbu.edu.cn
通信作者:张欣(1966—),男,河北大学教授,博士,主要研究方向为机器学习、图像处理等.E-mail:zhangxin@hbu.edu.cn
network was designed to obtain tumor boundary structure and maintain the consistency of tumor internal features. On this basis, a collaborative deep supervision network framework (CLT-ASegNet) was developed, which further improved the discriminant ability and convergence speed of the model by using hybrid multi-scale semantic feature fusion. CLT-ASegNet was evaluated on CLTCTI segmentation datasets and Lung16 datasets. The experimental results show that the proposed CLT-ASegNet can effectively segment lung tumors.
Key words: attention mechanism;complex lung tumor segmentation;semantic context prior; synergetic deep supervision
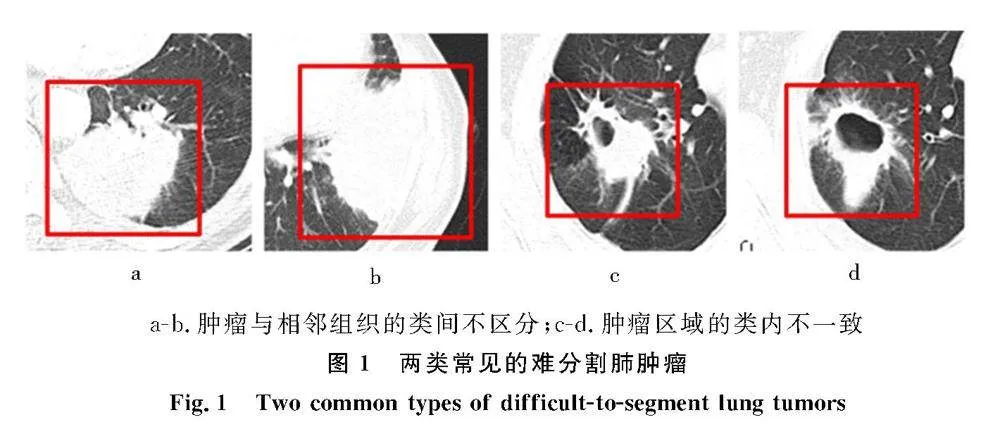
本研究的重点是计算机断层扫描(computed tomography,CT)图像中复杂肺肿瘤(complex lung tumor,CLT)的自动分割.传统的机器学习方法在处理医学图像分割时遇到了挑战,与肺结节[1]相比,肺肿瘤,特别是CLT,由于具有形态和结构的复杂性、边界信息较弱、背景和纹理的显著异质性等特征,使其难以取得令人满意的结果.近年来,基于深度卷积神经网络(deep convolutional neural networks,DCNN)的密集预测在医学成像领域取得了令人鼓舞的成果,如U-Net[2]和DeepLabv3+[3]的发展,促进了肺肿瘤[4-5]的分割.然而,这些方法得到的特征表示通常无法识别以下区域:1)肿瘤边界两侧相邻的区域,由于肿瘤与肺周围组织(如肺壁)的粘连较大,称为“肿瘤与相邻组织的类间不区分”(图1a-b);2)肿瘤内部的异质性块,应具有相同的语义标签,称为“肿瘤区域的类内不一致”(图1c-d).a-b.肿瘤与相邻组织的类间不区分;c-d.肿瘤区域的类内不一致
为了解决肿瘤和邻近组织之间的类间不区分问题,本研究将肺肿瘤和肺之间的语义上下文的先验知识纳入基于DCNN分割模型中,从而用特定领域知识指导训练过程[6].通过利用信息熵对肺形状的解剖先验进行建模,该信息熵是基于患者全部扫描数据的肺实质分割金标准(ground truth,GT)进行计算.此外,还设计了一个具有先验肺形状的注意模块(AMwPLS),并将其嵌入到三分类语义分割(semantic segmentation,SS)网络中,以便于AMwPLS学习肿瘤和肺之间的语义上下文特征.
另一方面,为了解决肿瘤内部类内不一致问题,并从宏观角度重新考虑CLT的分割,将肿瘤区域内的像素视为一个整体,为它们分配一致的语义标签.本研究建立了一个肿瘤边界增强辅助网络(TBE),在混合尺度上提取细粒度的肿瘤边界特征图(TBFM),TBFM既能代表肿瘤边界,又能保持肿瘤内部特征的一致性.
在大多数医学图像分割中很大概率会出现非感兴趣区域假阳性或感兴趣区域假阴性情况,这可能是由于网络模型浅层仍然保留着背景噪声信息,从而导致了分割效果不好.为了解决这一问题,在SS网络的输出端采用协同深度监督,以提升模型对隐层特征的识别能力.基于此,本研究设计了一个CLT分割的协同深度监督网络框架(CLT-ASegNet),它使用U型网络作为基线,使用多语义特征融合(MSFF)模块将AMwPLS细化的浅层特征、多尺度全局特征和TBE生成的肿瘤边界特征聚合起来,进一步提高模型的判别能力.
1相关工作
最近,许多基于深度学习的肺肿瘤和病变组织分割网络已经获得较好的分割结果.与传统方法(阈值化、区域增长、图切、主动轮廓模型)相比,深度学习方法对图像浅层特征(如强度、纹理、梯度等)表达方面表现出了更具竞争力的性能.
1.1基于深度学习的肺结节和肺肿瘤的分割
许多研究人员为提高肿瘤边界分割的精度做出了贡献.例如,Zhang等[4]增加了一个密集连接的条件随机场框架来勾画肺肿瘤的边界.Ni等[5]提出了一种从粗到细的2阶段框架:用于定位的三维多尺度U-Net和用于分割细化的2.5D多尺度可分离U-Net分割.Yu等[7]利用域先验知识分割肺实质区域,然后再进行肿瘤区域的分割,最后进行精细的校正.
1.2注意力机制在医学图像分割中的应用
注意力机制旨在通过引入一个聚焦于图像特定区域的模块来改善网络分割性能.Oktay等[8]提出了一种新的注意门模型用于医学图像,该模型自动学习关注不同形状和大小的目标结构.Wang等[9]应用了通道注意块和混合扩张注意卷积层,实现了适用于不同医学图像分割的目的.Lin等[10]设计了一种基于方差的不确定性机制,通过预测概率来评估每个体素的识别能力.Hu等[11]成功地在残差模块中提取了不同尺度的特征,并利用注意模块增强对多尺度特征的注意能力.
1.3医学图像语义分割网络架构
U-Net[2]和UNet++[12]等U型架构在医学图像分割领域得到了广泛的应用.另外,Cai等[13]设计了一种多尺度的目标预测机制,它聚合了由多个中间层生成的特征.An等[14]提出了一种基于U-Net的分割网络和多层边界检测网络组成的多层边界感知RU-Net分割模型.Zhang等[15]提出了一种新的模型BCU-Net,Li等[16]提出了一种结合Transformer和卷积线性注意块解决与正常组织密度相似的病变问题.Tang等[17]提出了一种不确定性引导网络,称为“UG-Net”,用于自动医学图像分割.
2CLT-ASegNet模型分割方法
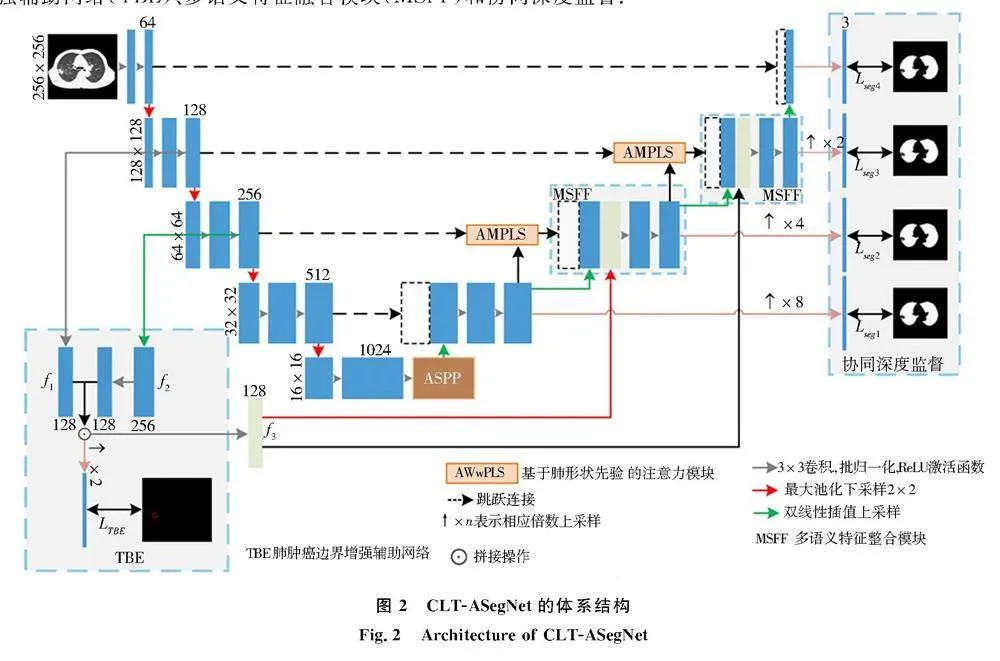
如图2所示,CLT-ASegNet总体框架包括4个方面:先验肺形状的注意模块(AMwPLS)、肿瘤边界增强辅助网络(TBE)、多语义特征融合模块(MSFF)和协同深度监督.图2CLT-ASegNet的体系结构
Fig.2Architecture of CLT-ASegNet2.1先验肺形状的注意模块(AMwPLS)
AMwPLS的目的是提高粘连于肺壁或纵隔的肿瘤与肺本身之间粘连边界的可区分性,从而减少肿瘤与邻近组织之间的类间不区分.通过利用信息熵对先验的肺形状进行建模,并将其纳入空间注意模块,以提高模型对肺轮廓的关注.同时,应用多分类损失函数来引导网络学习可区分肺和CLT的判别性特征.此外,由于CLT局限于特定肺区域,从AMwPLS中学习到的肿瘤和肺之间的语义上下文特征也有助于为CLT区域分配一致的语义标签,从而减少CLT内的类内不一致.
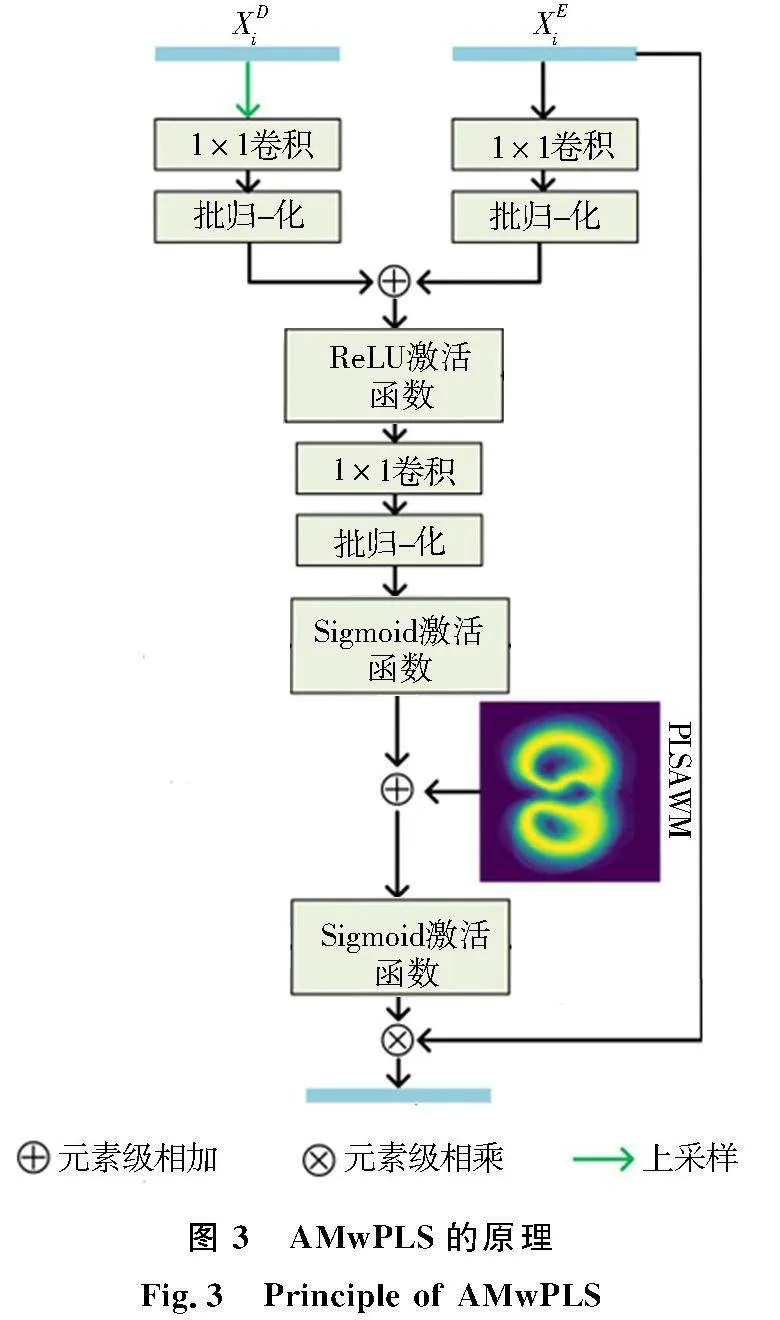
尽管在CT图像中对肺的形状、大小和位置有明显的解剖先验知识,但在同一数据集的不同切片之间存在相当大的差异.直观地说,信息熵为后续预测估计了特征的不确定程度.通过信息熵评估肺边界像素的可能位置,获得先验肺形状的注意力权重图(PLSAWM),并整合到空间注意力模块(图3),以引导网络关注肺边界,从而提高对肺边界附近潜在肿瘤的识别能力.此外,在解码器阶段部署了AMwPLS,以便指导编码器中低级滤波器学习与产生全局特征映射的滤波器相似且兼容的映射.具体来说,首先从公开的肺结节和肺实质分割数据集[18]中随机选择了800例肺实质的GT,然后对这些GT对应的像素位置先进行求和然后平均,得到先验的肺形状概率图(PLSPM).计算PLSAWM如下:
yi=-Pilog2Pi-(1-Pi)log2(1-Pi),(1)
其中,Pi为PLSPM中第i个位置的概率值,yi为PLSAWM中第i个位置的信息熵.w1表示通过计算从SS网络中提取的特征图得到的空间注意力权重图,将PLSAWM与w1相加之后经过激活函数可以得到AMwPLS注意力权重图w,将其与编码器输出的特征图进行多重叠加,以提取肿瘤和肺部的语义上下文特征(图3).AMwPLS注意矩阵w的计算公式如下:w1=φ2(C1(φ1(C1(xEi)⊙C1(Up(xDi))))),(2)
w=φ2(w1+PLSAWM).(3)图3AMwPLS的原理
Fig.3Principle of AMwPLSAMwPLS的输入是xEi、xDi和PLSAWM,xEi表示由编码器经过跳跃连接传输的特征,xDi(i=4)代表解码器特征,C1()表示卷积和批标准化操作,卷积核大小是1×1,步长是1.Up表示上采样,+ 是元素级加法,φ1表示ReLU激活函数,φ2=11+e-x表示S型激活函数,⊙表示通道拼接操作.
AMwPLS输出的x^Ei是通过w与xEi相乘得到的.计算公式如下:x^Ei=xEiw,(4)其中,“”是元素级乘法.
2.2肿瘤边界增强辅助网络(TBE)
TBE与SS网络共享少量特征提取过程,也允许学习不同于SS网络的特征表示,并可以为后续的MSFF模块提供专门的边界特征映射.同时,它还可以缓解由于使用耦合特征映射而引起2种网络次优解的问题.此外,为了减少肿瘤区域内严重的异质性对类内一致性的影响,将肿瘤区域内的像素作为整体来处理,并为它们分配一致的语义标签,目的是能够从宏观的角度来区分肿瘤内和肿瘤外的语义特征.为此,在目标函数中加入基于水平集的边界损失函数[19]来监督CLT边界的学习.
如图2所示,分别对编码器部分的2个浅层特征图进行卷积、批归一化和ReLU操作,得到了f1和f2,然后将f1和f2中的深层特征上采样之后进行拼接操作.随后,一方面对拼接后的特征图进行卷积、批处理归一化和ReLU操作,得到f3,f3将被聚合到高级解码器部分.另一方面,通过对拼接后的特征图进行卷积、上采样、批处理归一化和S型激活函数操作可以得到肿瘤边界预测结果.
边界损失用于增强肿瘤的边界特征,其表现如下:LB=∫ΩφGT(x)f(x)dp,(5)其中Ω表示整个图像区域,φGT是肿瘤区域GT的预先计算的水平集函数.零水平集表示该点在分割目标的边界上,f(x)表示像素x是通过TBE以及softmax概率输出得到的.
此外,利用了Xue等[20]提出的平滑损失来克服训练过程中的不稳定因素.Lreg=-∫ΩφGT(x)φp(x)φGT(x)φp(x)+φ2GT(x)+φ2p(x),(6)其中,φp(x)是预测的像素水平集.综上所述,TBE的总体损失如下:LTBE=LB+Lreg.(7)2.3多语义特征融合模块(MSFF)
为了探索准确的多语义融合特征是否是实现高性能协同深度监督语义分割的必要条件,提出了MSFF模块,通过合并细节特征、全局上下文特征和边界特征,以保持SS和TBE之间的相关性.如上所述,在传递到MSFF模块之前,通过AMwPLS对编码器浅层的细节特征进行了细化.全局上下文特征从解码器的前一层的输出中得到,而边界特征则由TBE模块产生.应用通道拼接、3×3卷积(步幅为1)、批归一化和ReLU激活函数得到低级细节特征与高级语义特征的互补信息xDi(i=2,3).这些操作被连续使2次,以获得融合特征Fi(i=2,3).
2.4协同深度监督
为了获得更准确的肿瘤边界特征和可判别的语义特征,采用协同深度监督体系结构.将解码器的不同尺度的特征映射表示为Fi(i=1,2,3,4).通过不同倍数的上采样操作,Fi恢复到原始图像大小.每个预测结果由3个通道组成,分别代表肺肿瘤、肺和背景的概率值.网络通过同时对肺肿瘤和肺进行分割增强了其对特征的判别能力,从而减少了肿瘤和肺之间边界模糊以及肿瘤内部的异质性对网络性能的影响.通过使用多分类Lovász-Softmax损失函数[21]来指导SS网络的学习,它是一个多类分割问题的Jaccard损失函数的分段线性凸代理.SS网络的分割损失函数表示如下:Lseg=1|C|∑c∈CΔJc(e(c)),(8)其中,e(c)∈RP+是c∈C的误差向量,P是图像中的像素数或批处理中所考虑的像素数,其中给定图像的每个像素i必须被分类为一个对象类c,所有的网络输出通过Softmax操作都被映射到概率si(c),C={肺肿瘤,肺和背景}.此外,ΔJC是基于Lovász子模集函数扩展的Jaccard损失的分段线性凸代理.c类的像素误差向量e(c)由下式定义:ei(c)=1-si(c),if c=GT class of pixelisi(c),otherwise.(9)CLT-ASegNet的总损失表示为Ltotal=Lseg+LTBE.(10)2.5CLT-ASegNet体系结构
CLT-ASegNet是一种端到端协同深度监督体系结构,由U型SS网络和TBE组成,它利用MSFF来驱动网络学习具有低级细节特征与高级语义特征的互补信息和更有判别性的特征.在SS网络的编码器中,从低层到高层提取的特征分别记为xEi(i=1,2,3,4).xEi=Cconv(Cconv(M(xEi-1))),(11)其中:M表示最大池化操作,每次执行1次,特征图变成原来的一半,通道数增加为原来的2倍;Cconv是指卷积核大小为3×3、步幅为2的卷积操作,再依次执行BN和ReLU激活函数.在SS网络的解码器中,只在第2层和第3层解码器中部署了MSFF模块.这是由于肿瘤边界特征f3是由编码器的第2层和第3层TBFMs中f1和f2计算出来的.其余解码器层的输出Fi由xEi和Fi-1计算得到,通过使用以下公式:Fi=C2(xEi⊙UM(Fi-1)).(12)3实验方法与结果分析
3.1数据集和预处理
本实验是基于河北大学附属医院放射科收集的复杂肺肿瘤CT成像(CLTCTI)数据集,它包括261例不同病例的复杂肿瘤的808例肺CT扫描,其中566例用于训练,242个用于测试.训练集和测试集来自于不同的病例.图像的平面分辨率为0.5~0.95 mm,切片间隔为1.00~8.00 mm,管电压为120~140 kV,电流为40~340 mA.CLT和肺分割由3位放射科专家注释.采用中心裁剪、旋转和翻转来扩展训练集.
CLT-ASegNet在Lung16数据集的一个子集上也测试了所提出的方法.Lung16数据集由肺图像数据库联盟和图像数据库资源倡议[22]提供,旨在促进肺癌相关研究和算法的发展.本实验对Lung16一个子集进行处理,通过数据预处理,得到678张CT图像,其中471张图像作为训练集,207张图像作为测试集.
3.2评价指标和实施细节
本研究采用Dice系数(DSC)、Hausdorff距离(HD)、灵敏度(Sen)、特异性(Spe)、精确率(Pre)对网络性能进行评价.
为了评价CLT-ASegNet的有效性,与TransUNet[23]、TFCNs[16]、BCU-Net[15]等7种主流分割网络进行比较.CLT-ASegNet和主流的分割方法都是通过使用Pytoush和NVIDIA RTX3090GPU进行训练,具有2.10 GHz 12核和256 GB内存.采用Adam优化器,学习速率为0.001,动量为0.9,权重衰减为10-4,进行300次迭代.使用随机初始化的权值开始训练所有的网络.
3.3与现有方法的比较
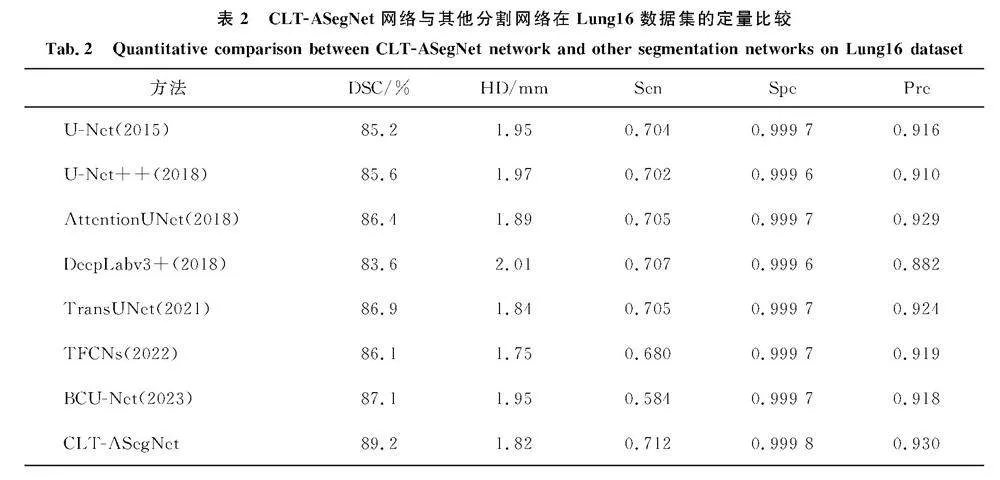
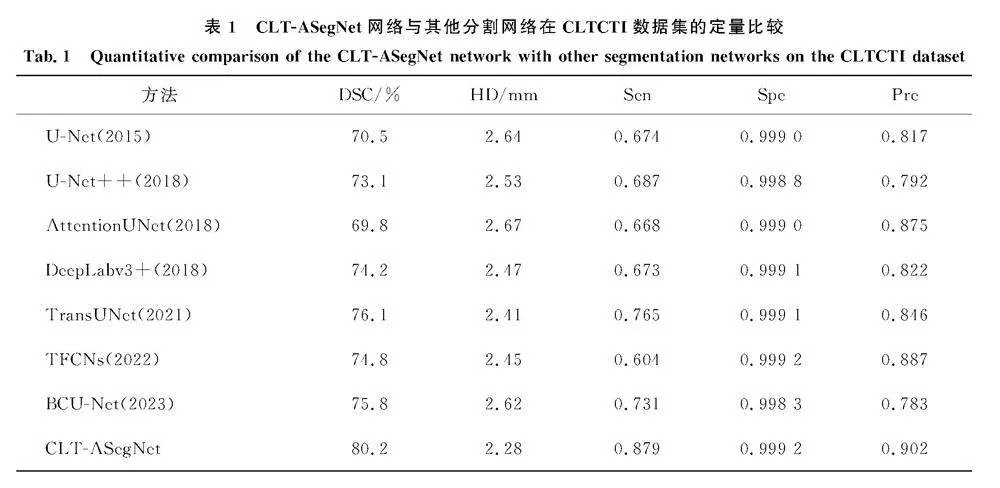
CLT-ASegNet与现有方法在CLTCTI数据集上的定量实验结果对比.如表1所示,DSC、HD、Sen、Spe和Pre分别为80.2%、2.28 mm、0.879、0.999 2和0.902.CLT-ASegNet与U-Net相比,DSC值提高了9.7%,HD值降低了0.36 mm.相比其他最好的分割方法TransUNet,DSC值提高了4.1%,HD值降低了0.13 mm.表1CLT-ASegNet网络与其他分割网络在CLTCTI数据集的定量比较
Tab.1Quantitative comparison of the CLT-ASegNet network with other segmentation networks on the CLTCTI dataset方法DSC/%HD/mmSenSpePreU-Net(2015)70.52.640.6740.999 00.817U-Net++(2018)73.12.530.6870.998 80.792AttentionUNet(2018)69.82.670.6680.999 00.875DeepLabv3+(2018)74.22.470.6730.999 10.822TransUNet(2021)76.12.410.7650.999 10.846TFCNs(2022)74.82.450.6040.999 20.887BCU-Net(2023)75.82.620.7310.998 30.783CLT-ASegNet80.22.280.8790.999 20.902
CLT-ASegNet与现有方法在Lung16数据集上的定量实验结果对比见表2.DSC、HD、Sen、Spe和Pre分别为89.2%、1.82 mm、0.712、0.999 8、0.930.与BCU-Net相比,CLT-ASegNet的DSC值提高了2.1%,HD值降低了0.13 mm.与最传统的医学图像分割网络U-Net相比,DSC值从85.2%提升到89.2%,HD值从1.95 mm降到了1.82 mm.表2CLT-ASegNet网络与其他分割网络在Lung16数据集的定量比较
Tab.2Quantitative comparison between CLT-ASegNet network and other segmentation networks on Lung16 dataset方法DSC/%HD/mmSenSpePreU-Net(2015)85.21.950.7040.999 70.916U-Net++(2018)85.61.970.7020.999 60.910AttentionUNet(2018)86.41.890.7050.999 70.929 DeepLabv3+(2018)83.62.010.7070.999 60.882TransUNet(2021)86.91.840.7050.999 70.924TFCNs(2022)86.11.750.6800.999 70.919BCU-Net(2023)87.11.950.5840.999 70.918CLT-ASegNet89.21.820.7120.999 80.930
图4展示了CLT-ASegNet和主流分割方法在CLTCTI数据集上的定性结果.与其他先进的方法相比,CLT-ASegNet在解决类间不区分和类内不一致问题上具有较好的效果.
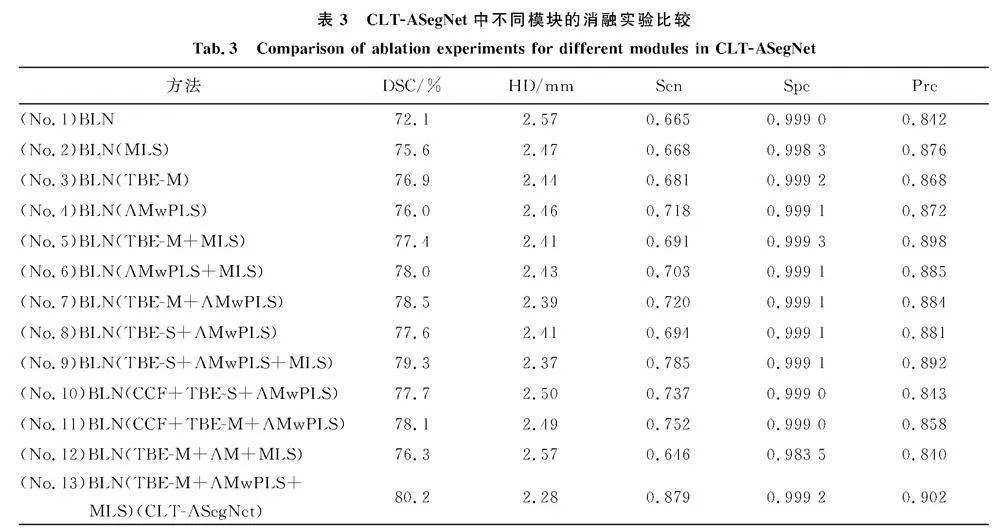
3.4消融分析
如表3所示,为了验证每个模块的有效性,CLT-ASegNet在CLTCTI数据集上进行了消融实验.首先,设计了一个基线网络(BLN).BLN(*)是指将指定的功能模块添加到BLN中,而多个功能模块则以一个加号连接.TBE-M表示肿瘤边界特征融合2层解码器,而TBE-S表示肿瘤边界特征仅融合一层解码器.此外,将解码器输出的多尺度特征的连接-卷积融合命名为“CCF”(没有深度监督),并将从AMwPLS中去掉先验肺形状的注意权重图的注意模块命名为AM.MLS代表深监督.通过使用序列号来表示相应的操作:例如,No.6表示BLN(AMwPLS+MLS).
从No.2、No.3、No.4可以看出,当将MLS、TBE或AMwPLS被添加到基线BLN中时,任何功能模块对CLT分割都有积极的影响.特别是No.3大大提高了CLT的分割性能,相比基线,DSC提高了4.8%,HD降低了0.13 mm, 其原因是由TBE网络生成的TBFMs能够有效地使该网络处理肿瘤与相邻组织之间的类间不区分以及肿瘤区域类内不一致.为了证明MLS的有效性,比较了No.5和No.6,在TBE和AMwPLS的基础上分别添加MLS相比较单个使用分割效果有所提升.为了评估MSFF的有效性,比较了No.7和No.8与No.4,实验结果证明多语义特征融合可以有效提高网络分割性能.有趣的是,No.7比No.8效果更好,这表明了混合尺度特征比单一尺度特征更有优势,No.10和No.11的比较结果中也得到相同结论.从No.8与No.9的比较中也证明了MLS对于分割效果有明显帮助.为了证明AMwPLS的有效性,比较了No.12与No.13(CLT-ASegNet)的性能.AMwPLS进一步提高了分割性能的原因是通过将先验的肺形状加入注意机制,可以有效地提取肿瘤的语义上下文信息,细化浅层特征图,从而突出病变区域.
图5为No.12与No.13(CLT-ASegNet)在训练过程中的收敛速度和稳定性的性能比较曲线.在DSC和HD的度量中,No.13(CLT-ASegNet)在第26个epochs和第15个epochs达到收敛,并保持着较小的波动,分别比No.12快了21个和11个epochs.研究结果表明,通过在注意机制中使用先验知识,可以大大提高模型的收敛速度和稳定性.a.DSC评价指标;b.HD评价指标
3.5讨论
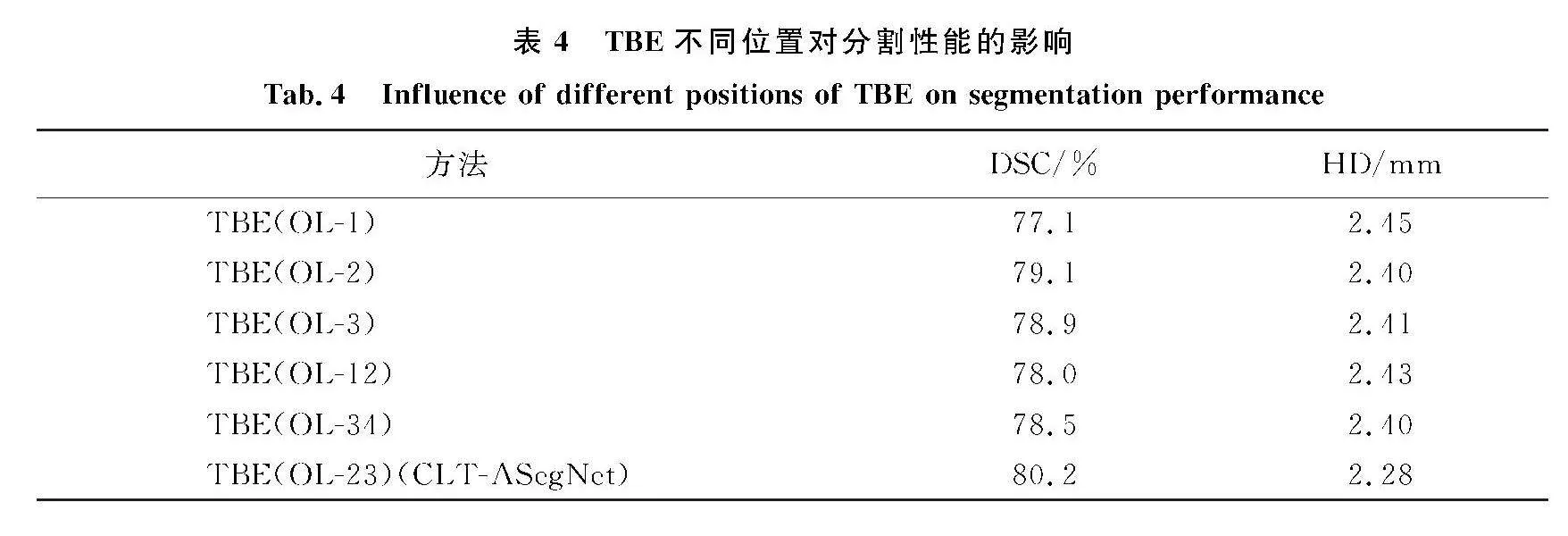
本节分析了TBE模块位置以及TBE和AMwPLS的组合对分割结果的影响.在CLT-ASegNet中,TBE的输入来自2个连续的不同层特征图,提供多尺度上下文表示,CLT-ASegNet使用了编码器的第2层和第3层.为了证明其合理性,将另外连续的2层特征图和单层特征图也作为TBE的输入,实验结果如表4所示.TBE(OL-1)是指CLT-ASegNet网络中的TBE模块只使用第1层特征图; TBE(OL-12)表示将第1层和第2层融合的边界信息应用于TBE.表4TBE不同位置对分割性能的影响
Tab.4Influence of different positions of TBE on segmentation performance方法DSC/%HD/mmTBE(OL-1)77.12.45TBE(OL-2)79.12.40TBE(OL-3)78.92.41TBE(OL-12)78.02.43TBE(OL-34)78.52.40TBE(OL-23)(CLT-ASegNet)80.22.28
TBE的目的是增强肿瘤边界信息,扩大肿瘤内外的区分度,从而以更高的精度指导解码器工作.从表4可以得出,第1层的分割效果比第2层和第3层差,证明从最浅的层获得边界特征并不是最好的选择,其原因是最浅层尚未产生有效的边界信息.然而,由于缺乏详细的信息,更高水平并不是生成独立肿瘤边界特征更好的选择.此外,多尺度肿瘤边界上下文特征可以提高网络分割性能.这是由于多尺度特征倾向于产生更鲁棒的特征表示.MSFF模块的目的是融合由AMwPLS细化的细节特征以及从TBE中获得的肿瘤边界特征.在表3中,由于No.13优于No.9,说明来自U型网络的相同尺度编码器和解码器层的特征映射表现出更一致的特征表示,从而使融合的特征能够有效地用于预测任务.
4结论
为了解决CT图像中复杂肺肿瘤的分割问题,提出了一种协同深度监督学习网络(CLT-ASegNet).语义分割网络采用了一个带有领域知识的注意模块(AMwPLS),以增强对肿瘤与邻近组织之间粘连边界的注意.TBE辅助网络提取肿瘤结构边界图,并将其融合到MSFF模块中,以增强网络的识别能力.CLT-ASegNet在2个数据集上的结果表明,通过将先验领域知识整合到分割模型设计以及重新考虑CLT分割,从宏观角度看,可有效提高密集预测性能.未来将不断扩展这些方法到其他医学图像的分割,希望研究新的方法使网络能够更有效地利用先验知识.
参考文献:
[1]WANG S, ZHOU M, LIU Z Y, et al. Central focused convolutional neural networks: developing a data-driven model for lung nodule segmentation[J]. Med Image Anal, 2017, 40: 172-183. DOI: 10.1016/j.media.2017.06.014.
[2]RONNEBERGER O, FISCHER P, BROX T. U-net: convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention, Cham: Springer, 2015: 234-241.10.1007/978-3-319-24574-4_28.
[3]CHEN L C, ZHU Y K, PAPANDREOU G, et al. Encoder-decoder with atrous separable convolution for semantic image segmentation[C]//European Conference on Computer Vision. Cham: Springer, 2018: 833-851.10.1007/978-3-030-01234-2_49.
[4]ZHANG B H, QI S L, WU Y N, et al. Multi-scale segmentation squeeze-and-excitation UNet with conditional random field for segmenting lung tumor from CT images[J]. Comput Methods Programs Biomed, 2022, 222: 106946. DOI: 10.1016/j.cmpb.2022.106946.
[5]NI Y F, XIE Z, ZHENG D Z, et al. Two-stage multitask U-Net construction for pulmonary nodule segmentation and malignancy risk prediction[J]. Quant Imaging Med Surg, 2022, 12(1): 292-309. DOI: 10.21037/qims-21-19.
[6]ZHOU Y Y, LI Z, BAI S, et al., Prior-aware neural network for partially-supervised multi-organ segmentation[C]//2019 IEEE/CVF International Conference on Computer Vision (ICCV), 2019: 10671-10680. DOI: 10.1109/ICCV.2019.01077.
[7]YU H, LI J Q, ZHANG L X, et al. Design of lung nodules segmentation and recognition algorithm based on deep learning[J]. BMC Bioinformatics, 2021, 22(Suppl 5): 314. DOI: 10.1186/s12859-021-04234-0.
[8]OKTAY O, SCHLEMPER J, LE FOLGOC L, et al. Attention U-net: learning where to look for the pancreas[EB/OL]. 2018: arXiv: 1804.03999. http://arxiv.org/abs/1804.03999.
[9]WANG Z K, ZOU Y N, LIU P X. Hybrid dilation and attention residual U-Net for medical image segmentation[J]. Comput Biol Med, 2021, 134: 104449. DOI: 10.1016/j.compbiomed.2021.104449.
[10]LIN H N, LI Z S, YANG Z F, et al. Variance-aware attention U-Net for multi-organ segmentation[J]. Med Phys, 2021, 48(12): 7864-7876. DOI: 10.1002/mp.15322.
[11]HU J F, WANG H, WANG J, et al. SA-Net: a scale-attention network for medical image segmentation[J]. PLoS One, 2021, 16(4): e0247388. DOI: 10.1371/journal.pone.0247388.
[12]ZHOU Z W, RAHMAN SIDDIQUEE M M, TAJBAKHSH N, et al. UNet++: A nested U-net architecture for medical image segmentation[C]//International Workshop on Deep Learning in Medical Image Analysis, International Workshop on Multimodal Learning for Clinical Decision Support. Cham: Springer, 2018: 3-11.10.1007/978-3-030-00889-5_1.
[13]CAI Y T, WANG Y. MA-Unet: an improved version of Unet based on multi-scale and attention mechanism for medical image segmentation[C]//Proc SPIE 12167, Third International Conference on Electronics and Communication; Network and Computer Technology (ECNCT 2021), 2022, 12167: 205-211. DOI: 10.1117/12.2628519.
[14]AN F P, LIU J E. Medical image segmentation algorithm based on multilayer boundary perception-self attention deep learning model[J]. Multimed Tools Appl, 2021, 80(10): 15017-15039. DOI: 10.1007/s11042-021-10515-w.
[15]ZHANG H, ZHONG X, LI G, et al. BCU-Net: bridging ConvNeXt and U-Net for medical image segmentation[J]. Comput Biol Med, 2023, 159: 106960. DOI: 10.1016/j.compbiomed.2023.106960.
[16]LI, Z H, LI D H, XU C B, et al. TFCNs: A CNN-Transformer Hybrid Network for Medical Image Segmentation[C]//In International Conference on Artificial Neural Networks, Cham: Springer Nature Switzerland, 2022: 781-792. DOI:10.1007/978-3-031-15937-4_65.
[17]TANG P, YANG P L, NIE D, et al. Unified medical image segmentation by learning from uncertainty in an end-to-end manner[J]. Knowl Based Syst, 2022, 241: 108215. DOI: 10.1016/j.knosys.2022.108215.
[18]CLARK K, VENDT B, SMITH K, et al. The cancer imaging archive (TCIA): maintaining and operating a public information repository[J]. J Digit Imag, 2013, 26(6): 1045-1057. DOI: 10.1007/s10278-013-9622-7.
[19]KERVADEC H, BOUCHTIBA J, DESROSIERS C, et al. Boundary loss for highly unbalanced segmentation[J]. Med Image Anal, 2021, 67: 101851. DOI: 10.1016/j.media.2020.101851.
[20]XUE Y, TANG H, QIAO Z, et al. Shape-aware organ segmentation by predicting signed distance maps[J]. Proc AAAI Conf Artif Intell, 2020, 34(7): 12565-12572. DOI: 10.1609/aaai.v34i07.6946.
[21]BERMAN M, TRIKI A R, BLASCHKO M B. The lovasz-softmax loss: a tractable surrogate for the optimization of the intersection-over-union measure in neural networks[C]//2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. June 18-23, 2018. Salt Lake City, UT, IEEE, 2018: 4413-4421. DOI: 10.1109/cvpr.2018.00464.
[22]ARMATO S G 3rd, MCLENNAN G, BIDAUT L, et al. The lung image database consortium (LIDC) and image database resource initiative (IDRI): a completed reference database of lung nodules on CT scans[J]. Med Phys, 2011, 38(2): 915-931. DOI: 10.1118/1.3528204.
[23]CHEN J N, LU Y Y, YU Q H, et al. TransUNet: transformers make strong encoders for medical image segmentation[EB/OL]. 2021: arXiv: 2102.04306. http://arxiv.org/abs/2102.04306.
(责任编辑:孟素兰)

