基于表型和基因型的藜麦种质资源遗传多样性分析
陶小所 姚晓华 吴昆仑 谢德庆 宋娇 姚有华
摘 要 为了解和掌握青海省引进和收集藜麦种质资源的遗传背景和多样性,本研究选择11个表型性状和221对SSR引物对114份藜麦种质资源进行遗传多样性分析。结果表明,114份藜麦种质资源的11个表型性状差异极显著(P<0.01),变异系数范围为4.57%~87.79%,主穗直径的变异最大(87.79%),籽粒直径的变异最小(4.57%);遗传多样性指数范围为0.47~2.29,单株产量的遗传多样性指数最大(2.29),茎秆长最小(0.47)。46对多态性较好的SSR引物扩增出165条多态性条带,每对引物的平均等位变异数为3.59,观测等位基因(Na)平均值为1.65,有效等位基因(Ne)平均值为1.50,Shannons信息指数(I)和Neis基因多样性指数(He)平均值分别为0.39和0.27,平均多态性比率为64.35%。通过聚类分析发现,114份藜麦种质基于表型性状和SSR分子标记的聚类结果存在一定的相似和差异,来源地相同的种质被分到了不同的类群,来源地不同的种质被分到了同一类或单独聚成一类,说明参试藜麦种质资源的表型性状和分子遗传信息多样性较丰富。
关键词 藜麦;种质资源;表型性状;SSR标记;遗传多样性
藜麦(Chenopodium quinoa. Willd.)(2n=4x=36)隶属于苋科藜属,一年生双子叶植物,起源于南美洲安第斯山、厄瓜多尔和秘鲁等地区,作为一种假谷物是当地土著居民的主要传统粮食之一[1-2]。藜麦具有广泛的遗传多样性,生长在海拔0~4 000 m,温度为-4~38 ℃的环境[3-6],具有耐寒、耐旱、耐盐碱、耐贫瘠等特性。藜麦籽粒含有丰富的蛋白质、赖氨酸、不饱和脂肪酸、维生素和矿物质成分,有利于平衡膳食营养、减肥、改善心脏病、治疗高血压等作用被逐步应用于食品加工和医疗领域[7]。中国自20世纪引进藜麦种质资源种植在西藏等高海拔地区,2013年在山西静乐县引种成功后,甘肃、内蒙古、河北、吉林、青海等省份也进行了藜麦资源的引进[8-9]。
种质资源遗传多样性可以通过表型性状和分子标记等方法评价[10]。表型性状是认识作物种质资源和培育新品种的基石[11],通过鉴定藜麦表型性状对其研究遗传变异规律和挖掘不同基因类型并加以利用具有重要意义。藜麦种质资源丰富多样,目前中国学者对藜麦种质资源进行表型性状鉴定和评价,以及利用分子标记SSR(Simple Sequence Repeat,SSR)进行遗传多样性分析,SSR具有重复性好、多态性高和特异性强等优点[12]。目前为止,陆敏佳等[13]首次用SSR引物研究44份藜麦种质资源多态性和亲缘关系,结果表明来自不同地区的藜麦种质资源遗传差异大,遗传距离较远。孙梦涵等[14]用65對SSR引物对163份藜麦种质资源和3份台湾红藜种质资源进行分析,结果表明来自玻利维亚和秘鲁种质资源与美国和智利种质资源的遗传信息存在明显区分,来自中国青海和云南的藜麦种质资源在亲缘关系上更接近安第斯高原型,来自中国河北和山西的藜麦种质资源更接近智利低海拔型。吴文强等[15]利用18对SSR引物对国内外收集的96份藜麦种质资源进行遗传多样性分析,聚类结果将种质资源按选系类型、来源地区、农艺性状等划分为不同的类群。叶君等[16]以国内外收集的101份藜麦种质资源进行表型性状的遗传多样性分析,结果表明藜麦表型变异系数较大,聚类分析将101份藜麦种质资源划分为特征明显的4类。表型性状和基因型是育种家获取不同藜麦种质资源遗传差异的主要途径,在水稻[17]、甜荞[18]、陆地棉[19]、菜豆[20]、板栗[21]、草地早熟禾[22]、红花[23]等作物中结合表型性状和分子标记开展遗传多样性分析已有较多相关研究,但结合表型性状和基因型的藜麦遗传多样性分析的研究较少。
本研究以藜麦11个表型性状和221对SSR引物对青海省引进和收集的114份藜麦种质资源进行多样性分析,旨在为现有藜麦种质资源在育种过程中的合理利用提供理论依据。
1 材料与方法
1.1 试验材料与试验地概况
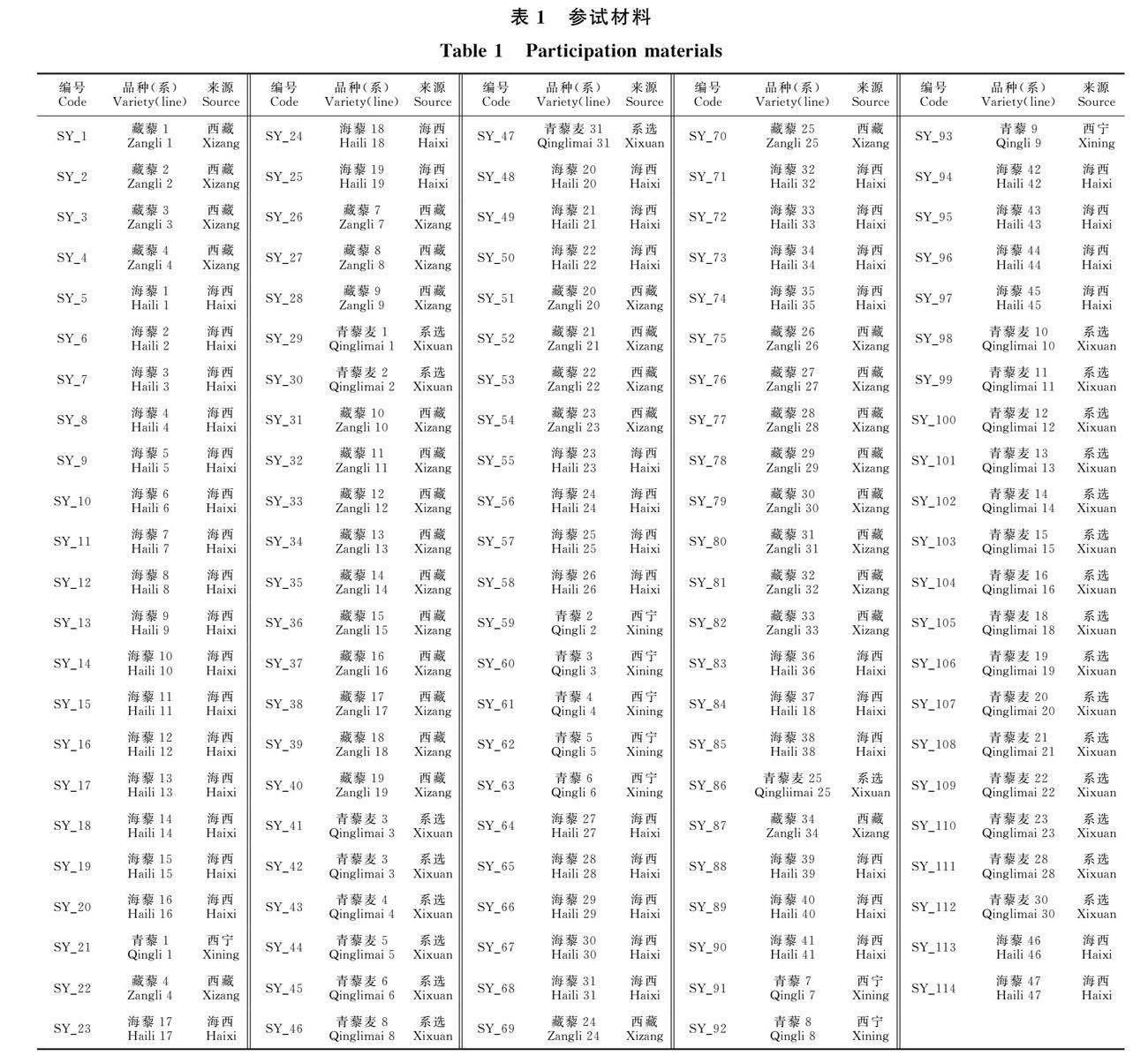
以国内114份藜麦资源为参试材料(表1),其中包含西藏资源(33份)、西宁资源(9份)、西宁系选材料(25份)和海西资源(47份),编号为 SY_1-114。试验在青海省农林科学院种质资源创新试验基地(101°49′17″E,36°34′03″N,海拔 2 309 m)进行,该地属于典型半干旱大陆性气候,土壤类型为栗钙土,质地沙壤土,土壤有机质含量为9.77 g/kg。
1.2 试验设计
2021年3月播种,每份藜麦材料种植2行,每行种植3.0 g,行距60 cm,行长6 cm。采用人工开沟溜播方式播种,田间管理采用常规大田管理。于2021年5月份每份材料分别选取植株6株并做标记,采集植株新鲜嫩叶提取DNA用于分子标记分析,标记植株用于后期表型性状调查。
1.3 试验方法
1.3.1 表型性状调查 收获期在田间对每份材料中挂牌的植株选取3株,参照宋娇等[24]研究方法对参试材料的表型性状:株高、主穗直径、穗长、有效穗数、茎秆长、千粒质量和单株产量进行调查;籽粒直径、面积、周长和长宽比采用万深检测科技的SC-G自动考种仪千粒质量仪形状分析 测量。
1.3.2 DNA提取与PCR扩增 每份参试材料取6株植株的顶部新鲜嫩叶2份,利用新型植物基因组DNA提取试剂盒(北京天根生化科技有限公司)进行DNA提取;提取的DNA首先使用核酸蛋白检测仪检测,若OD260/OD280比值大于1.6,小于1.9,稀释至最佳浓度后置于-20 ℃冰箱保存备用。
参照Jarvis等[25]、陆敏佳等[13]提供的PCR程序进行优化。随机选取32份材料对221对SSR引物进行初步筛选,得到46对多态性好、条带清晰的SSR分子标记。PCR扩增反应体系 20 μL,PCR反应体系为:94 ℃ 5 min;94 ℃ 1 min,68 ℃ 1 min,72 ℃ 1 min;(9个循环,每个循环将68-X/9 ℃);94 ℃ 30 s,(X+1) ℃ 30 s,72 ℃ 30 s(35个循环);72 ℃ 10 min;12 ℃保存。(X为上海生工合成的1对引物中退火温度较高的温度)。用8%非变性聚丙烯酰胺凝胶及银染法检测,通过银染显带读取条带并拍照记录。
1.4 数据分析
采用Excel 16.0和SPSS 19.0对表型数据进行统计和分析,遗传多样性指数的计算采用Shannon-Weaver多样性指数(H′),计算公式:
H′=-∑pi×Inpi(i=1,2,3,…)。式中:pi为某个性状第i个级别出现的概率[26]。SSR引物的扩增产物电泳图谱上同一位置上条带的有无用“1”和“0”进行统计。采用Popgene 32软件计算多态性位点数(NPL)、观测等位基因数(Na)、有效等位基因数(Ne)、Neis多样性指数(He)、Shannons信息指数(I)。采用NTSYS-PC 2.10e软件计算遗传相似系数并进行聚类分析和主成分分析。
2 结果与分析
2.1 藜麦表型性状的遗传多样性分析
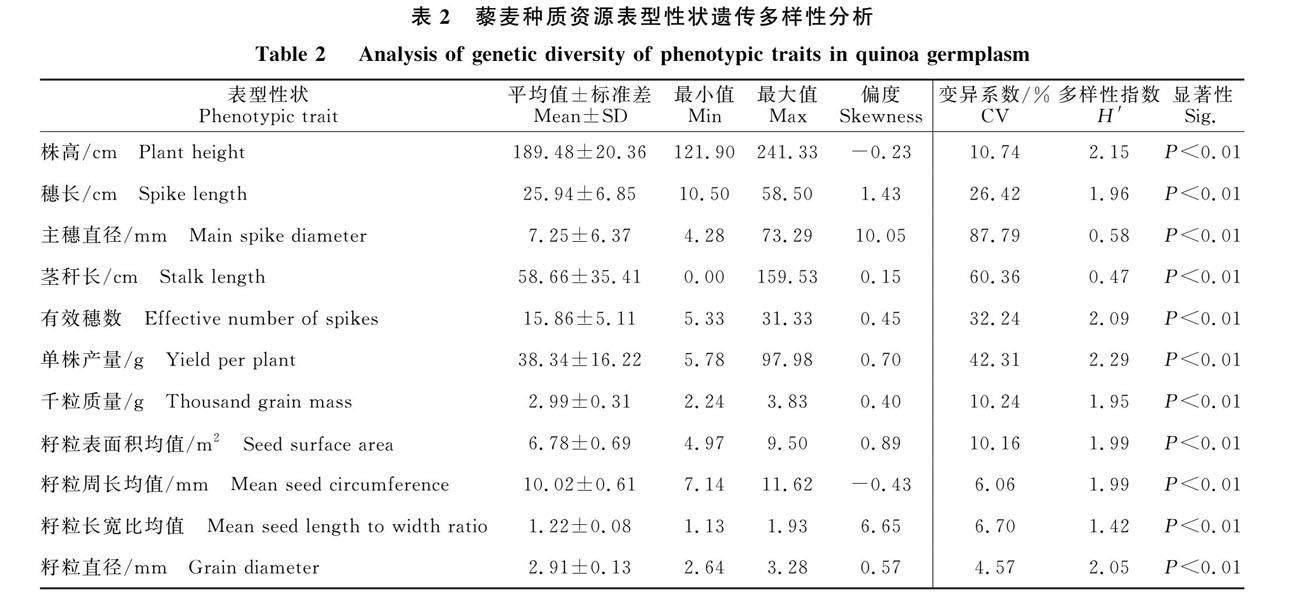
2.1.1 藜麥表型性状的遗传多样性分析 由表2可见,114份藜麦种质资源的每个表型性状之间差异极显著(P<0.01)。参试材料的11个表型性状变异系数范围为4.57%~87.79%,其中以主穗直径的变异系数最大为87.79%,以籽粒直径的变异系数最小为4.57%,从大到小排列顺序为主穗直径(87.79%)>茎秆长(60.36%)>单株产量(42.31%)>有效穗数(32.24%)>穗长 (26.42%)>株高(10.74%)>千粒质量 (10.24%)>籽粒表面积(10.16%)>籽粒长宽比 (6.70%)>籽粒周长(6.06%)>籽粒直径(4.57%)。11个表型性状的遗传多样性指数范围在0.47~2.29,其中以单株产量最大为2.29,以茎秆长最小为 0.47,从大到小排列顺序为单株产量(2.29)>株高(2.15)>有效穗数(2.09)>籽粒直径(2.05)>籽粒表面积均值和籽粒周长均值(1.99)>穗长(1.96)>千粒质量(1.95)>籽粒长宽比均值 (1.42)>主穗直径(0.58)>茎秆长(0.47)。
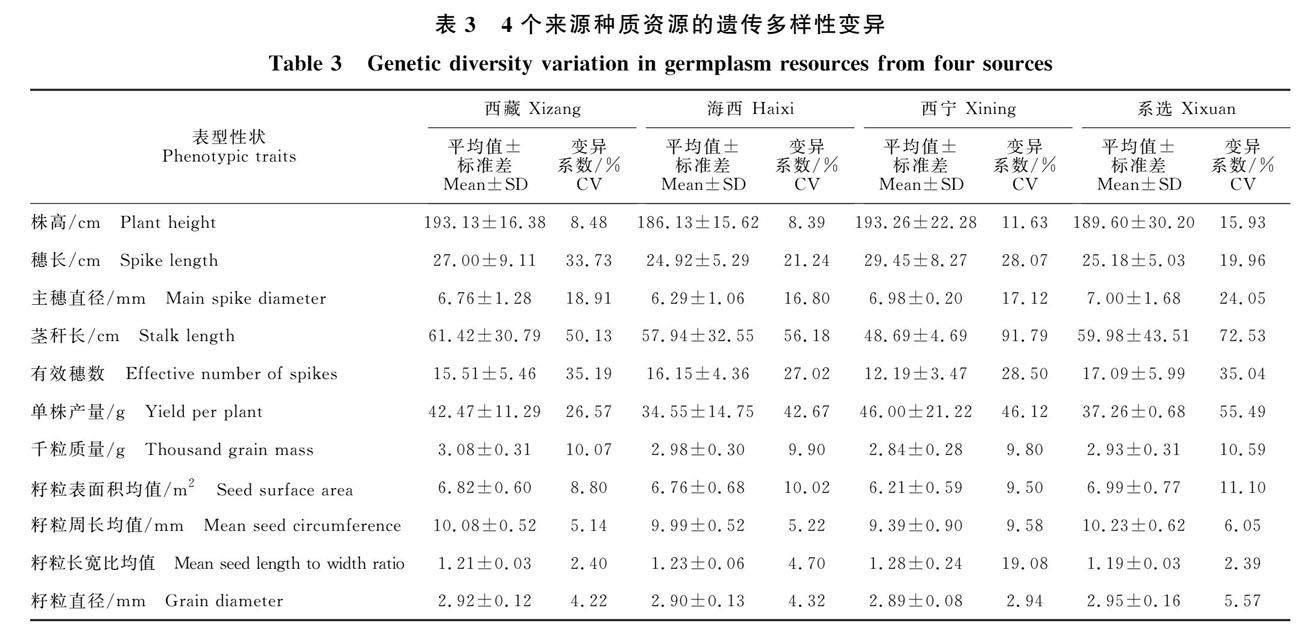
由表3可见,4个来源地的藜麦种质资源表型性状遗传变异丰富。西藏种质资源的茎秆长、单株产量和千粒质量指标高于海西、西宁和西宁系选的种质资源,西宁系选种质资源的有效穗数、籽粒面积、籽粒周长和籽粒直径指标高于西藏、海西和西宁的种质资源。因此,可以从西藏和西宁系选种质资源中根据育种目标进一步筛选藜麦新品种(系)。
2.1.2 表型数据的聚类分析 以株高等11个表型指标对114份藜麦种质资源进行系统聚类分析(图1)。聚类结果表明,当欧式距离10为阈值,可以将114份藜麦种质资源分为6大类群:I、Ⅱ、Ⅲ、Ⅳ、V、Ⅵ。第Ⅰ类包括来自不同地区的56份材料,分别为85、86、17、31、81、13、65、51、30、43、37、38、35、41、7、84、19、94、39、102、21、32、15、113、18、108、103、106、92、1、83、93、4、52、29、6、96、9、88、98、104、68、97、50、69、78、22、34、5、54、33、71、2、82、107、74,海西22份、西藏19份,西宁系选12份和西宁3份。第Ⅱ类包括来自不同地区的46份材料,分别为47、80、49、63、36、45、28、58、48、72、25、26、67、3、56、40、75、20、27、16、91、95、24、77、12、73、23、87、89、57、44、8、66、70、110、14、111、79、76、105、60、114、61、59、55、64,海西22份、西藏13份、西宁系选6份和西宁5份。第Ⅲ类包括5份材料,分别为10、99、109、100、101,海西1份和西宁系选4份。第Ⅳ类为11来自海西;第Ⅴ类为42来自西宁系选,主要特征在株高、茎秆长、有效穗数、籽粒周长、籽粒表面积、单株产量和千粒质量上最大;第Ⅵ类包括5份材料,分别为46、90、53、62、12,海西、西藏和西宁各一份,西宁系选2份,主要特征在株高、茎秆长、籽粒表面积、千粒质量和单株产量上最小。表型性状聚类结果表明,114份藜麦种质资源的表型性状的多样性与来源地并没有明显的相关性,不同来源地的材料表型均较为丰富。
2.2 基于SSR标记的遗传多样性分析
2.2.1 藜麦种质资源的分子标记多态性信息 221对SSR引物对114份藜麦种质资源进行DNA扩增,其中46对多态性丰富的SSR标记结果扩增带型清晰稳定、多态性丰富(图2),范围为100~370 bp,共检测165条多态性条带,平均等位变异数为3.59。遗传多样性分析结果如表4所示,46对引物的的多态性位点数为30~112,多态性位点数越大则引物的多态性越丰富,引物KAAT049的多态性最丰富,引物KCAA141、KAAT006、KAAT021、KAAT010、KAAT022、KGA156的多态性次于KAAT049;等位基因数在1.070 2~1.982 5;有效等位基因数在 1.049 6~1.849 0;Neis多样性指数在 0.029 1~0.443 2;Shannons信息指数在 0.042 4~0.622 5;多态性位点比率在 7.02%~98.25%,平均多态性位点比率为 64.35%大于50%。由以上多个遗传多样性指数结果表明,本研究所用的46对SSR引物具有较高多态性。同时,综合多个遗传多样性指数的结果发现,引物KAAT049、KCAA141、KAAT021、KAAT022和KGA156的多态性则较高,引物KCAA002的多态性最低。
2.2.2 藜麦种质资源遗传相似性分析 利用NTSYS-PC 2.10e软件计算114份藜麦种质资源的遗传相似系数。结果表明,114份材料的遗传相似系数分布为0.21~1.00,平均遗传相似系数为0.53。海西SY_96和西宁系选SY_99遗传相似系数最小(0.21),可见它们的遗传差异最大,亲缘关系最远。西宁SY_60和海西SY_58遗传相似系数较大(0.88),说明它们亲缘关系较近,因此推测它们来源地相同或相近。
2.2.3 藜麦种质资源聚类分析 UPGMA聚类分析表明,46对SSR引物可将114份藜麦种质资源分开,在遗传相似系数0.63处114份藜麦种质资源可分为7类(图3)。第Ⅰ类包括来自不同地区的106份材料,西藏32份,海西42份,西宁9份,西宁系选24份。第Ⅱ类是1份西宁系选材料单独聚成一份,说明该材料与其他种质亲缘关系较远。第Ⅲ类包括1份西藏材料和3份海西材料。第Ⅳ类和V类分别是1份海西材料单独聚成一类。第Ⅵ类和Ⅶ类分别是1份西宁系选材料单独聚成一类。西藏、海西、西宁和西宁系选材料主要集中在第Ⅰ类,因此,本研究探究的114份藜麦种质地区分类不明显,各地区的种质的遗传信息具有一定的相似性。同时也说明青海引进的藜麦种质混乱,在后期研究上需要进一步筛选和推进育种工作进程。
2.2.4 主成分分析(PCoA) 为了更好地验证114份藜麦种质资源间的遗传关系,基于SSR分子标记的遗传相似性系数矩阵,利用NTSYS-PC 2.10e对114份藜麦种质资源进行主成分分析。由图4可知,第一主坐标解释了种质资源间相关性的10.49%,第二主坐标解释了5.60%。114份藜麦种质资源在Dim-1和Dim-2主坐标上各被分成2组,综合起来可将114份藜麦种质资源划分成4组。其中,第一组包含22份材料,15份海西材料,5份西藏材料,2份西宁系选材料;与聚类分析结果相比,主要集中在聚类分析的第Ⅰ类,1份西藏材料在聚类分析第Ⅲ类。第二组包括58份材料,26份西藏彩礼,5份西宁材料,9份西宁系选材料,18份海西材料;与聚类分析结果相比,主要集中在聚类分析第Ⅰ类,1份西宁系选材料在聚类分析第Ⅱ类。第三组包括13份材料,5份海西材料,8份西宁系选材料;与聚类分析结果相比,2份海西材料在聚类分析第Ⅲ类,各1份海西材料在聚类分析第Ⅳ和V类,各1份西宁系选材料在聚类分析第Ⅵ和Ⅶ类。第四组包括21份材料,9份海西材料,4份西宁材料,2份西藏材料,6份西宁系选材料;与聚类分析结果相比,主要集中在聚类分析第Ⅰ类,1份海西材料在聚类分析第Ⅲ类。
2.2.5 基于表型性状和基因型的多样性分析 通过表型性状和SSR分子标记两种方法来研究藜麦种质资源的遗传多样性。从两种聚类结果来看,114份参试藜麦材料凌乱的分布在聚类树上,相同地区的种质资源没有在相应的组群上,且在表型性状分析上看不出具体的共性特征。按材料来源地在表型聚类分析中,47份海西材料均分布在6大类群,在分子标记聚类图中,材料主要集中在第Ⅰ类,第Ⅲ类包括3份材料,2份材料各单独聚成一类,说明海西材料表型和遗传信息多样性均较高。西藏33份材料在表型聚类中分布在第Ⅰ、Ⅲ和Ⅵ类,在分子聚类图中97%的材料集中在第Ⅰ类,1份材料单独具有一类,说明西藏材料表型多样性较高,遗传信息多样性较低。西宁9份材料分布在第Ⅰ、Ⅲ和Ⅵ类,在分子标记聚类图中均分布在第Ⅰ类,说明西宁材料表型多样性较高,遗传信息多样性较低。西宁系选25份材料主要集中在第Ⅰ类,2份材料各单独聚成一类,说明西宁系选材料表型和遗传信息多样性较低。
3 讨 论
为了解和掌握青海省引进的藜麦种质资源的遗传多样性,推进青海省藜麦育种进程。本研究对西藏资源、西宁资源、西宁系选材料和海西资源这4个地方的114份藜麦种质资源进行表型性状和SSR分子标记的遗传多样性分析。
3.1 基于表型性状的藜麦种质资源遗传多样性
表型性状是鉴定植物变异最直接的方法,本研究选取株高、穗长、主穗直径等11个表型性状对114份藜麦种质资源的遗传多样性进行了分析。变异系数可以从表型上明确不同种质的差异程度,吕伟等[27]认为变异系数大于10%时,种质间差异较大。本研究结果表明,114份藜麦种质资源的11个表型性状差异极显著,变异系数范围在4.57%~87.79%,主穗直径的变异系数最大,而籽粒直径的变异系数最小。11个表型性状的遗传多样性指数分布范围为0.47~2.29,平均值1.72。其中,多样性指数最大的是单株产量,最小的是茎秆长。由此可见,藜麦表型性状变异系数与遗传多样性指数没有正负相关关系,与赫卫等[28]、李宁等[29]研究一致。张锦豪等[30]研究表明藜麦种质资源表型性状变异系数为 13.75%~66.74%,平均遗传多样性指数为 1.65,与本研究结果基本相同。本研究表型性状聚类第V类是一份西宁系选材料,其中有效穗数、籽粒周长、籽粒表面积、单株产量和千粒质量最大。因此,要提高藜麦籽粒产量,可以用V类材料进行一步研究。而第Ⅵ类材料则与第V类材料相反,所以可以对这两个极端材料进行比较研究,发掘影响这两个藜麦籽粒产生差异的原因。本研究将114份藜麦种质聚成6大类可为青海省藜麦种质的有效利用和选品种选育提供参考依据,但是还需要进行多年多点研究。
3.2 基于SSR分子标记的藜麦种质资源遗传多样性
SSR分子标记已广泛应用在甜菜[31]、辣椒[32]、水稻[33]等作物上进行遗传多样性分析。本研究利用221对SSR引物对114份国内藜麦种质资源进行遗传多样性分析,46对多态性丰富的SSR引物扩增条带范围在100~370 bp,165条多态性条带,每对引物的平均等位变异数为 3.59。平均等位变异数低于陆敏佳等[13]、孙梦涵等[14]的研究,原因可能是不同的SSR分子标记数量存在差异。陈翠萍等[34]在藜麦SSR分子标记多态性信息中,引物KAAT021的多态性最丰富,引物KGA010、KGA100和BGA200的多态性最低,与本研究结果不一致,其原因可能是试验材料不同导致的结果。本研究基于SSR分子标记的聚类分别不明显,但个别材料被单独聚成一类,与陆敏佳等研究结果一致,所以推测相同的原因可能是引进材料的过程中存在交叉现象或研究使用的引物种类和数量较少有关。孙梦涵等[14]对166份藜麦种质资源进行聚类,结果为相同来源地的藜麦聚成一类,与本研究結果不一致,原因是引进的藜麦种质资源初始来源地相同。由此可见,这114份藜麦种质资源的遗传基础较广泛。
3.3 表型和基因型的藜麦种质资源遗传多样性差异结果分析
本研究114份藜麦种质资源从表型性状和基因型的聚类结果来看,既有存在一定的相似性和差异。原因一方面是藜麦本身具有广泛的农业生态多样性[35],表型性状聚类分析和SSR分子标记分析检测的目标不一致,数量性状是由多个基因控制,SSR标记可能只检测到了其中的一部分,或是检测到的多态性位点未在表型性状中表现出来;另一方面可能是引进的藜麦种质资源最初来源地不清楚,在引种过程中存在交叉渗透现象。赫卫等[28]研究结果表明,SSR分子标记将表型性状差异大的辣椒品种相互分开,与本研究结果不一致。本研究结果与徐福荣等[17]、傅鸿妃等[32]在水稻和辣椒上基于表型性状和SSR分子标记存在一定差异结果一致。不同物种在生长发育进程,表型性状受外界环境和基因影响,因此想要更准确、更全面的描述遗传多样性需要从2个方面上进行研究。
4 结 论
本研究发现,114份藜麦种质资源的11个表型性状差异极显著(P<0.01),其中46对多态性丰富的SSR引物扩增出165条多态性条带,每对引物的平均等位变异数为3.59,观测等位基因(Na)平均值为1.65,有效等位基因(Ne)平均值为1.50,Shannons信息指数(I)和Neis基因多样性指数(He)平均值为0.39和0.27,平均多态性比率为64.35%,大于50%。基于表型性状和SSR分子标记聚类分析表明,来源地相同的种质资源被分到了不同的类,来源地不同的种质资源被分到了同一类或单独聚成一类,表型性状和遗传信息具有一定的相似性,说明参试材料遗传多样性较丰富。研究结果为藜麦种质资源的应用和育种提供理论依据。
参考文献 Reference:
[1] 王创云,邓 妍,段鹏慧,等.藜麦生物学特性及精简栽培种植技术[J].山西农经,2017(23):88-89,139.
WANG CH Y,DENG Y,DUAN P H,et al.Quinoa biology and planting techniques for lean cultivation[J].Shanxi Agricultural Economy,2017(23):88-89,139.
[2] RISI J C,GALWEY N W.Chenopodium grains of the Andes:Inca crops for modern agriculture[J].Advances in Applied Biology,1984,10:145-216.
[3] SHAH S S,SHI L,LI Z,et al.Yield,agronomic and forage quality traits of different quinoa (Chenopodium quinoa Willd.) genotypes in northeast China[J].Agronomy,2020,10(12):1908.
[4] JACOBSEN S E,LIU F,JENSEN C R.Does root sourced ABA play a role for regulation of stomata under drought in quinoa(Chenopodium quinoa Willd.)[J].Scientia Horticulturae,2009,122(2):281-287.
[5] 杨利艳,杨小兰,朱满喜,等.干旱胁迫对藜麦种子萌发及幼苗生理特性的影响[J].种子,2020,39(9):36-40.
YANG L Y,YANG X L,ZHU M X,et al. Effects of drought stress on seed germination and seeding physiological characteristics of quinoa[J].Seeds,2020,39(9):36-40.
[6] 时丕彪,耿安红,李亚芳,等.江苏沿海地区12个藜麦品种田间综合评价及优良品种的耐渍性分析[J].江苏农业科学,2018,46(15):64-67.
SHI P B,GENG A H,LI Y F,et al.Comprehensive evaluation of twelve Chenopodium quinoa cultivars and analysis of waterlogging tolerance of excellent cultivars in Jiangsu coastal area[J].Jiangsu Agricultural Sciences,2018,46(15):64-67.
[7] VEGA G A,MIRANDA M,VERGARA J,et al.Nutrition facts and functional potential of quinoa (Chenopodium quinoa willd.),an ancient Andean grain:a review[J].Journal of the Science of Food and Agriculture,2010,90(15):2541-2547.
[8] 任贵兴,杨修仕,么 杨.中国藜麦产业现状[J].作物杂志,2015(5):1-5.
REN G X,YANG X SH,ME Y.Current situation of quinoa industry in China[J].Crops,2015(5):1-5.
[9] 李吉有.論藜麦在民和地区的推广[J].农业与技术,2017,37(19):179-184.
LI J Y.On the promotion of quinoa in the Minhe region[J].Agriculture and Technology,2017,37(19):179-184.
[10] 葛淑俊,孟义江,李广敏,等.我国药用植物遗传多样性研究进展[J].中草药,2006(10):1584-1589.
GE SH J,MENG Y J,LI G M,et al.Research progress on genetic diversity in Chinese medicinal plants[J].Chinese Traditional and Herbal Drugs,2006(10):1584-1589.
[11] 张爱民,阳文龙,方红曼,等.作物种质资源研究态势分析[J].植物遗传资源学报,2018,19(3):377-382.
ZHANG A M,YANG W L,FANG H M,et al.Current status of research on crop genetic germplasms[J].Journal of Plant Genetic Resources,2018,19(3):377-382.
[12] GROVER A,SHARMA P C.Development and use of molecular markers:past and present[J].Critical Reviews in Biotechnology,2016,36(2):290-302.
[13] 陆敏佳,蒋玉蓉,陆国权,等.利用SSR标记分析藜麦品种的遗传多样性[J].核农学报,2015,29(2):260-269.
LU M J,JIANG Y R,LU G Q,et al.Genetic diversity of quinoa germplasm assessed by SSR markers[J].Journal of Nuclear Agricultural Sciences,2015,29(2):260-269.
[14] 孫梦涵,邢 宝,崔宏亮,等.藜麦种质资源遗传多样性SSR标记分析[J].植物遗传资源学报,2021,22(3):625-637.
SUN M H,XING B,CUI H L,et al.Genetic diversity analysis of quinoa by SSR markers[J].Journal of Plant Genetic Resources,2021,22(3):625-637.
[15] 吴文强,杨 箐,陈天青,等.藜麦种质资源的遗传多样性分析[J].种子,2021,40(2):13-19.
WU W Q,YANG Q,CHEN T Q,et al.Genetic diversity analysis of germplasm resources of quinoa[J].Seeds,2021,40(2):13-19.
[16] 叶 君,吴晓华,李元清,等.基于表型性状的藜麦种质资源遗传多样性分析[J].北方农业学报,2020,48(1):1-6.
YE J,WU X H,LI Y Q,et al.Genetic diversity analysis of phenotypic traits in quinoa germplasm resources[J].Journal of Northern Agriculture,2020,48(1):1-6.
[17] 徐福荣,董 超,杨文毅,等.基于表型性状和SSR分子标记的云南省水稻主要育成品种(系)的遗传相似性分析[J].植物遗传资源学报,2011,12(5):700-708.
XU F R,DONG CH,YANG W Y,et al.Genetic similarity based on SSR markers and phenotypies traits of major improved rice (Oryza sativa L.) varieties in Yunnan province[J].Journal of Plant Genetic Resources,2011,12(5):700-708.
[18] 李金龙,范 昱,赵梦雨,等.基于表型性状和SSR分子标记构建甜荞初级核心种质[J].植物遗传资源学报,2021,22(5):1240-1247.
LI J L,FAN Y,ZHAO M Y,et al.Construction of primary pore collection of buckwheat germplasm resources based on phenotypic traits and SSR[J].Journal of Plant Genetic Resources,2021,22(5):1240-1247.
[19] LI C,SONG L,ZHU Y,et al.Genetic diversity assessment of upland cotton variety resources in China based on phenotype traits and molecular markers[J].Crop Science,2017,57(1):290-301.
[20] 汪宝根,董君暘,汪 颖,等.浙江省地方菜豆种质资源鉴定与遗传多样性分析[J].浙江农业学报,2022,34(11):2416-2427.
WANG B G,DONG J Y,WANG Y,et al.Evaluation and genetic diversity analysis of common bean germplasm in Zhejiang Province,China[J].Acta Agriculturae Zhejiangensis,2022,34(11):2416-2427.
[21] 白晓倩,陈 于,张仕杰,等.基于表型性状和SSR标记的板栗品种遗传多样性分析及分子身份证构建[J].植物遗传资源学报,2022,23(4):972-984.
BAI X Q,CHEN Y,ZHANG SH J,et al.Genetic diversity analysis and fingerprinting of chestnut varieties based on phenotypic traits and SSR markers[J].Journal of Plant Genetic Resources,2022,23(4):972-984.
[22] 程林涛,孙晓阳,谢福春,等.基于表型性状和SSR分子标记的草地早熟禾遗传多样性分析和分子身份证构建[J].中国草地学报,2022,44(9):1-10.
CHENG L T,SUN X Y,XIE F CH,et al.Genetic diversity analysis and molecular ID construction of kentucky bluegrass based on phenotypic traits and SSR molecular markers[J].Chinese Journal of Grassland,2022,44(9):1-10.
[23] HOUMANAT K,DOUAIK A,CHARAFI J,et al.Appropriate statistical methods for analysis of safflower genetic diversity using agglomerative hierarchical cluster analysis through combination of phenotypic traits and molecular markers[J].Crop Science,2021,61(6):4164-4180.
[24] 宋 嬌,姚有华,刘 洋,等.6个藜麦品种(系)农艺性状的主成分分析[J].青海大学学报,2017,35(6):6-10.
SONG J,YAO Y H,LIU Y,et al.Principal component analysis of agronomic traits of six quinoa varieties (or lines)[J].Journal of Qinghai University,2017,35(6):6-10.
[25] JARVIS D E,KOPP O R,JELLEN E N,et al.Simple sequence repeat marker development and genetic mapping in quinoa (Chenopodium quinoa Willd.)[J].Journal of Genetics,2008,87(1):39.
[26] 张金屯.数量生态学[M].2版.北京:科学出版社,2011.
ZHANG J T.Quantitative Ecology[M].2nd ed.Beijing:Science Press,2011.
[27] 吕 伟,刘文萍,任果香,等.不同浓度生根粉对芝麻生长及产量的影响[J].作物杂志,2017(5):100-105.
L W,LIU W P,REN G X,et al.Effects of different concentrations of ABT on growth and yield of sesame[J].Crops,2017(5):100-105.
[28] 赫 卫,张 慧.基于表型性状和SRAP标记的观赏用辣椒种质资源遗传多样性分析[J].中国瓜菜,2022,35(1):16-23.
HE W,ZHANG H.Analysis of genetic diversity of pepper germplasm resources for ornamental based on phenotypic traits and SRAP markers[J].China Cucurbits and Vegetables,2022,35(1):16-23.
[29] 李 宁,王 飞,姚明华,等.国内外辣椒种质资源表型性状多样性及相关性分析[J].辣椒杂志,2015,13(1):8-13.
LI N,WANG F,YAO M H,et al.Genetic diversity and correlation analysis of phenotype traits in Capsicum spp.germplasms from China and abroad[J].Journal of China Capsicum,2015,13(1):8-13.
[30] 张锦豪,王树彦,杨恩泽,等.藜麦种质资源表型性状遗传多样性分析[J].中国草地学报,2022,44(12):37-46.
ZHANG J H,WANG SH Y,YANG E Z,et al.Genetic diversity analysis of phenotypic traits in Chenopodium quinoa Willd. germplasm resources[J].Chinese Journal of Grassland,2022,44(12):37-46.
[31] 李乔乔,王宇晴,邳 植,等.基于SSR分子标记的甜菜种质资源遗传多样性分析[J].中国糖料,2023,45(1):1-7.
LI Q Q,WANG Y Q,PI ZH,et al.Genetic diversity analysis of sugar beet germplasm resources based on SSR molecular markers[J].Sugar Crops of China,2023,45(1):1-7.
[32] 傅鸿妃,吕晓菡,陈建瑛,等.辣椒种质表型性状与SSR分子标记的遗传多样性分析[J].核农学报,2018,32(7):1309-1319.
FU H F,L X H,CHEN J Y,et al.Genetic diversity analysis of capsicum germplasm based on phenotypic traits with SSR markers[J].Journal of Nuclear Agricultural Sciences,2018,32(7):1309-1319.
[33] 王晓映,张方玉,万 星,等.基于分子标记和表型性状的水稻地方品种遗传多样性研究[J].植物遗传资源学报,2023,24(3):636-647.
WANG X Y,ZHANG F Y,WAN X,et al.Diversity of rice landraces revealed by molecular markers and phenotypic traits[J].Journal of Plant Genetic Resources,2023,24(3):636-647.
[34] 陳翠萍,闫殿海,张书苗,等.藜麦SSR指纹图谱构建及遗传多样性分析[J].作物杂志,2023(3):35-42.
CHEN C P,YAN D H,ZHANG SH M,et al.Fingerprint construction and genetic diversity analysis of quinoa based on SSR markers[J].Crops,2023(3):35-42.
[35] PHARA B D,FILIP B V,HILDE M,et al.Yield and nutritional characterization of thirteen quinoa (Chenopodium quinoa Willd.) varieties grown in North-west Europe—Part I[J].Plants,2021,10(12):2689.
Genetic Diversity Analysis of Quinoa Germplasm
Resources Based on Phenotype and Genotype
TAO Xiaosuo1,YAO Xiaohua1,2,WU Kunlun1,2,
XIE Deqing1,2,SONG Jiao1 and YAO Youhua1,2
(1.Academy of Agricultural and Forestry Sciences,Qinghai University, Xining810016, China; 2.Qinghai-Tibet Plateau Germplasm Resources Research and Utilization Laboratory, Qinghai Province,Xining 810016,China)
Abstract To understand the genetic background and diversity of quinoa germplasm resources introduced and collected by Qinghai Province, 11 phenotypic traits and 221 pairs of SSR primers were selected to analyze genetic diversity of 114 quinoa germplasm resources.The results showed significant differences (P<0.01) among the 11 phenotypic traits observed in the 114 quinoa germplasm resources. The coefficient of variation ranged from 4.57% to 87.79%, with the largest variation found in main spike diameter (87.79%) and the least in seed diameter (4.57%).The genetic diversity index ranged from 0.47 to 2.29, with the highest genetic diversity index for yield per plant (2.29) and the lowest for stalk length (0.47). The 46 pairs of highly polymorphic SSR primers amplified 165 polymorphic bands. The mean variance allelic of each primers pair was 3.59, the average values for observed alleles (Na), effective alleles (Ne), Shannons information index (I) and Neis gene diversity (He) , were 1.65, 1.50, 0.39 and 0.27, respectively. The average polymorphism ratio was 64.35%. The clustering analysis revealed that 114 quinoa germplasm resources were similar and different based on phenotypic traits and SSR markers.The germplasm of the same origin were classified into different groups,while the germplasm from the different origins was either grouped together or formed a separate cluster. These results indicated the rich diversity of phenotypic characters and molecular genetic information of quinoa germplasm resources.
Key words Quinoa; Germplasm resources; Phenotypic traits; SSR markers; Genetic diversity
Received 2023-07-15 Returned 2023-07-28
Foundation item Special Project for Transformation of Scientific and Technological Achievements in Qinghai Province (No.2022-NK-112); Special Project for Science and Technology Specialists in Qinghai Province (No.2023-NK-P09).
First author TAO Xiaosuo,female,master student. Research area:genetic breeding of triticeae crops. E-mail:1245280378@qq.com
Corresponding author YAO Youhua,male,associate research fellow.Research area:breeding and cultivation of triticeae crops. E-mail:youhua8888@126.com
(责任编辑:成 敏 Responsible editor:CHENG Min)

